Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Biomédica
versión impresa ISSN 0120-4157
Biomédica vol.36 no.3 Bogotá jul./set. 2016
https://doi.org/10.7705/biomedica.v36i3.3098
NOTA TÉCNICA
doi: http://dx.doi.org/10.7705/biomedica.v36i3.3098
Luz Adriana Pérez 1, Freddy Rodríguez 1,2, Carl Henrik Langebaek 1,2, Helena Groot 1
1 Laboratorio de ADN, Universidad de los Andes, Bogotá, D.C., Colombia
2 Departamento de Antropología, Universidad de los Andes, Bogotá, D.C., Colombia
Contribución de los autores:
Luz Adriana Pérez: ensayos de laboratorio
Freddy Rodríguez: recolección de las muestras de museos y verificación de los resultados moleculares
Carl Henrik Langebaek: análisis antropológico e información arqueológica y antropológica
Helena Groot: supervisión de los ensayos de biología molecular y edición del manuscrito
Recibido: 13/10/15; aceptado: 29/03/16
Introducción. A diferencia de otro tipo de investigaciones, el análisis de ADN antiguo (ADNa) requiere la implementación de condiciones metodológicas y de infraestructura especializadas que garanticen la autenticidad de los resultados. Uno de los criterios de autenticidad para este tipo de muestras es la cuantificación del material genético, en la cual es común el uso de la reacción en cadena de la polimerasa (PCR) cuantitativa en tiempo real, por su sensibilidad y especificidad. La implementación de estas metodologías y de las condiciones necesarias para el cumplimiento de los requisitos de autenticidad hace que este tipo de investigación sea dispendioso y costoso.
Objetivo. Generar una estrategia de cuantificación del ADN mitocondrial de muestras seriamente degradadas mediante un sistema sencillo y de fácil implementación.
Materiales y métodos. El sistema se basa en el uso de iniciadores que posibilitan la amplificación específica de fragmentos cortos del ADN mitocondrial. La posterior purificación de este fragmento permite generar una curva estándar con concentraciones acordes al estado de degradación de la muestra.
Resultados. Se detectó ADN antiguo proveniente de restos óseos y tejidos momificados de diferentes fechas. Además, el sistema permitió detectar la presencia de agentes inhibidores del ADN.
Conclusión. La implementación de la estrategia aquí planteada es sencilla, puede reducir los costos de la investigación y, además, permite la detección de ADNa en muestras muy degradadas, así como la discriminación entre las muestras que no poseen material genético y aquellas que presentan agentes inhibidores.
Palabras clave: ADN, ADN mitocondrial, reacción en cadena de la polimerasa.
doi: http://dx.doi.org/10.7705/biomedica.v36i3.3098
Real-time quantification to analyze historical Colombian samples detecting a short fragment of hypervariable region II of mitochondrial DNA
Introduction: Unlike other molecular biology studies, the analysis of ancient DNA (aDNA) requires special infrastructure and methodological conditions to guarantee the quality of the results. One of the main authenticity criteria is DNA quantification, where quantitative real-time PCR is often used given its sensitivity and specificity. Nevertheless, the implementation of these conditions and methodologies to fulfill authenticity criteria imply higher costs.
Objective: To develop a simple and less costly method for mitochondrial DNA quantification suitable for highly degraded samples.
Materials and methods: The proposed method is based on the use of mini-primers for the specific amplification of short fragments of mitochondrial DNA. The subsequent purification of these amplified fragments allows a standard curve to be constructed with concentrations in accordance to the state of degradation of the samples.
Results: The proposed method successfully detected DNA from ancient samples including bone remains and mummified tissue. DNA inhibitory substances were also detected.
Conclusion: The proposed method represents a simpler and cost-effective way to detect low amounts of aDNA, and a tool to differentiate DNA-free samples from samples with inhibitory substances.
Key words: DNA; DNA, mitocondrial; polymerase chain reaction.
doi: http://dx.doi.org/10.7705/biomedica.v36i3.3098
El análisis de ADN antiguo (ADNa) es un área complementaria de otras disciplinas como la antropología y la arqueología, orientada a la reconstrucción y descripción de migraciones, de la estructura social y las costumbres funerarias de comunidades antiguas (1,2). Dicho tipo de análisis recurre a estrategias de la biología molecular, con el fin de recuperar material genético a partir de especímenes hallados en contextos arqueológicos o en museos (1,3).
A diferencia de otro tipo de muestras sometidas a análisis genéticos, las muestras antiguas, particularmente las recuperadas en contextos arqueológicos, han estado expuestas a agentes ambientales como el sol, los compuestos químicos del suelo y la humedad, entre otros, durante periodos prolongados, y esto genera daños en el ADN que dificultan su obtención y procesamiento para un posterior análisis (4,5). Además, como consecuencia de tales daños, este material genético es más propenso a la contaminación con ADN foráneo, particularmente ADN moderno, el cual puede provenir del personal encargado de la excavación o del laboratorio (5). Considerando estas dificultades, se ha planteado una serie de mínimos requisitos técnicos y de infraestructura a los laboratorios dedicados a este tipo de estudios, con el fin de garantizar que sus resultados provengan del análisis de los restos óseos antiguos y no sean producto de contaminación, es decir que sean auténticos (6).
Uno de los criterios de autenticidad más importantes en el desarrollo de proyectos con ADN antiguo es la cuantificación del número de copias de ADN, con lo cual se trata de relacionar el número de moléculas de ADN obtenidas con el estado de conservación de los restos humanos. En este sentido, lo usual es encontrar escasa cantidad de material genético en estas muestras muy degradadas, en tanto que hallar una gran cantidad de ADN podría suponer la presencia de moléculas contaminantes (7). La cuantificación de ADNa no solo permite inferir la autenticidad de las muestras, sino que aporta información relevante para la implementación o modificación de protocolos de extracción y amplificación acordes con la cantidad del material genético disponible (8).
Considerando la necesidad de implementar sistemas de cuantificación idóneos para la detección de material genético antiguo, se planteó una estrategia que integra el uso de miniiniciadores ( miniprimers ) para la amplificación de fragmentos cortos específicos de ADN mitocondrial, y la detección de dichos fragmentos en tiempo real mediante la determinación de su interacción con una sustancia fluorogénica (9,10).
Materiales y métodos
Selección de la muestra
Para el análisis genético de estas muestras se contó con la autorización de intervención arqueológica emitida por el Instituto Colombiano de Antropología e Historia (ICANH) (Nº 45 para Ubaté y Nº 4194 para Tibanica). Las 25 muestras de hueso analizadas provenían de dos hallazgos arqueológicos de la región central de Colombia (sabana de Bogotá). El material óseo recuperado del sitio arqueológico Tibanica (Soacha), se ha asociado a las siguientes fechas determinadas por 14 C: 940 ± 40 d. C., 1.180 ± 40 d. C. y 1.350 ± 40 d. C.. El material óseo de Ubaté se encontró en un sitio a cielo abierto habitado por cazadores recolectores, y se establecieron dos fechas determinadas por 14 C: 5.655 ± 30 AP y 5.545 ± 30 AP. Asimismo, se procesó tejido muscular proveniente de tres momias pertenecientes a la colección del Museo del Oro de Bogotá (cuadro 1).

Con el fin de construir una curva estándar específica para la cuantificación del ADN mitocondrial, se incluyeron en el estudio un grupo de muestras recientes y una línea celular linfoide (9947A), material genético que se procesó en un espacio diferente al área de trabajo con los especímenes antiguos.
Criterios de autenticidad
Se tuvieron en cuenta los siguientes criterios mínimos de autenticidad:
a) presencia de controles de extracción y amplificación;
b) áreas de trabajo separadas físicamente para los procesos previos y posteriores a la reacción en cadena de la polimerasa (PCR);
c) áreas y reactivos exclusivos para el trabajo con material antiguo;
d) verificación de la repetibilidad y la replicación de los resultados en un laboratorio independiente, y
e) la tipificación y el cotejo de las secuencias obtenidas con los perfiles de los investigadores (6,11).
Todas las posiciones polimorfas detectadas se compararon con los haplotipos de los investigadores encargados de la manipulación de las muestras, cuya verificación estuvo a cargo de grupos de investigación independientes o se hizo mediante la repetición de los ensayos a partir de, por lo menos, dos muestras diferentes del individuo.
Extracción del material genético
De las muestras de hueso se tomaron 3 cm del tercio medio de la diáfisis o fragmentos óseos con un peso aproximado de 3 g, y 30 mg de tejido momificado. En el caso del material óseo, este se sometió a un proceso de limpieza mecánica para remover por completo el periostio y el endostio mediante desgaste, usando lijas de grano de 120. Después de la limpieza mecánica, la pieza ósea se sometió a un proceso adicional de limpieza con luz ultravioleta durante 15 minutos, seguido de lavados con dodecilsulfato sódico (SDS) al 10 %, hipoclorito de sodio libre de metales pesados, etanol y agua Mili-Q ® . La pieza ósea se dejó secar y posteriormente se pulverizó con ayuda del programa automático de un equipo Freezer Mill 6750. El proceso de lavado y pulverización de los tejidos momificados fue igual al de las piezas óseas, pero la exposición a la luz ultravioleta se redujo a cinco minutos, de acuerdo con las modificaciones propuestas por el fabricante para el procesamiento de tejidos blandos (12).
Se tomaron alícuotas de 300 mg del material pulverizado para someterlas a un proceso de digestión con proteinasa K en presencia de agentes emulsificantes y decalcificantes, a una temperatura de 56 °C durante un lapso de tiempo superior a 12 horas. Para la obtención y la purificación del material genético, se consideraron dos estrategias de extracción: con fenol, cloroformo y alcohol isoamílico (25:24:1), según se describe en otro estudio (13), y mediante el uso de resinas de sílice en presencia de sales caotrópicas con el kit QIAamp DNA Investigator Kit ® de la casa comercial QIAGEN, aunque modificando las etapas previas a la interacción del ADN con la membrana para permitir la inclusión de 300 mg de tejido al protocolo descrito por los fabricantes (12).
El material genético reciente se obtuvo a partir de sangre de los investigadores involucrados en el proyecto y se preservó en tarjetas FTA ® siguiendo el protocolo de extracción del fabricante (12).
Cuantificación del material genético y análisis de datos
El material genético extraído a partir de las muestras antiguas y de las diluciones seriadas de la curva estándar se detectó mediante la amplificación de un fragmento específico de 123 pares de bases de la región hipervariable II, haciendo uso de iniciadores reportados por otros autores y del kit Eva Green Dye ®, el cual usa tintes fluorescentes que interactúan con el ADN de doble cadena (9,10). Los resultados se registraron con el sistema de detección 7500 Real-Time PCR System ® .
Las condiciones de termociclaje fueron las siguientes: 98 °C durante 30 segundos, 40 ciclos de 98 °C durante 5 segundos y 55,5 °C durante 30 segundos. Con el fin de evitar la detección de ADN no humano, que puede estar presente en este tipo de muestras, se usaron iniciadores específicos para la amplificación de un segmento de la región hipervariable II del ADN mitocondrial, el cual se verificó mediante una curva de fusión generada bajo las siguientes condiciones: 95 °C durante 15 segundos, 65 °C durante un minuto, 95 °C durante 15 segundos y 65 °C durante 15 segundos.
Se verificaron la veracidad de los resultados de los estándares y las muestras mediante el uso de réplicas en cada ensayo y su repetibilidad mediante múltiples réplicas independientes realizadas en diferentes días.
Diseño de la curva estándar
El material genético amplificado de los controles con ADN reciente se corrió en un gel de agarosa al 2,5 %; se recuperó una banda de 126 pb, la cual corresponde al fragmento de la región hipervariable II desde la posición 34 hasta la 159 del ADN mitocondrial. El ADN libre de dNTP e iniciadores se purificó de la matriz de agarosa mediante el estuche GeneJet Gel Extraction Kit (Thermo Scientific) siguiendo las recomendaciones del fabricante (14).
Este ADN de control purificado se cuantificó mediante espectrofotometría (Nanodrop 1000 , Thermo Scientific) y se lo consideró como el ADN de control para la generación de la curva estándar. A partir de una concentración inicial de 16,6 ng/µl, se generaron diluciones seriadas manualmente con un factor de 1:2 para generar siete puntos en la curva estándar, tal como se ha descrito otros estudios (15). Sin embargo, los ensayos iniciales de esta estrategia de cuantificación indicaron que las muestras procesadas presentaban concentraciones de ADN por debajo de los puntos generados en esta primera curva estándar; por esta razón, fue necesario incluir ocho puntos adicionales partiendo de una concentración de 0,8 ng/µl con un factor de dilución de 1:10. Se rechazaron las curvas estándar con coeficiente de correlación menor de 0,95 y los puntos que se desviaban significativamente de la línea de tendencia.
Resultados
Cuantificación del material genético mitocondrial
Con la metodología propuesta se obtuvo una curva estándar con 15 puntos que abarcaban concentraciones desde 16 hasta 8 x 10 -9 ng/µl (siete copias por µl), con una correlación superior al 95 % (figura 1) y una eficiencia de reacción de 1,5. Debido a que las diluciones seriadas se hicieron manualmente, la curva que se presenta carece de cierta linealidad, sin embargo, el coeficiente de correlación lineal obtenido se ajusta a los estándares sugeridos por otros autores (16,17).
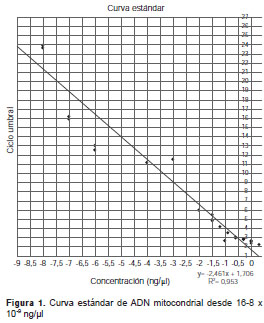
Cuantificación de muestras óseas antiguas
De las dos estrategias de extracción consideradas, solo fue posible detectar material genético a partir de muestras biológicas procesadas con la estrategia de resinas de sílice, sistema de extracción cuya idoneidad ha sido reportada por otros autores (18-20). Con este sistema de extracción y la estrategia de cuantificación propuesta, se logró detectar señales replicables para la mayoría de las muestras con valores de desviación menores de tres ciclos. Estos valores, conjuntamente con las estimaciones obtenidas en la curva estándar, indicaron que las muestras contaban con una concentración promedio de 8,6 x 10 -14 ng/µl; las muestras con concentraciones inferiores no pudieron replicarse (cuadro 2 y cuadro 3).
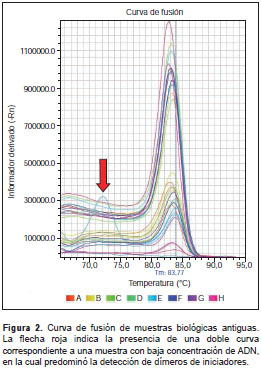
Se determinó la especificidad de la amplificación mediante la determinación de temperaturas de fusión. Las curvas que muestran una temperatura de fusión del fragmento igual a 83,3 °C corresponden a fragmentos con valores de fusión específicos para el segmento de 126 pb amplificado en este ensayo. Además, se detectaron fragmentos con temperaturas de fusión de 68,3 °C, los cuales corresponden a dímeros de iniciadores. Estos artefactos han sido detectados en reacciones previas de PCR, en las que se verificó el producto con geles de agarosa y se evidenció la preferencia por su amplificación en los casos en los que la muestra antigua se caracterizaba por una baja concentración de ADN (figura 2).
Detección de inhibidores
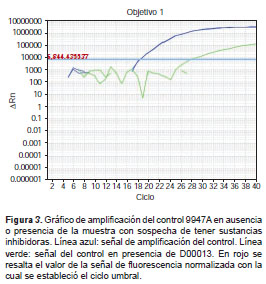
Para la detección de inhibidores en la muestra se agregó 1 µl de una de las muestras con detección o amplificación negativa a la reacción de cuantificación de la línea celular 9947A. Este ensayo se hizo en muestras con resultado negativo, en tres de las cuales se logró detectar el efecto de sustancias que reducen la capacidad de amplificar correctamente el control positivo (figura 3).
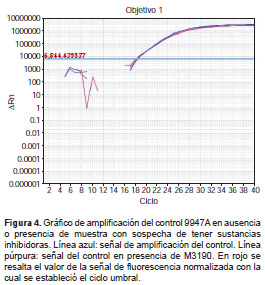
La muestra 3190 no presentó ningún efecto sobre el control positivo, por lo que se consideró que no presentaba agentes inhibidores, y no se amplificó debido a la ausencia de ADN o a su muy baja concentración (figura 4).
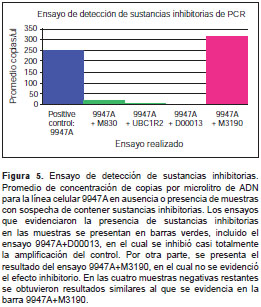
En las muestras que presentaron evidencia de sustancias inhibidoras se detectó una reducción de la amplificación de 97 %, aproximadamente (figura 5).
En ninguno de los ensayos con muestras antiguas o controles negativos se evidenciaron señales que sugirieran la contaminación de alguna de ellas o de los reactivos con ADN reciente.
Discusión
Una de las principales fuentes de información en los análisis de poblaciones antiguas es el ADN mitocondrial, lo cual se debe, en gran medida, a la gran cantidad de copias de este genoma, que pueden llegar a cientos y miles en una célula humana (21), y a la alta tasa de mutaciones, lo cual otorga gran diversidad a esta molécula (22). Pese a estas ventajas, la manipulación del genoma mitocondrial en muestras óseas antiguas también tiene un inconveniente evidente por su propensión a la contaminación con ADN reciente foráneo o con material ambiental. Aunque la cuantificación del material genético obtenido a partir de muestras biológicas antiguas no permite determinar qué moléculas corresponden a contaminantes, sí logra establecer un número inicial de copias y detectar agentes inhibidores. Esto, a su vez, permite plantear estrategias de extracción que maximicen la cantidad de ADN que se puede obtener, así como el uso de agentes potenciadores de la PCR (23).
Las técnicas asociadas a la detección espectrofotométrica y al uso de geles de agarosa son relativamente económicas y de ejecución sencilla, sin embargo, sus limitaciones para detectar material genético escaso o agentes contaminantes que interfieren con la interpretación de los resultados son evidentes; en este sentido, las técnicas asociadas a la PCR son mucho más sensibles y específicas (24,25).
Pese a la gran cantidad de opciones comerciales disponibles para la cuantificación de material genético, no existe un producto que satisfaga la necesidad de determinar la concentración del ADN mitocondrial presente en la muestra, como tampoco existe una alícuota comercial de ADN mitocondrial extraído, que sirva como referente para plantear una estrategia de cuantificación específica.
En vista de esto, otros autores han propuesto una estrategia que involucra la amplificación específica de un fragmento de ADN mitocondrial seguida de su purificación y posterior cuantificación por espectrofotometría, con el fin de generar una curva estándar específica para cuantificar el ADN mitocondrial (15). Pese a ello, las técnicas que dependen de la detección de sondas marcadas con flúor requieren de una inversión adicional, lo cual eleva los costos de la investigación. Además, los sistemas propuestos por otros autores dependen de la amplificación de fragmentos mayores de 200 pb, los cuales no logran ser detectados en muestras seriamente degradadas (26,27).
En el presente trabajo se consideró el uso de curvas estándar a partir de muestras amplificadas y posteriormente purificadas; sin embargo, se incorporó el uso de amplicones más cortos que favorecen la detección de ADN a partir de muestras muy degradadas, y se logró un sistema de detección más económico y asequible como lo es la metodología basada en la detección de ADN de doble cadena mediante el uso de tintes fluorescentes.
La validez de los resultados obtenidos se estimó verificando su replicación, tal como lo recomiendan otros autores para la detección de ADN de muestras muy degradadas (16,17). Se ha reportado, asimismo, que el caso de las muestras en las que no se obtuvieron resultados replicables coincide con el de aquellas en las que se reportan menos de 100 copias por µl (28), como en este estudio. La baja concentración de dichas muestras también afectó su eficiencia en las etapas posteriores de amplificación y secuenciación; sin embargo, la autenticidad de este ADN pudo verificarse mediante el cotejo de los haplotipos mitocondriales obtenidos de las muestras con los perfiles de los investigadores, así como mediante la verificación de las secuencias de ADN mitocondrial en ensayos independientes hechos por el mismo laboratorio y por otros grupos de investigación (29).
Además de los beneficios de sensibilidad y especificidad de esta estrategia, el sistema permitió la detección de agentes inhibidores, los cuales también representan un reto al momento de procesar este tipo de muestras, lo cual representa un gran beneficio metodológico, pues permite implementar estrategias de amplificación diferenciales acordes con la dificultad particular que presente la muestra.
A la Universidad de los Andes, especialmente a la Rectoría, al Laboratorio de Genética Humana de la Facultad de Ciencias y al Grupo de Arqueología de la Facultad de Ciencias Sociales, por su apoyo incondicional en el desarrollo del proyecto, así como en su financiación y la adecuación de un espacio especialmente diseñado para el trabajo con este tipo de muestras. Al Banco de ADN de la Universidad del País Vasco (Vizcaya, España), por su colaboración en los ensayos de repetibilidad de los resultados de secuenciación, y a María Antonieta Corcione, por la información antropológica aportada.
Los autores de este trabajo declaramos que no se presentó ningún tipo de conflicto de intereses que pudiera afectar los resultados del presente trabajo.
Este trabajo fue financiado en su totalidad por el Fondo de Investigaciones de la Facultad de Ciencias, con apoyo de la Vicerrectoría de Investigaciones y el Laboratorio de Genética Humana de la Universidad de los Andes.
Correspondencia:
Luz Adriana Pérez, Carrera 51A N° 127-75, apartamento 1201, interior 1, Bogotá, D.C., Colombia
Teléfono: (1) 339 4949, extensión 2790
1. Hagelberg E, Hofreiter M, Keyser C. Ancient DNA: The first three decades. Philos Trans R Soc Lond B Biol Sci. 2015;370:20130371. http://dx.doi.org/10.1098/rstb.2013.0371. [ Links ]
2. Kaestle FA, Horsburgh A. Ancient DNA in anthropology: Methods, applications, and ethics. Yearb Phys Anthr. 2002;130:92-130. [ Links ]
3. Willerslev E, Cooper A. Ancient DNA. Proc Biol Sci. 2005;272:3-16. http://dx.doi.org/10.1098/rspb.2004.2813 [ Links ]
4. Holliday R, Grigg GW. DNA methylation and mutation. Mutat Res. 1993;285:61-7. http://dx.doi.org/10.1016/0027-5107(93)90052-H [ Links ]
5. Höss M, Jaruga P, Zastawny TH, Dizdaroglu M, Pääbo S. DNA damage and DNA sequence retrieval from ancient tissues. Nucleic Acids Res. 1996;24:1304-7. http://dx.doi.org/10.1093/nar/24.7.1304 [ Links ]
6. Gilbert MTP, Bandelt HJ, Hofreiter M, Barnes I. Assessing ancient DNA studies. Trends Ecol Evol. 2005;20:541-4. http://dx.doi.org/10.1016/j.tree.2005.07.005 [ Links ]
7. Cooper A, Poinar H. Ancient DNA, do it right or not at all. Science. 2000;289:1139. http://dx.doi.org/10.1126/science.289.5482.1139b [ Links ]
8. Krings M, Stone A, Schmitz RW, Krainitzki H, Stoneking M, Pääbo S. Neanderthal DNA sequences and the origin of modern humans. Cell. 1997;90:19-30. http://dx.doi.org/10.1016/S0092-8674(00)80310-4 [ Links ]
9. Gabriel MN, Huffine EF, Ryan JH, Holland M, Parsons T. Improved MtDNA sequence analysis of forensic remains using a " mini-primer set " amplification strategy. J Forensic Sci. 2001;46:247-53. [ Links ]
10. Bio-Rad Laboratories. SsoFast TM EvaGreen ® Supermix 200. Hercules, CA, USA: Bio-Rad Laboratories; 2014. [ Links ]
11. Butler J. Forensic DNA typing. Biology, technology, and genetics of STR markers. Second edition. Burlington, MA: Academic Press; 2005. [ Links ]
12. QIAGEN. QIAamp® DNA Investigator. Sample and Assay Technologies. Valencia, CA: QIAGEN; 2010. [ Links ]
13. Loreille OM, Diegoli TM, Irwin JA, Coble MD, Parsons TJ. High efficiency DNA extraction from bone by total demineralization. Forensic Sci Int Genet. 2007;1:191-5. http://dx.doi.org/10.1016/j.fsigen.2007.02.006 [ Links ]
14. Thermo Fisher Scientific Inc. GeneJET Gel Extraction Kit. Waltham, MA, USA: Thermo Fisher Scientific Inc; 2012. [ Links ]
15. Alonso A, Albarrán C, Martín P, García P, García O, De la Rúa C, et al . Multiplex – PCR of short amplicons for mtDNA sequencing from ancient DNA. Int Congr Ser. 2003;1239:585-8. http://dx.doi.org/10.1016/S0531-5131(02)00401-6 [ Links ]
16. Kavlick MF, Lawrence HS, Merritt RT, Fisher C, Isenberg A, Robertson JM, et al . Quantification of human mitochondrial DNA using synthesized DNA standards. J Forensic Sci. 2011;56:1457-63. http://dx.doi.org/10.1111/j.1556-4029.2011.01871.x [ Links ]
17. Sprouse ML, Phillips NR, Kavlick MF, Roby RK. Internal validation of human mitochondrial DNA quantification using real-time PCR. J Forensic Sci. 2014;59:1049-56. http://dx.doi.org/10.1111/1556-4029.12477 [ Links ]
18. Young H, Jin M, Young N, Eun J, Ick W, Shin K. Simple and highly effective DNA extraction methods from old skeletal remains using silica columns. Forensic Sci Int Genet; 2010;4:275-80. http://dx.doi.org/10.1016/j.fsigen.2009.10.014 [ Links ]
19. Yang DY, Eng B, Waye JS, Dudar JC, Saunders SR. Technical note: Improved DNA extraction from ancient bones using silica-based spin columns. Am J Phys Anthropol. 1998;105:539-43. http://dx.doi.org/10.1002/(SICI)1096-8644(199804)105:4<539::AID-AJPA10>3.0.CO;2-1 [ Links ]
20. Anderung C, Persson P, Bouwman A. Fishing for ancient DNA. Forensic Sci Int Genet. 2008;2:104-7. http://dx.doi.org/10.1016/j.fsigen.2007.09.004 [ Links ]
21. Robin ED, Wong R. Mitochondrial DNA molecules and virtual number of mitochondria per cell in mammalian cells. J Cell Physiol. 1988;136:507-13. http://dx.doi.org/10.1002/jcp.1041360316 [ Links ]
22. Stoneking M. Hypervariable sites in the mtDNA control region are mutational hotspots. Am J Hum Genet. 2000;67: 1029-32. http://dx.doi.org/10.1086/303092 [ Links ]
23. Keyser-Tracqui C, Ludes B. Methods for the study of ancient DNA. Methods Mol Biol. 2005;297:253-64. [ Links ]
24. Lee SB, Mccord B, Buel E. Advances in forensic DNA quantification. Electrophoresis. 2014;35:3044-52. http://dx.doi.org/10.1002/elps.201400187 [ Links ]
25. Nielsen K, Smidt H, Hedman J, Parson W, Morling N, Niedersta H. Comparison of five DNA quantification methods. s.f 2008;2:226-30. http://dx.doi.org/10.1016/j.fsigen.2008.02.008 [ Links ]
26. Lamers R, Hayter S, Matheson CD. Postmortem miscoding lesions in sequence analysis of human ancient mitochondrial DNA. J Mol Evol. 2009;68:40-55. http://dx.doi.org/10.1007/s00239-008-9184-3 [ Links ]
27. Gotherstrom A, Collins MJ, Angerbjorn A, Liden K. Bone preservation and DNA amplification. Archeometry. 2002;44:395-404. http://dx.doi.org/10.1111/1475-4754.00072 [ Links ]
28. Handt O, Krings M, Ward RH, Pääbo S. The retrieval of ancient human DNA sequences. Am J Hum Genet. 1996;59:368-76. [ Links ]
29. Pérez LA, Groot H, Langebaek CH. Aportes genéticos para el entendimiento de la organización social de la comunidad muisca Tibanica (Soacha, Cundinamarca) (tesis). Bogotá: Universidad de los Andes; 2016. [ Links ]














