La aparición y diseminación de cepas resistentes a los principales fármacos disponibles para el tratamiento de la tuberculosis constituyen un gran problema para el control de la enfermedad a nivel mundial, debido al aumento de casos de tuberculosis multirresistente (TB-MDR), de aquellos causados por Mycobacterium tuberculosis con resistencia in vitro a isoniacida y rifampicina, y de casos extremadamente resistentes (TB-XDR), en los cuales se presenta, además, resistencia a las fluoroquinolonas y a los medicamentos inyectables de segunda línea.
La Organización Mundial de la Salud (OMS) estimó que en el 2014 se diagnosticaron 9,6 millones de casos nuevos de tuberculosis, de los cuales 480.000 serían multirresistentes, pero las dos estimaciones resultaron inferiores a los casos efectivamente notificados a nivel mundial 1. En ese mismo año, en Colombia se reportaron 12.824 casos de tuberculosis en todas sus formas, de los cuales 103 fueron multirresistentes y, dos, extremadamente resistentes 2,3.
En su plan de respuesta a esta amenaza, la OMS sugiere la implementación de pruebas de laboratorio rápidas para la tamización de la resistencia. En el 2008, recomendó el uso de métodos moleculares del tipo de sondas en línea, entre los cuales se cuenta la prueba comercial GenoType(r) MTBDRplus (Hain Lifescience GmbH, Nehren, Germany), con sensibilidad de 97 % y especificidad de 99 % para rifampicina, y de 90 % y 99 % para isoniacida en muestras pulmonares con baciloscopia positiva o aislamientos de M. tuberculosis en medios de cultivo sólidos o líquidos, con lo cual se obtienen resultados rápidos, en especial, cuando se trabaja directamente con muestras 4,5.
La metodología de GenoType(r) MTBDRplus V.2 se basa en la amplificación del ADN mediante reacción en cadena de la polimerasa (PCR) e hibridación inversa, lo cual permite detectar el complejo M. tuberculosis, y las mutaciones responsables de la resistencia a la rifampicina en el gen rpoβ y las determinadoras de resistencia a la isoniacida en los genes katG e inhA. Esta técnica ha sido evaluada por los programas nacionales de control de la tuberculosis en varios países de la región, pues, además de la presencia de resistencia, su uso permite determinar la variabilidad genética de M. tuberculosis a nivel mundial.
El diagnóstico de tuberculosis en Colombia se hace principalmente mediante baciloscopia y cultivo, según lo establecido por el Laboratorio Nacional de Referencia para la Red Nacional de Laboratorios. Desde el 2012, la Red ha venido incorporando los nuevos métodos moleculares y hoy el país cuenta con 22 laboratorios de salud pública y de instituciones prestadoras de servicios de salud que cuentan con este tipo de pruebas, así como el Laboratorio Nacional de Referencia, lo cual favorece la oportunidad en la detección de los casos resistentes.
El objetivo de este trabajo fue describir las mutaciones del gen rpoβ que confieren resistencia a la rifampicina y las de los genes katG e inhA de resistencia a la isoniacida, detectadas mediante la técnica GenoType(r) MTBDRplus V.2 en muestras de esputo y aislamientos de M. tuberculosis procesadas en el Laboratorio Nacional de Referencia durante el 2014.
Materiales y métodos
Este es un estudio de tipo retrospectivo descriptivo.
Muestras de esputo
El Laboratorio Nacional de Referencia recibió 167 muestras de esputo para diagnóstico de tuberculosis resistente procedentes de 27 laboratorios de salud pública del país, las cuales se descontaminaron y se concentraron mediante el método de NaOHN- acetil-L-cisteína. La baciloscopia se hizo con la coloración de Ziehl-Neelsen, cultivo en medio de Löwenstein-Jensen y en medio líquido BBLTM MGITTM (Mycobacteria Growth Indicator Tube), siguiendo las recomendaciones de los manuales de la Organización Panamericana de la Salud (OPS) para baciloscopia y cultivo sólido, y las indicaciones de la casa comercial (Becton-Dickinson) para el cultivo en medio líquido.
Aislamientos de Mycobacterium tuberculosis
Se recibieron 670 aislamientos de M. tuberculosis de muestras sembradas en los laboratorios de la Red según los protocolos y manuales de procesamiento de cada institución, los cuales deben enviarse a los laboratorios de salud pública y, de ahí, al Laboratorio Nacional de Referencia cuando hay crecimiento, para hacer la prueba de sensibilidad. Todos los resultados obtenidos mediante la prueba molecular fueron confirmados por la técnica Bactec MGITTM, considerada como la prueba de referencia.
Extracción del ADN
El ADN se extrajo utilizando el estuche l GenoLyse(r) DNA Extraction, según instrucciones del fabricante.
Genotype MTBDR plus V.2
Se empleó la prueba siguiendo las instrucciones descritas en el inserto del estuche comercial. Para la amplificación por PCR se utilizó un termociclador de HAIN Lifesciences con los protocolos "MDR DIR" para muestras y "MDR CUL" para cultivos. La hibridación se hizo en un equipo Twincubator(r). Se aplicaron controles internos para descartar contaminación con el ADN mediante la evaluación de los reactivos del GenoLyse y las condiciones previas de mezcla de la PCR.
En la prueba se emplean tiras de nitrocelulosa, las cuales tienen 23 sondas; las primeras tres son los controles internos de la prueba (control de conjugado, de amplificación y de detección del complejo M. tuberculosis). Para la detección de la resistencia a rifampicina, hay un control de locus y ocho sondas silvestres (wildtype, WT) que permiten analizar el segmento entre el codón 505 y el 533, y cuatro sondas específicas para las mutaciones (MUT): Asp516Val, His526Tyr, His526Asp y Ser531Leu. Para la identificación de la resistencia a la isoniacida de alto nivel hay un control de locus, una sonda silvestre que analiza el codón 315 del gen katG y dos sondas específicas para las mutaciones Ser315Thr1 y Ser315Thr2. Para determinar la resistencia a isoniacida de bajo nivel, hay un control de locus, dos sondas silvestres y cuatro sondas para las mutaciones C15T, A16G, T8C y T8A. La lectura de los resultados de las pruebas, así como la interpretación de los patrones de bandas, se hicieron de forma visual utilizando la plantilla incluida en el estuche.
Análisis estadístico de los datos
Se recolectaron y analizaron los datos en una base de datos en Excel(r), en la cual se registró el patrón de bandas observado.
Resultados
Se hicieron 837 pruebas, 670 de cultivos y 167 de muestras de esputo. Se obtuvieron 689 resultados de pruebas, de las cuales 581 (84,3 %) resultaron sensibles, 58 (8,4 %), resistentes, y 50 (7,2 %), multirresistentes; 28 pruebas no pudieron interpretarse debido a irregularidades en el patrón de las bandas y en 120 no hubo amplificación del ADN (cuadro 1).
Cuadro 1. Resultados obtenidos con la prueba GenoType(r) MTBDRplus V.2
Mutaciones detectadas en los casos resistentes a rifampicina
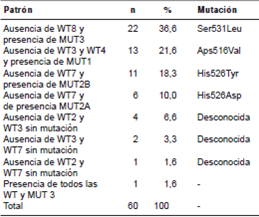
Se consideraron como resistentes los patrones de bandas en los que hubo ausencia de una sonda silvestre y presencia de una sonda de mutación, y los patrones de bandas que presentaron ausencia de dos bandas silvestres sin mutación. En 60 de los aislamientos y muestras, se detectaron mutaciones resistentes a rifampicina.
Se observaron siete patrones de banda diferentes en las tiras de prueba, siendo la ausencia de WT8 y la presencia de MUT3 el patrón más frecuente (22 casos, 36,6 %), el cual corresponde a la mutación Ser531Leu. En 13 (21,6 %) de los aislamientos, se evidenció la ausencia de WT3 y de WT4 con presencia de MUT1, siendo este el segundo patrón más frecuente. Se observó un patrón de heterorresistencia en un aislamiento que presentaba todas las sondas WT y una sonda MUT3 (cuadro 2).
Cuadro 2. Patrones de bandas de resistencia a rifampicina
Mutaciones detectadas en los casos resistentes a isoniacida
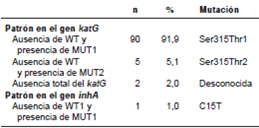
Se encontraron 98 aislamientos que presentaron resistencia a isoniacida. El 99 % de las mutaciones ocurrió en el gen KatG. El patrón más frecuente fue la ausencia de WT y la presencia de MUT1 en 90 aislamientos (91,9 %), el cual corresponde a la mutación Ser315Thr1 con resistencia de alto nivel.
Se observó un patrón con ausencia total del gen katG, el cual se interpretó como resistente, y un aislamiento con mutación en el gen inhA y un patrón de ausencia de WT1 y presencia de MUT1, el cual corresponde a la mutación C15T (cuadro 3).
Cuadro 3. Patrones de bandas de resistencia a isoniacida
Resultados no interpretables
Se obtuvieron 28 pruebas con resultados no interpretables. Para rifampicina, se encontraron patrones de bandas que solo presentaban ausencia de una sonda WT y ausencia de sondas MUT, o con alguna sonda WT de poca intensidad en el color del revelado, es decir, muy tenue. Se observaron patrones con ausencia de WT8, WT7, WT6 y WT2, que tampoco fueron concluyentes.
Como parte de la metodología de procesamiento en el LNR, todas las pruebas moleculares se confirmaron con el sistema Bactec MGITTM, en tanto que en los casos no interpretables el reporte de resultados se hizo mediante el método convencional.
Discusión
La implementación de técnicas moleculares para el diagnóstico de la tuberculosis resistente y multirresistente es relativamente reciente en el país. Es importante que la Red Nacional de Laboratorios continúe promoviendo su adopción en aquellas instituciones que cuentan con la capacidad técnica y el recurso humano, pues este proceso de descentralización fortalece las actividades de detección y diagnóstico del programa, ya que se emplean métodos que cuentan con el aval de la OMS y la OPS por haber comprobado su idoneidad a nivel mundial y sus ventajas, tales como la rapidez en la obtención de resultados, aproximadamente seis horas en muestras con baciloscopia positiva. En aquellos casos con uno a nueve bacilos ácidoalcohol resistentes en 100 campos microscópicos observados, o con baciloscopia negativa, no se recomienda el uso de esta prueba, ya que no se logra la amplificación del ADN. La implementación de este tipo de pruebas de forma rutinaria favorecería la tamización de la resistencia y, en consecuencia, el inicio del tratamiento adecuado.
En varios países se ha implementado este tipo de pruebas con base en los datos de concordancia con las técnicas convencionales y la frecuencia de las mutaciones evaluadas 6-9. La divulgación de tales resultados ha permitido compartir la información sobre la variabilidad genética de las cepas circulantes en las regiones del mundo, la presencia de diferentes mutaciones y si confieren resistencia a los fármacos o no. Por esta razón, es importante que las redes nacionales de laboratorios que decidan comenzar a aplicar estas técnicas evalúen la pertinencia de su uso, determinando si las mutaciones reconocidas corresponden a las mismas que el estuche comercial detecta para, así, validar su utilidad como método de diagnóstico 10.
Con respecto a la resistencia a la rifampicina, en este estudio se encontró que el 36,6 % (22/60) de los casos resistentes presentaban el patrón de ausencia de WT8 y presencia de MUT3, lo cual corresponde a la mutación Ser531Leu. Se observó, igualmente, el patrón de ausencia de WT3 y WT4 y presencia de MUT1 en 21,6 % (13/60) de las muestras, patrón que corresponde a la mutación Asp516Val, en tanto que en el 18,3 % (11/60) de las muestras se presentó el patrón de ausencia de WT7 y presencia de MUT2B, es decir, la mutación His526Tyr.
En estudios en otros continentes se ha evidenciado que la mutación Ser531Leu es la más frecuente, con porcentajes entre 41 y 73 % 11-13. En la región de las Américas, Chile, Argentina y Perú, reportan frecuencias entre 56 y 67 % 14,15. En Colombia, Ferro, et al., registraron una frecuencia de 64 % para esta misma mutación en aislamientos provenientes de la región del Valle del Cauca 16).
Al comparar estos datos con los obtenidos en nuestro estudio, es claro que hay diferencias en las proporciones encontradas, lo cual demuestra que es necesario verificar la utilidad de estas pruebas como un ejercicio rutinario para decidir con respecto a su implementación como método de diagnóstico.
Dicha evaluación debe considerar la epidemiología de la enfermedad, pues es posible que existan variaciones, incluso entre zonas con alta prevalecía de resistencia a fármacos, como puede verse por las diferencias entre los resultados de este trabajo y lo reportado por Ferro, et al.16.
La resistencia a isoniacida se presentó en 99 % de los aislamientos debido a mutaciones en el gen katG, siendo la mutación Ser315Thr1 la más frecuente (91,9 %; 90/98). Al comparar estos datos con los obtenidos por Asencios, et al., en Perú 15, donde la mutación en katG más frecuente fue la MUT1, con 71,2 % de los casos, los datos del presente estudio fueron similares en términos de frecuencia. En el estudio de Tessema 11, se encontró que el 100 % de las mutaciones estaban en la MUT1, lo cual coincide con lo reportado en el presente trabajo y resalta la variabilidad genética.
Solo se presentó un aislamiento con resistencia en el gen inhA. En otros estudios en la región de las Américas, esta mutación se ha reportado con poca frecuencia, sin embargo, es fundamental que los programas realicen un estudio más detallado de su prevalencia y relevancia dado que el profesional clínico debe definir un esquema de tratamiento del caso con base en tales resultados 14,15.
Se reportaron 23 (3,34 %) casos no interpretables en cuanto a su resistencia a rifampicina debido a la ausencia de una sonda WT (WT8, WT7, WT6 y WT2) y de la sonda MUT, lo cual sugiere la presencia de mutaciones en el gen rpoβ que no están presentes en la tira reactiva de este estuche comercial, y plantea la necesidad de hacer estudios para determinar mejor su frecuencia. Alonso, et al.17, reportaron que la ausencia de la WT3 sin mutación puede presentarse cuando hay mutaciones silenciosas; puesto que esta es una de las limitaciones de los métodos estudiados, en estos casos es fundamental hacer una prueba fenotípica, idealmente con técnicas de secuenciación para determinar si se trata de mutaciones poco frecuentes y cuál es su relevancia clínica.
En Colombia, se identificaron diversas mutaciones del gen rpoβ que generaban resistencia a la rifampicina, de las cuales la más frecuente fue la Ser531Leu, aunque en menor porcentaje que en otros países. En cuanto a la resistencia a isoniacida, la mutación más común asociada a la resistencia de alto nivel fue la Ser315Thr1 en el gen katG.
Con estos datos puede concluirse que la técnica de GenoType(r) MTBDRplus V.2 permite reconocer las mutaciones más frecuentes asociadas a la resistencia y que, en los casos con resultados no interpretables, debe considerarse la confirmación mediante pruebas fenotípicas e incluir también a aquellos pacientes cuya evolución clínica y bacteriológica sugiere resistencia.
Una característica importante de M. tuberculosis es que los mecanismos de su resistencia a los fármacos no están completamente descritos y, aunque la presencia de mutaciones es el principal, deben considerarse otros como las bombas de expulsión activa, en el cual el diagnóstico de farmacorresistencia solo se logra mediante una prueba convencional 18.
Se evidencian, así, nuevas oportunidades de investigación para evaluar las mutaciones que no se habían detectado antes, así como su relevancia clínica para el país.