La tuberculosis es un problema de salud pública a nivel mundial. Durante el 2012, la Organización Mundial de la Salud (OMS) estimó 8,6 millones de casos a nivel mundial, y 1,3 millones de muertes por esta causa 1. Muchos factores se han asociado durante la última década al incremento en el número de casos de la enfermedad, una tercera parte del cual se atribuye a la propagación del virus de la inmunodeficiencia humana (HIV) 2. Durante ese año, en el mundo se presentaron 1,1 millones de casos de infección concomitante de tuberculosis y HIV, de los cuales 31.000 se registraron en Latinoamérica.
En el 2012, se diagnosticaron en Colombia 11.990 casos de pacientes con tuberculosis en todas las formas, de los cuales 7.572 (63,2 %) habían solicitado la prueba voluntaria para HIV, y 1.397 (11,8 %) presentaban infección concomitante de tuberculosis y HIV 3.
Desde 2009, el Programa Nacional de Control de Tuberculosis definió los grupos de riesgo de tuberculosis mediante la circular 058, entre ellos, los casos de personas con HIV, cuyo diagnóstico se debe hacer por baciloscopia, cultivo y prueba de sensibilidad a fármacos antituberculosos 4. En el Grupo de Micobacterias de la Dirección de Redes en Salud Pública del Instituto Nacional de Salud, se reciben los cultivos de casos de tuberculosis provenientes de algunos de los entes territoriales, con el fin de hacer las pruebas de sensibilidad en el marco de las actividades de vigilancia en salud pública.
Además, en el Grupo de Micobacterias, se hacen estudios de epidemiología molecular para conocer la cadena de transmisión de la enfermedad, establecer las causas de las recurrencias y proveer información para determinar la transmisión activa, evidenciada por la circulación de genotipos y la presencia de agrupamientos, información que contribuye al control de la enfermedad en diferentes poblaciones, especialmente en los grupos de riesgo, como es el caso de pacientes con infección concomitante de tuberculosis y HIV 5-8. En los países en donde la tuberculosis y la infección por HIV son endémicas, es común encontrar casos agrupados en los análisis de epidemiología molecular, lo cual demuestra la expansión epidémica de la tuberculosis asociada al HIV 6,9-10.
Por lo tanto, es importante el desarrollo de estudios que analicen la repercusión de la infección por el HIV en la transmisión y la adquisición de la resistencia a los medicamentos, como parte de las prioridades en investigación cuando los recursos son limitados y dicha infección es prevalente 11. En este contexto, los análisis de transmisión mediante estudios de epidemiologia molecular y la vigilancia de la resistencia a los fármacos deben hacerse rutinariamente en este grupo de pacientes.
El objetivo de este trabajo fue caracterizar aislamientos clínicos de Mycobacterium tuberculosis de individuos positivos para el HIV, recibidos en el Laboratorio Nacional de Referencia de Micobacterias del Instituto Nacional de Salud durante el primer semestre de 2012.
Materiales y métodos
Tipo de estudio
Se hizo un estudio observacional y descriptivo para caracterizar los aislamientos de casos con infección concomitante de tuberculosis e infección por HIV en Colombia, recibidos en el primer semestre de 2012. En el análisis se tuvieron en cuenta las variables sociodemográficas relacionadas con el paciente, los perfiles de resistencia a fármacos antituberculosos y la genotipificación de los aislamientos.
Aislamientos de Mycobacterium tuberculosis
Se analizaron 63 aislamientos provenientes de individuos con tuberculosis e infección por HIV, registrados en el formato único de vigilancia de micobacterias en la categoría de "persona que convive con el HIV", y con resultado en la prueba de sensibilidad a los fármacos antituberculosos. Dichos registros fueron remitidos al Instituto Nacional de Salud por los laboratorios de salud pública de aquellos entes territoriales que no cuentan con estas pruebas.
Pruebas de laboratorio
Identificación de especie y sensibilidad. Los aislamientos se sometieron a identificación fenotípica, mediante pruebas bioquímicas y enzimáticas que detectan el complejo M. tuberculosis12. Para determinar los patrones de sensibilidad, se utilizó la técnica semiautomatizada en medio líquido Bactec MGITTM, en la cual se emplea un tubo que contiene caldo Middlebrook 7H9 y un compuesto de rutenio fluorescente incluido en silicona, y se fundamenta en la acción de consumo de oxígeno por parte de la micobacteria, lo cual libera la fluorescencia del compuesto y permite detectar su viabilidad al ser expuesta a los fármacos para determinar si es sensible o resistente. Las concentraciones críticas usadas fueron las siguientes: estreptomicina, 1 y 4 μg/ml; isoniacida, 0,1 y 0,4 μg/ml; rifampicina, 1 μg/ml, y etambutol, 1 y 7,5 μg/ml. En la interpretación de los resultados se utilizó el programa Epicenter 13,14.
La calidad de las pruebas de sensibilidad a los fármacos fue evaluada por el Instituto de Salud pública de Chile, el cual lleva a cabo un control anual a los laboratorios nacionales en el marco del programa mundial de vigilancia de la resistencia.
Tipificación mediante spoligotyping y Mycobacterial interspersed repetitive unitvariable number (MIRU-VNTR)
La extracción de ADN y la aplicación de la metodología de spoligotyping se hicieron según lo expuesto por Kamerbeek, et al., en 1997. La tipificación con oligonucleótidos espaciadores (spoligotyping) es una técnica basada en la reacción en cadena de la polimerasa (PCR), útil para la discriminación de especies del complejo M. tuberculosis mediante la detección de espaciadores entre las repeticiones directas del locus DR 15. La metodología de MIRUVNTR de 24 loci se hizo de forma manual mediante la amplificación por PCR de los 24 minisatélites y la visualización de los amplificados en geles de agarosa, según el protocolo descrito por Supply, et al., en 2006 16. Desde 2013, el Laboratorio Nacional de Referencia del Instituto Nacional de Salud garantiza la calidad de las pruebas de MIRUVNTR mediante el control de calidad del Laboratorio de Referencia para Tuberculosis del Ministerio de Salud, Bienestar y Deportes de Holanda.
Análisis de agrupamientos
El agrupamiento se definió como dos o más aislamientos de M. tuberculosis con un patrón idéntico determinado mediante spoligotyping y MIRU.
Análisis molecular y designación de familias genéticas
Los patrones obtenidos por spoligotyping se compararon con la base de datos internacional SITVIT del Instituto Pasteur de Guadalupe (http://www.pasteur-guadeloupe.fr:8081/SITVIT_ONLINE), y se designaron las familias genéticas y los códigos SIT (Spoligo International Type) 17.
La comparación de los resultados obtenidos mediante las dos metodologías, así como la generación de los dendrogramas y los árboles de expansión mínima (Minimum Spanning Tree, MST), se hicieron con el programa VNTR Plus. Para la construcción de los árboles MST, se usó el algoritmo neighbor-joining18.
Los patrones de spoligotyping que no se encontraron en la base de datos SITVIT (patrones huérfanos), se analizaron mediante la herramienta SpotClust (http://tbinsight.cs.rpi.edu/run_spotclust. html SPOTCLUST), la cual hace una predicción de las familias genéticas de M. tuberculosis mediante el uso de un modelo derivado de la base de datos SpolDB3, con base en el análisis de los algoritmos de evolución de los patrones de spoligotyping19.
Análisis de los datos
Se utilizaron tres fuentes de información. La primera correspondió al formato único de vigilancia de las micobacterias, documento establecido por el Instituto Nacional de Salud para el procesamiento de las pruebas de sensibilidad, el cual se recibe con cada cultivo y contiene información sobre el sexo y la edad de los pacientes, la forma clínica de la enfermedad, el ente territorial remitente y la condición de ingreso al programa de cada paciente (nuevo o previamente tratado), datos que deben ser recopilados por el personal de salud en la institución donde se los atiende. La segunda fueron los resultados de las pruebas de sensibilidad para la detección de los patrones de resistencia a los fármacos, y la tercera, la base de datos de los resultados de la genotipificación específica para este estudio.
Se describieron las variables sociodemográficas de los casos y su distribución geográfica empleando el programa EPI Info(tm), versión 7.1.3.
Se calculó la proporción de resistencia a fármacos, teniendo en cuenta la condición de ingreso de la persona al programa de tuberculosis (nuevo o previamente tratado), y los porcentajes de aislamientos sensibles y resistentes en cada una de las familias genéticas identificadas, con un intervalo de confianza de 95 %. Las diferencias entre las proporciones de resistencia a fármacos según familias genéticas se establecieron mediante la prueba exacta de Fisher, tomando como referencia la familia genética de M. tuberculosis con mayor número de aislamientos resistentes en el estudio, con un intervalo de confianza de 95 %, y una significación estadística de p<0,05.
Resultados
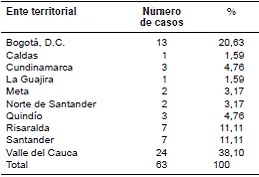
El análisis de las variables sociodemográficas evidenció que 37 (58,7 %) casos correspondían a pacientes de sexo masculino; el rango de edad osciló entre 19 y 59 años, con una mediana de 32. En 51 (80,9 %) casos se presentaron formas pulmonares y en 60 (95,2 %) la condición de ingreso al programa correspondió a pacientes nuevos, es decir, sin antecedente de tratamiento antituberculoso. Los casos analizados provenían de diez entes territoriales, principalmente del departamento del Valle del Cauca, con 24 (38,1 %) y Bogotá, con 13 (20,6 %) (cuadro 1).
Cuadro 1 Procedencia de los 63 casos de infección concomitante de tuberculosis y HIV incluidos en el estudio
Todos los aislamientos de los casos en estudio pertenecían al complejo M. tuberculosis, según las pruebas fenotípicas utilizadas. Al evaluar los patrones de resistencia en los 60 aislamientos provenientes de casos nuevos, se encontró que 54 (90 %) eran sensibles, dos (3,3 %) presentaron resistencia solo a la rifampicina, uno (1,6 %), resistencia a isoniacida, y tres (5 %), resistencia combinada a la estreptomicina y la isoniacida. La resistencia global para este grupo fue de 10 %. Los tres aislamientos (4,7 %) de casos previamente tratados fueron sensibles.
Los aislamientos de los 63 casos pertenecían a 11 familias genéticas de M. tuberculosis (cuadro 2). Diecinueve (30,1 %) de los resultados obtenidos por spoligotyping no mostraron similitud con los patrones de la base de datos SITVIT, y se consideraron patrones huérfanos. Entre las familias descritas en la base de datos mundial, se encontraron la LAM9, la H1 y la T1, con una frecuencia de 20 (31,7 %), ocho (12,7 %) y siete (11,1 %) casos, respectivamente. Los códigos SIT más frecuentes fueron el 42 (LAM9), el 53 (T1) y el 62 (H1).
Cuadro 2 Frecuencia de las familias de complejo M. tuberculosis encontradas en 63 aislamientos de personas con infección concomitante de tuberculosis y HIV, y análisis de las diferencias entre las proporciones de resistencia a fármacos según familias genéticas
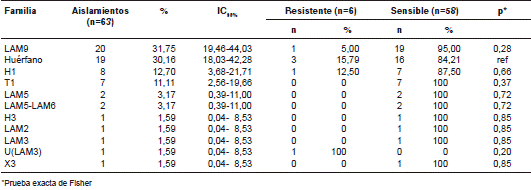
Mediante el uso de SpotClust para la predicción de las familias genéticas de M. tuberculosis, los 19 casos huérfanos se clasificaron como sigue: ocho pertenecían a la familia H1, cinco, a LAM9, y dos aislamientos, a cada una de las familias T1, S y LAM2 (cuadro 3). No se encontraron diferencias estadísticamente significativas entre las proporciones de resistencia a los fármacos entre las familias genéticas de M. tuberculosis encontradas al tomar como referencia el patrón huérfano (cuadro 2). El análisis de los aislamientos por spoligotyping y MIRU-VNTR en el MST evidenció una gran prevalencia de la familia LAM (figura 1).
Cuadro 3 Resultados del análisis de los 19 patrones huérfanos de aislamientos de M. tuberculosis procedentes de pacientes con infección concomitante de tuberculosis y HIV mediante la herramienta SpotClust
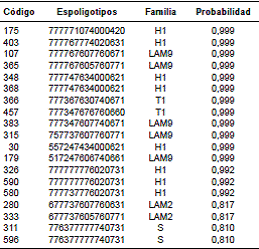
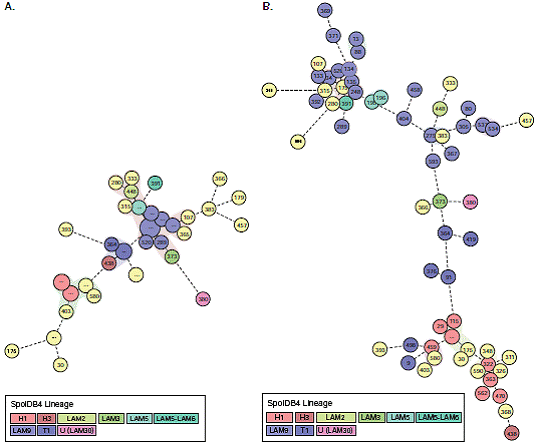
Figura 1 Árbol de expansión mínima (Minimum Spanning Tree, MST) que ilustra la relación entre los 63 genotipos identificados en aislamientos de pacientes con infección concomitante de tuberculosis y HIV. El MST ilustra la relación entre los 63 genotipos identificados en el estudio. A. Árbol construido con spoligotyping. B. Árbol construido con MIRU-VNTR. El árbol filogenético conecta cada genotipo con base en los cambios necesarios para ir de un alelo a otro. La longitud entre las ramas representa la distancia entre los genotipos, y el color diferencia las familias genéticas de M. tuberculosis descritas. Los genotipos sin código representan los patrones huérfanos.
Al analizar los aislamientos con spoligotyping y MIRU-VNTR, solo se halló el agrupamiento de dos de ellos correspondientes a pacientes de Bogotá y Bucaramanga; uno de dichos pacientes tenía tuberculosis pulmonar y, el otro, tuberculosis extrapulmonar, y no fue posible establecer ningún vínculo epidemiológico entre ellos (figura 2).
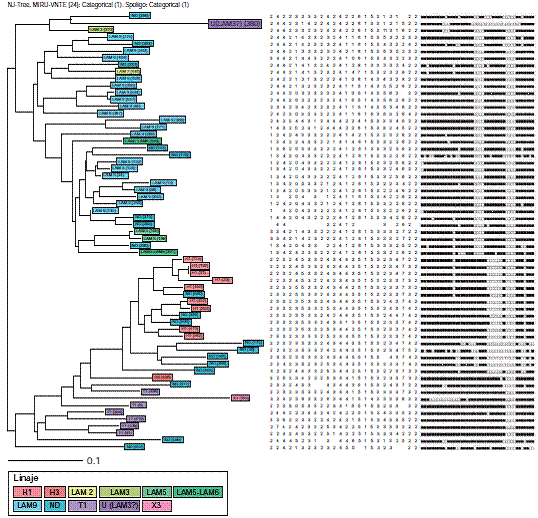
Figura 2 Dendrograma de los agrupamientos de los 63 aislamientos de pacientes con infección concomitante de tuberculosis y HIV obtenido mediante las metodologías de spoligotyping y MIRU-VNTR. Se observa un agrupamiento correspondiente a dos pacientes de Bogotá y Bucaramanga con idéntico genotipo, uno con tuberculosis pulmonar y el otro con tuberculosis extrapulmonar.
Discusión
El análisis de los datos demográficos de sexo, edad y tipo de tuberculosis de los pacientes incluidos en el estudio arrojó proporciones similares a lo reportado por el Sivigila en el mismo año 3, a pesar de que el número de casos analizados en este trabajo fue mucho menor al de la notificación.
Los entes territoriales que más casos aportaron según la notificación del Sivigila fueron Antioquia, Valle del Cauca y Bogotá, con 44,3 %, en tanto que en este trabajo el mayor número de casos provenía del Valle del Cauca y Bogotá; no obstante, debe señalarse que la información de Antioquia no se analizó porque en este departamento se cuenta Cuadro 1. Procedencia de los 63 casos de infección concomitante de tuberculosis y HIV incluidos en el estudio con un laboratorio de salud pública que lleva a cabo la vigilancia de la resistencia y no envía datos al Instituto Nacional de Salud 3.
Son varias las razones por las cuales se documenta la presencia de resistencia en personas con HIV; se ha demostrado su mayor riesgo de desarrollar enfermedades de tipo gastrointestinal que reducen los niveles séricos de la rifampicina; asimismo, en individuos con acetilación rápida, la concentración de isoniacida, etambutol y pirazinamida es variable. Estos factores contribuyen a generar resistencia y están presentes en alrededor de 5,0 % de los casos de infección concomitante de tuberculosis y HIV 20. En este trabajo la resistencia global en los casos nuevos fue de 10 %, principalmente a rifampicina e isoniacida, es decir, a los dos medicamentos más eficaces en el esquema de tratamiento de la tuberculosis. En el grupo de los pacientes previamente tratados no se evidenció resistencia, lo que puede deberse al número de cultivos analizados.
En un estudio en un hospital de tercer nivel de atención en Camboya, se evaluó la sensibilidad a los fármacos antituberculosos en 236 personas con infección concomitante de tuberculosis y HIV, y se encontró una resistencia global en casos nuevos y previamente tratados de 29,4 % y 63,2 %, respectivamente. En los casos nuevos la resistencia a isoniacida fue de 13,9 % y a la rifampicina fue de 1,6 %, con una multirresistencia de 3,7 %. En los pacientes previamente tratados, la resistencia a la isoniacida y la rifampicina fue la misma (7,9 %), y se presentó multirresistencia en 28,9 % de los casos. Las diferencias entre los resultados obtenidos podrían deberse al comportamiento epidemiológico de la enfermedad en ambos países y a las diferencias en el tamaño de la muestra en cada uno de los trabajos 21.
En España, en una cohorte de pacientes con HIV, se encontró que el 5 % de los pacientes había desarrollado tuberculosis; a 175 se les hizo prueba de sensibilidad y se detectó una resistencia global en pacientes nuevos y previamente tratados de 11,1 % y 36,4 %, respectivamente. En los casos nuevos, la resistencia a isoniacida fue de 5,9 % y a rifampicina fue de 1,5 %, y hubo un caso de tuberculosis multirresistente. En pacientes previamente tratados, la resistencia a isoniacida fue de 27,3 % y a rifampicina fue de 9,1 %, y se presentó un caso multirresistente. Los datos correspondientes a pacientes nuevos son similares a los del presente trabajo, y en los previamente tratados las diferencias se explicarían por el número de casos evaluado en cada estudio 22.
En un estudio en Perú para establecer la resistencia a los medicamentos antituberculosos en 523 personas con tuberculosis e infección por el HIV, se encontró una resistencia global de 55,8 % y 93 %, y una multirresistencia de 32,1 % y 74,8 % en casos nuevos y previamente tratados, respectivamente. En ese país se ha registrado una alta prevalencia de la resistencia a fármacos, razón por la cual no hay similitud entre los datos reportados en ese estudio y el presente 23.
En dos estudios en personas con tuberculosis e infección por el HIV en Colombia, se evaluó la sensibilidad a los fármacos teniendo en cuenta el antecedente de tratamiento. Uno de dichos estudios se hizo en el Valle del Cauca en 25 pacientes, y en él se encontró resistencia a isoniacida en 28,5 % de los casos nuevos, en tanto que no se encontró resistencia a la rifampicina ni multirresistencia; en pacientes previamente tratados, se detectó resistencia a la isoniacida (50 %) y a la rifampicina (25 %). En ese trabajo se analizó el 37 % de los casos de infección concomitante de tuberculosis y HIV que se presentaron a lo largo de cuatro años en una institución de tercer nivel de atención, por lo cual es posible que la mayoría provinieran de zonas con alta prevalencia de resistencia, como Cali y Buenaventura; además, el número de muestras analizadas fue inferior, razón por la cual los porcentajes de resistencia evidenciados fueron superiores a los informados en el presente trabajo 24.
El segundo estudio se hizo en Medellín, en 22 personas positivas para HIV, en las cuales la resistencia global fue de 18,2 %; se detectó resistencia a rifampicina en un paciente y multirresistencia en otro (4,5 %), ambos correspondientes a casos nuevos. Medellín es uno de los municipios con altas tasas de tuberculosis resistente, por lo cual los porcentajes obtenidos en ese estudio fueron superiores a los de este, en el cual, como ya se mencionó, no se incluyeron aislamientos de Antioquia 25.
Las familias genéticas encontradas correspondieron a las de mayor prevalencia en casos de tuberculosis en la población general del país 26-28. Además, se trata de familias genéticas de M. tuberculosis prevalentes en los demás países de Latinoamérica, con frecuencias muy similares a las reportadas en Perú, Argentina y, recientemente, Honduras 29-31. Puerto, et al., describieron las familias genéticas prevalentes en 741 aislamientos de M.tuberculosis circulantes en Colombia entre 1999 y 2012, y al igual que en este estudio, las familias LAM9, los genotipos huérfanos, y las familias H1 y T1 presentaron las más altas frecuencias, con 27, 17, 11,9 y 6,9 %, respectivamente 27. Asimismo, Realpe, et al., en Medellín, Cali y poblaciones del Cauca, y Cerezo, et al., en Bogotá, encontraron que las familias genéticas de M. tuberculosis más frecuentes correspondían a LAM9, H1 y T1 (más del 70 % de las familias encontradas) 26,28; sin embargo, a diferencia de los resultados de Puerto, et al., y los obtenidos en este estudio, el número de patrones huérfanos apenas representó un porcentaje de 4,8 y 3,9, respectivamente.
La identificación de aislamientos con patrones huérfanos utilizando SpotClust evidenció su pertenencia a las familias de M. tuberculosis descritas como prevalentes en Colombia.
En el país se han hecho varios estudios de caracterización molecular de casos de tuberculosis, en los que se ha evidenciado la asociación de la infección concomitante de tuberculosis y HIV con la transmisión reciente y los patrones agrupados 32, así como una asociación significativa entre aislamientos y familias genéticas específicas 26. En este estudio no se observó una asociación entre los aislamientos o las familias genéticas descritas, probablemente debido a que los aislamientos provenían de varios departamentos del país, lo cual refleja un panorama general de la circulación de genotipos y no de la transmisión confinada a una sola región. La observación de patrones únicos no agrupados puede deberse a la reactivación de infecciones latentes, o, según los hallazgos de estudios similares sobre pacientes con tuberculosis con HIV en regiones de alta incidencia, corresponderían a infecciones recientes adquiridas después de la infección por HIV 5,33.
En el estudio de Realpe, et al., en el cual se genotipificaron siete aislamientos de pacientes con tuberculosis e infección por HIV, se encontró que las familias H1, código SIT 727, y T1, código SIT 53, tenían una mayor prevalencia, y en los 407 aislamientos de pacientes negativos para el HIV se encontró una asociación significativa del SIT 45, perteneciente a la familia H1, con multirresistencia 26. En este estudio no se encontró relación significativa entre los genotipos y la presencia de resistencia a los fármacos.
En China, Liu, et al., analizaron 70 aislamientos de personas con HIV y establecieron que los genotipos de M. tuberculosis circulantes en la población en general eran los mismos que en personas con tuberculosis e infección por HIV; además, encontraron una tasa de prevalencia de casos de tuberculosis extremadamente resistente mayor en estos individuos que en la población general 33.
Los casos analizados se recibieron en el Instituto Nacional de Salud durante un periodo de seis meses, y no se consideró la información sobre la notificación de casos de infección concomitante de tuberculosis y HIV en ese mismo periodo; sin embargo, al revisar todos los casos con infección concomitante en el 2012, se encontró que los 63 analizados correspondían a 4,5 % del total (63/1.397). Este bajo porcentaje se debe a que Antioquia y Atlántico tienen la capacidad de hacer las pruebas de sensibilidad a fármacos y no envían los aislamientos al Instituto; por otra parte, ante la baja carga bacilar de las muestras procedentes de pacientes con infección concomitante de tuberculosis y HIV, no siempre se logra la comprobación bacteriológica y el diagnóstico se hace con base en el criterio clínico o por nexo epidemiológico. Algunos estudios indican que el porcentaje de casos de tuberculosis en personas con HIV que acceden a un diagnóstico por pruebas de laboratorio es de 65 % 22. Otra de las causas podría ser que, a pesar de contar con un lineamiento nacional, el médico tratante no solicita las pruebas de sensibilidad, lo cual evidencia debilidades en la vigilancia de la resistencia en este grupo de pacientes. Las razones mencionadas fueron limitaciones del estudio, por lo tanto, no se puede excluir la posibilidad de transmisión activa en estos pacientes.
La selección de los casos también representó una limitación, ya que la fuente primaria de información fueron los formatos únicos que se emplean para remitir los cultivos al Laboratorio Nacional de Referencia, en los cuales se indica que la muestra proviene de un paciente con HIV, y es posible que durante el periodo de estudio llegaran más cultivos de personas con esta condición, pero, al no estar registradas en este formato, no fueron incluidos en el trabajo.
Los resultados de este estudio evidencian la necesidad de fortalecer las actividades de vigilancia de la tuberculosis en personas con HIV. Se requieren estudios más completos que permitan evaluar una proporción representativa de aislamientos durante un mayor periodo, con la participación de otros grupos de investigación del país y con una supervisión de los datos registrados en los formatos únicos, lo cual permitiría conocer la situación real de la dinámica de transmisión de la tuberculosis en pacientes con HIV en el país para, así, proponer estrategias de intervención a los programas nacionales de tuberculosis y de HIV.














