El virus del dengue (DENV) es un miembro del género Flavivirus y es el patógeno viral humano transmitido por mosquitos más importante en el mundo. En la actualidad, la incidencia del dengue es 30 veces mayor que hace 50 años y se estima que se presentan anualmente 390 millones de infecciones 1. A pesar de los esfuerzos, no existen vacunas o medicamentos para tratar esta enfermedad.
Hasta el momento se han identificado cuatro serotipos del DENV (DENV 1-4) y todos ellos tienen un genoma de ARN de cadena sencilla y sentido positivo, el cual codifica para una única poliproteína que es procesada para dar lugar a tres proteínas estructurales (C, prM y E) y siete proteínas no estructurales (NS1, NS2A, NS2B, NS3, NS4A, NS4B y NS5) 2,3. El procesamiento de la poliproteína es llevado a cabo por proteasas de la célula huésped y por la proteasa viral, la cual está constituida por el dominio N-terminal de la proteína NS3, que actúa con la proteína NS2B como cofactor 4.
Por su parte, la región C-terminal de la proteína NS3 alberga actividades de la enzima helicasa del ARN, del nucléotido del trifosfato de adenosina (NTPasa) (nucleótido de trifosfatasa) 5 y la RTPasa (ARN 5'-trifosfatasa) 6, las cuales convierten la proteína NS3 en una proteína con múltiples funciones durante el ciclo de replicación del DENV. Además, se ha demostrado que la proteína NS3 es una proteína inmunodominante y uno de los blancos más significativos de la reacción de los linfocitos T durante la infección con el DENV 7,8. Todas estas características la han convertido en un blanco terapéutico valioso y, por lo tanto, en un objeto interesante de estudio.
El objetivo de este trabajo fue expresar proteínas recombinantes de NS3 y a partir de ellas producir anticuerpos policlonales útiles en ensayos de Western blot, inmunofluorescencia e inmunoprecipitación. Estas proteínas recombinantes y los anticuerpos producidos pueden llegar a ser herramientas valiosas en el estudio de las interacciones entre la NS3 y las proteínas celulares, así como en la detección de la replicación viral in vivo.
Materiales y métodos
Clonación
El virus DENV2 empleado (INS-2009) es el pasaje 6 de un aislamiento colombiano adaptado a células C6/36, gentilmente cedido por Jairo Méndez del Grupo de Virología del Instituto Nacional de Salud de Colombia. Antes del inicio de este trabajo, se confirmó que se trataba de un DENV2 mediante una reacción en cadena de la polimerasa (PCR) semianidada para los genes de cápside y premembrana (C-PreM), según lo descrito por Chien, et al.9.
Para obtener el ARN viral, se infectaron células C6/36 (ATCC) con el virus INS-2009 y, a partir de los sobrenadantes de los cultivos, se hizo la extracción utilizando el estuche QIAamp Viral RNA(r) (Qiagen). Para la síntesis del ADN complementario se usó 1 μg del ARN extraído, 200 U de transcriptasa inversa del virus de la leucemia de ratón de Moloney (Moloney Murine Leukemia Virus, MMLV) (Promega), 200 U del inhibidor de la ribonucleasa RNAsin (Promega), 0,6 μg de iniciadores aleatorios (Promega) y 0,5 mM de desoxirribonucleótidos de trifosfato (dNTP).
El ADN complementario obtenido se usó como plantilla para amplificar mediante PCR dos regiones del extremo C-terminal de NS3, dominio en el cual se encuentran los epítopos más antigénicos de la proteína (10). Para ello, se usó el genoma de la cepa de referencia 16681 de DENV2 (NC_001474.2), del cual se extrajo la secuencia de nucleótidos que correspondía a la proteína NS3.
Con esta secuencia se alimentó el programa Primer Designer (versión 1.01) (http://www.scied.com/pr_ cmpro.htm), conservando todos los parámetros establecidos por defecto y se eligieron dos parejas de oligonucleótidos. Los oligonucleótidos diseñados daban lugar a dos amplicones de 399 y 606 pb, los cuales correspondían a los aminoácidos 176 a 303 y 403 a 599 de NS3. Como la cepa de referencia 16681 es de origen tailandés, se hizo un alineamiento entre cada uno de los oligonucleótidos diseñados y los genomas de 11 aislamientos colombianos de DENV2 (números de acceso en Genbank: EU854294.1, FJ024473.1, FJ024474.1, FJ024475.1, FJ182012.1, GQ868553.1, GQ868555.1, GQ868556.1, GQ86 8557.1, GQ868558.1 y GU131947.1). Dos de los oligonucleótidos presentaron una identidad del 100 % con las 11 secuencias analizadas, otro presentó identidad en 14 de 15 residuos y, el otro, en 14 de 17 residuos.
Las secuencias de estos dos últimos se ajustaron para obtener una identidad del 100 % con las secuencias de los aislamientos colombianos, y los cuatro oligonucleótidos fueron sintetizados por Macrogen. Así, el amplicón de 399 pb se obtuvo con el oligonucleótido sentido CATATGCCAGAGA TCGAAGATGAC y antisentido GGATCCTAATCG AGTTGAAATGTA, y el amplicón de 606 pb se obtuvo con el oligonucleótido sentido CATATGG ACTTTGTGGTCACGAC y antisentido GGATCC TAACCTAGCATCCAACC (las secuencias de reconocimiento para las enzimas NdeI y BamHI aparecen subrayadas).
Las condiciones de la PCR fueron las siguientes: 1 mM de cloruro de magnesio (MgCl2), 200 μM de dNTP, 0,5 U de ADN polimerasa TaqPlatinum (Invitrogen) y 0,5 μM de cada oligonucleótido. El programa de amplificación comprendió una desnaturalización inicial a 95 °C durante tres minutos, seguida de 35 ciclos de desnaturalización (a 94 °C durante 30 segundos), hibridación (a 50 °C durante 30 segundos) y extensión (a 72 °C durante 30 segundos), y una extensión final a 72 °C durante cinco minutos.
Los amplicones se purificaron de geles de agarosa con el estuche Wizard(r) SV Gel and PCR Clean- Up System (Promega) y se clonaron en el vector pJET1.2/blunt (ThermoScientific). Luego, los fragmentos se liberaron por digestión con las enzimas NdeI y BamHI (New England Biolabs, NEB), y se subclonaron en el vector pMALc5x (NEB). En este vector, los fragmentos se insertan corriente abajo del gen malE que codifica para una proteína de unión a maltosa (MBP) de 42,5 kDa, lo cual resulta en la expresión de proteínas de fusión MBP. Los dos constructos pMALc5x se usaron para transformar bacterias Escherichia coli NEB Express (NEB) y luego se detectaron las colonias positivas mediante PCR. Para confirmar su identidad se purificó ADN 'plasmídico' (WizardPlus SV Minipreps DNA Purification(r), Promega) y se usó un servicio de secuenciación (Macrogen). La secuencia se envió al GenBank (número de acceso: KX303476.
Por último, el peso molecular teórico de las proteínas recombinantes denominadas como NS3176_303 y NS3403_599, se calculó con la herramienta ExPASy compute pI/Mw (http://web.expasy.org/compute_pi/).
Producción de las proteínas recombinantes
La expresión de NS3176_303 y NS3403_599 se hizo en 10 ml de medio Luria Bertani (LB, Sigma) con suplemento de 100 μg/ml de ampicilina (Amresco) (LB+Amp). Los cultivos se incubaron hasta alcanzar una densidad óptica (DO600) de ≈0,4, momento en el cual se añadió isopropil-beta-tiogalactopiranósido (IPTG) (Promega) a una concentración final de 0,3 mM, y se permitió el crecimiento a 37 °C con agitación a 200 rpm.
De cada cultivo se tomaron muestras de 1 ml justo antes de la inducción (tiempo 0), y una, dos, tres y 16 horas después de la inducción. Posteriormente, de cada muestra se tomó una alícuota que se preparó con solución tampón Laemmli y se sometió a electroforesis en gel de poliacrilamida con dodecilsulfato sódico (SDS-PAGE) para monitorizar la expresión.
El volumen restante se usó para establecer si las proteínas recombinantes NS3176_303 y NS3403_599 se encontraban en la fracción soluble o en la insoluble. Para esto, la suspensión bacteriana se centrifugó y los sedimentos se sometieron de nuevo a suspensión en solución tampón de lisis (5 mM de imidazol; 0,5 M de NaCl; 10 % de glicerol; 20 mM de Tris-HCl, pH 7,9; 1 % de Tritron X-100), a choque térmico (10 minutos en nitrógeno líquido seguidos de una incubación a 37 °C), a sonicación y a centrifugación.
Los sobrenadantes y los sedimentos resultantes se sometieron a SDS-PAGE (10 %). A continuación se hizo una expresión a mayor escala inoculando una colonia de cada proteína recombinante en 500 ml (para NS3176_303) o 250 ml (para NS3403_599) de medio LB+Amp. La expresión se indujo con las mismas condiciones ya descritas y la cosecha de las bacterias se hizo a las tres horas de la inducción para NS3176_303 y, a las 16, para NS3403_599. Las bacterias se sometieron a lisis como se describió anteriormente y los sobrenadantes se usaron para hacer una cromatografía de afinidad sobre resina de amilosa (NEB), siguiendo las recomendaciones del fabricante. Las proteínas se liberaron de la resina con solución tampón maltosa (10 mM) y se recolectaron fracciones (500 μl) que se cuantificaron mediante el método de Bradford.
Producción de los anticuerpos
Cada proteína recombinante se inoculó por vía intraperitoneal en dos ratas macho Wistar de cuatro semanas de edad. El esquema de inmunización comprendió una dosis de sensibilización con Imject Alum Adjuvant(r) (ThermoScientific), una primera dosis de 100 μg de proteína recombinante con adyuvante ocho días después, y dos refuerzos de 50 μg, 15 y 30 días después de la primera dosis. Las ratas se sacrificaron ocho días después de la última inoculación, los sueros se recuperaron y a partir de ellos se purificaron por afinidad los anticuerpos anti- NS3176_303 y anti-NS3403_599, siguiendo el protocolo descrito por Smith, et al.11).
Cultivo celular en infección
Las células BHK-21 (ATCC) y A549 (ATCC) se mantuvieron en medio Eagle modificado de Dulbecco (DMEM) (Sigma) con suplemento de 10 % de suero fetal bobino (FBS) (Hyclone), a 37 °C en una atmosfera de 5 % de CO2. Cuando las células alcanzaron una confluencia de ≈80 %, se infectaron con DENV2 (aislamiento colombiano INS-2009) (MOI: 1) suspendido en DMEM con FBS al 2 %. Las células se mantuvieron en cultivo hasta 24, 48 o 72 horas después de la infección.
Ensayos de Western blot
A las 48 horas de la infección, las células BHK- 21 y A549 se trataron con tripsina (0,25 % de tripsina, 0,5 mM de EDTA), se centrifugaron, y el sedimento se sometió a lisis con solución tampón RIPA (25 mM de Tris-HCl, pH 7,6; 150 mM de NaCl; 1 % de detergente NP-40; 1 % de deoxicolato de sodio; 0,1 % de dodecilsulfato sódico, SDS) y con suplemento de inhibidores de proteasas (Sigma).
Los extractos celulares se cuantificaron con el método del ácido bicinconínico y se almacenaron a -80 °C hasta su uso.
Para los ensayos de Western blot con los sueros crudos de las ratas, se sometieron a SDS-PAGE (10 %) 25 μg de cada extracto celular y se transfirieron a membranas de fluoruro de polivinilideno (PVDF) (ThermoScientific), que luego se bloquearon con solución tampón salina con Tween 20 y leche (0,1 % de TBST y 5 % de leche descremada).
A continuación, las membranas se incubaron durante toda la noche a 4 °C con diferentes diluciones de los sueros crudos (1:500, 1:1000 y 1:2000 en TBST y leche). Al día siguiente, las membranas se lavaron con TBST y se incubaron con el anticuerpo secundario (1:10.000) (anti-rata, conjugado con biotina, Pierce 31830) durante una hora a temperatura ambiente y luego se reveló mediante la adición de estreptavidina- HRP (1:10.000) (ThermoScientific), peróxido de hidrógeno (0,02 %) y diaminobencidina (DAB) (0,05 %). Como control, se incubó una membrana con un anticuerpo policlonal anti-NS3 comercial producido en conejo (1:1.000) (GTX124252, GeneTex) y luego con un anticuerpo anti-conejo de unión a biotina (1:2.000) (Sigma B8895), bajo las mismas condiciones ya descritas.
Para los ensayos de Western blot con los anticuerpos purificados, se sometieron a SDS-PAGE (8 %) 20 μg de cada extracto celular, y se hizo la transferencia, el bloqueo y la incubación con los anticuerpos anti-NS3176_303 y anti-NS3403_599 purificados (1:1000), y con los sueros preinmunes (1:1.000), bajo las mismas condiciones ya descritas.
Al día siguiente, las membranas se lavaron y se incubaron con el anticuerpo secundario acoplado a peroxidasa (1:5000) (anti-rata_HRP, Thermo Scientific, 31470) durante una hora a temperatura ambiente. El revelado se hizo con peróxido de hidrógeno (0,02 %) y DAB (0,05 %).
Ensayos de inmunofluorescencia (IF)
Se sembraron células BHK-21 sobre laminillas previamente tratadas con poli-L-lisina (10 μg/ml) e infectadas con DENV2, como ya se describió.
Las células se mantuvieron en cultivo durante 24, 48 o 72 horas, tras lo cual se fijaron con paraformaldehído al 4 %, se hicieron permeables con Tritón X-100 (0,3 %, Sigma) durante 30 minutos y se incubaron con suero de cabra (Gibco) al 10 % durante tres horas.
A continuación, las laminillas se incubaron durante una hora a 37 °C con el anticuerpo anti-NS3176_303 (1:250), el anticuerpo anti-NS3403_599 (1:500) o el anticuerpo GTX124252 (1:500), y después, con los respectivos anticuerpos secundarios biotinilados.
Las células se lavaron con solución tampón fosfato salino y se incubaron con estreptavidina acoplada al colorante Alexa 594 (Molecular Probes).
Después, se contrastaron los núcleos con una solución de Hoechst 33342 (Sigma), las laminillas se montaron con Vectashield (Vector) sobre una lámina portaobjeto y se observaron las células bajo microscopio (Zeiss Axioimager A2) utilizando un sistema de fluorescencia X-Cite serie 120Q y el programa ZEN 2012.
Como control positivo, se prepararon laminillas con un anticuerpo monoclonal dirigido contra la proteína de envoltura (E) del virus de la encefalitis de San Luis (1:500, Millipore MAB8744), el cual tiene reacción cruzada con la proteína E del DENV2. Como anticuerpo secundario en este experimento, se usó un anticuerpo anti-ratón conjugado con biotina (1:200, Vector BA9200).
Como controles negativos, se usaron los sueros preinmunes NS3176_303 (1:250) y NS3403_599 (1:500) en células infectadas, y los anticuerpos anti- NS3176_303 (1:250) y anti-NS3403_599 (1:500) en células no infectadas.
Ensayos de inmunoprecipitación
Los ensayos de inmunoprecipitación se hicieron con el anticuerpo anti-NS3403_599 como modelo de experimentación. Para esto, 10 μl del anticuerpo se agregaron a 200 μg de extracto de células BHK-21 infectadas con DENV2 (obtenido según se describió antes) y la mezcla se incubó en agitación orbital sobre hielo durante cuatro horas. A continuación, se añadieron proteínas A y G inmovilizadas sobre perlas de agarosa (ThermoScientific) y se incubó en agitación toda la noche a 4 °C. Al día siguiente, las perlas se recolectaron por centrifugación, se lavaron con solución tampón de inmunoprecipitación (50 mM de Tris HCl, pH 7,5; 100 mM de NaCl; 0,1 % de Tritón X-100) y se sometieron de nuevo a suspensión con solución tampón Laemmli para ser analizadas por Western blot. Se hicieron dos experimentos de control bajo las mismas condiciones ya descritas: en uno se usaron 10 μl de suero preinmune en la inmunoprecipitación y en el otro no se agregó anticuerpo.
Los proyectos en los cuales se enmarca este trabajo (130852128608 y UB.270-2010) cuentan con el aval ético del Comité Institucional de Ética en Investigaciones de la Universidad El Bosque, del 29 de septiembre de 2010.
Resultados
Expresión de las proteínas recombinantes y producción de los anticuerpos
El peso molecular teórico de NS3176_303 y NS3403_599 se calculó en 58.381 Da (≈58 kDa) y 66.068 Da (≈66 kDa), respectivamente. La expresión de cada proteína recombinante se hizo primero en 10 ml de medio LB+Amp para establecer el tiempo adecuado de cosecha de las bacterias y la solubilidad de cada proteína recombinante.
La expresión de la proteína NS3176_303 fue detectable una hora después de la inducción y alcanzó su máximo a las tres horas. La expresión de la proteína NS3403_599 fue detectable a las dos horas de la inducción y alcanzó su máximo a las tres horas; sin embargo, se obtuvo más proteína NS3403_599 soluble a las 16 horas (no se presentan los datos), razón por la cual se eligió ese tiempo para hacer la expresión a mayor escala. En las figura 1A y 1B se observa que las dos proteínas migraron a la altura esperada y se detectaron tanto en la fracción soluble (S) como en la insoluble (P).
A continuación, se hizo una expresión a mayor escala seguida por una cromatografía de afinidad sobre resina de amilosa. En las figura 1C y 1D se observa la SDS-PAGE de las fracciones recuperadas en las cromatografías para las proteínas recombinantes NS3176_303 y NS3403_599, respectivamente. Se observaron las bandas esperadas y una banda de menor peso molecular (≈42 kDa) que en los ensayos de Western blot con un anticuerpo anti-MBP (NEB E8032S), la cual se identificó como la etiqueta MBP (no se presentan los datos).
Para la inmunización de las ratas, se usó como antígeno la mezcla de las fracciones 4 y 5 de la cromatografía para NS3176_303 (figura 1C, carriles 4 y 5) y la mezcla de las fracciones 6 y 7 de la cromatografía para NS3403_599 (figura 1D, carriles 6 y 7). La concentración de proteína en esas mezclas, determinada mediante el método de Bradford, fue de 650 μg/ml y 740 μg/ml, respectivamente.
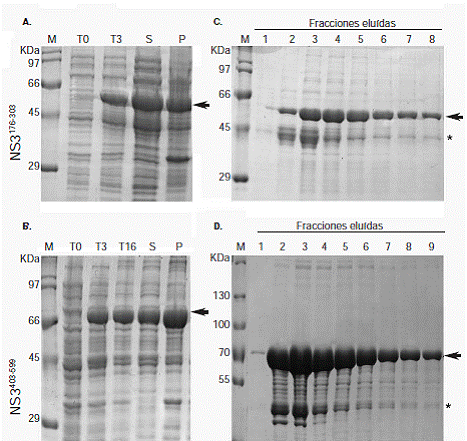
Figura 1 Expresión y purificación de las proteínas recombinantes NS3176_303 y NS3403_599. Estas se expresaron como proteínas de fusión MBP y se purificaron mediante cromatografía de afinidad. A. y B. SDS-PAGE (10 %) en la cual se observa la expresión de las proteínas recombinantes. M: marcador SDS6H2 (Sigma). T0: resultado de la lisis de bacterias antes de la inducción de la expresión. T3: resultado de la lisis de bacterias a tres horas de la inducción. T16: resultado de la lisis de bacterias a 16 horas de la inducción. S: sobrenadante producto de la lisis celular. P: sedimento producto de la lisis celular. S y P corresponden al sobrenadante y al sedimento de T3 en la figura 1A, y de T16 en la figura 1B. Las flechas señalan las proteínas recombinantes. C. y D. SDS-PAGE (10 %) en la cual se observan las fracciones de la cromatografía de afinidad para NS3176_303 y NS3403_599. M: SDS6H2 (Sigma) en la figura C y PageRuler Plus (Fermentas) en la figura D. Carriles 1 al 8 en C, y 1 al 9 en D: fracciones eluídas. Las flechas señalan las proteínas recombinantes. El asterisco señala la etiqueta MBP.
Ensayos de Western blot
Los sueros recuperados de las ratas inmunizadas y los anticuerpos purificados se evaluaron en ensayos de Western blot sobre extractos de células BHK-21 y A549 infectadas con DENV2. En la figura 2A se observa la inmunodetección de NS3 (≈70 kDa) sobre extractos de células BHK-21 y A549 infectadas con DENV2, usando tres anticuerpos diferentes: el anticuerpo comercial GTX124252 (carril 1), el suero crudo anti-NS3176_303 (carril 2) y el suero crudo anti-NS3403_599 (carril 3).
Todos los anticuerpos se usaron en una dilución de 1:1000 y reconocieron la proteína NS3 de manera específica y con igual intensidad de señal. La figura 2B muestra la detección de NS3 usando el suero crudo anti-NS3176_303, el anticuerpo anti-NS3176_303 purificado y el suero preinmune NS3176_303. La proteína NS3 fue visible a la altura esperada (≈70 kDa) en los extractos de células BHK-21 infectadas con DENV2 e indetectable en los extractos de células no infectadas o al usar como anticuerpo primario el suero preinmune. Los mismos resultados se obtuvieron al hacer la detección de NS3 con el suero crudo anti-NS3403_599, el anticuerpo anti-NS3403_599 purificado y el suero preinmune NS3403_599 (figura 2C).
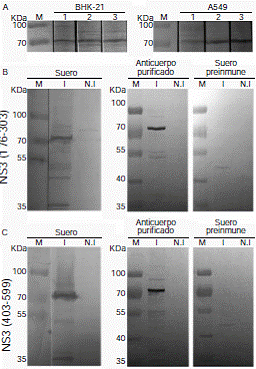
Figura 2 Inmunodetección de la proteína NS3 viral en extractos de células infectadas con DENV2. A. Detección de NS3 (≈70 kDa) en extractos de células BHK-21 y A549 infectadas con DENV2. Inmunodetección con anticuerpo comercial (1:1000) (GeneTex. GTX124252), carril 1; suero anti-NS3176_303 (1:1000), carril 2; suero anti-NS3403_599 (1:1000), carril 3. B. Detección de NS3 sobre extractos de células BHK-21 infectadas y no infectadas con DENV2. La inmunodetección se hizo con suero crudo anti-NS3176_303, anticuerpo anti-NS3176_303 purificado y suero preinmune NS3176_303. M: marcador PageRuler Plus (Fermentas). I: extracto de células BHK-21 infectadas con DENV2. N.I: extracto de células BHK-21 no infectadas. C. Igual que en B, pero la inmunodetección se hizo con suero crudo anti-NS3403_599, anticuerpo anti-NS3403_599 purificado y suero preinmune NS3403_599.
Ensayos de inmunofluorescencia
Se infectaron con DENV2 células BHK-21 mantenidas en cultivo por 24, 48 o 72, fijadas e incubadas con anti-NS3176_303 (1:250), anti-NS3403_599 (1:500), anti-proteína E (1:500) o el anticuerpo GTX124252 (1:500). La fluorescencia específica para todos los anticuerpos fue detectable en los tres tiempos de cultivo ensayados (no se presentan los datos). La figura 3 corresponde a células fijadas a las 48 horas de la infección y muestra que los anticuerpos anti- NS3176_303, anti-NS3403_599 y GTX124252 tuvieron una reacción similar. No hubo detección en las células no infectadas al usar anti-NS3176_303 (1:250) o anti- NS3403_599 (1:500), ni en las células infectadas al emplear usándolos sueros preinmunes NS3176_303 (1:250) y NS3403_599 (1:500). Las células totales e infectadas se contaron en las imágenes utilizando la herramienta cell counter (software Image J) y se determinaron porcentajes de infección de ≈30 % al hacer la inmunodetección con anti-NS3176_303 o anti-NS3403_599.
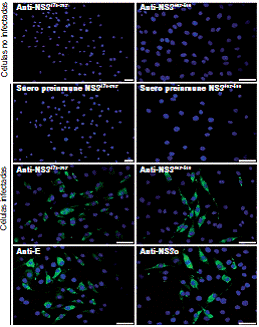
Figura 3 Detección de la proteína NS3 en células BHK-21 infectadas con DENV2. Detección de NS3 en células BHK-21 infectadas con DENV2 (MOI: 1) durante 48 horas. La detección se hizo con los sueros preinmunes NS3176_303 (1:250) y NS3403_599 (1:500), y con los anticuerpos anti-NS3176_303 (1:250), anti- NS3403_599 (1:500) y anti-NS3 comercial (NS3c) (1:500) (GeneTex. GTX124252). Como control positivo de la infección, se usó un anticuerpo monoclonal dirigido contra la proteína de envoltura (E) (1:500) (Millipore). Los dos paneles superiores corresponden a la detección con los anticuerpos anti-NS3176_303 y anti-NS3403_599 en células no infectadas. Barra: 50 μm.
Ensayos de inmunoprecipitación
Los ensayos de inmunoprecipitación se hicieron con el anticuerpo anti-NS3403_599 como modelo de experimentación. En la figura 4A se observa que el anticuerpo anti-NS3403_599 purificado permitió la inmunoprecipitación de la proteína NS3 en extractos de células BHK-21 infectadas con DENV2. No hubo detección de NS3 al hacer la inmunoprecipitación con el suero preinmune NS3403_599 y el Western blot con el anticuerpo anti-NS3403_599 purificado, y tampoco cuando esta se hizo con el anticuerpo anti-NS3403_599 purificado y el Western blot con el suero preinmune NS3403_599. En la figura 4B puede observarse que en el ensayo sin el anticuerpo, la proteína NS3 apareció en la fracción no unida y no hubo señal de ella en la inmunoprecipitación.
En la figura 4C se ven los resultados de un ensayo de inmunoprecipitación con el anticuerpo anti- NS3403_599, pero esta vez en extractos de células A549 infectadas con DENV2. Los resultados fueron similares a los descritos antes para las células BHK-21, ya que el anticuerpo permitió la inmunoprecipitación de la proteína NS3 presente en las células A549 infectadas de forma específica.
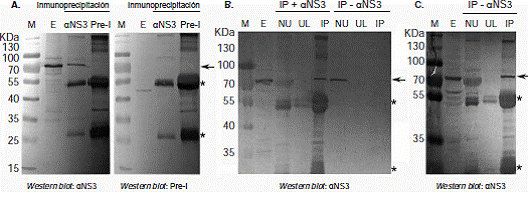
Figura 4 Desempeño del anticuerpo anti-NS3403_599 en ensayos de inmunoprecipitación. A. Western blot con el anticuerpo anti- NS3403_599 (α-NS3) o con el suero preinmune NS3403_599 (Pre-I), sobre la inmunoprecipitación (IP) con el anticuerpo anti-NS3403_599 (IP/α-NS3) o con el suero preinmune NS3403_599 (IP/Pre-I). B. Western blot con anti-NS3403_599 sobre las inmunoprecipitaciones con (IP + α-NS3) o sin (IP - α-NS3) anticuerpo anti-NS3403_599. NU: fracción no unida. UL: último lavado de las perlas. C. Como en B, pero usando extractos de células A549 infectadas con DENV2. En A, B y C. Las inmunoprecipitaciones se hicieron con 200 μg de extracto de células infectadas con DENV2. La flecha indica la posición de la proteína NS3, y los asteriscos señalan las cadenas pesadas y livianas de las inmunoglobulinas. E: 20 μg de extracto de células BHK21 infectadas. M: marcador PageRuler Plus (Fermentas).
Discusión
En este trabajo se logró la expresión soluble de proteínas recombinantes de dos regiones del dominio helicasa NS3 del DENV2, las cuales se usaron para inmunizar ratas y producir anticuerpos policlonales. Los dos anticuerpos anti-NS3 demostraron su eficacia en ensayos de Westernblot e inmunofluorescencia, y uno de ellos (anti- NS3403_599), en ensayos de inmunoprecipitación.
La proteína NS3176_303 contenía los residuos 176 a 303 de NS3, los cuales corresponden a gran parte del subdominio 1 (residuos 168 a 322) del dominio helicasa de la proteína viral. Por su parte, la proteína recombinante NS3403_599 contenía los residuos 403 a 599, los cuales corresponden a parte del subdominio 2 (residuos 323-488) y a gran parte del subdominio 3 (residuos 489 a 618). Las proteínas NS3176_303 y NS3403_599 se expresaron como proteínas de fusión MBP y ambas se obtuvieron abundantemente en la fracción soluble (figura 1A y 1B).
Este fue un resultado notable, porque en un trabajo previo en el que se buscaba la expresión individual de los subdominios 1 o 2 del dominio helicasa de NS3, se produjeron proteínas solubles al usar DENV4, pero insolubles con el DENV2. Más aún, en ese mismo estudio la expresión del subdominio 3 produjo proteínas insolubles para ambos serotipos y esa dificultad solo pudo resolverse para DENV4 al expresar una proteína recombinante que contenía los subdominios 2 y 3 12. Por el contrario, en este trabajo las dos proteínas recombinantes del dominio helicasa de NS3 de DENV2 se obtuvieron de forma soluble y se alcanzaron producciones de ≈2 mg de NS3176_303 a partir de 500 ml de cultivo (3 horas después de la inducción) y ≈4 mg de NS3403_599 a partir de 250 ml de cultivo (16 horas después de la inducción). Las proteínas recombinantes purificadas se usaron para inmunizar ratas y los anticuerpos, purificados por afinidad, se emplearon en los ensayos de Western blot, inmunofluorescencia e inmunoprecipitación.
En los ensayos de Western blot e inmunofluorescencia se usó como control el anticuerpo GTX124252 (GeneTex), el cual se produce con una proteína recombinante que comprende los aminoácidos 354 al 568 de NS3.
En los ensayos de Western blot, se usaron los sueros crudos anti-NS3176_303 y anti-NS3403_599 para detectar la proteína viral (≈70 kDa) sobre extractos de células BHK-21 y A549 infectadas con DENV2. El desempeño de estos sueros, medido por la intensidad y la especificidad de la señal y la dilución de trabajo (1:1.000), fue comparable con el del anticuerpo GTX124252 (figura 2A). En los ensayos de Western blot en células BHK-21 no infectadas, no hubo detección de NS3, así como tampoco con los sueros crudos, ni con los anticuerpos anti- NS3176_303 y anti-NS3403_599 purificados (figura 2B y 2C). Tampoco hubo detección alguna en la región de ≈70 kDa al hacer el Western blot con los sueros preinmunes (figura 2B y 2C).
Con respecto a la localización celular, además de su ubicación en el citoplasma, NS3 también se asocia a membranas mediante su cofactor NS2B 13. En los ensayos de inmunofluorescencia, tanto el anticuerpo control como los anticuerpos anti- NS3176_303 y anti-NS3403_599 detectaron la proteína NS3 en el citoplasma y, más intensamente, en la región perinuclear de las células infectadas (figura 3). Esta misma ubicación subcelular había sido descrita en células CV-1 infectadas con DENV2 (14), en células BHK-21 transfectadas con un constructo de NS3 15 y en células Huh-7.5 y HEK 293T transfectadas con los constructos NS3 o NS2B-NS3 16. El anticuerpo anti-NS3403_599 (1:500) produjo una intensidad y una distribución de fluorescencia comparables a las del anticuerpo GTX124252 (1:500), mientras que el anticuerpo anti-NS3176_303 (1:250) produjo una marcación más débil que hizo menos apreciables las prolongaciones citoplásmicas de las células (figura 3).
Por último, aunque en el mercado existen anticuerpos comerciales contra NS3, los fabricantes declaran no haber comprobado su utilidad en experimentos de inmunoprecipitación. Un ensayo de este tipo demanda cerca de 10 μl de anticuerpo primario por reacción y eso hace particularmente costosa esa verificación. Ya que en este trabajo se produjeron aproximadamente 5 ml de cada anticuerpo anti-NS3 por rata, y la dilución de trabajo determinada para el anticuerpo anti-NS3403_599 fue igual a la del anticuerpo comercial (GTX124252) en los ensayos de Western blot (1:1000) e inmunofluorescencia (1:500), el paso siguiente fue establecer la eficacia de este anticuerpo en ensayos de inmunoprecipitación.
En experimentos descritos en otros estudios, este ensayo se hizo con anticuerpos antietiqueta 15, con sueros anti-NS3 o con fluido ascítico hiperinmune 17,18, usando extractos de células en las que se indujo la sobreexpresión de NS3 o que están marcadas metabólicamente con radioisótopos. Aunque la sobreexpresión facilita la detección de interacciones en las que participa una proteína determinada, los resultados podrían sugerir la existencia de artefactos debidos a la sobreexpresión y no necesariamente a interacciones biológicas reales.
Por otra parte, aunque la marcación metabólica le da al sistema una muy alta sensibilidad y le permite revelar interacciones indetectables con otras técnicas, tiene desventajas como los riesgos de seguridad, la generación de desechos radiactivos y la toxicidad para las células. Por eso, es importante contar con un anticuerpo que en condiciones de cultivo e infección estándar, permita la inmunoprecipitación y detección de proteínas virales de interés, para hacer el posterior análisis de las proteínas celulares que pudieran precipitarse conjuntamente con ella.
En este caso, el anticuerpo anti-NS3403_599 permitió la inmunoprecipitación de NS3 en extractos de células BHK-21 infectadas con DENV2 (figura 4A y 4B). La interacción fue específica, pues no hubo detección de NS3 en el sistema donde se hizo la inmunoprecipitación con el suero preinmune (figura 4A), o en el que no se agregó anticuerpo anti-NS3403_599 (figura 4B). El anticuerpo anti- NS3403_599 también permitió la inmunoprecipitación de la proteína NS3 sobre extractos de células A549 infectadas con DENV2 (figura 4C).
Comparados con un anticuerpo monoclonal, los anticuerpos descritos en este trabajo pueden producirse más fácilmente, de forma más económica y en menor tiempo. Además, los anticuerpos anti- NS3176_303 y anti-NS3403_599 fueron eficientes para detectar la proteína NS3 en ensayos de Western blot (ambos), inmunofluorescencia (ambos) e inmunoprecipitación
(anti-NS3403_599), y no produjeron ruido de fondo con ninguna de las técnicas examinadas.
En conclusión, las proteínas recombinantes expresadas y los anticuerpos policlonales producidos constituyen herramientas valiosas para detectar la replicación viral in vivo, estudiar los procesos infecciosos del DENV que involucren la NS3 y evaluar las pruebas encaminadas a interferir con las funciones de esta proteína viral.














