Las nitrorreductasas son un grupo de enzimas que catalizan la reducción de grupos nitro de compuestos estructuralmente diversos que incluyen los nitrotoluenos, los nitrofuranos y los nitroimidazoles. Estas enzimas se pueden subdividir en dos clases generales: sensibles al oxígeno e insensibles al oxígeno 1.
En Helicobacter pylori, bacteria que infecta a los seres humanos y se asocia con el desarrollo de gastritis, úlcera péptica, linfoma gástrico de tejido linfoide asociado a mucosa (Mucosa-Associated Lymphoid Tissue,MALT) y cáncer gástrico 2,3, la nitrorreductasa RdxA insensible al oxígeno cataliza la reducción del medicamento antimicrobiano metronidazol y genera productos intermedios y finales, como la hidroxilamina y el peróxido de hidrógeno, y radicales libres de oxígeno que producen daño en el ADN y la muerte celular del microorganismo 1.
Esta nitrorreductasa es esencial para la activación del metronidazol en H. pylori4, el cual se considera un antimicrobiano efectivo contra organismos con poca reacción de oxidorreducción (redox) intracelular, como las bacterias anaerobias, los protozoos y los microrganismos microaerófilos, entre ellos, H. pylori5.
Uno de los medicamentos utilizados en los esquemas de tratamiento para la erradicación de esta infección es el metronidazol; sin embargo, la aparición de aislamientos que no responden al medicamento ha fomentado el interés en las investigaciones para comprender las causas principales de la resistencia de la bacteria a su acción 4-6.
En varias revisiones sistemáticas se ha reportado una gran heterogeneidad en las frecuencias de prevalencia de la resistencia primaria al metronidazol en los países latinoamericanos, y cabe resaltar que Colombia presenta la tasa de resistencia más alta, 83 % 7,8.
Los resultados de diversos estudios han demostrado que la resistencia de H. pylori al metronidazol se debe a la presencia de diferentes mutaciones en el gen rdxA, las cuales se evidencian como inserciones o deleciones en diferentes sitios del gen. Estos cambios en la secuencia del genoma llevan a que la nitrorreductasa RdxA codificada por el gen se inactive y no logre reducir el metronidazol a nivel celular, generando así la resistencia de la bacteria al antimicrobiano 4,9,10. Las mutaciones que inactivan el gen rdxA originan codones de parada o sustituciones que tornan disfuncional la proteína. En algunos casos, el gen frxA promueve la aparición de resistencia y en otros muta con el gen rdxA9,11.
La participación del gen rdxA en el desarrollo de la resistencia al metronidazol se ha estudiado mediante modelos en ratones para determinar su secuencia en cepas sensibles y resistentes al medicamento. Se ha encontrado que las cepas resistentes presentan mutaciones por el cambio en el marco de lectura resultado de la pérdida o ganancia de adenina (A) o de timina (T), mutaciones de sentido erróneo que contienen la sustitución de aminoácidos como la prolina (P) por la leucina (L), la tirosina (Y) por la histidina (H) y la alanina (A) por la valina (V) 12,13. Aldana, et al. 9, utilizaron muestras de pacientes sometidos a endoscopia, cultivadas y posteriormente inducidas a resistencia al someterlas a diversas concentraciones de metronidazol. En los análisis de la secuencia del gen rdxA, se han encontrado sustituciones de aminoácidos, como el cambio de serina (S) por asparagina (N).
Según las recomendaciones del Consenso de Maastricht IV 14,15, la secuenciación es una técnica utilizada con éxito en la determinación de mutaciones, en especial en áreas geográficas donde los cultivos de la bacteria no están disponibles. El uso de los tratamientos de erradicación de la infección debe basarse en estudios de prevalencia de la resistencia a los antimicrobianos en cada región 2,7.
Puesto que en Colombia son pocos los estudios en que se ha determinado la presencia de mutaciones en la nitrorreductasa RdxA y, dado que en el departamento del Cauca no existe un laboratorio con la infraestructura para cultivar H. pylori, el objetivo de este estudio fue determinar la frecuencia de mutaciones de RdxA y describir los cambios que, según los reportado en varios estudios, estarían asociados con la resistencia al metronidazol.
Materiales y métodos
Población de estudio
Se incluyeron 170 pacientes remitidos a las unidades de gastroenterología del Hospital Universitario San José y el Susana López de Valencia, y a la Unidad ENDOVIDEO 2000 (29, 40 y 101, respectivamente) de Popayán, entre octubre de 2011 y junio de 2013.
Los criterios de inclusión fueron los siguientes: pacientes mayores de 18 años, nacidos en el departamento del Cauca, con síntomas de dispepsia, y que no hubieran recibido tratamiento de erradicación de H. pylori, ni antibióticos ni sales de bismuto durante el último año.
Los pacientes aceptaron su participación en el estudio mediante la firma de un consentimiento informado, según lo dispuesto en la Resolución N° 008430 de 1993 del Ministerio de Salud. Esta investigación fue aprobada por los comités de ética de la Universidad del Cauca y de la Pontificia Universidad Javeriana. Los participantes respondieron un cuestionario estandarizado sobre algunas variables sociodemográficas y clínicas.
Se tomaron ocho biopsias de cada paciente: tres del antro, tres del cuerpo y dos de la incisura angular, según los criterios de Sydney 16. Para el análisis histopatológico, se utilizaron dos biopsias de antro y cuerpo, y una de incisura, todas examinadas por un único patólogo experto, siguiendo los criterios establecidos internacionalmente 17. Las biopsias restantes se utilizaron para la extracción del ADN.
Extracción del ADN y amplificación mediante PCR
Las muestras de las biopsias gástricas (antro, incisura y cuerpo) de cada paciente se colocaron en recipientes independientes y se transportaron en medio de cultivo de caldo de Brucella spp. Con 20 % de glicerol para la extracción genómica, usando el estuche Wizard Genomic DNA Purification® (Promega, Madison, WI, USA) de acuerdo con las recomendaciones del fabricante. El ADN se reconstituyó en 100 µl de solución de rehidratación y se almacenó a -30 °C hasta su uso.
La calidad del ADN se determinó estableciendo la relación de absorbancia A260/A280 con un espectrofotómetro Thermo Scientific NanoDrop 2000™.
La presencia de H. pylori se determinó por los alelos s y m del gen vacA, según la metodología descrita previamente 18. Se utilizó una prueba de PCR múltiplex (Qiagen Multiplex PCR®) para amplificar los genes vacA y cagA, con las siguientes secuencias de cebadores: vacA s F 5’ - ATGGAAATACAACAAACACAC - 3’; R 5’ - CTGCTTGAATGCGCCAAAC - 3’; vacA m F 5’ - CAA TCT GTC CAA TCA AGC GAG - 3’; R 5’ - GCG TCT AAA TAA TTC CAA GG - 3’ y cagA F5’-GTTGATAACGCTGTCGCTTC-3’, R 5-GGGTT GTATGATATTTTCCATAA’3’. La reacción incluyó una mezcla de 2 µl de ADN genómico bacteriano, 1X de Qiagen Master Mix®, 0,3 pmol/µl de cada cebador, y 2 µl de agua libre de ARNasa en un volumen de 12 µl de reacción.
Los productos de amplificación se evidenciaron en geles de agarosa al 1,5 % por tinción con bromuro de etidio bajo luz ultravioleta, tomando como referencia las bandas de amplificación de las cepas NCTC 11637 (genotipo: cagA, vacA s1/m1) y 3062 (genotipo: cagA negativo, vacA s2/m2) de H. pylori.
Análisis del gen rdxA
Se amplificó una región de 460 pb del gen rdxA en las muestras positivas para H. pylori determinadas por histopatología y mediante la genotipificación del gen vacA. Se usaron los iniciadores rdxA F 5’- GACAATTATTAAACGAGCGC-3’ y rdxA R 5’- CCTCCAATAATGCAACTATC-3’ correspondientes a las posiciones 1013697 a 1014157 de la cepa de referencia 26695 (acceso en Genbank: gb| CP003904.1) (19), reportados por Kwon, et al.20.
Para la PCR, se usó una mezcla con 2,7 U de Taq ADN polimerasa recombinante (Invitrogen™, Brasil), 5 µl de ADN genómico bacteriano, 0,5 pmol/µl de cada cebador, 1,0X de solución tampón de reacción proporcionada por el fabricante (Invitrogen™, Brasil), 2 mmol/L de MgCl2, y 0,5 mmol/L de cada deoxinucleótidotrifosfato (dNTP) (Amersham Pharmacia Biotech, Inc.) en un volumen final de 50 µl de reacción.
Las muestras fueron llevadas al termociclador (MyCycler Termal Cycler™, BIORAD) bajo las siguientes condiciones: desnaturalización inicial durante 9 minutos a 95 °C, 40 ciclos de desnaturalización a 95 °C durante 30 segundos, hibridación a 50 °C durante 45 segundos, extensión a 72 °C durante 45 segundos, y una extensión final a 72 °C durante 5 minutos.
Los productos de amplificación de 460 pb se evidenciaron en geles de agarosa al 2 % por tinción con bromuro de etidio bajo luz ultravioleta y por comparación con la banda de amplificación de la cepa de H. pylori NCTC 11638.
Los productos de la PCR se enviaron a Macrogen® Inc. Company (Corea) para el análisis de secuenciación del gen rdxA. Posteriormente, las secuencias obtenidas se evaluaron usando la herramienta BLASTx (Basic Local Alignment Search Tool) 21. Para determinar los cambios de la nitrorreductasa RdxA, se comparó con la región amplificada de la cepa de referencia 26695 (Genbank HP26695 ) 22 sensible al metronidazol según estudios previos 23. Además, se compararon con las secuencias de las cepas 11638 (identificación de la secuencia: emb|CAC83809.1| y gi|9858509|gb|AF180408.1|). Los cambios reportados fueron relacionados luego con los codones de parada y las mutaciones de cambio de aminoácidos. Se utilizó el programa CLUSTAL W (24) para el análisis de los cambios más significativos en la nitrorreductasa RdxA.
Resultados
Características sociodemográficas de la población
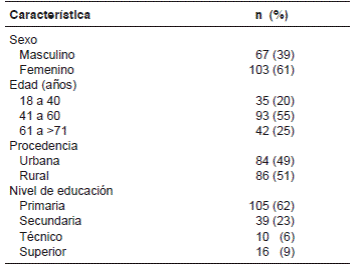
Se incluyeron 170 pacientes diagnosticados por histopatología con infección por H. pylori, y muestras sometidas a genotipificación y análisis del gen rdxA. La distribución de las características de sexo, edad, procedencia y escolaridad de los participantes se muestra en el cuadro 1. La edad de la población de estudio fluctuaba entre los 18 y los 84 años, y la edad promedio fue de 51 ± 13,6 años; la mayoría eran mujeres (103, 60,6 %). El 51 % de la población provenía del área rural y el 62 % tenía un nivel educativo menor o igual a primaria.
Se hizo la genotipificación de todas las muestras y el genotipo más frecuente en esta población fue el vacA s1/m1 negativo para cagA (49,4 %), seguido por el genotipo vacA s1/m1 positivo para cagA (39,7 %), otros genotipos s2/m2 negativos para cagA (5,3 %), el s1m2 negativo para cagA (1,8 %), y el s1m2 positivo para cagA (0,6 %); en 3,2 % de los pacientes se detectaron infecciones múltiples.
Análisis de secuencia de la proteína RdxA
En 92,9 % de las secuencias de la proteína RdxA se detectaron cambios en, al menos, un aminoácido en comparación con la cepa de referencia 26695. En el cuadro 2 se presentan todas las mutaciones distribuidas en codones de parada y mutaciones puntuales desde la posición 46 hasta la posición 160 de la proteína RdxA. Por otra parte, el 78,2 % de las muestras presentó mutaciones relacionadas con la resistencia a metronidazol (49,10,13,22, 25), específicamente mutaciones puntuales tales como R131K (en 101 muestras), R90K (en 97 muestras), en A118T (en 42 muestras), H97T (en 26 muestras), E133K (en 11 muestras), y S108A (en 9 muestras), en tanto que el 21,8 % de las mutaciones puntuales no habían sido reportadas en aislamientos resistentes: la D59N (en 153 muestras), la G98S (en 27 muestras), la H97Y (en 18 muestras), la A118S (en 5 muestras) y la I160F (en 32 muestras) 4,6,12,22,26. Se encontraron, además, mutaciones relacionadas con los siguientes codones de parada: Q50*(en cinco muestras); I160* en seis muestras, E75* en tres, C159* en diez, y en la posición I62* en dos muestras.
Cuadro 2 Frecuencia de mutaciones de aminoácidos en la nitrorreductasa RdxA de Helicobacter pylori
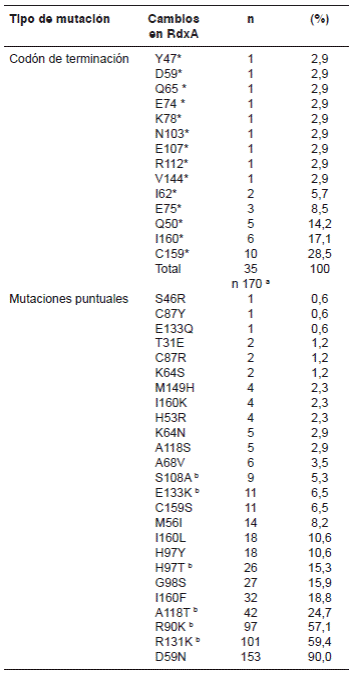
a: número de muestras analizadas para mutaciones puntuales en RdxA
b: cambios asociados a resistencia según los estudios consultados (8,10)
*: codón de terminación en la proteína RdxA
En la figura 1 se observan los tres sitios con mayor variabilidad en la proteína RdxA (R90, H97, R131), así como los codones de terminación en las posiciones 50, 78 y 102. En la posición 133, se encontró una doble sustitución asociada a un cambio en el marco de lectura. En el cuadro 3 se relacionan las mutaciones de inserción en las posiciones Q50*, D59* y E74*, y de sustitución en las posiciones E75*, K78*, N103* y C159*, las cuales generaron codones de terminación y posibles interrupciones del marco abierto de lectura.
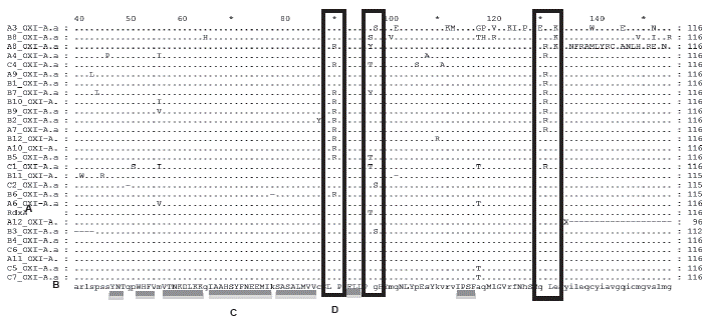
Figura 1 Posiciones variables relacionadas con los cambios en la proteína RdxA de Helicobacter pylori a partir del alineamiento de la secuencia traducida y analizada. A: secuencia rdxA de referencia 11638; B: secuencia de consenso, las letras en minúscula reflejan cambios en los aminoácidos y las letras en mayúscula muestran las zonas conservadas de RdxA; C: regiones poco variables de la proteína RdxA; D: regiones hipervariables de la proteína RdxA (R90, H97, R131)
Cuadro 3 Mutaciones de cambio de lectura (inserciones y sustituciones) relacionadas con la presencia de codones de parada en la proteína RdxA de Helicobacter pylori en la población de estudio
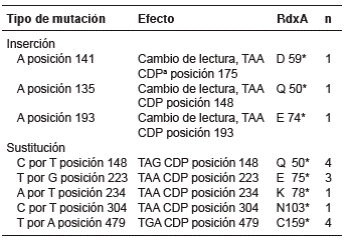
CDPa: codón de terminación
*: codón de terminación en la proteína RdxA
Por otro lado, se compararon los tipos de mutaciones distribuidos en las lesiones gástricas (cuadro 4) en las que fueron más frecuentes las mutaciones puntuales en las diferentes enfermedades gástricas, aunque no se registraron diferencias estadísticamente significativas (p=0,74) en comparación con las mutaciones en los codones de parada.
Discusión
La resistencia de H. pylori al metronidazol se ha atribuido a la inhibición de la actividad de las nitrorreductasas, especialmente por mutaciones en el gen rdxA, las cuales se relacionan con la estabilidad de la proteína, en tanto que la dimerización y la actividad enzimática dependen de la unión al cofactor fosfato de lactoflavina (FMN) 1,5. Las mutaciones que afectan las posiciones A68V, A118T, R90K, R131K, R16H, S88P y H97T, y las que introducen un codón de parada, como N73* y Q50*, se han correlacionado con la resistencia al metronidazol, especialmente cuando hay valores altos en las concentraciones inhibitorias mínimas (CIM) (13,20,27) y cuando la CIM por dilución en agar es igual o superior a 12 µg/ml 13,22,25.
Dichos resultados coinciden con los hallazgos del presente estudio, en el cual las mutaciones R90K, R131K, D59N y H97T fueron las más representativas, en tanto que las más frecuentes fueron la D59N (153/170), seguida por la R131K (101/170), la R90K (97/170), y la A118T (42/170). Mendz, et al., reportaron una compilación de mutaciones en RdxA, incluidas aquellas que representan pérdida de la función de la proteína 4, datos que coinciden con los encontrados en este estudio, especialmente en cuanto a las mutaciones en las posiciones 56, 59, 90, 97, y 118, en las cuales se han documentado cambios en los diferentes aislamientos.
Las mutaciones H53R, M56I, L62V, A68V,H97T y G98S halladas en este estudio concuerdan con lo reportado en otros estudios en aislamientos resistentes al metronidazol 12,13,22; sin embargo, los aislamientos sensibles también han presentado estas mutaciones, lo cual confirmaría las limitaciones del empleo de las mutaciones en RdxA como único marcador de resistencia al metronidazol en H. pylori22. Por ello, se necesitan estudios posteriores de transformación con cepas resistentes y sensibles, para avanzar en la comprensión del papel del gen rdxA en la resistencia a este medicamento 27.
Se ha reportado que las sustituciones e inserciones que causan un codón de terminación prematuro en la proteína RdxA están asociadas con el fenotipo de resistencia 27. Las mutaciones de los codones de parada encontradas en el presente estudio podrían estar asociadas con dicho fenotipo.
En un estudio reciente en Kuala Lumpur, en el cual se emplearon métodos fenotípicos, se estableció que la resistencia primaria al metronidazol en aislamientos clínicos fue de 32,3 %. Al evaluar las variaciones en el gen rdxA y la correspondiente alteración de la proteína RdxA se encontraron cambios en los aminoácidos en 70,2 % de las cepas resistentes y mutaciones asociadas con codones de parada en 56,8 % 28, lo cual evidencia la importancia de evaluar la resistencia mediante pruebas moleculares 29.
En contraposición a estos hallazgos, en otros estudios se ha reportado la presencia de aislamientos resistentes en los cuales las proteínas RdxA estaban intactas, lo cual sugiere que habría otros genes involucrados en la resistencia al metronidazol, como el gen frxA, el cual participa en su activación en el interior de la célula 20,26. Se ha postulado que las mutaciones en la proteína FrxA pueden aumentar los valores de la CIM en los aislamientos resistentes y que las que ocurren en los codones de parada reforzarían el fenotipo resistente 25. Por consiguiente, no hay que desconocer el papel que cumplen otras nitrorreductasas como la frxA, la fdxA y la rpsU en la resistencia al metronidazol 26,27,30.
También, se ha postulado que existen otros mecanismos de resistencia diferentes a las proteínas RdxA o FrxA. Algunas mutaciones en genes relacionados con el potencial de oxidorreducción asociado con el metabolismo del metronidazol y sus productos intermedios activos contra la bacteria, por ejemplo, en la proteína Fur de H. pylori, pueden afectar la sensibilidad al metronidazol al alterar el equilibrio de oxidorreducción mediado por esta proteína, y detener la activación de los productos bactericidas 31.
La combinación de metronidazol con otros antibióticos se utiliza ampliamente en el tratamiento estándar contra la infección por H. pylori y otras infecciones bacterianas anaerobias 32-37. Sin embargo, la resistencia de la bacteria al metronidazol es una de las causas del fracaso del tratamiento de dolencias gástricas 7. En los estudios en los que se emplearon métodos fenotípicos (dilución en agar) en Bogotá, Trespalacios, et al., encontraron una prevalencia de resistencia al metronidazol de 81 % 38; en otro estudio en Pereira, la prevalencia fue de 88 % 29. Estos valores en diferentes poblaciones colombianas indican que la prevalencia de la resistencia a este nitroimidazol supera los límites máximos de 40 % recomendados por el consenso europeo de Maastricht IV 14,39. Según dichas recomendaciones, en áreas geográficas en donde la prevalencia de resistencia sea superior a 40 %, el metronidazol no puede utilizarse en los tratamientos empíricos 39.
A diferencia de los estudios mencionados en Colombia, en otros países latinoamericanos se han reportado datos distintos 8,34: en Chile, se encontró una resistencia al metronidazol de 44,9 %, en una región de México, del 37,1 %, detectada mediante la técnica de E-test 40, y en Brasil se reportaron valores de resistencia de 40,2 % 41. Estas diferencias entre poblaciones indican la importancia de vigilar los niveles de resistencia regional y local, ya que existen diferencias en la prevalencia de la resistencia según las áreas geográficas, lo cual significa que los resultados obtenidos en una región no pueden extrapolarse a otras y, por lo tanto, es fundamental disponer de información específica sobre los niveles de resistencia del microorganismo a los antibióticos usualmente utilizados en los esquemas de erradicación, con el fin de elegir aquellos eficaces y replantear el uso de algunos en el tratamiento empírico 7.
A nivel local no hay publicaciones sobre la resistencia al metronidazol, y aun así, se sigue utilizando frecuentemente en la práctica clínica como tratamiento de primera línea. Lo más preocupante es la ausencia de protocolos para verificar la eficacia de los tratamientos en la erradicación del microorganismo; de hecho, en un estudio en el cual se hizo una encuesta entre médicos generales, especialistas y gastroenterólogos, se encontró que todavía se prescribe el metronidazol como tratamiento de primera línea para la infección por H. pylori42.
En el presente estudio, se determinó por primera vez la frecuencia de las mutaciones en la nitrorreductasa RdxA en pacientes infectados por H. pylori en el departamento del Cauca. En esta zona del país, se ha reportado una gran prevalencia de H. pylori, y se ha sugerido que los aislamientos circulantes en su población están expuestos a la presión de selección dado el uso indiscriminado de antimicrobianos del tipo de los nitroimidazoles como tratamiento antiparasitario, así como contra infecciones gastrointestinales 42 y, especialmente, en infecciones urinarias y vaginales 8,34. Por ello, debe implementarse la vigilancia de las tasas de erradicación de H. pylori en poblaciones con riesgo de cáncer gástrico. La gran frecuencia de mutaciones (78 %) en la nitrorreductasa RdxA podría estar relacionada con la resistencia al metronidazol.
Se sugiere hacer estudios fenotípicos adicionales para relacionar los resultados de los análisis genéticos obtenidos con las CIM del antibiótico. En los eventuales estudios que se lleven a cabo, debería hacerse el antibiograma de las muestras seleccionadas en pacientes con recurrencia de la infección para establecer la relación con los hallazgos que aquí se presentan. Por ello, se propone emplear el cultivo bacteriano como una herramienta necesaria en pacientes en quienes el tratamiento ha fallado, según lo recomendado por Malfertheiner, et al.14. Por lo pronto, el análisis de las mutaciones de la proteína RdxA es una excelente aproximación para analizar la resistencia en un gran número de muestras en poblaciones como la de Popayán, en donde los estudios son escasos y, además, no existen las condiciones para cultivar la bacteria. Dada la alta tasa de incidencia de la enfermedad gastrointestinal en Popayán, se deberían proponer programas de prevención de la infección por H. pylori y de promoción del uso adecuado de medicamentos.
En conclusión, este es el primer estudio en el departamento del Cauca en el cual se han evaluado las mutaciones de la nitrorreductasa RdxA, asociadas en diversos estudios con la resistencia de H. pylori al metronidazol en pacientes con dispepsia funcional. Se requieren nuevos estudios para evaluar el impacto de la resistencia en el fracaso del tratamiento en la población de esta región y para establecer la prevalencia de la resistencia a los otros antimicrobianos utilizados para combatir la infección.