La tuberculosis es una enfermedad infecciosa causada por Mycobacterium tuberculosis que compromete principalmente los pulmones, pero puede afectar otros órganos y tejidos (1). La infección suele ser asintomática en personas sanas, pues su sistema inmunológico las protege de la bacteria. Actualmente, la tuberculosis es una de las enfermedades infecciosas con mayor morbilidad y mortalidad en el mundo (2,3). Aproximadamente, un tercio de la población mundial está infectada con el bacilo que la causa (4) y en 22 países de ingreso medio y bajo se acumula cerca del 80 % de los casos en el mundo (5).
Aunque la enfermedad es tratable, el mal uso y el poco cumplimiento del tratamiento antibiótico han generadoresistencia, lo cual ha dado lugar a la aparición de la tuberculosismultirresistente, considerada hoy como un problema de salud pública a nivel mundial (6,7). La Organización Mundial de la Salud (OMS) estima que entre 2011 y2015 más de dos millones de personas desarrollaron la enfermedad multirresistente (8).
Aunque la morbilidad de la tuberculosis ha decrecido en los últimos años, la incidencia y la prevalencia de la forma multirresistente se han incrementado (8,9). Sus patrones de transmisión varían según las diferencias geográficas, el desarrollo socio-económico de los territorios y las epidemias de la enfermedad en áreas y poblaciones específicas (10). La OMS estima que anualmente aparecen 650.000 casos de tuberculosis multirresistente (al menos a isoniacida y rifampicina, que son los medicamentos de primera línea más eficaces), y casi 50 % de ellos se registran en India, China y Rusia (6).
En algunos casos, la resistencia a los fármacos puede provocar la aparición de tuberculosis extremadamenteresistente (11). Se estima que cerca de 9,6 % de los casos de tuberculosis multirresistente también son extremadamente resistentes (8). Según datos de la OMS para el 2014, el 3,3 % de los nuevos casos de tuberculosis y el 20 % de los casos previamente tratados, correspondían a tuberculosis multirresistente, en tanto que 190.000 personas murieron.
Uno de los principales obstáculos para detener la aparición de la resistencia a los medicamentos es la falta de acceso a pruebas diagnósticas. Se estima que solo 58 % de los pacientes previamente tratados y 12 % de los pacientes nuevos, han tenidoacceso a una prueba que permita detectar la resistencia a medicamentos (12). La detección oportuna de la tuberculosis multirresistente y de la extremadamente resistente permitiría un tratamiento más efectivo y, por lo tanto, reduciría la carga de la enfermedad, y su impacto social y económico en los sistemas de salud (13).
En 2008, la OMS validó el uso del ensayo GenoType MTBDRplus(r) (GTPlus) (Life Science, Nehren, Alemania, V1.0) para la detección rápida y simultánea del complejo M. tuberculosis, y la resistencia a rifampicina e isoniacida a partir de cultivos o muestras clínicas pulmonares con baciloscopia positiva (14,15). La prueba se basa en una reacción en cadena de la polimerasa múltiple (PCR múltiple), la cual genera diversos productos de amplificación (sondas) que reconocen las mutaciones génicas más frecuentes asociadas con la resistencia a isoniacida y rifampicina mediante hibridación inversa; los resultados se obtienen en ocho horas (16-18).
En 2009, la compañía Hain Life Science introdujo la prueba comercialGenoType MTBDRsl(r) (GTSL) para detectar la resistencia a medicamentos de segunda línea contra la tuberculosis. La prueba detecta las mutaciones más comunes en los genes gyrA, rrs y embB, relacionadas con la resistencia afluoroquinolonas, aminoglucósidos y etambutol (19,20).
En 2010, la OMS autorizó el uso de la prueba Gene Xpert MTB/RIF(r) (GX) (Cepheid, Inc., CA, USA) en países endémicos (21). Este es un método molecular automatizado que integra la extracción de ADN, la amplificacióngenómica por PCR en tiempo real, la detección semicuantitativa y la detección de la resistencia a la rifampicinadebida a mutaciones en el gen rpoB, con lo cual el diagnóstico de tuberculosis y la detección de resistencia a estemedicamento se obtienen en dos horas (22).
Según los lineamientos de la OMS para el 2015, las pruebas de diagnóstico molecular deben utilizarse en donde haya alta incidencia de tuberculosis, en pacientes con infección simultánea con HIV y cuando exista gran sospecha de tuberculosis multirresistente. Estas pruebas tienen la ventaja de brindar resultados rápidos, comparadas con los métodos tradicionales de cultivo, con los cuales se requieren, en promedio, cuatro semanas para obtener un resultado. Sin embargo, se recomienda que se haga una prueba fenotípica para confirmar el resultado de la prueba molecular (23).
En Colombia, se notificaron 12.720 casos de tuberculosis en 2014, es decir, una incidencia de 26 por 100.000 habitantes (24). Según la última encuesta publicada sobre prevalencia de tuberculosis multirresistente en 2005, la incidencia era de 2,38 % en los casos nuevos y de 31,44 % en los previamente tratados (25). En 2013, el Instituto Nacional de Salud reportó cerca de 378 casos de tuberculosis multirresistente, lo cual evidenció un significativo aumento en la incidencia de la resistencia a los fármacos antituberculosos (26).
En este contexto, se hizo una revisión sistemática de la literatura científica para establecer la precisión diagnóstica de tres pruebas moleculares (GTPlus, GTSL y GX) de detección de tuberculosis multirresistente y tuberculosis extremadamente resistente en estudios en población inmunocompetente.
Materiales y métodos
Búsqueda electrónica y criterios de selección
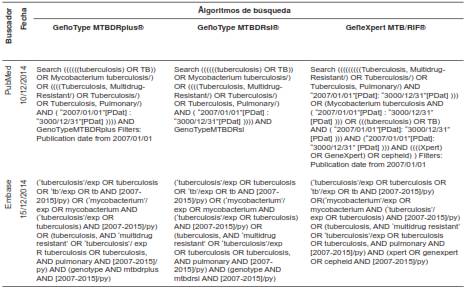
Se buscaron en las bases de datos de Medline de PubMed y en Embase los estudios publicados desde el 2007, por ser este el año de inicio del uso de las pruebas moleculares, y hasta el 15 de diciembre de 2014. En el anexo 1, disponible en https://doi.org/10.7705/biomedica.v34i2.3437, se resumen los algoritmos de búsqueda utilizados en la revisión.
En la selección de los estudios, se incluyeron los ensayos clínicos aleatorios y los estudios observacionales que evaluaron las pruebas GTPlus, GTSL y GX, ya fuera por separado o en conjunto, o que las hubieran comparado con los estándares de referencia para el diagnóstico de tuberculosis multirresistente o tuberculosis extremadamente resistente en población adulta sin diagnóstico de infección por HIV. Solo se incluyeron los estudios con acceso al texto completo.
Definición de tuberculosis multirresistente y tuberculosis extremadamente resistente
La tuberculosis multirresistente se define como la causada por un organismo resistente, por lo menos, a los dos medicamentos más utilizados en el tratamiento de esa enfermedad (isoniacida y rifampicina) (27) y la tuberculosis extremadamente resistente, como aquella con resistencia a isoniacida y rifampicina, a una fluoroquinolona (levofloxacina, moxifloxacina u ofloxacina) o, por lo menos, a uno de tres aminoglucósidos inyectables (amikacina, kanamicina o capreomicina) (27).
Selección de estudios
Después de la búsqueda inicial para excluir duplicados, dos revisores exploraron de forma independiente los títulos y los resúmenes de los artículos para detectar los estudios elegibles con acceso al texto completo. Las discrepancias fueron resueltas por otros dos revisores. Los estudios se etiquetaron tomando como referencia el apellido del autor y el año de publicación.
Selección de variables y reporte de datos
Las variables de análisis fueron la temporalidad del estudio (prospectivo, retrospectivo), el año de publicación, el país y la clasificación del ingreso según el Banco Mundial (28), el tipo de selección de los participantes (consecutiva, por conveniencia, aleatoria) y las medidas de precisión diagnóstica (sensibilidad, especificidad, valores predictivos positivos y negativos). Los datos se almacenaron y seprocesaron en Microsoft Excel(r) 2013.
La precisión diagnóstica de las pruebas se determinó con base en los valores máximos y mínimos de sensibilidad y especificidad, y los valores predictivos positivos y negativos.
Resultados
Estudios incluidos en las revisiones sistemáticas
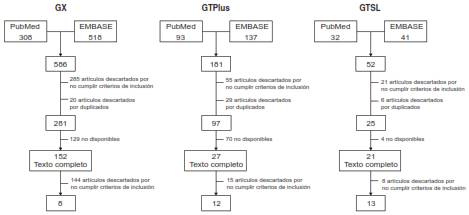
El flujograma de inclusión y exclusión de estudios se describe en la figura 1.
Capacidad diagnóstica de GTPlus, GTSL y GX
GeneXpert MTB/RIF(r) (GX). Con los algoritmos de búsqueda se encontraron 826 artículos (240 duplicados). Se revisaron los títulos y los resúmenes de los 586 restantes y, finalmente, se escogieron 281. De estos, 152 tenían el texto completo, y ocho (22,27,29-34) cumplían con los criterios de inclusión para la evaluación de la tuberculosis multirresistente. De los ocho incluidos, dos eran estudios multicéntricos (22,29) para evaluar la tuberculosis multirresistente en cinco países cada uno, y otro era una revisión sistemática (34) de la cual se extrajo la información de 12 estudios para evaluar la prueba GX en diversos países del mundo.
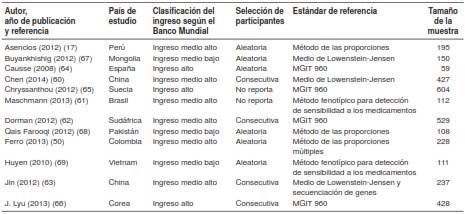
Se analizaron en total 28 estudios en sendos países, de los cuales 12 (42,9 %) se realizaron en países de ingreso alto (27,30,31,35-43), diez (35,7 %), en países de ingreso medio alto (22,29,33,44,45), cuatro (14,3 %), en países de ingreso medio bajo (22,29,32), y dos (7,1 %), en países de ingreso bajo (29,46). Veinte estudios (71,4 %) eran prospectivos, cuatro (14,3 %) eran retrospectivos y cuatro (14,3 %) combinaban el diseño retrospectivo y el prospectivo. Según el tamaño de la muestra, el estudio con la muestra más pequeña fue el de Malbruny (39) y el de la muestra más grande, el de Hanrahan (44) (cuadro 1).
Cuadro 1 Estudios que cumplieron con los criterios de inclusión en la revisión sistemática de las pruebas GX y GTSL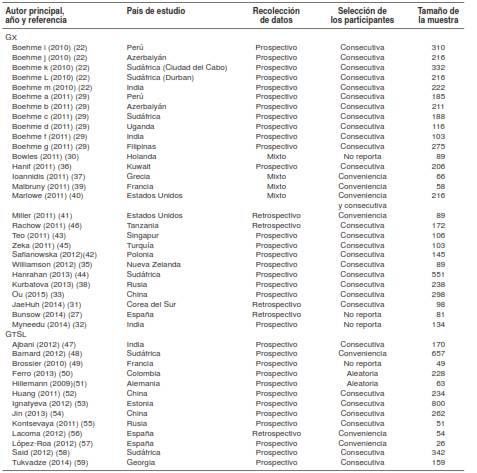
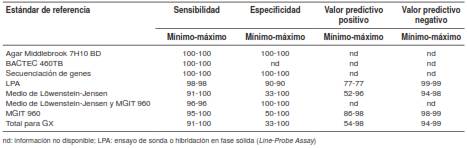
Con respecto al estándar de referencia para la tuberculosis multirresistente en los estudios de evaluación de la prueba GX, el más utilizado fue el Mycobacteria Growth Indicator Tube (MGIT960), empleado como referencia en la mitad de los estudios (22,29-32,35,38-41,44,46). El segundo más frecuente fue el medio de Löwenstein-Jensen(32,1 %), utilizado en nueve estudios (22,29,33,37, 42). En otros estudios se utilizó el agar Middlebrook 7H10 (45), el BACTEC 460TB (36), la secuenciación de genes (43) y la prueba de sonda o hibridación en fase sólida (Line-Probe Assay, LPA) (29).
La especificidad de la prueba osciló entre 91 y 100 %, y la sensibilidad, entre 33,3 y 100 %. La precisión diagnóstica de esta prueba se resume en el cuadro 2.
Cuadro 2 Sensibilidad, especificidad y valores predictivos positivos y negativos de la prueba GX en comparación con el estándar de referencia
GenoType MTBDRplus(r) (GTPlus). Se detectaron 181 artículos (49 duplicados), y se escogieron finalmente los 97 que se ajustaban a los criterios de inclusión. De estos, 27 tenían el texto completo y 12 cumplían con los criterios de inclusión para la evaluación de la tuberculosis multirresistente y de la extremadamente resistente.
De los 12 estudios incluidos, la mitad se llevó a cabo en países de ingreso medio alto (17,50,60-63), tres, en países de ingreso alto (64-66), y los restantes, en países de ingreso medio bajo (67-69), según la clasificación de ingresos del Banco Mundial.
En cuanto al tamaño de la muestra, el estudio de Causee (64) tuvo el menor número de participantes y el de Dorman (62), el mayor (cuadro 3).
En más de la mitad de los estudios se utilizaron el MGIT960 (62,64-66) y el medio de Löwenstein-Jensen (60,63,67) como estándares de referencia. En el 50 % de los estudios se reclutó a los pacientes o se seleccionaron las muestras de forma aleatoria(17,50,64,67-69), en el 33 %, de forma consecutiva (60,62,63,66), y en el resto no se reportó esta información (cuadro 3).
En el cuadro 4 se presenta el resumen de los valores mínimos y máximos reportados en los 12 estudios incluidos. Estos valores correspondían a la especificidad y la sensibilidad de la prueba GTPlus para detectar la resistencia a isoniacida y rifampicina, así como la multirresistencia general, y se presentan según los diferentes estándares de comparación utilizados en los estudios.
Cuadro 4 Valores máximos y mínimos de sensibilidad y especificidad, y valores predictivos positivos y negativos de la prueba molecular GTPlus
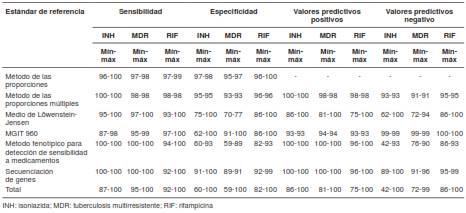
La sensibilidad de la GTPlus para evaluar la resistencia a la rifampicina varió entre 92 y 100 %, y la especificidad frente a la isoniacida fue mayor (60 a 100 %), es decir, hubo estudios en los que la especificidad de la prueba fue de 60 %, como en el caso del estudio de Maschmann (61).
Los valores predictivos positivos y negativos de la GTPlus reportados en los estudios seleccionados se presentan de forma detallada y agrupada en valores máximos y mínimos en el cuadro 4.
GenoType MTBDRSL(r) (GTSL). Se encontraron 52 artículos después de la revisión por títulos y resúmenes. Se seleccionaron 25, de los cuales 21 estaban disponibles en texto completo. Se incluyeron 13 estudios, seis (46,2 %) realizados en países de ingreso medio bajo (47,52-55,59), cuatro (30,8 %) en países de ingreso alto (49,51,56,57) y tres (23,1 %) en países de ingreso medio-alto (48,50,58). Se destacó el hecho de que no se encontraron estudios en países de ingreso bajo. Los países con más estudios de esta prueba fueron China, España y Sudáfrica, con dos estudios cada uno.
En un poco más de la mitad de los estudios (53,8 %) se utilizó el reclutamiento de pacientes o la selección de muestras de forma consecutiva (47,52-55,58,59), en tres (23,1 %), por conveniencia (48,56,57), en dos (15,4 %), de manera aleatoria, y en uno no se reportó esta información. Solo un estudio fue de tipo retrospectivo (56), en el resto se utilizó un diseño de recolección de datos prospectivo (cuadro 1). Según el número de participantes, en el estudio de López-Roa (57) se reclutaron 26, y en el de Ignatyeva (53), en Estonia, la muestra fue de 800 (cuadro 1).
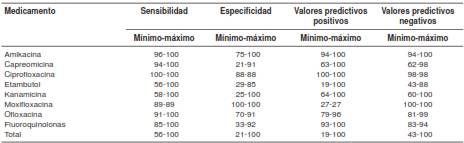
En los estudios incluidos para evaluar la precisión diagnóstica de la GTSL se estimaron la especificidad, la sensibilidad, y los valores predictivos positivos y negativos de diferentes tratamientos utilizados para combatir la tuberculosis extremadamente resistente. En ellos, el medicamento en el cual hubo menor precisión diagnóstica fue el etambutol, puesto que hubo estudios como el de Said (58) en los que la especificidad y la sensibilidad, así como los valores predictivos positivos, registraron valores bajos comparados con los de los demás medicamentos. De otra parte, en el estudio de Said (58), la prueba presentó una baja sensibilidad para la capreomicina, comparada con otros medicamentos. La sensibilidad y los valores predictivos positivos presentaron un mayor rango entre los valores mínimos y máximos (cuadro 5).
Discusión
La implementación de una nueva tecnología diagnóstica debe garantizar, como mínimo, el aumentode la proporción de casos oportunamente diagnosticados, lo cual puede facilitar la reducción de la carga de la enfermedad y el aumento de los pacientes curados (70).
En la novena reunión del Comité Técnico Asesor para el Control de la Tuberculosis de la OMS, se recomendó que los países deberían fortalecer la capacidad instalada de sus laboratorios para hacer diagnósticos oportunos de la tuberculosis multirresistente. Se reconoció que las pruebas moleculares presentan considerables ventajas para dicho diagnóstico debido a su gran sensibilidad y especificidad, su capacidad para entregar resultados rápidamente y su bioseguridad (71).
En el presente estudio se describió la precisión diagnóstica de las pruebas GX, GTSL y GTPlus, utilizadas en muchos países por su efectividad para detectar la tuberculosis multirresistente y la extremadamente resistente.
El uso de pruebas moleculares para confirmar la presencia de la tuberculosis multirresistente o la extremadamente resistente de forma rápida, es muy importante, especialmente en países con alta frecuencia de tuberculosis resistente a medicamentos. Aunque en Colombia la frecuencia de casos de tuberculosis multirresistente y extremadamente resistente no sea alta, se debe conocer la precisión diagnóstica de estos métodos para estimar su costo-efectividad e incorporarlos en los correspondientes esquemas o guías de manejo.
En la presente revisión se constató que la aplicación de estas tres pruebas fue eficaz para el diagnóstico de la tuberculosis multirresistente y de la extremadamente resistente en la mayoría de los estudios evaluados. La sensibilidad de la prueba GX fluctuó entre 33 y 100 %, y la especificidad, entre 91 y 100 %; la sensibilidad de la GTPlus estuvo entre 59 y 100 %, y la especificidad, entre 87 y 100 %para la detección de resistencia a isoniacida y rifampicina, así como de multirresistencia, en tanto que en la GTLS fluctuaron entre 21 y 100 % y 56 y 100 %, respectivamente.
El objetivo de la presente revisión sistemática no se limitó a analizar la precisión diagnóstica de las pruebas moleculares, pues también se describieron las características de los estudios. La mayoría de ellos se hicieron en países de ingreso alto y medio alto, lo cual evidencia la falta de investigación en esta área en países pobres.
La selección de la mejor prueba diagnóstica de la tuberculosis en un país no puede depender únicamente de su efectividad y su costo. Se deben tener en cuenta, asimismo, los datos epidemiológicos existentes, el modelo de provisión de servicios de salud, el recurso humano disponible y la infraestructura de los servicios de atención (72). La existencia de estudios hechos en Colombia es un buen antecedente para precisar el alcance diagnóstico de estas pruebas y orientar su uso en nuestro medio (50).
Entre las fortalezas del presente estudio, se destaca la exhaustiva estrategia de búsqueda que permitió obtener resultados afinados. Otra fortaleza fue la participación de varios autores en las etapas de revisión y extracción de los datos de los estudios, pues ello permitió resolver por consenso las discrepancias. Además, la revisión sobre la precisión diagnóstica de tres pruebas moleculares resulta un aporte importante al estudio de la resistencia a los fármacos para la tuberculosis, especialmente cuando la evaluación de las pruebas médicas está rezagada con respecto a la evaluación de los tratamientos (73).
En la presente revisión no se hizo el metaanálisis de los resultados de los estudios. Sin embargo, en las tres pruebas evaluadas, los valores mínimos de especificidad y sensibilidad eran valores extremos, los cuales no reflejaban la distribución de las estimaciones en la mayoría de los casos. Ello pudo deberse a las características de la población con tuberculosis o tuberculosis multirresistente y a la complejidad de la casuística, o a aspectos metodológicos específicos de cada uno de los estudios, lo cual tiene un efecto más patente en la sensibilidad de los procedimientos diagnósticos. Por ejemplo, en las pruebas GX, la sensibilidad mínima encontrada fue de 33 %, pero en el estudio con la siguiente sensibilidad mínima, esta fue de75 %, lo cual permitiría especular que este valor fue atípico, ya que los rangos de sensibilidad suelen oscilar entre 75 y 100 %.
Otra posible limitación de la revisión fue que no se hizo búsqueda de la literatura gris, ya que se consideró que, al no ser evaluada por pares, la mayoría de tales estudios no era adecuada para incluirlos en una revisión sistemática de calidad.
En conclusión, según los estudios revisados, los tres métodos de diagnóstico evaluados (GTPlus, GTSL y GX) presentaban un perfil diagnóstico adecuado para detectar la tuberculosis multirresistente y la extremadamente resistente, así como la resistencia a medicamentos de segunda línea en el caso de la GTSL. Sin embargo, es importante tener presente que la implementación de las pruebas GTPlus y GTSL requiere de un laboratorio con equipamiento especial y personal capacitado, y que su rendimiento está determinado, entre otros factores, por la incidencia de la enfermedad. En Colombia, los laboratorios de la red pública y de la privada ya procesan muestras utilizando estas tres pruebas, pero todavía no existen datos concluyentes sobre su desempeño.