Se denomina leishmaniasis a un grupo de antropozoonosis causadas por diferentes especies de parásitos del género Leishmania y trasmitidas principalmente por la picadura de insectos hematófagos de los géneros Lutzomyia spp. para el Nuevo Mundo y Phlebotomus spp. para el Viejo Mundo (1,2). Estas enfermedades son endémicas en más de 98 países, y afectan a 12 millones de personas, en tanto que más de 350 millones están en riesgo de infección en el mundo (3). En Colombia, la leishmaniasis es endémica en casi todo el territorio y representa un problema significativo de salud pública debido a su alta morbilidad. En el país se han estimado alrededor de 10.000 casos por año, y casi 10 millones de personas en riesgo de adquirir la infección, principalmente en áreas rurales (4).
En el Nuevo Mundo se encuentran distribuidas 16 especies de Leishmania, las cuales son causantes de una gran diversidad de síntomas clínicos (3). En Colombia se ha registrado la circulación de las especies L. panamensis, L. braziliensis, L. amazonensis, L. mexicana, L. guyanensis y L. infantum,siendo las dos primeras las de más alta distribución a lo largo del territorio nacional y, por consiguiente, las principales responsables de la alta prevalencia de la leishmaniasis cutánea en el país (4).
El diagnóstico acertado de la infección con Leishmania sp, así como la identificación de la especie infecciosa, son críticos en el momento de escoger e iniciar el tratamiento apropiado. En este sentido, la Organización Mundial de la Salud (OMS) ha recomendado que el tratamiento solo se debe administrar tras la confirmación de la enfermedad, y en algunas regiones del Nuevo Mundo el diagnóstico debe considerar la especie, ya que se ha demostrado que las especies infecciosas y la región geográfica influyen en la eficacia de los tratamientos y, por consiguiente, en las recomendaciones terapéuticas. Esto es de gran importancia debido a la alta toxicidad de los medicamentos disponibles, la resistencia observada en algunas especies, especialmente las causantes de la leishmaniasis cutánea, en la cual parece haber mayor riesgo de selección de parásitos farmacorresistentes, y el uso restringido de los medicamentos en los pacientes confirmados (3). Asimismo, debido a la gran similitud entre los síntomas causados por Leishmania spp. y otros grupos de enfermedades (5,6), y la gran cantidad de especies de Leishmania que circulan en el continente americano, el diagnóstico rápido y certero debe mejorarse (7-9).
En la actualidad no se dispone de un método de gran poder discriminatorio que pueda aplicarse de a lo largo del territorio nacional y, por consiguiente, manera universal al diagnóstico y a la identificación de las especies causantes de la leishmaniasis cutánea (10). El diagnóstico se basa en la descripción de las características clínicas, además del análisis de los datos epidemiológicos y las pruebas de laboratorio, así como en métodos de observación directa de las formas parasitarias mediante microscopía (11). En este contexto, los nuevos métodos basados en blancos moleculares han ido cobrando relevancia por sus valores de sensibilidad y especificidad (12). Tales métodos se basan principalmente en la amplificación de regiones genómicas, como las de los genes hsp70 (13) y del citocromo b (14), o de espaciadores intergénicos ribosomales como el ITS-1 y el ITS-2 rRNA (2,15), o el ADN del cinetoplasto. Además, se dispone de métodos moleculares adicionales, como la electroforesis de enzimas multilocus (MLEE) o la PCR-RFLP, que también pueden emplearse en la identificación de la especie infecciosa (10,11).
Dado lo anterior, se requiere el diseño de estrategias novedosas para la identificación de regiones específicas en las especies de Leishmania, que permitan tanto el diagnóstico como la identificación de la especie. En los últimos años se han logrado grandes avances en el conocimiento de la estructura y la composición genómica de los tripanosomátidos gracias al desarrollo de las técnicas de secuenciación masiva (16,17). Para el género Leishmania se han reportado hasta ahora al menos 14 genomas secuenciados (http://tritrypdb.org/tritrypdb/), entre los cuales se encuentran el de L. panamensis y L. braziliensis, responsables del mayor número de casos de leishmaniasis cutánea en Colombia. A partir de estas bases de datos es posible comparar la estructura y la composición genómica de estas especies, con el fin de identificar regiones específicas que puedan ser útiles para su diagnóstico e identificación, lo cual puede validarse experimentalmente mediante técnicas de PCR avanzadas de alto poder resolutivo como la HRM. En este sentido, en el presente trabajo se propuso la búsqueda y selección de nuevos marcadores moleculares específicos de especie derivados del análisis genómico comparativo de L. panamensis, L. braziliensis y L. guyanensis, que permitan el diagnóstico y la identificación de las especies causantes de la enfermedad en nuestro país.
Materiales y métodos
Material biológico y extracción del ADN
El ADN de las cepas de L. panamensis (cepa UA946, Universidad de Antioquia), L. guyanensis (MHOM/BR/75/M4147), y L. braziliensis (MHOM/ BR/75/M2904) se utilizó para validar los marcadores seleccionados. Se utilizaron muestras del ADN de L. major (MHOM/IL/81/FE/BNI), Trypanosoma cruzi (MRAT/COL/Gal61), T. evansi y T. rangeli (2XeqC2) para evaluar las reacciones cruzadas con otros tripanosomátidos. El ADN se extrajo a partir de cultivos in vitro siguiendo el protocolo del estuche DNeasy Blood & Tissue (Qiagen). La calidad del ADN obtenido se analizó por electroforesis en geles de agarosa al 0,8 % teñidos con bromuro de etidio, y la concentración se determinó mediante la lectura de la absorbancia a 260 nm en un espectrofotómetro NanoDrop 2000 (Thermo Scientific).
Identificación de regiones genómicas específicas en Leishmania spp
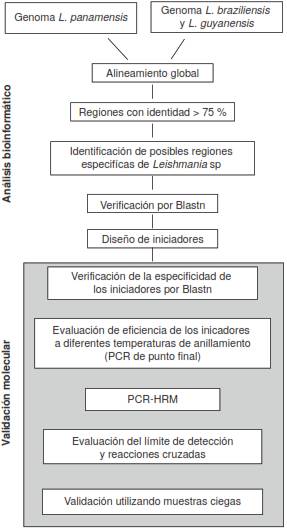
Con el fin de seleccionar regiones genómicas específicas para las tres especies de Leishmania causantes de la leishmaniasis cutánea en Colombia, se hizo un análisis comparativo entre los genomas de L. panamensis y L. braziliensis (publicados en la base de datos de TriTryp: http://tritrypdb.org/tritrypdb/) con los datos crudos del genoma de L. guyanensis (número de acceso en el Genbank: PRJEB168). Un esquema general de la metodología empleada se muestra en la figura 1; el alineamiento global de los tres genomas completos ensamblados se realizó usando el programa MUMmer (18), con el fin de hallar regiones genómicas con identidades mayores de 75 % entre pares de especies. Se seleccionaron las regiones con una identidad menor de 25 % yposteriormente se validaron como regiones específicas de especie mediante BLASTN(r) (blast.ncbi.nlm.nih.gov), contrastándolas con las bases de datos de Assembly, BioProject y SRA del NationalCenter for Biotechnology Information (NCBI). Una vez confirmada la ausencia de ortología entre las especies evaluadas, se diseñaron iniciadores paracada región específica usando la herramienta Primer3 (http://bioinfo.ut.ee/primer3-0.4.0/).
Amplificación de marcadores específicos en L. panamensis, L. braziliensis y L. guyanensis
La amplificación por PCR convencional de los marcadores de cada especie se hizo en un volumen final de 25 µl que contenía 1X de solución tampón Taq, 2,5 mM de MgCl2 , 0,2 mM de dNTP, 0,4 µM de cada cebador, 1 U de Taq polimerasa y 10 ng del molde de ADN. La PCR se hizo en un termociclador T1 (Biometra) bajo las siguientes condiciones: 9 5 °C durante 10 minutos, seguido por 35 ciclos de desnaturalización a 95 °C durante 15 segundos, una temperatura de anillamiento desde 58 °C hasta 64 °C durante 15 segundos , y una extensión a 72 °C durante 20 segundos, con una extensión final a 72 °C durante 10 minutos. Los productos amplificados se visualizaron mediante electroforesis en geles de agarosa al 2 % (w/v). Para cada uno de los marcadores analizados por PCR se utilizó el ADN de L. panamensis, L. guyanensis y L. braziliensis como control.
Curvas de fusión en Leishmania spp. de Colombia
Los marcadores que amplificaron regiones específicas mediante la PCR convencional se evaluaron seguidamente mediante PCR en tiempo real complementada con curvas de fusión de alta resolución en el equipo para PCR en tiempo real Rotor-Gene Q (Qiagen). Cada reacción se llevó a cabo en un volumen total de 10 µl, el cual contenía 5 µl de HRM PCR master mix Type-it(r) 2X (Qiagen), 0,4 µM de cada cebador y 10 ng de molde de ADN. El perfil térmico consistió de un ciclo de desnaturalización y activación de la enzima a 95°C durante 10 minutos, seguido de 40 ciclos de amplificación incluida la desnaturalización de ADN a 95 °C durante 15 segundos y anillamiento a 64 °C durante 1 minuto. Los productos de PCR se sometieron a un programa de curvas de fusión que consistió en la desnaturalización a 95 °C durante 10 segundos, hibridación de ADN de doble cadena a 64 °C durante 1 minuto, seguida de un aumento gradual de la temperatura hasta 95 °C durante 120 minutos, y unión final de ADN a 64 °C durante 15 segundos. Para determinar las temperaturas medias de fusión (Tm) para cada especie se utilizó el programa Rotor-Gene Q v 2.0.2 (Qiagen).
Límite de detección de los marcadores
Con el fin de estimar el límite de detección de los marcadores seleccionados, cada una de las regiones específicas de especie elegidas se clonó en el vector pTZ57R/T (ThermoFisher Scientific) bajo las condiciones previamente reportadas (19).
A partir de estos, se hicieron diluciones seriadas (1:10) del plásmido que contenía la secuencia blanco de los cebadores de cada una de las especies de Leishmania y se estimó la concentración mínima de amplificación. Para ello se utilizaron siete diluciones seriadas por triplicado del ADN de los controles positivos L. panamensis, L. guyanensis y L. braziliensis, que iban desde 1.000.000 hasta una copia del plásmido que contenía la región de interés. Para determinar la eficiencia de cada ensayo se estimaron las curvas de calibración a partir de los ciclos de umbral (threshhold cycles) Ct de las diluciones utilizando el programa Rotor-Gene Q v 2.0.2 (Qiagen).
Reacciones cruzadas
Para corroborar que los marcadores seleccionados no amplificaran en otras especies de tripanosómatidos, el ADN de L. major, T. cruzi, T. evansi y T. rangeli se utilizó como molde para la reacción de PCR bajo las mismas condiciones descritas previamente. Este ensayo se realizó por triplicado con 10 ng del ADN de cada especie.
Utilidad de los marcadores para el diagnóstico de las muestras de los pacientes
Para evaluar la utilidad de los marcadores seleccionados, se probaron en total 41 muestras ciegas que contenían ADN de cepas de cultivo o de muestras de ADN aisladas de biopsias de pacientes de Colombia, así como muestras de humanos negativas (nunca habían estado en contacto con el parásito) y muestras de pacientes presuntivas de infección con leishmaniasis, pero que por otros métodos fueron negativas en los distintos análisis. Todas las muestras positivas se caracterizaron previamente mediante PCR-RFLP utilizando el marcador hsp70 (13,20-22). Este conjunto de muestras ciegas se evaluó mediante PCR-HRM por triplicado, utilizando como control en todas las reacciones el ADN de L. panamensis, L. guyanensis y L. braziliensis. A partir de esta información y con base en los resultados, se determinó la concordancia entre las pruebas PCRRFLP y la PCR-HRM para las muestras positivas.
Validez y seguridad de los marcadores
A partir de los resultados obtenidos se estimaron la sensibilidad (S), la especificidad (E), el valor predictivo positivo (VPP) y el valor predictivo negativo (VPN), que dan cuenta de la validez y la seguridad de los nuevos marcadores. Estos parámetros se calcularon con el paquete estadístico epiR, del programa R, versión 3.2.2 (The R Foundation for Statistical Computing Platform), con intervalos de confianza de 95 % (23,24).
Resultados
Identificación de regiones específicas, validación mediante PCR convencional y determinación de la especificidad analítica
Las comparaciones pareadas entre el genoma de L. panamensis y los de L. guyanensis y L. braziliensis mostró 4.422 y 10.700 regiones genómicas con identidad menor de 25 %, respectivamente. El 98% de las diferencias entre estas regiones en las especies correspondió a deleciones, y solo 2 % a regiones con diferencias específicas en los nucleósidos. Se seleccionaron 24 regiones como específicas de especie, las cuales se emplearon en el diseño de los cebadores para validar la especificidad mediante PCR. Solo tres pares de cebadores amplificaron únicamente en la especie de Leishmania para la cual se diseñaron (figura 2A, cuadro 1). Los otros 21 cebadores amplificaron en más de una especie de Leishmania (figura 2B; figura suplementaria 1 disponible en: https://doi.org/10.7705/biomedica.v34i2.3634; cuadro 1).

Figura 2 A: Productos de la amplificación de los marcadores seleccionados (Pan081, Guy901 y Bra1C). Se observa la amplificación única en la especie de Leishmania para la que el marcador fue diseñado. En los carriles se muestran los siguientes: Lp: L. panamensis, Lb: L. braziliensis, Lg: L. guyanensis, Lm: L. major, Tc: T. cruzi, Te: T. evansi, Tr: T. rangeli y Cn: control negativo. B: Gel representativo de los productos de PCR de los marcadores que amplificaron en más de una especie de Leishmania. En los carriles se muestran los de Lp: L. panamensis, Lb: L. braziliensis, Lg: L. guyanensis, Lm: L. major y Cn: control negativo.
Límite de detección
Los tres marcadores específicos de especie seleccionados se utilizaron en la PCR en tiempo real, con el fin de evaluar su utilidad en el diagnóstico y tipificación en cepas y muestras clínicas. En la figura 3 se observa que cada uno de los marcadores fue capaz de detectar hasta una copia de la secuencia blanco. Este ensayo se hizo por triplicado siempre con el mismo resultado. Además, a partir de los valores de Ct de cada una de las diluciones, se estimaron los porcentajes de eficiencia de la amplificación en cada una de las especies evaluadas: 94,33, 95,73 y 90,92 % para L. panamensis, L. guyanensis y L. braziliensis, respectivamente (cuadro 2).

Figura 3 A. Diluciones seriadas del plásmido que contiene la secuencia blanco de L. panamensis utilizando el marcador Pan081. B. Gel de agarosa al 2 % con los productos de PCR de cada una de las diluciones
Cuadro 2 Valores de temperatura de fusión (Tm), coeficiente de correlación (R 2 ) y porcentajes de eficiencia de L. panamensis, L.guyanensis y L. braziliensis en el ensayo de PCR-HRM

Curvas de fusión
Los tres marcadores que se seleccionaron también se analizaron mediante curvas de fusión. Los resultados indicaron que estos marcadores diferenciaron claramente las especies de L. panamensis, L. guyanensis y L. braziliensis (figura 4). Las temperaturas de fusión (Tm) para cada marcador se muestran en el cuadro 2; se observa que luego de tres corridas estas fueron altamente reproducibles (no se muestran los datos).

Figura 4 A. Temperatura de fusión (Tm) obtenida para cada uno de estos marcadores (Pan081: 86,55 °C para L. panamensis, Guy901: 82,21 °C para L. guyanensis y Bra1C: 85,68 °C para L. braziliensis). B. Curvas de fusión normalizadas para las especies de Leishmania analizadas utilizando los marcadores Pan081, Bra1c y Guy901
Utilidad de los marcadores en muestras de pacientes
De las 32 muestras clasificadas previamente como positivas con otros marcadores, 29 amplificaron con los marcadores seleccionados, lo que significó una concordancia de 90,63 % con la PCR-RFLP del gen hsp70 que se utilizó previamente para caracterizar los aislamientos. Las nueve muestras negativas se clasificaron correctamente (cuadro 3). El análisis estadístico mostró valores de sensibilidad de 91,43 % (IC95% : 7 5 - 9 8 % ) y d e e s p e c i fi c i d a d d e 100 % (IC95% : 55-100 %), un valor predictivo positivo de 100 % (IC95% : 83-100 %) y un valor predictivo negativo de 75 % (IC95%: 43-95 %).
Discusión
En Colombia la leishmaniasis cutánea es causada principalmente por L. panamensis, L. braziliensis y L. guyanensis, especies que responden diferencialmente al tratamiento con glucantime, por lo cual el diagnóstico y la tipificación apropiada de su agente causal son aspectos críticos en el momento de decidir el tratamiento de elección, pues evita someter al paciente a tratamientos que podrían ser ineficaces y muy tóxicos (7,25).
El diagnóstico de la leishmaniasis se ha basado tradicionalmente en la clínica y en criterios epidemiológicos, y ciertas manifestaciones clínicas de la enfermedad se han asociado con complejos de especies o especies particulares, aunque se han reportado infecciones atípicas que cuestionan esta afirmación. Por ejemplo, se han registrado nuevas variantes de la leishmaniasis cutánea en Honduras y Nicaragua causadas por L. infantum, y hay algunos reportes de leishmaniasis visceral causada por L. braziliensis (26). Dado que estas infecciones atípicas son clínicamente idénticas a las típicas y pueden ocurrir en áreas donde se encuentren varias especies de Leishmania, es necesaria la implementación de métodos capaces de distinguir entre varias especies de Leishmania.
Actualmente, la mayoría de métodos de diagnóstico molecular aplicados a la identificación de especies de Leishmania se basan en la técnica de PCR-RFLP, que aunque menos laboriosa que el análisis de isoenzimas (considerado como el método de referencia), presenta problemas de reproducibilidad y su análisis es complejo (12). Para la PCR-RFLP se han utilizado varios blancos moleculares como el gen ssu rRNA, el gen hsp70, microsatélites o ADN mitocondrial (kDNA) (15,27,28), algunos de los cuales son menos sensibles que otros debido a su bajo número de copias. Se han descrito varios ensayos para la detección de parásitos y la discriminación de especies de Leishmania en el Nuevo Mundo (29,30), sin embargo, ninguno de estos métodos cumple con los dos objetivos de manera simultánea y, en ocasiones, pueden carecer de especificidad (31). Un diagnóstico molecular rápido específico de especie permite elegir el tratamiento más adecuado y hacer un seguimiento del parásito para fines epidemiológicos (investigación de brotes o seguimiento de cepas de parásitos resistentes a los medicamentos) (2). El análisis genómico comparativo de estas especies es una herramienta alternativa para la identificación de regiones específicas de especie y para el diseño de nuevos marcadores. En este sentido, el presente trabajo seleccionó regiones ortólogas altamente divergentes en estas tres especies para proponer nuevos marcadores moleculares que permitan el diagnóstico y la tipificación en una prueba simple pero de gran resolución de la PCR, como las curvas de fusión.
La PCR-HRM es una herramienta atractiva para el diagnóstico de diferentes enfermedades. Esta técnica puede evitar el aislamiento de los parásitos en grandes cultivos celulares y métodos posteriores a la PCR, como las electroforesis, los cortes con enzimas de restricción, la hibridación y los métodos de secuenciación, lo cual disminuye los tiempos del diagnóstico y las posibilidades de contaminación (32). Este tipo de prueba también puede arrojar resultados tres veces más rápidamente y cinco veces más económicamente que otro tipo de análisis (33).
En este trabajo se estudiaron una serie de regiones a lo largo del genoma de las diferentes especies de Leishmania distribuidas en Colombia para encontrar las que se ajustaran adecuadamente a los parámetros necesarios para el ensayo de PCR-HRM. Se seleccionaron aquellas que cumplían con los criterios requeridos, entre los cuales se destaca el tamaño de la región (156-200 pb), y que se encontraran de manera específica en cada una de las especies. Así, un total de 24 regiones seleccionadas mediante una serie de análisis bioinformáticos se sometieron a diferentes ensayos de PCR para finalmente seleccionar tres regiones que podían ser potenciales blancos moleculares para el diagnóstico y la tipificación. Esto indica que, a pesar de la rigurosidad con la que se seleccionaron dichas regiones mediante análisis bioinformáticos, siempre es necesario confirmarlas experimentalmente. Los marcadores seleccionados fueron altamente específicos, ya que ningún otro tripanosomátido se amplificó utilizando estos cebadores. Esto es importante porque existe la posibilidad de encontrar infecciones mixtas de Leishmania y T. cruzi (34).
Con los tres marcadores seleccionados se encontró una clara diferencia entre las temperaturas de fusión de las tres especies de Leishmania, lo cual permitiría su identificación incluso si no se cuenta con la herramienta de HRM. Además, los perfiles de las curvas de punto de fusión también sirven para diferenciar entre las especies, lo cual facilita el diagnóstico.
Para validar la utilidad de la PCR-HRM se hizo el diagnóstico de 33 muestras provenientes de pacientes colombianos, las cuales habían sido identificadas previamente a nivel de especie mediante PCR-RFLP del gen hsp70. Los resultados obtenidos mediante las dos técnicas fueron similares en un 90,6 %, lo cual demuestra que los nuevos marcadores son comparables con otro tipo de técnicas utilizadas comúnmente en la actualidad. Es importante resaltar que la prueba que se propone evita el uso de técnicas adicionales como la RFLP para el diagnóstico, lo cual permite que este sea más rápido y facilita la adopción de decisiones. Recientemente se reportó el uso de una nueva prueba molecular para el diagnóstico de leishmaniasis cutánea causada por Leishmania (Viannia) spp, y aunque los resultados son de utilidad para estudios en campo por su capacidad para diferenciar especies del subgénero Leishmania, tiene la limitación de no diferenciar entre las especies del subgénero Viannia (35). La prueba empleada en este estudio tiene la ventaja de ser rápida y diferenciar estas especies, por lo que podría combinarse con la prueba en campo para hacer un diagnóstico diferencial más rápido y beneficioso para el paciente.
Por último, la prueba mostró una especificidad de 100 % y una sensibilidad de 91,4 %, lo que lleva a pensar en el mejoramiento de los métodos previos a la PCR, tales como la toma de muestras, la extracción de ADN y su posterior almacenamiento, de manera que se pueda obtener una mayor concentración de material genético y conservarlo durante mucho tiempo para aumentar el porcentaje de muestras positivas y mejorar la prueba.
La imposibilidad de tener una mayor cantidad de muestras para el análisis fue una gran limitante investigaciones posteriores se valide la utilización de estos marcadores en la PCR-HRM como una herramienta de diagnóstico directa en una mayor cantidad de muestras, no solo de pacientes sino también de reservorios y vectores del parásito. Además, se podría explorar la posibilidad de la búsqueda de un mayor número de marcadores con mayor número de copias, lo cual mejoraría la sensibilidad del método. Todo ello generaría más información para mejorar el diagnóstico de la enfermedad y proponer estrategias para la prevención y el control de la leishmaniasis. Por otro lado, con la PCR-HRM y los marcadores descritos se podría cuantificar la carga parasitaria en las muestras en futuros estudios, lo que es de gran importancia para la vigilancia de la progresión de la enfermedad y el resultado final después del tratamiento (36).
En conclusión, estos resultados indican que el método permite la identificación de Leishmania en muestras clínicas, y es reproducible.
La PCR-HRM representa una herramienta alternativa para la identificación de las especies causantes de leishmaniasis cutánea directamente en las muestras clínicas. Hasta donde se sabe, este es el primer estudio en el que se describen nuevos marcadores para la detección e identificación de especies de Leishmania, y fue posible discriminar tres especies de Leishmania presentes en América. Además, los resultados concordaron con los obtenidos anteriormente mediante la técnica de PCRRFLP, lo que sugiere que esta técnica puede ser de potencial aplicación en la detección de parásitos. No obstante, aún quedan pendientes ensayos queayuden a dilucidar el verdadero poder de diagnóstico y tipificación del método, de manera que se pueda utilizar en los centros de salud de una manera rápida y efectiva y mejorar así el diagnóstico y la administración segura de los tratamientos a los pacientes, disminuyendo su duración, así como los problemas que se puedan presentar por una posible resistencia de los parásitos a los diferentes medicamentos.