Existen de 130 a 150 millones de personas crónicamente infectadas con el virus de la hepatitis C (HCV) 1. Esta infección crónica puede conllevar cirrosis, cáncer hepático y muerte 2-4. El HCV muestra una gran variabilidad genética, con siete genotipos y numerosos subtipos con una divergencia que alcanza el 30 % en genotipos y alrededor del 15 % en los subtipos 5,6. Los genotipos y subtipos del HCV difieren en su distribución geográfica 7.
En Latinoamérica, la prevalencia de la infección por hepatitis C oscila entre 0,3 y 0,9 %, con predominio de la circulación de los genotipos 1 (6-81 %), 2 (0-33 %) y 3 (0-30 %) (7-9). Cabe destacar que la prevalencia más alta para el genotipo 2 en la región, se encuentra en Venezuela 8,10. La diversidad genética del HCV en Latinoamérica y el Caribe es más restringida que la que se observa en el continente africano, en general, con una menor variedad de subtipos y menor diversidad entre los aislamientos circulantes 11.
En Venezuela, los subtipos predominantes del HCV son el 1a, el 1b y el 2j; el genotipo 2j es el más frecuente, aun considerando el número relativamente grande de subtipos circulantes del genotipo 2. En países vecinos, como Colombia y Brasil, el subtipo 2j no se ha identificado y se reporta la circulación de subtipos más comunes, como el 2a, el 2b y el 2c 10,11.
Un factor ampliamente reconocido implicado en la patogénesis viral es el genotipo del HCV involucrado en la infección, según el cual la reacción al tratamiento puede variar 12. El tratamiento de la infección crónica por el HCV se ha revolucionado con el surgimiento de los antivirales de acción directa, en un intento por optimizar un tratamiento efectivo para todas las variantes del virus 13.
Como sucede con el tratamiento con el interferón ‘pegilado’ y la ribavirina, la selección de un antiviral de acción directa, la duración del tratamiento y la reacción virológica sostenida dependen del genotipo y el subtipo del HCV 14,15. Poco se conoce sobre la distribución de más de 67 subgenotipos del HCV. Potencialmente, la diversificación del virus podría afectar las tasas de reacción virológica sostenida si surgen sustituciones asociadas con la resistencia. Estas mutaciones dependen del subtipo y afectan la efectividad del tratamiento 16.
Dado el impacto mundial de la infección por el HCV y debido a que los estudios sobre la diversidad de sus subtipos son escasos en Latinoamérica, es importante contar con herramientas para determinar el genotipo, que permitan la búsqueda de información más precisa y correcta sobre la dinámica de la distribución de los genotipos y subtipos del virus.
En este sentido, el objetivo de este estudio fue evaluar el desempeño de distintos métodos, la secuenciación de ácidos nucleicos y la hibridación inversa 17, para determinar el genotipo del HCV e identificar el subtipo 2j, muy prevalente en Venezuela.
Materiales y métodos
Población de estudio
Previa autorización y consentimiento informado por parte de los participantes, se analizaron 65 muestras de suero o plasma provenientes de pacientes infectados con el HCV que acudieron al servicio diagnóstico entre los años 2009 y 2012. La mayoría de los pacientes eran crónicos, con diagnóstico previo de infección por el HCV y con seguimiento médico especializado para ser sometidos a tratamiento antiviral. Las muestras se procesaron inmediatamente o se mantuvieron a una temperatura de -70 °C hasta su posterior análisis.
Determinación del genotipo viral
Las muestras se evaluaron mediante el ensayo de hibridación inversa Versant HCV Genotype 2.0 Assay (LiPA)™ (Innogenetics, distribuido por Siemens, Healthcare Diagnostic, USA). Asimismo, se evaluaron el genotipo y el subtipo mediante la secuenciación de ácidos nucleicos a partir de las regiones genómicas 5’NC y NS5B del HCV 10. Las secuencias de nucleótidos obtenidas fueron alineadas con un panel de secuencias de referencia depositadas en la base de datos de GenBank.
Se hizo el análisis filogenético mediante el método neighbor-joining, basado en matrices de distancias estimadas por Kimura de dos parámetros, utilizando el programa DNA man, versión 5.2.2 (Lynnon Biosoft, Canadá). Los análisis fueron sustentados con valores de confianza (bootstrap) de 1.000 réplicas y se crearon árboles filogenéticos basados estrictamente en el porcentaje de divergencia genética.
Análisis estadístico
El grado de concordancia entre las metodologías empleadas se estableció mediante el cálculo del coeficiente kappa (κ) 18.
Resultados
En este estudio, se analizaron 65 muestras provenientes de sueros de pacientes que presentaron anticuerpos contra el HCV (anti-HCV). A partir de una reacción en cadena de la polimerasa (PCR) anidada en la región 5´NC del HCV (5´NC), se logró identificar el genotipo circulante en el 100 % de las muestras, a diferencia del 91 % (59/65) con el ensayo de hibridación inversa (LiPA) y el 77 % (50/65) al emplear la región NS5B del HCV (NS5B).
El análisis de los resultados reveló la circulación predominante de dos genotipos representados por tres subtipos, específicamente 1a, 1b y 2j. Sin embargo, la asignación del subtipo 2j se determinó únicamente a partir de la amplificación de la región NS5B, mientras que, con los métodos 5´NC y LiPA, los aislamientos del subtipo 2j fueron clasificados en su mayoría como subtipo 2ac o, en su defecto, como genotipo 2. Los métodos mostraron una concordancia del 100 % en cuanto a la asignación de genotipos, a diferencia de lo observado en la asignación de los subtipos circulantes. Al comparar la asignación de subtipos de los métodos 5´NC y LiPA con NS5B, se observó una concordancia de 70 y 66 %, respectivamente. En el cuadro 1 se muestra la correlación de las asignaciones de subtipos entre los métodos LiPA y NS5B. La discrepancia entre subtipos del HCV se detectó, particularmente, para aquellos subtipos poco comunes del genotipo 2.
Cuadro 1 Concordancia entre los métodos NS5B y LiPA
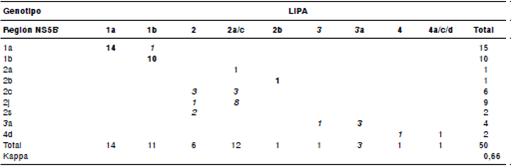
Las letras en negrilla corresponden a una concordancia total y, las letras en cursiva, a discrepancias en subtipo.
En la figura 1 se muestra el árbol filogenético usando las secuencias de la región NS5B. Se observó una gran diversidad de subtipos dentro del genotipo 2, con cinco subtipos (2a, 2b, 2c, 2j y 2s), algunos de los cuales no fueron identificados por los otros métodos de determinación de genotipo. En cuanto a los genotipos 4, el análisis en la región NS5B permitió clasificarlos como subtipo 4d.

Figura 1 Árbol filogenético de la región NS5B de aislamientos del HCV. Se estimó la distancia genética por el método de Kimura de dos parámetros y se construyó el árbol filogenético con el método de neighbor-joining. Se muestran los valores de bootstrap por encima de 80 %. Los aislamientos están designados con su número de acceso en el GenBank y su genotipo, salvo los aislamientos venezolanos analizados en este estudio, que se muestran en negrilla. Se muestra el patrón de bandas obtenidas por LiPA para las muestras de genotipo 2.
Debido a la gran frecuencia del subtipo 2j en el grupo estudiado, lo cual es poco común fuera de Venezuela, se quiso evaluar si existía un patrón distintivo de hibridación para este subtipo mediante el LiPA. Sin embargo, no se observó un patrón sugestivo de la presencia del genotipo 2j, ya que los aislamientos 2j presentaron cuatro patrones distintos de hibridación, no pudiéndose discriminar las muestras con genotipo 2j de las otras con genotipo 2. Ninguna de las muestras con genotipo 2j mostró banda de hibridación en la región de la cápside. De la misma manera, las dos muestras clasificadas como genotipo 2s por la región NS5B, mostraron el mismo patrón de bandas en la tira de nitrocelulosa con el método LiPA, siendo asignadas como genotipo 2 (figura 1).
Discusión
El presente estudio permitió hacer un análisis comparativo de diferentes metodologías para determinar el genotipo circulante del HCV. La amplificación del ARN viral por los métodos LiPA y 5´NC mostró la mayor sensibilidad, de 91 % y 100 %, respectivamente, comparada con una de 77 % obtenida por el método NS5B. Estos resultados concuerdan con los reportados en otros estudios, en los cuales los métodos basados en la amplificación de la región 5’NC mostraron un alto grado de sensibilidad 17,19-21. No se puede descartar que el almacenamiento de las muestras o una baja carga viral hayan afectado el éxito de amplificación por medio de alguna de estas técnicas. Sin embargo, la baja sensibilidad del método NS5B ha sido reportada previamente, en particular, para aislamientos virales del genotipo 2 10.
En términos de calidad de asignación de genotipos, los resultados mostraron el 100 % de correlación por los diferentes métodos empleados. Este alto grado de correlación es similar a lo reportado por otros autores 17,22. La mayoría de las discrepancias observadas en los subtipos con las determinaciones por LiPA y 5´NC respecto al método de referencia, se presentaron en aislamientos del genotipo 2, lo que concuerda con los reportes de Cai, et al., en 2013, en los cuales la identificación de los subtipos con la técnica LiPA y la secuenciación de la región NS5B alcanzó una concordancia de solo 45 %, en el caso del genotipo 2 23. Welzel, et al., en 2017, observaron una mayor discordancia en la genotipificación de aislamientos del genotipo 2, al comparar el método LiPA con la secuenciación, probablemente debido a la gran diversidad de sub-tipos dentro de este genotipo 16.
El análisis filogenético de la región NS5B permitió identificar una gran diversidad de subtipos presentes en los aislamientos venezolanos del genotipo 2; además de la asignación de subtipos comunes, como 2a, 2b y 2c, se identificó la circulación de los subtipos 2j y 2s. La bondad de la técnica de NS5B para identificar subtipos poco comunes ha sido descrita por varios autores; por ejemplo, Margall, et al., detectaron los subtipos 2j y 2q en muestras circulantes en España 24. En Venezuela, la alta prevalencia del subtipo 2j permitió la secuenciación del genoma completo de este subtipo para su identificación 10. En Europa, se ha encontrado el genotipo 2j en España y Francia, con prevalencias que oscilan entre 0,9 y 2,3 %. 24. En Argentina, se identificó un aislamiento de subtipo 2j en una población de donantes voluntarios 25. El subtipo 2s, aunque no es muy frecuente en Venezuela, ha mostrado ser recurrente en distintas regiones del país 10,26. Las secuencias venezolanas del HCV de genotipo 2j se agruparon dentro de un cladomonofilético 10; a pesar de ello, se observaron diversos patrones de hibridación mediante la técnica LiPA.
La relativa alta prevalencia del genotipo 2 en Venezuela, con respecto a otros países suramericanos, podría deberse a varios factores. En primer lugar, podría deberse a la relativa baja frecuencia de subtipos que circulan entre usuarios de drogas intravenosas, como el subtipo 3a, ya que la frecuencia de su uso es relativamente menor en Venezuela 27. En segundo lugar, no se puede descartar una ventaja en la diseminación del subtipo 2j en Venezuela por haber sido introducido en la época colonial, mientras que los otros sub-tipos, como 1a y 1b, fueron introducidos el siglo pasado 10. Finalmente, se desconoce si existe un factor de riesgo asociado con la infección por el subtipo 2j; no se descarta que la transmisión sexual pueda influir 10, lo cual podría estar afectando la prevalencia de este subtipo.
El conocer el subtipo del HCV para el manejo clínico de los pacientes está cobrando importancia con el advenimiento de fármacos antivirales de acción directa ya que, por ejemplo, los pacientes infectados con el subtipo 1a tienden a mejorar de forma más lenta que los infectados con el subtipo 1b 14,28. Se desconoce si esta variación podría asociarse con una mayor resistencia a los nuevos tratamientos 29. La asignación de genotipos y subtipos del virus es también importante para diversos propósitos de investigación, incluyendo estudios filogenéticos, epidemiológicos y de la historia natural de la infección.
Por otra parte, se desconoce la diversidad de sub-tipos del HCV en muchos países latinoamericanos. Así como ocurre en Venezuela, en otros países de la región podrían presentarse subtipos poco frecuentes del HCV. Es por ello que se recomienda que en cada país se evalúe la posibilidad de estudiar la secuenciación de las variantes circulantes, bien sea con fines epidemiológicos o de rutina clínica, usando, por ejemplo, la secuenciación en la región NS5B, para tener una visión de la variedad de sub-tipos que circulan, ya que esto podría tener implicaciones de adecuación del tratamiento antiviral.














