La región HLA (Human Leukocyte Antigen, HLA) del complejo mayor de histocompatibilidad en humanos comprende aproximadamente 4 Mb de ADN, y en ella se codifican alrededor de 200 genes. Se encuentra localizada en el brazo corto del cromosoma 6, región 6p21.3. Esta región se ha subdividido en tres con base en las proteínas codificadas: la HLA de clase I, en la que se codifican las moléculas clásicas HLA-A, HLA-B y HLA-C; la HLA de clase II, en la que se codifican las moléculas clásicas HLA-DR, HLA-DQ y HLADP, y la HLA de clase III, en la cual se codifican otras proteínas involucradas en la inmunidad 1.
La diabetes mellitus de tipo 1 es una enfermedad compleja causada por la destrucción autoinmunitaria de las células β de los islotes pancreáticos. Esta destrucción se ha descrito como un proceso crónico que, generalmente, se inicia en la infancia o la adolescencia, el cual genera deficiencia en la producción y la liberación de insulina, así como pérdida de la regulación del metabolismo de la glucosa 2.
Como sucede en la mayoría de las enfermedades autoinmunitarias, las variaciones en la región del HLA representan el mayor riesgo genético para el desencadenamiento de la diabetes mellitus de tipo 1. De hecho, se ha descrito que esta región aporta cerca del 50 % del riesgo genético de desarrollarla 3.
En la población caucásica, dicho riesgo es conferido por las moléculas de HLA de clase II, especialmente los alelos DRB1*03 y DRB1*04 cuando se encuentran en haplotipos con los alelos DQB1*02:01 y DQB1*03:02, respectivamente 4. Asimismo, se ha observado que ciertos alelos HLA de clase I confieren el riesgo de ser afectado por la enfermedad, siendo el alelo HLA-A*24:02 el de mayor asociación reportada 5.
También, se ha observado que algunos alelos, como el HLA-DQB1*06:02 y el HLA-A*03:01, confieren protección frente al riesgo de padecer esta enfermedad. Además, se ha observado que los alelos HLA-I, en conjunto con los HLA-II, pueden conferir un alto grado de protección. Por ejemplo, se ha determinado que el aleloA*32:01y e lDRB1*11:01 en combinación con el DQB1*03:01, así como el A*11:01 y el DRB1*15:01 en combinación con el DQB1*06:02 o con el DRB1*07:01 y el DQB1*02, configuran haplotipos que confieren protección frente al riesgo de desarrollar diabetes mellitus de tipo 1 6,7.
En Antioquia, Colombia, el único reporte sobre la asociación entre el desarrollo de esta enfermedad y la región HLA, ha sido el de Montoya, et al., quienes llevaron a cabo un estudio de casos y controles en 1996 para establecer la posible asociación con la enfermedad de los alelos clásicos de los HLA de clases I y II 8. En este estudio no se encontró asociación estadísticamente significativa entre los alelos clásicos de los HLA de clase I y la enfermedad, pero sí entre el alelo HLA-DRB1*03:01 y un riesgo cuatro veces mayor de desarrollarla.
Se sabe que los genes del complejo mayor de histocompatibilidad se heredan como bloques de haplotipos, lo cual se traduce en un gran desequilibrio de ligamiento en la región. Este hecho favorece los estudios basados en metodologías como la de los polimorfismos representativos de un solo nucleótido (tag Single Nucleotide Polymorphism, tag SNP), pues con ellas no se requiere caracterizar todas las variantes presentes en la región y es posible obtener información acerca de la mayor parte de ellas e, incluso, asignar los alelos clásicos de los HLA presentes en una población 9.
En su estudio, de Bakker, et al., genotipificaron más de 7.500 SNP dentro de la región HLA de clase I y de clase II en cuatro poblaciones, africana (YRI), europea (CEU), china (CHB) y japonesa (JPT)), y determinaron la variación dentro de esta región genética. En ese estudio se identificaron los tag SNP informativos para la captura de variantes en la región HLA en cada una de estas poblaciones 9, con lo cual se pudieron capturar los alelos clásicos del HLA.
Esta metodología ofrece una alternativa atractiva cuando no se requiere una exactitud del 100 %, lo que permite analizar la asociación entre la región del HLA y las enfermedades autoinmunitarias y, además, llevar a cabo estudios epidemiológicos en los cuales el tipo de HLA puede ser la causa potencial de una enfermedad. Además, es una alternativa económica para predecir el riesgo de recurrencia en los familiares de pacientes con enfermedades como la diabetes mellitus tipo 1 10,11.
El propósito de este estudio fue validar los tag SNP de los alelos HLA clásicos reportados para población europea 9 y evaluar su asociación en una muestra de 200 familias antioqueñas nucleares con integrantes afectados por la diabetes mellitus de tipo 1.
Materiales y métodos
Muestras
Se contó con una muestra de 200 familias antioqueñas conformadas por 192 tríos familiares (los dos padres y un hijo con diabetes mellitus de tipo 1), y ocho familias con dos hijos afectados. Los pacientes fueron seleccionados en el Servicio de Endocrinología Pediátrica de la Universidad de Antioquia, el Hospital Universitario San Vicente Fundación, la Clínica UPB-Medellín, la IPS Universitaria-Universidad de Antioquia y el Instituto Antioqueño de Diabetes, Metabolismo y Nutrición.
Los criterios de inclusión fueron los siguientes: 1) tener un diagnóstico clínico acorde con los criterios de la American Diabetes Association (ADA) y confirmado por el laboratorio 12; 2) haber sido diagnosticado antes de los 15 años de edad; 3) contar con ambos padres vivos, y 4) tener ancestro ‘paisa’ según la información de la propia familia en la encuesta que se hizo para recopilar los datos demográficos y sobre la procedencia de los bisabuelos del paciente. Se asumió que si al menos seis de los ocho bisabuelos eran de ascendencia paisa, el caso podría ingresar al estudio.
Elección y genotipificación de los tag SNP
Se genotipificaron 13 marcadores dentro de la región HLA, así: rs2844821, rs762324, rs9380122, rs1611430, rs2523933, rs29243, rs2844535, rs3094117, rs3819294, rs1265100, rs7745906, rs2524074, para la región HLA de clase I, y rs2854275 para la región HLA de clase II. Estos marcadores fueron propuestos por de Bakker, et al., como tag SNP para alelos clásicos de HLA en la población europea asociados con la protección o el riesgo de desarrollar diabetes mellitus de tipo 1 9.
La genotipificación se hizo mediante el ARMSPCR (Amplification Refractory Mutation System- Polymerase Chain Reaction), o mediante la PCR-RFLP (PCR-Restriction Fragment Length Polymorphism). La elección de los iniciadores se hizo con la aplicación diseñada por Ye, et al.13, y la de las enzimas de restricción, con el programa NEBcutter™, versión 2.0. (New England Biolabs, Inc.) diseñado por Vincze, et al. 14. Estos dos métodos se basan en las PCR, las cuales se realizaron en un termociclador Dyad Dual-Bay™ (BioRad). El cuadro suplementario1 presenta la secuencia de oligonucleótidos y las enzimas de restricción usadas para el proceso de genotipificación.
Análisis por simulación computacional y elección de los tag SNP para la población antioqueña
Se tomaron los datos de 60 individuos pertenecientes a la población de colombianos que viven en Medellín participantes en la fase 3 del proyecto de mil genomas (1000 Genomes Project) iniciada en mayo de 2013 15. Se contaba con la información de los alelos de estos individuos para los SNP mencionados, así como de los alelos HLA clásicos 16. El objetivo era validar que estos tag SNP estuvieran en desequilibrio de ligamiento con los alelos clásicos de HLA que se querían analizar en esta población. La información sobre la diversidad de HLA para esta población, se tomó de los ensayos de tipificación realizados por Gourraud, et al., en el 2013 16, cuyos datos están disponibles en la página del 1000 Genomes Project (http://www.internationalgenome.org/). Los cálculos y gráficos del desequilibrio de ligamiento se hicieron con el programa Haploview™, versión 4.2 (Broad Institute of MIT and Harvard) 17.
La búsqueda de los SNP en acentuado desequilibrio de ligamento con los alelos clásicos HLA, se hizo en la población colombiana registrada en la base de datos del 1000 Genomes Project. Para ello, se tomaron todos los SNP reportados en dicha base de datos ubicados dentro de la región HLA de clases I y II, clásica y extendida. En total, se tomaron 133.289 SNP que abarcan una región de 6,86 Mb.
Dado el gran número de SNP, primero se calculó el desequilibrio de ligamento de los SNP con los alelos clásicos reportados para 60 individuos de esta población usando el paquete Plink™, versión 1.07 (http://pngu.mgh.harvard.edu/purcell/plink/) 18, y así, se hallaron los marcadores con un desequilibrio de ligamiento moderado (R2>0,5). Posteriormente, usando la función tagger del programa Haploview™, versión 4.2 17, se obtuvieron los tag SNP (R2>0,8) más apropiados para la población antioqueña.
Análisis de asociación e interacción
El análisis de asociación entre los marcadores y la diabetes mellitus de tipo 1, se hizo usando la prueba no paramétrica de desequilibrio de transmisión (Transmission Disequilibrium Test, TDT), la cual mide las transmisiones desde los padres heterocigotos hacia los hijos afectados 19, con el programa Unphased™, versión 3.1.7(S. Karger AG, Basel) 20. La interacción se analizó con el paquete Trio logic regression implementado por el R Project™, versión 3.2.0 (The R Foundation) 21, entre los 13 marcadores de HLA y los siguientes nueve marcadores en otros genes asociados con la diabetes mellitus de tipo 1 genotipificados en un estudio previo 22: rs2476601, rs1217418 y rs2488457 para el gen PTPN22; rs1990760, rs3747517 y rs10930046 para el gen IFIH1, y los SNP rs231775, rs3087243 y rs231779 para el gen CTLA4.
El valor corregido de p se asumió como 0,0023 para mantener una significación alfa de 0,05, y se usó tanto para los análisis de asociación como para los de interacción.
Resultados
En el cuadro 1 se presentan los valores de desequilibrio de ligamiento entre los tag SNP 9 y los alelos clásicos en la población del 1000 Genomes Project. Ninguno de los SNP (solos o en haplotipos) obtuvo un R2>0,8. Por lo tanto, no se puede decir que estos marcadores sean tags apropiados para inferir los alelos clásicos del HLA en esta población. Por su parte, el SNP analizado para la región de HLA-II (rs2854275) registró un acentuado desequilibrio de ligamiento (R2=0,826) con el alelo clásico HLA-DRB1*03:01 para esta población.
Cuadro 1 Medidas del desequilibrio de ligamiento D’ y r2 entre los tag SNP reportados y los alelos clásicos de HLA en población colombiana en la base de datos del 1000 Genomes Project
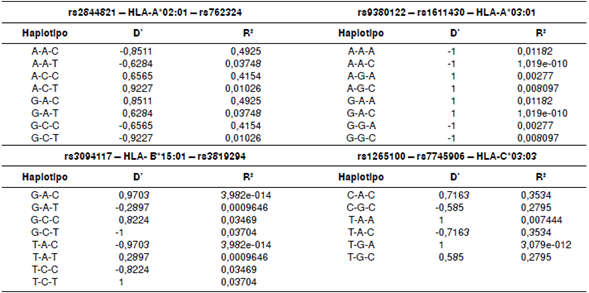
Los haplotipos se componen de dos tag SNP reportados para la población europea (CEU) y el alelo HLA clásico con el cual fueron asociados en dicha población. En la posición del alelo clásico se colocó A/C para la presencia o ausencia del alelo analizado. Se presentan los resultados para la población de estudio con base en cuatro alelos HLA clásicos y sus tag SNP según de Bakker, et al. (9).
Los tag SNP basados en la CLM del 1000 Genomes Project
Dados los resultados que se presentan en el cuadro 1, se hizo necesario identificar los tag SNP propios de la población en estudio. Para ello, se usó la información de los SNP presentes en la región HLA de clase I y II, clásica y extendida.
Para la región clásica y la extendida de los HLA de clase I, se analizaron 98.743 marcadores comprendidos entre la posición 25.730.000 y la 31.480.000, la cual abarca una región de 5,75 Mb. Para la región clásica y la extendida de los HLA de clase II, se analizaron 34.546 SNP comprendidos entre las posiciones 32.260.000 y 33.370.000, las cuales abarcan una región de 1,11 Mb.
Tres (HLA-DRB1*04:01, HLA-DRB1*04:02, HLADRB1* 04:05) de los 19 alelos clásicos de interés por su asociación con la diabetes mellitus de tipo 1, no se pudieron analizar por su baja frecuencia en la población objeto. Para otros cuatro alelos (HLA-A*02:01, HLA-B*15:01, HLA-C*03:04 y HLADQB* 06:02) no se encontraron SNP que tuvieran un acentuado desequilibrio de ligamento (R2>0,8). Para los restantes doce alelos HLA clásicos, se pudieron obtener tags con un r2>0,8 para esta población (cuadro 2).
Cuadro 2 Los polimorfismos de un solo nucleótido (SNP) en desequilibrio de ligamiento con alelos clásicos de HLA en población colombiana
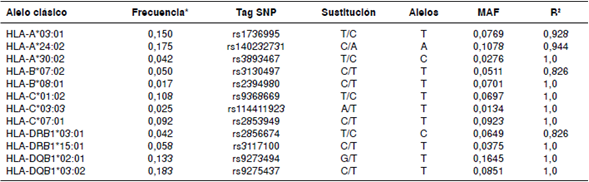
* Frecuencia de alelos clásicos en la población de colombianos de Medellín; MAF: frecuencia del alelo menor; R²: coeficiente de determinación Los datos que dieron origen a este cuadro provienen de www.1000genomes.org, particularmente los de la población de colombianos de Medellín.
La figura 1 muestra el desequilibrio de ligamiento entre el alelo HLA-B*07:02 y doce marcadores (SNP) dentro de la región de HLA-I, los cuales tienen un perfecto desequilibrio de ligamiento entre sí, es decir, cualquiera de estos marcadores serviría como tag para este alelo HLA clásico en la población de estudio.
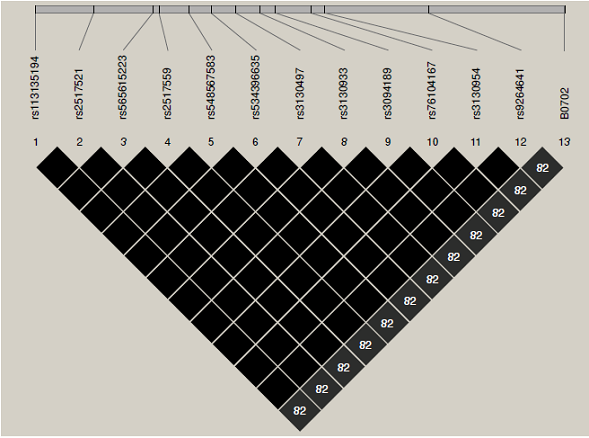
Figura 1 Desequilibrio de ligamiento entre el alelo HLA-B*07:02 y doce marcadores en la región HLA de clase I. Valores expresados en R2. El intervalo entre los polimorfismos de un solo nucleótido incluye una región física de 248,322 pb. El alelo clásico se localiza en posición centromérica (identificado con la marca # 13).
Análisis de asociación
En el cuadro 3 se presentan los resultados de asociación de los 13 SNP probados en las 200 familias nucleares antioqueñas. Se observó que los SNPrs29243 (p=2,634e-5; odds ratio (OR)=2,85 [1,67-4,85]) y rs2854275 (p=1,27e-7; OR=2,71 [1,82-4,02]) contribuyen al riesgo genético de padecer la enfermedad en la población analizada.
Cuadro 3 Asociación alélica de los tag SNP con la diabetes mellitus de tipo 1 establecida mediante la prueba TDT
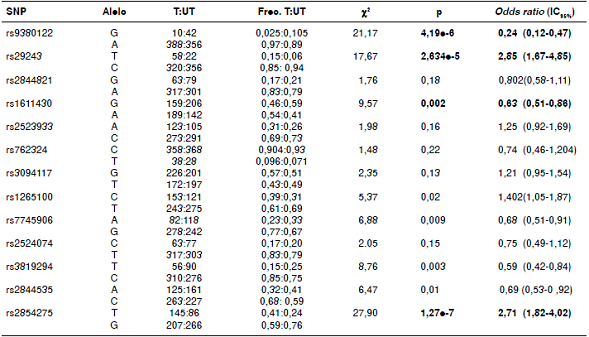
SNP: Single nucleotide polymorphism; T:U: transmisiones Vs. no transmisiones; Frec. T:UT: frecuencias de transmisiones Vs. no transmisiones. Las negrillas representan los resultados significativos.
Los marcadores rs9380122 (p=4,19e-6; OR=0,24 [0,12-0,47]) y rs1611430 (p=0,002; OR=0,63 [0,51-0,86]) están asociados con la protección frente al riesgo de desarrollar diabetes mellitus de tipo 1. Los demás marcadores no superaron el nivel de confianza del 95 % después de la corrección para múltiples comparaciones.
El análisis de interacción entre los marcadores analizados para el HLA no evidenció interacciones estadísticamente significativas, pues ninguna de ellas superó el valor de p corregido (0,0023) para múltiples comparaciones.
Discusión
Las causas del desequilibrio de ligamiento son diversas. Una de ellas es la cercanía física entre los loci, por lo cual la probabilidad de que ocurra un evento de recombinación entre ambos disminuye. Otra fuente de este desequilibrio es la mezcla de dos poblaciones que pueden estar inicialmente en equilibrio de Hardy-Weinberg y que poseen diferentes frecuencias alélicas 23.
En este estudio se estimó el desequilibrio de ligamiento entre los tag SNP analizados (reportados por de Bakker, et al.) y los alelos clásicos de HLA reportados para la población CLM en el 1000 Genomes Project para validar si estos SNP eran tags apropiados de los alelos clásicos en la población de estudio.
Teniendo en cuenta la ascendencia predominantemente europea de los antioqueños 24,25, se eligieron los marcadores reportados por de Bakker, et al., en el 2006 para población europea residente entonces en los Estados Unidos (CEU) 9.
El análisis reveló diferencias en el desequilibrio de ligamiento en la región de HLA entre la población europea y la antioqueña. Tales diferencias pueden estar relacionadas con el efecto de mezcla y con el crecimiento poblacional de esta última en relativo aislamiento. Además, la ascendencia amerindia y africana de los antioqueños también contribuye a diferenciar los bloques de haplotipos definidos por eventos de recombinación 23,26.
Con el propósito de identificar los tag SNP de los alelos HLA clásicos para esta población de Antioquia, se usó la información disponible en la base de datos del 1000 Genomes Project. Se logró la identificación de los tag SNP de 12 de 19 alelos HLA clásicos. A diferencia de lo observado por de Bakker, et al., en población europea, en la cual se requerían dos SNP para alcanzar un valor de R2 por encima de 0,8, se observó que en la población del presente estudio un SNP era suficiente para alcanzar dicho valor.
Este hallazgo puede explicarse por el reciente cuello de botella experimentado por la población antioqueña. Con la colonización europea se diezmó la población amerindia y llegaron esclavos africanos. Entre estos tres grupos étnicos ocurrieron cruces que originaron la nueva población 27. Durante las migraciones, no todos los cromosomas de la población parental han de quedar representados en la población naciente. Se acepta que ha existido un mayor número de generaciones desde el origen de la población europea que desde el origen de la población antioqueña, lo cual implica que ha ocurrido un mayor número de meiosis en aquella población que en la antioqueña. A su vez, este fenómeno aumenta la probabilidad de que hayan ocurrido más eventos de recombinación en la región HLA de los europeos que en la de los antioqueños, y explicaría por qué el desequilibrio de ligamiento es mayor en estos, además con alelos diferentes 28.
Llama la atención que varios marcadores se encuentran en perfecto desequilibrio de ligamiento (R2=1,0) con los alelos clásicos HLA y entre ellos (figura 1). Esta observación concuerda con el fuerte desequilibrio de ligamiento que se ha descrito previamente para la región de HLA y con el bajo número de generaciones después de ocurrida la mezcla.
El análisis de asociación entre los marcadores analizados y la diabetes mellitus tipo de 1 evidenció que estos marcadores influyen en el riesgo genético para desarrollar la enfermedad. Cuatro de los 13 marcadores analizados mostraron asociación estadísticamente significativa al aplicar la prueba no paramétrica de desequilibrio de transmisión (Transmission Disequilibrium Test, TDT). De los doce marcadores de HLA de clase I analizados, rs29243(T) se asoció con un aumento de más de dos veces en el riesgo de desarrollar la enfermedad, en tanto que los marcadores rs9380122(G) y rs1611430(G) se asociaron con una disminución importante del riesgo de padecerla.
También, se observó que el haplotipo G-G de los marcadores rs9380122 y rs1611430 es un elemento claramente protector frente al riesgo de desarrollar la enfermedad (no se presentan los datos). Estos marcadores no se encuentran dentro de un gen, pero pueden estar en desequilibrio con otros SNP dentro de los genes del HLA-I y, probablemente, con un alelo HLA clásico.
Según de Bakker, et al., estos tres SNP serían marcas apropiadas para los alelos clásicos HLA-A*30:02 y HLA-A*03:01. Los SNP rs9380122 y rs1611430 aparecen como tags de HLA-A*03:01, en tanto que el SNP rs29243 aparece como tag del alelo clásico HLA-A*30:02 9. Según nuestros análisis, esta inferencia habría sido imprecisa para nuestra población.
En el estudio de Montoya, et al., en 1996, no se encontró asociación estadísticamente significativa entre los alelos clásicos de HLA de clase I y la diabetes mellitus de tipo 1 8. Sin embargo, estos autores sí encontraron una asociación del alelo HLA-DRB1*03:01 con un riesgo cuatro veces mayor de desarrollar la enfermedad 28.
El presente análisis de asociación no permite concluir cuáles alelos HLA clásicos estarían implicados en la asociación identificada. La imposibilidad de inferirlos se debió al bajo desequilibrio de ligamiento (R2<0,8) de los tag SNP usados como referencia 9, y los alelos HLA clásicos reportados para la población CLM 16. No obstante, es evidente la fuerte asociación entre la región HLA y la predisposición o la protección frente a la diabetes mellitus de tipo 1.
En la región HLA de clase II solo se analizó un marcador (rs2854275), el cual está en un alto desequilibrio de ligamiento (R2=0,83) con el alelo clásico HLA-DRB1*03:01 según los datos del 1000 Genomes Project. En la prueba TDT este marcador evidenció una asociación estadísticamente significativa con el riesgo de desarrollar diabetes mellitus de tipo 1, resultado que concuerda con lo reportado en otros estudios, en los cuales este alelo clásico se ha asociado claramente con el riesgo de desarrollar la enfermedad en varios grupos poblacionales, incluidos algunos de Colombia y Latinoamérica 7,8,29-31.
No se hallaron interacciones estadísticamente significativas entre los marcadores de HLA analizados. Se ha reportado que los alelos HLA de clase I influyen en el riesgo de padecer diabetes mellitus de tipo 1 cuando se expresan conjuntamente con alelos HLA de clase II. Es el caso del HLA-A*24, que aumenta en un 2,04 el riesgo de padecer la enfermedad cuando se expresa conjuntamente con el haplotipo DQA1*03-DQB1*03:02 (DQ8) 32. Tampoco se encontró evidencia de interacción entre los marcadores de HLA y los de otros genes (PTPN22, IFIH1 y CTLA4) asociados con esta enfermedad 22.
En conclusión, el desequilibrio de ligamiento en la región HLA para la población antioqueña (CLM) analizada no se comportó de manera similar a lo reportado para la población europea (CEU). Por lo tanto, es importante tener en cuenta los tag SNP específicos aquí reportados para la población de estudio. Asimismo, dicho comportamiento debería ser objeto de otros estudios que incluyan el análisis de HLA en población latinoamericana. En el presente estudio, además, se confirmó que la región HLA contribuye a la predisposición o protección frente a la diabetes mellitus de tipo 1 en la población analizada.
El equipo de investigación está actualmente en proceso de evaluar los tag SNP aquí reportados para los alelos HLA clásicos en todo el conjunto de familias con diabetes mellitus de tipo 1.















