El cáncer primario de hígado es la segunda causa más común de muerte por cáncer en el mundo, con alrededor de 746.000 muertes en el 2012 (9,1 % del total de muertes; tasa de mortalidad: 9,5/100.000 habitantes), y es el quinto cáncer más común en hombres y el noveno en mujeres 1,2. Entre el 75 y el 90 % del total de los casos corresponden a carcinoma hepatocelular 3. Los principales factores de riesgo de este cáncer son la infección crónica por el virus de la hepatitis B (HBV) y el virus de la hepatitis C (HCV), el consumo crónico de alcohol, la exposición a aflatoxinas en la dieta y la esteatohepatitis no alcohólica 2.
La International Agency for Research on Cancer (IARC) estima que la incidencia de cáncer primario de hígado en Colombia (2012) fue de 3,2 por 100.000 habitantes en la población masculina y de 2,8 por 100.000 en la femenina, y la tasa de mortalidad, de 4,3 y 3,7 por 100.000 habitantes en la población masculina y la femenina, respectivamente 1,2. Dicha estimación se basó en los registros poblaciones de cáncer de Cali, Bucaramanga, Manizales y Pasto, entre 2003 y 2007, datos que representan el 8 % de la población total del país y no incluyen las dos ciudades más importantes, Bogotá y Medellín, en términos de población y de centros hospitalarios de máximo nivel de atención. La incongruencia entre los datos de incidencia y de mortalidad en Colombia, indica que hay problemas de calidad de los datos, subregistro de casos y errores en la clasificación de las muertes asociadas con tumores hepáticos malignos debido a la inclusión de casos de metástasis en hígado.
En el periodo de 2010 a 2014, el Observatorio Nacional de Salud del Instituto Nacional de Salud estimó que el 6 % de las 155.542 muertes por cáncer en Colombia se podían atribuir a tumores malignos de hígado: 53,5 % en mujeres y el 46,5 % en hombres 4. Además, según el Departamento Administrativo Nacional de Estadística (DANE), en este periodo, la causa de la muerte en 1.726 individuos fue el tumor maligno de hígado, en tanto que las “enfermedades del hígado” produjeron 2.465 muertes 5.
La información disponible en Colombia sobre los factores de riesgo del carcinoma hepatocelular es escasa. En un estudio retrospectivo realizado en laboratorios de patología de referencia de Bogotá, Medellín y Cali, se analizaron 178 casos de este carcinoma diagnosticados entre el 2000 y el 2007: 64 % de ellos diagnosticados en hombres, con una edad media de 62 años; los biomarcadores de infección por HBV se analizaron en el 58,1 % (25/43), la infección por HCV, en el 37 % (10/27), y la exposición a la aflatoxina, en el 10,5 % (4/38) de los casos analizados de carcinoma hepatocelular. En este estudio, no se registró la información sobre el consumo de alcohol de los pacientes 6.
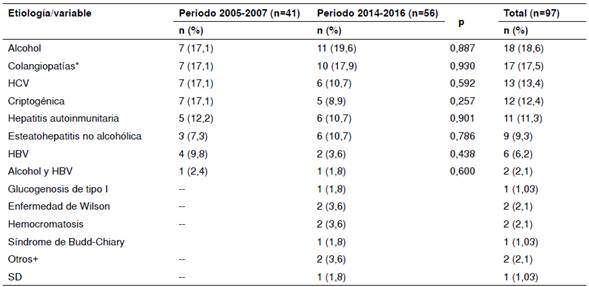
Entre octubre de 2005 y noviembre de 2007, se hizo un estudio prospectivo en la Unidad de Hepatología del Hospital Pablo Tobón Uribe de Medellín, uno de los principales centros de trasplante hepático del país. En el periodo de estudio, se atendieron 131 pacientes con diagnóstico de enfermedad hepática terminal (cirrosis y carcinoma hepatocelular) y se estableció el consumo de alcohol como el principal factor de riesgo (37,4 %), seguido de la infección crónica por HBV (13 %), las enfermedades auto inmunitarias (9,9 %) y la esteatohepatitis no alcohólica (7,6 %). El 10,7 % de los casos se catalogaron como de etiología criptogénica 7. El consumo de alcohol, como el más importante factor de riesgo, corresponde a un patrón epidemiológico que no se esperaba, dadas las regiones de alta prevalencia de hepatitis B en Colombia.
Los hallazgos de este estudio contrastaron con lo descrito por Prieto, et al., en pacientes con diagnóstico de cirrosis hepática atendidos en dos centros de hepatología de Bogotá, entre el 2010 y el 2014. La principal causa etiológica en el grupo de estudio en Bogotá, fue la esteatohepatitis no alcohólica (25,5 %), en tanto que el consumo de alcohol se estableció en el 14,8 % de los casos de cirrosis 8.
El riesgo de enfermedades hepáticas asociado con el consumo crónico de alcohol parece estar relacionado con los polimorfismos en genes que codifican enzimas del metabolismo del alcohol 9-15. El hígado es el órgano encargado del metabolismo del 90 % del alcohol una vez este es absorbido en el tubo digestivo. Cuatro enzimas intervienen en el metabolismo hepático del alcohol: la alcohol deshidrogenasa (ADH1) y la acetaldehído deshidrogenasa (ALDH2), las cuales metabolizan el 80 % del alcohol, la citocromo p450 2E1 (CYP2E1), que hace parte del sistema inducible microsómico de oxidación del alcohol (Microsomal Etanol Oxidizing System, MEOS) y se encarga de la oxidación del 10 % del etanol, y la catalasa, que metaboliza el 2 % del alcohol ingerido 16,17.
El gen ADH1B codifica la subunidad ß de la enzima ADH1 18 y el alelo ADH1B*1 codifica el aminoácido arginina en las posiciones 48 y 370 (Arg48, Arg370), en tanto que el alelo ADH1B*2 codifica una histidina en la posición 48 (His48, Arg48), la cual da lugar a una enzima con una actividad enzimática 100 veces mayor que el alelo ADH1B*1 19. El alelo ADH1B*3 codifica el aminoácido cisteína en la posición 370 (Arg48, Cys370), en lugar de la histidina, que codifica el alelo ADH1B*1.
El gen ADH1C codifica la subunidad gamma de la ADH1 18; el alelo ADH1C*1 codifica los aminoácidos arginina e isoleucina en las posiciones 272 y 350 (Arg48, Ile370), los cuales son reemplazados por el ácido glutámico y la valina (Gln48, Val370) en el alelo ADH1C*2, que presenta una actividad enzimática 2,5 veces menor que el ADH1C*1 20. En el caso del gen CYP2E1, la transición C por T en la región reguladora (posición -1053) se asocia con un incremento de la actividad de transcripción del alelo CYP2E1*5B con respecto a la del alelo CYP2E1*5A 21.
En estudios poblaciones, se ha demostrado que las variantes genéticas con mayor actividad metabólica o de transcripción se asocian con un riesgo mayor de alcoholismo y de desarrollo de la enfermedad hepática alcohólica en la población caucásica 12,13, la india 11 y la mexicana 14, aunque los resultados han sido contradictorios en algunas poblaciones 9,10.
Recientemente, se publicó el primer estudio sobre la adicción al alcohol en la población colombiana. En él se describe el predominio de los alelos ADH1B*1, ADH1C*1, CYP2E1*5A y ALDH2*1 (Gln504) en adictos atendidos en centros de rehabilitación de Bogotá 24. Sin embargo, no se dispone de información sobre la frecuencia genotípica en pacientes con enfermedad hepática. En este contexto, el objetivo de este estudio fue identificar y caracterizar las variantes alélicas de los genes ADH1B, ADH1C y CYP2E1 por primera vez en pacientes colombianos con diagnóstico de cirrosis y carcinoma hepatocelular.
Materiales y métodos
Población de estudio
Se incluyeron pacientes con diagnóstico de cirrosis y carcinoma hepatocelular atendidos en la Unidad de Hepatología del Hospital Pablo Tobón Uribe durante los periodos de febrero de 2005 a febrero de 2007 y de junio de 2014 a agosto de 2016. Los pacientes del primer periodo habían sido sometidos a trasplante de hígado; las muestras de tejido hepático se obtuvieron en el marco del estudio prospectivo 7.
En el período de 2014 a 2016, el grupo de investigación hizo un estudio de casos y controles para estimar el peso del alcohol como factor de riesgo de hepatopatías. El tamaño de la muestra se calculó en 80 casos y 80 controles. Por cada caso con diagnóstico de cirrosis y carcinoma hepatocelular, se incluyeron dos controles; el primer grupo de control incluía a pacientes sometidos a resecciones parciales de hígado en el Hospital para el estudio histopatológico y serológico, con el fin de descartar enfermedad hepática. El segundo grupo de control incluía pacientes atendidos en la unidad de urgencias del Hospital por enfermedades no relacionadas con el hígado, ni con trauma ni consumo de alcohol. En el marco de este proyecto de casos y controles, se incluyeron 22 pacientes sometidos a trasplante durante esta etapa y a 24 pacientes seleccionados aleatoriamente de la base de datos de la Unidad de Hepatología; estos últimos fueron contactados telefónicamente e invitados a participar en el estudio.
Todos los pacientes y controles fueron entrevistados por investigadores del proyecto previamente capacitados. Se utilizó una encuesta sobre la historia familiar y de exposición a factores de riesgo, como el consumo de alcohol y el tabaquismo, y se utilizó el Cuestionario de Identificación de los Transtornos debidos al Consumo de Alcohol (Alcohol Use Disorders Identification Test, AUDIT) de la Organización Mundial de la Salud (OMS) y la encuesta de seguimiento del consumo de alcohol a lo largo de la vida (life drinking history), gentilmente donada por el doctor Harvey A. Skinner, profesor de la Universidad de Toronto 25-27.
El diagnóstico de cirrosis hepática de los pacientes atendidos en los dos periodos de estudio, se estableció con base en criterios clínicos, como hipertrofia de parótidas, ictericia, nevos en araña, ginecomastia y eritema palmar, o de complicaciones asociadas, como encefalopatía hepática, ascitis, sangrado digestivo por várices esofágicas, coagulopatía, síndrome hepatorrenal y peritonitis bacteriana espontánea. Todos los casos se confirmaron mediante imágenes diagnósticas y, en algunos casos, por histopatología. El diagnóstico de carcinoma hepatocelular se estableció siguiendo los criterios de la European Association for the Study of the Liver (EASL) y de la American Association for the Study of Liver Diseases (AASLD).
A partir de la historia clínica y de la encuesta, se obtuvo información sobre los datos demográficos, el diagnóstico y la etiología de la enfermedad de los pacientes y los controles. A todos los participantes se les explicó el estudio y se solicitó su consentimiento informado. El estudio fue aprobado por el Comité de Bioética del Instituto de Investigaciones Médicas de la Universidad de Antioquia y por el del Hospital Pablo Tobón Uribe.
Muestras
Las muestras de los explantes hepáticos se conservaron en tubos estériles en el Hospital a 4 °C durante un periodo máximo de 24 horas, hasta el momento de transportarlas al Laboratorio del grupo de Gastrohepatología de la Universidad de Antioquia, donde se almacenaron a -70 °C. Las muestras de sangre se recogieron en tubos con EDTA, se centrifugaron y el plasma se almacenó a -20 °C en el Hospital para, luego, transportarlas en hielo seco a este mismo laboratorio y almacenarlas a -70 °C.
Extracción del ADN
A partir de las muestras de tejido hepático, se obtuvieron fragmentos de, aproximadamente, 500 mg para la extracción del ADN total, empleando el método TRIzol-cloroformo (Ambion™, Thermo Fisher Scientific). A partir de la muestra de sangre periférica, se extrajo el ADN utilizando un estuche comercial (QIAamp DNA BloodMini Kit™ de Qiagen) según las instrucciones del fabricante. El ADN se cuantificó mediante espectrofotometría (NanoDrop 2000™, Thermo Fisher Scientific).Una vez determinada la concentración de ADN, se prepararon alícuotas en una concentración de 40 ng/μl para los experimentos.
Amplificación de ADH1B, ADH1C y CYP2E1
Se estandarizaron las condiciones para la amplificación de los genes ADH1B, ADH1C y CYP2E1, adaptando los protocolos descritos por Khan, et al. 11. Se amplificó un fragmento de 107 pb del exón 3 del gen ADH1B mediante PCR de touchdown (TD-PCR). La reacción se llevó a cabo en un volumen final de 25 μl con solución tampón 1X, MgCl2 2,5 M, dNTP 0,2 mM, 1 U de BiolasaADN polimerasa™ (Bioline, USA), 80 ηg de ADN y 0,2 μM de los oligonucleótidos sentido P1 (5-AATCTTTTCTGAATCTGAACAG-3) y antisentido P2(5-GAAGGGGGGTCACCAGGTTGC-3) 9. Las condiciones de los ciclos fueron las siguientes: 5 minutos a 95 °C, 15 ciclos a 95 °C durante 30 segundos, a 65 °C durante 45 segundos hasta disminuir a 0,5 °C por ciclo y a 72 °C durante 1 minuto, seguidos de 20 ciclos a 95 °C durante 30 segundos, a 55 °C durante 45 segundos y a 72 °C durante 1 minuto, con una extensión final a 72 °C durante 5 minutos.
En el caso del ADH1C, se amplificó un fragmento de 146 pb del exón 8 mediante PCR convencional, bajo las siguientes condiciones: solución tampón 1X, MgCl2 2,5 mM, dNTP 0,2 mM, 1 U de ADN Taq polimerasa (Thermo Fisher Scientific™) y 80 ηg de ADN y 0,2 μM de los oligonucleótidos sentido P3 (5-GCTTTAAGAGTAAAGAATCTGTCCCC-3) y antisentido P4 (5-AATCTA CCTCTTTCCAGAGC-3) 9 en un volumen final de 25 μl. El protocolo de los ciclos fue el siguiente: un ciclo a 94 °C durante 5 minutos, 30 ciclos a 94 °C durante 30 segundos, a 55,9 °C durante 30 segundos, a 72 °C durante 30 segundos y una extensión final a 72 ºC durante 5 minutos.
El fragmento de 413 pb de la secuencia 5’ del gen CYP2E1, se amplificó mediante PCR convencional en un volumen final de 25 μl de solución tampón 1X, MgCl2 2,5 mM, dNTPs 0,2 mM, 1 U de ADN Taq Polimerase™ (Thermo FisherScientific, USA), 80 ηg de ADN y 0,2 μM de los oligonucleótidos sentido P5 (5-CCAGTCGAGTCTACATTGTCA-3) y antisentido P6 (5-TTCATTCTGTCTTCTAACTGG-3) 11. Los ciclos incluyeron un ciclo a 94 °C durante 5 minutos, 35 a 94 °C durante 30 segundos, a 55,9 °C durante 45 segundos, a 72 °C durante 1 minuto y una extensión final a 72 °C durante 5 minutos.
Los productos de la amplificación se analizaron mediante electroforesis en gel de agarosa al 1,5 % y tinción con bromuro de etidio, y se visualizaron en un transiluminador MultiDoc-It Digital Imaging System™ (UVP). En cada ensayo de PCR, se incluyó como control positivo ADN extraído de la línea celular HepG2 y, como control negativo, agua libre de nucleasas.
Genotipificación mediante RFLP
Para identificar los genotipos de ADH1B, ADH1C y CYP2E1, los productos de amplificación se sometieron a digestión, adaptando los protocolos descritos por Khan, et al., Silva, et al., y Kato, et al. 11,21,28.
Para el ADH1B, la restricción se hizo con la enzima MaeIII (Roche, USA) durante 3 horas a 55 °C en un termociclador C1000 Touch™ (BioRad) y en un volumen final de 20 μl (1X de solución tampón, 0,75 U de enzima y 4 μl del producto de la PCR). La ausencia de digestión del producto de la PCR correspondió al genotipo homocigótico silvestre ADH1B*1/*1, en tanto que la obtención de tres fragmentos (de 96, 60 y 35 pb) correspondió al genotipo heterocigótico ADH1B*1/*2, y la obtención de dos fragmentos de 60 y 35 pb, al genotipo homocigótico mutante ADH1B*2/*2. El producto amplificado presentó un sitio de corte en el extremo 3’ no relacionado con el genotipo.
La restricción del producto de amplificación del ADH1C se hizo con la enzima SspI (Jena Bioscience, Alemania) a 37 °C durante 3 horas, seguido por inactivación a 65 °C durante 20 minutos. El volumen final de la reacción fue de 20 μl (1X de solución tampón, 3 U de enzima y 4 μl del producto de la PCR). La obtención de dos fragmentos de 83 y 63 pb correspondió al genotipo homocigótico ADH1C*1/*1, en tanto que la de tres fragmentos (de 146, 83 y 63 pb) correspondió al genotipo heterocigótico ADH1C*1/*2; la ausencia de corte en el producto de la PCR correspondió al genotipo homocigótico ADH1C*2/*2.
Para el CYP2E1, la restricción se hizo con la enzima RsaI (Jena Bioscience, Alemania); se incubó a 37 °C durante 3 horas y luego se inactivó a 65 °C durante 20 minutos. El volumen final de la reacción fue de 20 μl (1X de solución tampón, 2 U de enzima y 7,0 μl del producto de la PCR). La obtención de dos fragmentos de 352 y 61 pb indicó la presencia del genotipo homocigótico silvestre CYP2E1*C/C (CYP2E1*5A), en tanto que la generación de tres fragmentos de 413, 352 y 61 pb correspondió al genotipo heterocigótico CYP2E1*C/T; la ausencia de digestión del producto de la PCR de 413 pb correspondió al genotipo homocigótico mutante CYP2E1*T/T (CYP2E1*5B).
En todas las reacciones se utilizó el producto de la PCR del ADN de la línea celular HepG2 como control positivo y, como control negativo, los productos amplificados de la línea celular en ausencia de la enzima correspondiente. El producto de restricción de ADH1B y ADH1C se visualizó en gel de poliacrilamida al 15 %, teñido luego del corrido con bromuro de etidio; la restricción del CYP2E1 se visualizó en gel de agarosa al 3 % teñido con bromuro de etidio.
Análisis estadístico
Se utilizó la herramienta en línea SNPStats Lts (29) para calcular las frecuencias alélicas y genotípicas para ADH1B, ADH1C y CYP2E1, y se verificó que se encontraran en equilibrio de Hardy-Weinberg. El análisis incluyó las frecuencias alélicas descritas en poblaciones de otras regiones del mundo. Para esto, se descargaron los genotipos reportados para los polimorfismos de interés en los genes ADH1B (rs1229984), ADH1C (rs698) y CYP2E1 (rs2031920) de la base de datos del 1000 Genomes Project provenientes de población africana (Yoruba, Nigeria), caucásica (Utah, ancestro del norte y occidente de Europa, CEU), asiática (sur de China), peruana, mexicana, y de población de Medellín.
Con el programa EpiDat 3.1 30, se compararon las proporciones de las frecuencias alélicas y genotípicas del grupo de pacientes con condiciones debidas al consumo crónico de alcohol y en el de pacientes con enfermedades hepáticas terminales asociadas con otra causa etiológica, así como en la población de estudio, los grupos de control y poblaciones de diversas regiones del mundo. Se aplicó una corrección de Bonferroni en la comparación de las frecuencias alélicas entre la población de estudio y las otras poblaciones 31.
Además, con el programa SPSS™, versión 21, se analizaron las siguientes variables: consumo de alcohol mensual, consumo excesivo de alcohol, consumo de cigarrillos durante más de seis meses y consumo de más de cinco paquetes de cigarrillos a lo largo de la vida. El consumo excesivo de alcohol se calculó utilizando la fórmula de la guía del doctor Harvey A. Skinner (lifetime drinking history), con las siguientes variables: frecuencia de consumo de alcohol al mes en días, cantidad promedio de bebidas alcohólicas por día y la constante, una bebida de alcohol de 13,6 g (el consumo excesivo de alcohol en hombres corresponde a 60 g o más y, en mujeres, a 40 g o más).
Para el análisis descriptivo de los casos y de los controles, se utilizaron frecuencias absolutas y relativas. La asociación entre las diferentes variables se estableció mediante la prueba U de Mann-Whitney y se consideró estadísticamente significativo un valor de p menor de 0,05.
Resultados
Población de estudio
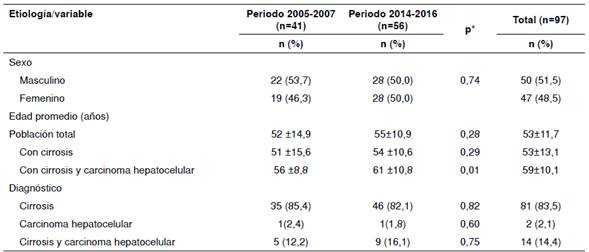
Se analizaron las muestras de tejido hepático o de sangre de 97 pacientes con diagnóstico de cirrosis y carcinoma hepatocelular. El rango de edad de los pacientes fluctuó entre los 22 y los 78 años, con una media de 53 años y una mediana de 56; el 51,5 % de ellos era de sexo masculino. La edad promedio de los controles del grupo 1 fue de 57±10,7 años y, los del grupo 2, de 52±11,3 años. Se observó una diferencia significativa en la edad de los pacientes con diagnóstico de carcinoma hepatocelular entre los periodos de 2005 a 2007 y de 2014 a 2016 (56 años y 61 años, respectivamente) (cuadro 1).
Cuadro 1 Características demográficas de la población de estudio
* Valor de p reportado en la comparación de los periodos
Al comparar los pacientes con condiciones debidas al consumo crónico de alcohol y aquellos con otra causa etiológica, no se encontró diferencia con respecto a la edad, pero sí con respecto al sexo. Como se esperaba, la mayoría de los pacientes con enfermedad hepática por consumo de alcohol eran hombres (94,4 %) (cuadro 2).
Cuadro 2 Características demográficas de los pacientes según la etiología de su condición
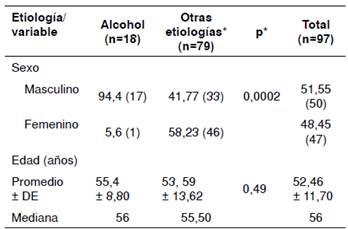
*Incluidos dos pacientes con doble causa etiológica: consumo crónico de alcohol e infección por HBV
Con respecto al diagnóstico, el 83,5% (81/97) de los pacientes presentaba cirrosis, el 14,4 % (14/97), cirrosis y carcinoma hepatocelular, y el 2,1 % (2/97), carcinoma hepatocelular. La etiología de la enfermedad hepática terminal se presenta en el cuadro 3; las más frecuentes fueron el consumo crónico de alcohol (18,6 %), las colangiopatías (17,5 %) y la infección crónica por el HCV (13,4 %).
Genotipificación de ADH1B, ADH1C y CYP2E1
Los genotipos se determinaron mediante PCRRFLP y se establecieron las frecuencias alélicas y genotípicas de los genes ADH1B, ADH1C y CYP2E1 (cuadro 4). Los genotipos más frecuentes en la población de estudio fueron el ADH1B*1/*1, cADH1C*1/*1 y el CYP2E1*C/C, presentes en más del 80 % de las muestras analizadas: ADH1B, 82 %; CYP2E1, 84 %, y ADH1C, 59 %.
Cuadro 4 Frecuencias genotípicas y alélicas de ADH1B, ADH1C y CYP2E1 en la población de estudio
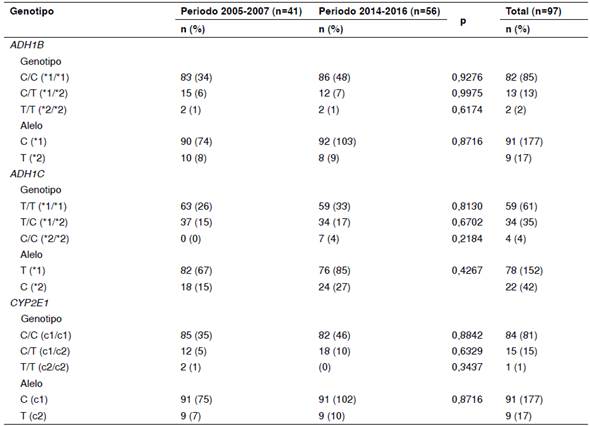
* Valor de p<0,05 reportado en la comparación de cada período
Las frecuencias genotípicas de la población total de estudio (n=97) y de los grupos discriminados según el período de estudio (2005-2007 y 2014-2016) estaban en equilibrio de Hardy-Weinberg (p>0,05), lo que indica que no había habido fuerzas evolutivas, como la selección, actuando sobre ellas.
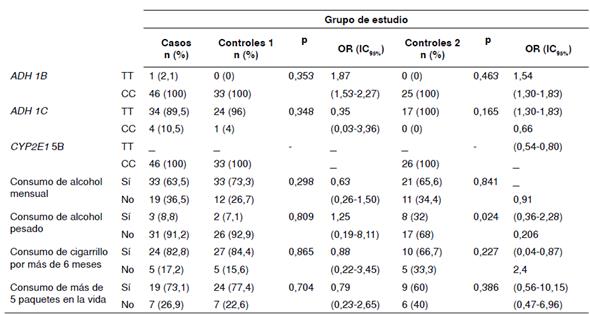
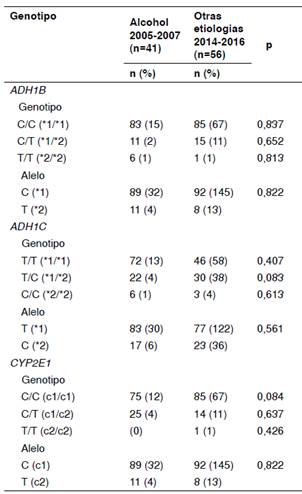
No se encontraron diferencias estadísticamente significativas en la frecuencia de los genotipos y los alelos del grupo de pacientes con condiciones debidas al consumo crónico de alcohol, comparado con otras causas (cuadro 5).
Cuadro 5 Frecuencias genotípicas y alélicas de ADH1B, ADH1C y CYP2E1 en pacientes con condiciones debidas al consumo crónico de alcohol y a otras causas etiológicas
Al comparar las frecuencias genotípicas de la población de estudio y los grupos de control 1 (cuadro 6) y 2 (cuadro 7), se observaron diferencias en las frecuencias alélicas del CYP2E1 en los controles del grupo 1 después de aplicar la corrección de Bonferroni.
Cuadro 6 Frecuencias genotípicas y alélicas de ADH1B, ADH1C y CYP2E1 en pacientes con diagnóstico de cirrosis y carcinoma hepatocelular, y en el grupo de control 1
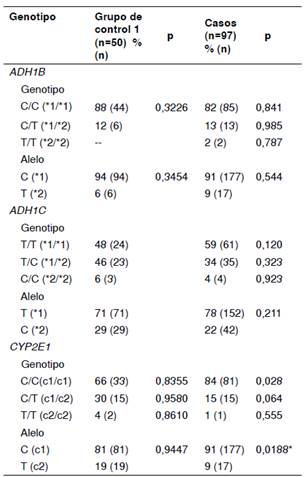
* Corrección de Bonferroni con dos comparaciones, p=0,025
Cuadro 7 Frecuencias genotípicas y alélicas de ADH1B, ADH1C y CYP2E1 en pacientes con diagnóstico de cirrosis y carcinoma hepatocelular, y en el grupo de control 2
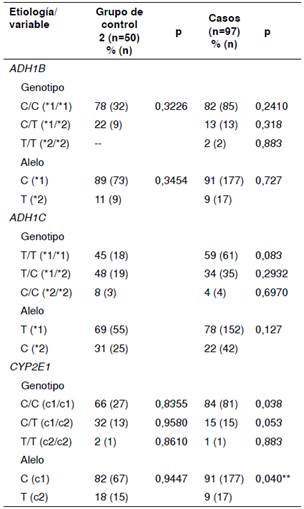
Las frecuencias alélicas de la población de estudio comparadas con las de otras poblaciones, evidenciaron diferencias estadísticamente significativas en el alelo del ADH1B*2: con la población africana (p<0,05), la caucásica (p< 0,05), la asiática (p<0,05) y la peruana (p=0,0026); en el alelo ADH1C*1: con la población africana (p< 0,05), la caucásica (p< 0,05) y la asiática (p= 0,0001), y en el CYP2E1*T: con la población africana (p< 0,05) y la asiática (p=0,0023). Al realizar la corrección de Bonferroni, se conservó la significación estadística de estas comparaciones (figura 1).
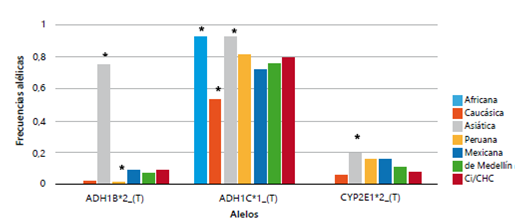
Figura 1 Frecuencias de los alelos de ADH1B, ADH1C y CYP2E1 en pacientes con diagnóstico de cirrosis y carcinoma hepatocelular y en poblaciones continentales (en ADH1B*2 y CYP2E1 la población africana presentaba los alelos ADH1B*1 y CYP2E1 c1, por lo que no se observan en la gráfica.) * p<0,05
El riesgo de cirrosis y carcinoma hepatocelular según el consumo de alcohol o tabaco, y por la presencia de los genotipos ADH1B, ADH1C y CYP2E1, se presenta en el cuadro 8. No se observaron diferencias significativas para ninguna de las variables. La odds ratio de la variable de consumo excesivo de alcohol tuvo un efecto protector en el grupo de control 2 (p=0,024), resultado que, aunque contradictorio, se explica por el elevado consumo de alcohol, lo cual no se esperaba en ese grupo de control.
Discusión
La asociación de las variantes genéticas de enzimas que metabolizan el alcohol, es una de las hipótesis para explicar por qué menos del 20 % de los individuos que consumen alcohol de manera crónica desarrolla cirrosis hepática. La actividad de las enzimas está regulada por los polimorfismos presentes en las regiones codificadoras y no codificadoras del gen respectivo. En estudios en poblaciones de diversas regiones del mundo con características genéticas diferentes, se ha sugerido que algunas variantes pueden implicar el riesgo de adquirir cirrosis en individuos que consumen alcohol de manera crónica, aunque los resultados son contradictorios.
En este estudio, se describen por primera vez las variantes genéticas de los genes ADH1B, ADH1C y CYP2E1 en pacientes con diagnóstico de cirrosis y carcinoma hepatocelular en Colombia.
La edad promedio de la población de estudio fue de 53 años y, al discriminar según el diagnóstico, fue de 53 años en pacientes con cirrosis y de 59 en aquellos con carcinoma hepatocelular (con cirrosis o sin ella). Estos resultados coinciden con lo reportado en casos de carcinoma hepatocelular en Brasil (55,9 años), en Chile (56 años), en población hispana de Estados Unidos (62,5 años) y en el estudio retrospectivo sobre este carcinoma en Bogotá, Medellín y Cali (61 años), pero difiere de los hallazgos de un estudio en Perú, en el que la edad promedio fue de 41,4 años 6,32-35. Estas variaciones pueden deberse a los patrones epidemiológicos y factores de riesgo predominantes en cada país, así como al nivel de complejidad del hospital en el que se trató a los pacientes. El 48,5 % de los casos incluidos en este estudio correspondía a mujeres. El predominio del sexo masculino en los casos se ha descrito a nivel global, con algunas excepciones, como el caso de Latinoamérica 7. Los genotipos más frecuentes fueron ADH1B*1/*1, ADH1C*1/*1 y CYP2E1*C/C, tanto en la población total de pacientes como en el análisis discriminado según si la condición se debía al consumo de alcohol. Estos genotipos concuerdan con lo reportado en poblaciones caucásicas 18,36, y es un hallazgo que se esperaba en la población de estudio, ya que más del 60 % de la población de Antioquia tiene una proporción de 0,4 o más de ancestro europeo 37.
Aunque no se observó una diferencia significativa, sí fue más frecuente el genotipo ADH1C*1/*1 en los pacientes cuya condición se debía al consumo crónico de alcohol (72 %), comparado con aquellos con una diferente causa etiológica (46 %). El genotipo ADH1C*1/*1 también fue más frecuente en los casos (59 %) que en los grupos de control 1 (48 %) y 2 (45 %), pero la diferencia no tuvo significación estadística (p=0,120 y p=0,083, respectivamente).
Asimismo, se observó una mayor frecuencia del genotipo ADH1B*2/*2, aunque discreta, en los pacientes cuya condición se debía al consumo crónico de alcohol (6 %), comparados con aquellos con otras causa etiológicas y con los grupos de control (1 % y 0 %, respectivamente).
Es de anotar que los genotipos ADH1C*1/*1 y ADH1B*2/*2 están asociados con una actividad catalítica de oxidación del alcohol 2,5 veces y 100 veces mayor, respectivamente (19,20), lo cual podría estar asociado con un riesgo de enfermedad hepática por acumulación de acetaldehído y por estrés celular.
Las diferencias en las frecuencias de los alelos ADH1B*2, ADH1C*1 y CYP2E1*T observadas en la población de estudio con respecto a poblaciones de otras regiones del mundo, pueden deberse a la composición étnica de la población colombiana (caucásica, amerindia y africana). Sin embargo, se deben considerar sesgos del análisis como el tamaño de la muestra, la representatividad de la población de procedencia (para evitar el efecto de selección natural o deriva génica, entre otros), así como el número de marcadores genéticos analizados 38,39.
Se esperaba una proporción similar de los dos alelos del gen ADH1C; sin embargo, se observó un franco predominio del alelo ADH1C*1 en los pacientes colombianos cuya condición se debía al consumo crónico de alcohol (83 %), así como en los pacientes con otras causas etiológicas (77 %) 17,40. Este hallazgo amerita estudios adicionales para establecer la relación entre el alelo ADH1C*1 y el desarrollo de cirrosis hepática por alcohol en la población colombiana. En diversos estudios se han explorado el riesgo de enfermedad hepática y los polimorfismos en el gen ADH1C con resultados contradictorios. Según un metaanálisis de 16 estudios de casos y controles publicado por He, et al., en pacientes asiáticos, los polimorfismos ADH1C*1/*2 se asociaron con un riesgo de 1,63 de desarrollar cirrosis alcohólica, en tanto que, en población caucásica, se asociaron con un bajo riesgo (0,76) 9. En el presente estudio, el genotipo ADH1C*1/*2 fue más frecuente en los grupos de control 1 (46 %) y 2 (48 %), que en los casos (34 %).
En un estudio reciente, se pudo analizar el riesgo de enfermedad hepática por alcohol en una población de India utilizando 14 marcadores genéticos. El estudio demostró una asociación significativa de los polimorfismos rs2066701CC en el ADH1B, rs 1693425TT en el ADH1C, rs 4880TT en el gen de la superóxidodismutasa de manganeso (MnSOD) y el alelo M1 del de la glutatión S transferasa (GSTM1), con la enfermedad hepática por alcohol en esta población. Los resultados del estudio permitieron establecer la interacción de los genes que codifican enzimas que participan en el metabolismo del alcohol (ADH1B y ADH1C) y los genes de reacción al estrés oxidativo (MnSOD y GSTM1), así como el efecto sinérgico en el riesgo de la enfermedad hepática por alcohol 41.
El riesgo de dependencia del alcohol también se ha explorado en algunas poblaciones. Los polimorfismos rs1229984 en el gen ADH1B y rs698 en el ADH1C presentaron asociación con el riesgo de dependencia en una población de Taiwán 42. Sin embargo, en un estudio en Turquía no fue posible establecer una asociación entre el riesgo de dependencia y los polimorfismos en ADH1B y ADH1C42. En el estudio en población colombiana de Méndez, et al., no se observaron diferencias significativas con respecto a la población de control; sin embargo, dado que la frecuencia de los alelos ADH1B*2, CYP2E1*1 y ALDH2*1 fue mayor en el grupo de individuos con dependencia del alcohol que en el grupo de control, los autores sugirieron que sí había una tendencia a la asociación 24.
Un resultado similar al descrito por Méndez, et al., se observó en el presente estudio con las frecuencias alélicas de ADH1B*2 en el 9 %, de ADH1C*1 en el 78 % y de CYP2E1*T en el 9% de los pacientes colombianos con diagnóstico de cirrosis y carcinoma hepatocelular, comparado con 11,5 %, 70,8 % y 6,2 %, respectivamente, en los individuos con adicción al alcohol del estudio de Méndez, et al. 24.
La población de estudio presentó frecuencias alélicas del CYP2E similares a las de la población de ancestro europeo, pero diferentes a las de poblaciones de África y Asia. El alelo CYP2E1*T, que presenta mayor actividad de transcripción, se detectó en el 9 % de los pacientes colombianos con diagnóstico de cirrosis y carcinoma hepatocelular; sin embargo, la frecuencia de este alelo fue mayor en los grupos de control 1 (19 %) y 2 (18 %). La frecuencia de este alelo en otras poblaciones de pacientes con cirrosis por consumo de alcohol, ha sido de 4 % en India, 6,1 % en México y 3,8 % en España 11,14,43. Los resultados del presente estudio sugieren que el alelo CYP2E1*T es frecuente en la población colombiana, incluso más en los grupos de control que en los casos. Este hallazgo es contradictorio y requiere confirmación en futuros estudios con un mayor tamaño de muestra en otras poblaciones colombianas.
En un reciente estudio en pacientes alcohólicos e individuos con consumo esporádico en Brasil, se sugiere que las variantes de CYP2E1 con mayor capacidad de transcripción estarían asociadas con un mayor daño del ADN debido a una tasa más alta en el metabolismo del alcohol y, por lo tanto, mayor producción y acumulación de acetaldehído, lo que genera el daño del ADN 44.
Este es el primer estudio sobre las frecuencias alélicas y genotípicas de enzimas que participan en el metabolismo del alcohol en pacientes con diagnóstico de cirrosis y carcinoma hepatocelular en Colombia. Los genotipos más frecuentes en los casos y en los grupos de control fueron el ADH1B*1/*1, el ADH1C*1/*1 y el CYP2E1*C/C, pero no se encontraron diferencias estadísticamente significativas en el grupo de paciente clasificados según si la causa etiológica de su condición respondía al consumo crónico de alcohol, ni en los grupos de control 1 y 2. El tamaño de la muestra, la inclusión de pacientes tratados en un único hospital y la ausencia de información sobre los ancestros, limitan las conclusiones del presente estudio, por lo que se requieren estudios adicionales para explorar la asociación de los polimorfismos en los genes ADH1C, ADH1B y CYP2E1, con el riesgo de enfermedad hepática en individuos colombianos que consumen alcohol de manera crónica, así como las variantes en otros genes que codifican enzimas que participan en el metabolismo del alcohol y en el control del estrés celular asociadas con la acumulación de los metabolitos del alcohol.