Los gliomas son los tumores primarios más frecuentes del sistema nervioso central. La Organización Mundial de la Salud (OMS) los clasifica de I a IV según su agresividad. Sin embargo, su comportamiento biológico, el pronóstico y la respuesta al tratamiento varían, incluso, dentro de una misma categoría histológica1.
Los recientes avances en el conocimiento de la biología molecular de los gliomas han contribuido a precisar mejor su diagnóstico y su pronóstico, a predecir la respuesta al tratamiento y a desarrollar nuevas estrategias específicas de tratamiento2-4. En el glioblastoma múltiple, por ejemplo, el pronóstico, la respuesta al tratamiento y la supervivencia, varían según si se trata de tumores primarios (de novo) o secundarios (progresión a partir de un glioma de menor grado), diferencias que se han correlacionado con la frecuencia de las diferentes alteraciones moleculares que estén presentes. Por ejemplo, las mutaciones en el gen supresor tumoral p53 se observan principalmente en glioblastomas múltiples secundarios, en tanto que la sobre expresión del factor de crecimiento epidérmico predomina en los primarios5. También, se han empleado otros marcadores para mejorar la clasificación histológica y la graduación de los gliomas, pero su utilidad es limitada5.
Recientemente, se han identificado mutaciones que determinan la pérdida de función de la enzima deshidrogenasa de isocitrato (IDH), una enzima involucrada en el proceso de metabolismo celular y en la respuesta al estrés oxidativo. Estas mutaciones ocurren tanto en la IDH de tipo 1 (IDH1) (citoplásmica y del peroxisoma) como en la IDH de tipo 2 (IDH2) (mitocondrial), y se han descrito principalmente en gliomas de bajo grado (50 a 80 % de los casos) y en glioblastomas múltiples secundarios (75 %), lo cual sugiere su importancia como marcadores en este tipo de tumores6. En general, las mutaciones en los genes IDH1 e IDH2 se han asociado con un mejor pronóstico, una mayor supervivencia global libre de progresión y una mejor respuesta a la quimioterapia7-9.
Las IDH son homodímeros que catalizan la descarboxilación oxidativa del isocitrato (IC) a ɑ-ceto-glutarato (ɑ-CG) y a dióxido de carbono con la reducción de la deshidrogenasa de alcohol (NADP+) a fosfato de dinucleótido nicotinamida adenina (NADPH), el cual se utiliza para la generación de glutatión reducido, el principal antioxidante celular que favorece la supervivencia celular y la antiapoptosis. Las mutaciones delIDH1 (395G→A: residuo Arg 132) y el IDH2 (515G→A: residuos Arg 172) inhiben su función, dando lugar a la generación de 2 hidroxiglutarato (2-HG), considerado un oncometabolito, y causando una disminución en los niveles de ɑ-CG y un incremento en los niveles de radicales libres.
Además de estabilizar al factor inductor de hipoxia-1a (HIF-1a), el 2-HG actúa como inhibidor competitivo de ɑ-CG, interfiriendo con su unión a demetilasas de histonas y, por ende, cambia el perfil de metilación a un fenotipo metilador de islas CpG (G-CIMP), lo cual favorece la inhibición de genes supresores tumorales. Asimismo, altera otra serie de vías, incluido el incremento del número de células madre, el compromiso en la diferenciación celular y la promoción de la transformación, las cuales favorecen el proceso de formación de tumores10,11. En general, los mecanismos por los cuales el 2-HG promueve la formación de tumores son variados y contradictorios, y es mucho lo que todavía se desconoce de ellos11.
Con la incorporación de esta información molecular, la última revisión de la clasificación de tumores del sistema nervioso central de la OMS (2016) incluyó gliomas u oligodendrogliomas difusos con base en parámetros moleculares, entre los cuales la presencia o la ausencia de mutaciones en la IDH aparecen como un factor importante de ayuda en el diagnóstico12.
El objetivo del presente trabajo fue definir la frecuencia de mutaciones en los genesIDH1 e IDH2 en una serie de casos de glioma en Colombia y comparar los resultados con los obtenidos enotros estudios.
Materiales y métodos
Pacientes y controles
Se analizaron 26 muestras de ADN obtenido de tejido tumoral de pacientes con diagnóstico de glioma (cuadro 1). Las muestras se obtuvieron del Grupo de Neurooncología del Instituto Nacional de Cancerología y se almacenaron en el Instituto de Genética de la Universidad Nacional de Colombia. Se analizaron, además, 100 muestras de ADN de individuos sanos sin historia familiar de gliomas, obtenidas del banco de ADN del Instituto de Genética de la Universidad Nacional de Colombia, con el objetivo de determinar la frecuencia poblacional de las mutaciones de interés.
Extracción y amplificación de ADN
Para la extracción de ADN a partir de las muestras, se utilizó el estuche GeneJET Genomic DNA Purification Kit™ (Thermo Scientific, USA), siguiendo las especificaciones del fabricante. En la amplificación mediante PCR del exón 4 de los genes IDH1 e IDH2, se emplearon los iniciadores (5′AAT GAGCTCTATATGCCATCACTG 3′) sentido y (5′TTCATACCTTGCTTAATGGGTGT 3′) antisentido; (5’ AATTTTAGGACCCCCGTCTG 3) sentido y(5’ TGTGGCCTTGTACTGCAGAG3’) antisentido, respectivamente, bajo las siguientes condiciones:95 °C durante 4 minutos, 30 ciclos a 95°Cdurante 30 segundos, a 59 °C durante 30 segundos y a72 °C durante 25 segundos, y un ciclo final a 72 °C durante 5 minutos.
Los fragmentos amplificados se comprobaron mediante electroforesis en geles de agarosa al 1,5 % de tampón salino (SB 1X), usando como revelador la SYBR SafeDNA Gel Stain™ (Thermo Fisher Scientific, USA). Los fragmentos obtenidos se purificaron con etanol y, posteriormente, se corrieron mediante electroforesis en un gel de agarosa al 1,5 % para evaluar su calidad.
Secuenciación de ADN
Los fragmentos de PCR purificados se secuenciaron con los mismos iniciadores sentido y antisentido usados para la amplificación. Las reacciones de secuencia se hicieron en un volumen total de 5 µl (4,5 µl del fragmento amplificado y 0,5 µl del iniciador) empleando el estuche de secuenciación BigDye Terminator™, 3.1 v. (Applied Biosystems, USA) y se corrieron en el secuenciador automático ABI PRISM 3500™ (Applied Biosystems, USA) del Instituto de Genética de la Universidad Nacional de Colombia.
Análisis de las secuencias
Las secuencias de nucleótidos obtenidas de las 26 muestras de tejido tumoral y de las 100 muestras de la población general, se visualizaron con el programa 4 Peaks (http://nucleobytes.com/index.php/4peaks) y se alinearon empleando el programa MAFFT (http://mafft.cbrc.jp/alignment/software/). Las secuencias reportadas en el GenBank del exón 4 de cada gen, se emplearon como referencia (números de acceso: Exón 4 IDH1: NM005896; Exón 4 IDH2: NM002168.2).
Resultados
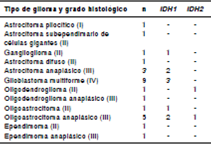
Se estudiaron muestras de 26 pacientes con diferentes tipos de gliomas, 17 hombres y 9 mujeres, con un rango de edad entre los 10 y los 58 años (cuadro 1). El análisis de las secuencias del exón 4 de los genesIDH1 e IDH2 reveló mutaciones en 11 casos (42,3 %): 9 en IDH1 y 2 en IDH2 (cuadro 2), comparadas con las secuencias de referencia de IDH1 (figura 1A) e IDH2 (figura 2A).
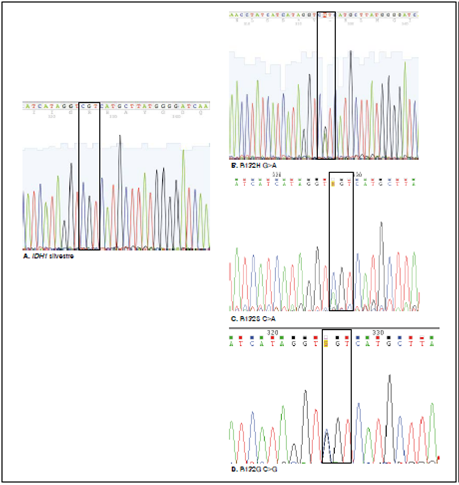
Figura 1 Electroferogramas de secuenciación del exón 4 del gen IDH en gliomas. A. Se muestra la secuencia de referencia en laque el codón 132 (CGT) codifica para el aminoácido arginina (R). B. En este codón se determinaron nueve mutaciones: siete casoscon mutación no sinónima heterocigota (G/A) en la segunda posición del codón, en la que se cambia la arginina (R) por histidina (H)(R132H); y, C. dos mutaciones en la primera posición del codón 132: una no sinónima heterocigota (C/A) que genera un cambio de arginina (R) por serina (S) (R132S), y, D. una no sinónima heterocigota (C/G) en la primera posición del codón 132, que produce elcambio de arginina (R) por glicina (G) (R132G).
Cuadro 2 Distribución de la frecuencia y el tipo de mutaciones en IDH1 e IDH2 según el grado histológico
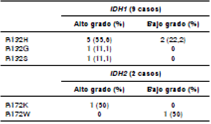
En el codón 132 (CGT) del gen IDH1, se presentaron siete casos con mutación no sinónima heterocigota (G/A) en la segunda posición del codón, la cual cambia arginina (R) por histidina (H) (R132H) (figura 1B). Dicha mutación se detectó en cinco tumores de alto grado (tres glioblastomas múltiples, un astrocitoma anaplásico de grado III y un oligoastrocitoma de grado III) y en dos tumores de bajo grado (un oligoastrocitoma de grado II y un ganglioglioma de grado II). Además, se detectaron dos mutaciones en la primera posición del codón 132: una no sinónima heterocigota (C/G) en la primera posición del codón 132, la cual produce el cambio de arginina (R) por glicina (G) (R132G), en un astrocitoma anaplásico (grado III) (figura 1C), y una no sinónima heterocigota (C/A) que genera un cambio de arginina (R) por serina (S) (R132S) en un oligoastrocitoma anaplásico (grado III) (figura 1D).
Por otro lado, en el análisis de secuencias del exón 4 del genIDH2, se identificó un paciente con oligoastrocitoma anaplásico (grado III) con una mutación no sinónima heterocigota (G/A) en la segunda posición del codón 172 (AGG), la cual produce el cambio de arginina (R) por lisina (K) (R172K) (figura 2B), y una mutación no sinónima heterocigota (A/T) en la primera posición del codón 172, la cual produce un cambio de arginina (R) por triptófano (W) (R172W) en un oligodendroglioma de grado I (figura 2C).
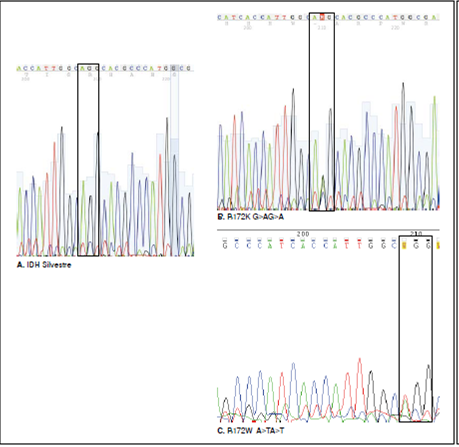
Figura 2 Electroferogramas de secuenciación del exón 4 del gen IDH2 en gliomas. A. Se muestra la secuencia de referencia en la que aparece el codón 172 (AGG) que codifica para una arginina (R), en la cual se determinaron dos mutaciones: B. una nosinónima heterocigota (G/A) en la segunda posición, la cual produce el cambio de arginina (R) por lisina (K) (R172K) y, C. una mutación no sinónima heterocigota (A/T) en la primera posición del codón 172, la cual produce un cambio de arginina (R) por triptófano (W) (R172W).
En ninguna de las 100 muestras de controles secuenciadas (80 mujeres y 20 hombres, con un rango de edad entre 38 y 89 años), se detectaron mutaciones en el exón 4 de los genes IDH1 e IDH2.
Discusión
En el presente estudio se determinó la presencia de mutaciones no sinónimas en el exón 4 del genIDH1 (codón 132) y del genIDH2 (codón 172). Estas mutaciones se detectaron en 11 de los 26 casos con algún tipo de glioma (42,3 %; IDH1: 34,6 % e IDH2: 7,7 %), y no así en los 100 individuos de la población general, lo cual sugiere que su frecuencia es muy baja en individuos sin gliomas y sin antecedentes familiares de este tipo de cáncer. Debe tenerse en cuenta que en el presente estudio se utilizó la clasificación del 2007, la cual difiere de la reciente clasificación del 2016 que, además de la histología y de la inmunohistoquímica (GFPA, S100, Oligo, Ki- 67, NF, sin aptofisina, etc.), incorpora pruebas moleculares (codeleción 1p19q, p53, ATRX, estado de metilación de MGMT) y la evaluación de las mutaciones de IDH, con lo cual es posible detectar tumores con IDH silvestre o mutada. De esta manera, se facilita su pronóstico y su posible empleo predictivo en un futuro. Cuando no se evalúa la presencia de mutaciones en IDH1 e IDH2, se recomienda usar el término NOS (Non Other wise Specified).
De las 11 mutaciones encontradas, ocho se presentaron en tumores de alto grado con diferenciación astrocitaria (astrocitoma anaplásico, oligoastrocitoma anaplásico y glioblastoma múltiple) y tres en tumores de bajo grado(ganglioglioma, oligodendroglioma y oligoastrocitoma). Con respecto a los oligoastrocitomas, es importante señalar que esta categoría fue suprimida en la clasificación de la OMS del 2016, pues el uso de las pruebas moleculares ha permitido diferenciar astrocitomas de oligodendrogliomas en casos que presentan aspecto histológico mixto, y solo dejan una mínima proporción de casos con características de diferenciación dual.
Teniendo en cuenta los resultados de este y otros estudios, se ha sugerido que las mutaciones enIDH1 e IDH2 se presentan desde fases tempranas del proceso carcinogénico, y podrían contribuir a la progresión tumoral, por lo cualse consideran como marcadores de glioblastoma múltiple secundario6,10,13,14. Estos resultados también destacan la importancia de estas mutaciones en los gliomas en Colombia. No obstante, se requieren estudios a mayor escala y un tamaño mayor de la muestra, para determinar con precisión la real contribución de estos cambios genéticos en la carcinogénesis de los gliomas.
En estudios previos en otras poblaciones, se ha reportado una frecuencia de mutaciones en el IDH1 de 71 % en los gliomas de grado II, de 64 % en gliomas de grado III, de 76 % en glioblastomas múltiples secundarios y solo de 6 % en gliomas primarios. La frecuencia de mutaciones en el IDH2 es de 2,4 % en promedio en gliomas de grado II, de 4 % en gliomas de grado III, y no se ha informado en glioblastomas múltiples13.
Hay pocos estudios en población latinoamericana. En Brasil se demostró una frecuencia global de 11,8 % (19 de 161 casos) en glioblastomas múltiples: 66,7 % en los secundarios y 9,7 % en los primarios, la cual se asoció con una mayor supervivencia15. Recientemente, en un estudio en población colombiana con gliomas de bajo grado (astrocitomas, oligodendrogliomas y oligoastrocitomas), se reportó una frecuencia de 57,1 % de mutaciones en los genes IDH1 e IDH216. En el presente estudio, la mutación más frecuentemente observada en el gen IDH1 fue la R132H, que ha sido reportada con frecuencia hasta en el 90 % en otras series de casos13,14,17, en tanto que en el genIDH2 se encontraron las mutaciones R172K y R172W, informadas previamente13,17.
De las 11 mutaciones encontradas, ocho se presentaron en tumores de alto grado con diferenciación astrocitaria y de oligodendroglía (astrocitomas y oligoastrocitomas de grado III y glioblastoma múltiple de grado IV), y tres de ellas en tumores de bajo grado con esta misma diferenciación (oligodendroglioma, oligoastrocitoma y un ganglioglioma). No se detectaron mutaciones en un astrocitoma pilocítico (grado I), un astrocitoma subependimario de células gigantes y dos ependimomas (grados II y III).
Debido a que el estudio de Cardona, et al., se circunscribió a gliomas de bajo grado en los que se encontró una frecuencia de dichas mutaciones más alta de la reportada aquí16, nuestros hallazgos refuerzan la importancia del estudio de mutaciones enIDH1 e IDH2 en casos de glioblastoma múltiple. Precisamente, en este estudio la presencia de mutaciones en gliomas de alto grado sugiere su progresión a partir de gliomas de bajo grado, lo cual se correlaciona también con la edad de los pacientes analizados.
Además de los gliomas, se han identificado mutaciones en el exón 4 de los genes IDH1 e IDH2 en otros tumores, como los cartilaginosos (54 % en condrosarcomas esporádicos centrales, 71 % en condrosarcomas parostales y 57 % en condrosarcomas no diferenciados, en leucemia mieloide aguda (15 a 30 %) (9,13), en colangiocarcinomas intrahepáticos (10-23 %) y en adenocarcinomas prostáticos (2 %)9. Estas mutaciones se asocian a alteraciones metabólicas y epigenéticas de peor pronóstico en estos tumores18.
Sin embargo, el valor predictivo de las mutaciones en los genesIDH no es claro aún, lo cual es particularmente importante cuando se ha empezado a evaluar el uso de inhibidores para revertir su efecto transformador, promover la diferenciación celular9 y aumentar la sensibilidad a la quimioterapia y a la radioterapia10.
Debido a que las mutaciones del IDH1 en el laboratorio de patología se exploran mediante el estudio de la expresión de la proteína mutada mediante inmunohistoquímica utilizando el anticuerpo (mIDH1R132H) en cortes de tejido incluidos en parafina19, y teniendo en cuenta que su valor es limitado, ya que no identifica la expresión de productos de otras mutaciones deIDH 1 e IDH2, se requieren análisis más precisos que identifiquen mutaciones alternativas y relevantes mediante el análisis del ADN20. Esto es importante porque, además de su valor como factor pronóstico, se están empezando a evaluar inhibidores de las IDH mutadas y silvestres como alternativas terapéuticas que reversan sus efectos transformadores, promueven la diferenciación celular9 y aumentan su sensibilidad a la quimioterapia y la radioterapia10.
En conclusión, en el presente trabajo se reporta una frecuencia de mutaciones enIDH1 e IDH2 de 42,3 % en una serie de 26 gliomas de pacientes colombianos, frecuencia similar a la encontrada en estudios previos. Teniendo en cuenta la ausencia de estas mutaciones en la población general analizada, su estudio puede ser importante para determinar su valor como factor pronóstico y como potenciales blancos terapéuticos en gliomas.