A pesar de los estrictos controles que se imponen a la industria alimentaria, cada año mueren, aproximadamente, tres millones de personas como consecuencia de enfermedades transmitidas por alimentos 1,2, siendo la causa principal el consumo de agua o alimentos contaminados 3,4. Se han descrito más de 250 enfermedades transmitidas por alimentos 5, lo que exige llevar a cabo controles microbiológicos y sanitarios cada vez más estrictos durante los procesos de producción de alimentos.
Según la Norma General del Codex Alimentarius, el queso fresco se define como el producto higienizado sin madurar que está listo para el consumo después de su elaboración. El queso se clasifica según su consistencia, es decir, el valor correspondiente al porcentaje de humedad sin materia grasa, en extraduro, duro, firme semiduro y blando 6,7. El queso que se produce y distribuye en la región Caribe colombiana es un producto autóctono de los departamentos de Córdoba, Sucre, Bolívar, Atlántico, Magdalena, Cesar y La Guajira. Se elabora con leche de vaca no pasteurizada utilizando una tecnología muy rústica y se somete a un periodo de salado de dos horas para prensarlo posteriormente; es de color blanco y no es madurado ni ácido. El procedimiento para su elaboración no está estandarizado ni responde a las normas higiénicas y, además, el almacenamiento del producto no es adecuado 8,9.
Dado su consumo masivo, el riesgo de listeriosis, salmonelosis y brucelosis, producidas por Listeria monocytogenes y microorganismos de los géneros Salmonella spp. y Brucella spp., es alto. Dichas infecciones hacen parte de las enfermedades transmitidas por alimentos, cuyo impacto en la salud pública es innegable 10-12.
La transmisión de Brucella spp. puede darse por contacto directo con secreciones de animales infectados 13 o, indirectamente, por el consumo de productos lácteos no pasteurizados 14. Sin embargo, los reportes de transmisión de persona a persona de Brucella spp. son escasos 15, en tanto que Salmonella spp. y L. monocytogenes se transmiten principalmente por la ingestión de alimentos contaminados. El desarrollo de estas enfermedades depende de varios factores, como la patogenia del microorganismo, la dosis infecciosa y el estado de salud del consumidor 16.
El objetivo de este trabajo fue detectar mediante qPCR Salmonella spp., Listeria spp. y Brucella spp. en quesos artesanales procedentes de los departamentos de la costa Atlántica colombiana que se distribuyen en expendios de Barranquilla.
Materiales y métodos
Origen y características de los quesos
Se encontraron 27 expendios en la base de datos de la Secretaría de Salud de Barranquilla. Esta información se complementó buscando en el directorio telefónico y mediante un sondeo directo. Diecisiete de los expendios aceptaron participar. El origen y el tipo de los quesos expendidos en la ciudad, se determinaron entrevistando a los distribuidores mediante una encuesta estructurada en la cual se clasificaba el tipo de queso en blando, semiduro y duro, y se consignaba su procedencia y el volumen de queso acopiado para la venta.
Muestreo de los quesos frescos
Mediante un muestreo no probabilístico entre los meses de enero y mayo del 2013, se recolectaron muestras de queso costeño fresco en 17 expendios mayoristas de Barranquilla, y se tomaron 27 muestras. Los expendios incluidos vendían queso costeño fresco producido en la costa Caribe colombiana y sus distribuidores aceptaron participar libremente en el estudio mediante consentimiento informado. De cada muestra se tomaron 100 g que fueron procesados en el Laboratorio de Investigación de la Universidad Libre de Barranquilla.
Extracción, purificación y cuantificación del ADN
Se pesaron 25 g de cada muestra y se homogenizaron en 225 ml de agua con peptona al 0,1 % en un equipo Stomacker 400 Circulator™ (Seward Ltd.) a 260 rpm durante 30 segundos.
Con el propósito de permitir que las partículas sólidas en suspensión se decantaran, los homogenizados permanecieron a temperatura ambiente durante dos horas y, posteriormente, se mezclaron 500 μl del sobrenadante con 200 μl de la solución tampón TE 1X (10 mM Tris-HCl, 1 mM EDTA, pH 8,0). En la lisis celular se emplearon 10 mg/m con pH 8,0 de lisozima A y 0,5 % v/v de dodecilsulfato sódico (SDS). La mezcla se incubó durante 90 minutos a 37 °C.
Para eliminar los péptidos y lípidos residuales, se adicionaron 80 μl de una solución de 5 M de cloruro de sodio (NaCl) más 100 μl de 3M de acetato de amonio y se incubó nuevamente durante 10 minutos a 65 °C. La suspensión resultante se trató con una mezcla de fenol, cloroformo y alcohol isoamílico (25:24:1), y el sobrenadante se tomó para una segunda purificación con cloroformo y alcohol isoamílico (24:1).
El ADN se sometió a precipitación con isopropanol frío durante 30 minutos y, posteriormente, se lavó con etanol al 70 %. Después de la evaporación del etanol, se hidrató con 20 μl de agua libre de desoxirribonucleasas (DNasa) y ribonucleasas (RNasa). La pureza y concentración del ADN se determinaron en un espectrofotómetro NanoDrop 2000c™ de Thermo Scientific, Waitham, MA, USA (17,18).
Reacción en cadena de la polimerasa en tiempo real
La amplificación mediante qPCR para la detección del género Salmonella spp. se realizó con el estuche comercial iQ-Check Bio-Rad Salmonella II™, la del género Listeria spp., con el estuche comercial iQ-Check Listeria spp.™ (Bio-Rad), siguiendo las instrucciones del fabricante, y la de Brucella spp., con el estuche comercial Quantification of Brucella genus (all species)™ (PrimerDesign, Ltd.), el cual emplea una sonda TaqMan™ leída en el canal FAM.
Se preparó la mezcla de reacción siguiendo las indicaciones del fabricante para un volumen final de 25 μl, con una concentración de ADN de 150 ng. El protocolo de amplificación se llevó a cabo con el estuche CFX96 Touch Real-Time PCR Detection System™ (Bio-Rad, Hercules, California) en un termociclador C-1000™.
En todas las muestras se agregó un control interno en el momento de la lisis celular durante la extracción del ADN, el cual se amplificó conjuntamente con el ADN de la muestra utilizando otra sonda TaqMan™ detectada en el canal VIC. En cada ensayo se incluyeron los respectivos controles positivos, negativos y los blancos, según las instrucciones del fabricante.
Análisis de resultados
El porcentaje de muestras positivas para los géneros Salmonella, Listeria y Brucella se determinó mediante el cociente entre el número de muestras positivas y el número total de muestras multiplicado por 100. El cálculo se hizo considerando como unidad de muestreo el expendio y, como unidad objeto de análisis, el tipo de queso.
Resultados
Origen y características de los quesos
Según la encuesta aplicada, el queso distribuido en expendios mayoristas de Barranquilla procedía de 36 municipios y corregimientos de la región Caribe colombiana. El departamento del que provenía la mayor parte (58 %) de este producto fue Magdalena. Los departamentos de Atlántico y Bolívar aportaron 19 y 14 %, respectivamente. Los departamentos con los menores porcentajes fueron Sucre (6 %) y Córdoba (5 %). Los volúmenes distribuidos oscilaban entre 50 y 2.000 kg por día. En cuanto al tipo de queso, el de mayor distribución fue el queso blando (58 %), en tanto que los quesos semiduro y duro registraron un porcentaje de 21 % cada uno.
Características de los quesos frescos analizados
Los porcentajes de muestras positivas para Salmonella spp., Listeria spp. y Brucella spp. fueron de 63 % (17/27), 70,4 % (19/27) y 22,2 % (6/27), respectivamente. De las 27 muestras recolectadas, 17 (63 %) eran de queso blando, 5 (18,5 %) de queso semiduro y 5 (18,5 %) de queso duro.
Once de 17 (64,7 %) muestras de queso blando fueron positivas para Salmonella spp., 13 de 17 (76,5 %) lo fueron para Listeria spp. y 6 de 17 (35,3 %) para Brucella spp., es decir, el total de muestras positivas para Brucella spp. en este estudio, se encontraron en queso blando. Tres de las muestras fueron positivas simultáneamente para los tres microorganismos. En cuatro de las 27 muestras analizadas, las cuales fueron positivas para Brucella spp. (sin determinar), no se logró la amplificación del control interno.
Cuatro de 5 (80 %) de las muestras de queso duro fueron positivas para Salmonella spp., 3 de 5 (60 %) para Listeria spp. y ninguna fue positiva para Brucella spp., en tanto que 2 de 5 (40 %) muestras de queso semiduro fueron positivas para Salmonella spp., 3 de 5 (60 %) para Listeria spp. y ninguna para Brucella spp.
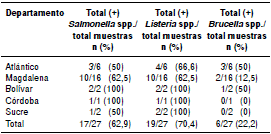
Porcentaje de muestras positivas para Salmonella spp., Listeria spp. y Brucella spp. por departamento de procedencia del queso
De las muestras de quesos procedentes de Atlántico, 3 de 6 (50 %) fueron positivas para Salmonella spp., 4 de 6 (66,6 %) para Listeria spp. y 3 de 6 (50 %) para Brucella spp. De las de quesos procedentes del Magdalena, 10 de 16 (62,5 %) fueron positivas para Salmonella spp., 10 de 16 (62,5 %) para Listeria spp. y 2 de 16 (12,5 %) para Brucella spp. Las dos muestras de quesos procedentes de Bolívar fueron positivas para Salmonella spp. y para Listeria spp., y una de ellas para Brucella spp.
En las muestras de los quesos de Córdoba y Sucre, no se encontró Brucella spp., pero la única muestra de Córdoba fue positiva para Salmonella spp. y Listeria spp.; las dos muestras de quesos provenientes de Sucre fueron positivas para Listeria spp., pero solo una lo fue para Salmonella spp. (cuadro 1).
Discusión
En Barranquilla confluyen varias rutas de comercialización del queso costeño proveniente de los departamentos del Caribe colombiano, principalmente de Magdalena, que en este estudio aportó más del 50 % del queso distribuido en la ciudad, en tanto que Sucre y Córdoba tuvieron una participación menor en dicha comercialización, aunque son departamentos con una alta prevalencia de brucelosis en los hatos lecheros 19.
En el presente estudio se reporta la presencia de Brucella spp. en quesos distribuidos en Barranquilla. Este agente patógeno se ha encontrado previamente en las etapas de producción primaria de leche en otras ciudades de Colombia 20,21. En el estudio se reconfirmó, además, la presencia de Salmonella spp. 22,23 en quesos artesanales distribuidos en el Caribe colombiano y de Listeria spp. en queso costeño. Como en el caso de Brucella spp., este último patógeno se ha encontrado anteriormente en la materia prima para la elaboración de los quesos 24. Puede concluirse, entonces, que en Barranquilla el consumo de queso costeño estaría asociado con el riesgo de contagio por Salmonella spp. y Brucella spp.
Sin embargo, el riesgo también debe asociarse con la viabilidad de las bacterias en el alimento y el tipo de queso costeño consumido. El tiempo de supervivencia de los microorganismos en el queso depende del proceso de elaboración y de factores como la concentración de sal, el pH, las condiciones de maduración y las cepas bacterianas empleadas en la transformación de la leche 25.
En este estudio se encontró ADN de Salmonella spp., Listeria spp. y Brucella spp. simultáneamente en tres muestras de queso fresco costeño blando, lo cual coincide con lo reportado en otros estudios en los cuales se ha demostrado que estos microorganismos sobreviven más tiempo en los quesos blandos que en los duros 26.
Por otra parte, la ausencia de Brucella spp. en quesos duros con un contenido de sal de alrededor de 3,47 % 27, puede explicarse por la sensibilidad de Brucella spp. a las altas concentraciones de sal 26. Se ha encontrado que la sal utilizada en los procesos de salmuera o salado directo del queso reducen el crecimiento de los microorganismos patógenos y disminuyen su actividad metabólica (28,29). Cinco de las muestras de queso blando analizadas fueron positivas tanto para Brucella spp. como para Listeria spp., 12 fueron positivas para Salmonella y Listeria spp., y de estas, ocho eran de queso blando, tres de queso duro y una de queso semiduro.
Se ha reportado la viabilidad de algunas cepas de Listeria spp. aisladas de quesos con altas concentraciones de cloruro de sodio 30, lo cual explicaría el mayor porcentaje de muestras positivas de Listeria spp. frente a los otros microorganismos evaluados en este estudio.
Otro factor que se debe tener en cuenta es el crecimiento de microorganismos que hacen parte de la microflora del alimento y que compiten por los nutrientes, hecho que aumenta la probabilidad de reportar falsos negativos atribuibles principalmente al método microbiológico tradicional, el cual determina cualitativamente la presencia o ausencia de microorganismos y, además, emplea medios de cultivo selectivos a lo largo de muchas etapas, como las de caracterización bioquímica y serológica de colonias bacterianas 31.
Aunque el método estándar o cultivo microbiológico tradicional tiene un gran valor en los estudios epidemiológicos, su sensibilidad es baja y la confirmación de los resultados tarda entre cuatro y cinco días 32, por lo tanto, es necesario emplear metodologías más sensibles de diagnóstico, como la qPCR 21. Aunque la técnica de qPCR detecta el ADN de los microorganismos y no determina su viabilidad, se pudo confirmar su aplicabilidad para detectar microorganismos de difícil aislamiento por su elevado límite de detección.
En diversas investigaciones, se han reportado límites de detección para el ADN de Brucella spp. del orden de 16 a 1.600 ng con diferentes protocolos de qPCR 33. En este estudio fue posible la detección con 83,25 ng de ADN total extraído de una muestra positiva, lo que evidencia el aumento de la probabilidad de detección en muestras de alimentos en las cuales estos agentes patógenos se encuentran generalmente en pequeñas cantidades. En un estudio de 30 muestras de queso, realizado en México, no se detectaron bacterias mediante el método microbiológico, lo cual se explicó por el bajo número de microorganismos presente en el alimento 34. En un estudio en Brasil, solo mediante PCR convencional fue posible detectar la bacteria en quesos ilegales 35.
El hecho de que en cuatro de las muestras de queso en este estudio no hubo amplificación del control interno, se explicaría, según estudios previos, por la presencia de inhibidores de la PCR. En tales estudios se han reportado variaciones en la inhibición de polimerasas en diferentes concentraciones de la muestra 36,37. Entre los inhibidores de polimerasas presentes en alimentos como el queso, se han reportado el sodio y el calcio.
En este estudio se presentó un mayor porcentaje de inhibición en muestras de queso duro, lo cual se puede asociar a su alto contenido de cloruro de sodio, ya que las polimerasas Ultma, rTth, Taq, HotTub, Tfl y Taq son sensibles a concentraciones mayores de 60 mM de Na+2. También, se ha reportado un efecto inhibitorio de la Taq polimerasa con concentraciones mayores de 0,2 mM de Ca+2, lo que puede explicar la inhibición en algunas muestras de queso blando, pues se ha demostrado que en productos lácteos el calcio es la mayor fuente de inhibición, aunque también el magnesio utilizado en la reacción de PCR puede tener efecto inhibitorio inverso de los iones calcio 38.
En cuanto a la presencia de bacterias como Salmonella spp. y Listeria spp., que son de difícil aislamiento, su gran frecuencia sugeriría su presencia a lo largo de toda la cadena de producción del queso, desde la obtención de la leche en las fincas hasta la distribución del producto. La presencia de dichas bacterias en estos productos se ha reportado en otros trabajos en Colombia, en los cuales se ha determinado que el queso fresco es uno de los alimentos con mayor presencia de L. monocytogenes entre los de venta callejera en Bogotá 39, así como en las ciudades de Montería y Cereté, donde se confirmó que los quesos caseros frecuentemente están contaminados con Listeria spp. 40,41. A nivel internacional, se han reportado Salmonella spp. y Listeria spp. en quesos frescos de venta callejera 42.
El presente estudio fue un ensayo piloto en el que se demostró la utilidad de la técnica de qPCR como método alternativo para determinar el estado microbiológico del queso y para el desarrollo de estudios de vigilancia epidemiológica, al demostrar la presencia de microorganismos de difícil aislamiento, como Brucella spp., Salmonella spp. y Listeria spp., en muestras de queso costeño procedentes de diferentes departamentos de la costa Caribe colombiana.
Estos resultados constituyen una alerta de salud pública en torno al queso como producto que puede transmitir estas bacterias, y plantea la necesidad de implementar planes de vigilancia y control.