Helicobacter pylori es una bacteria microaerófila Gram negativa capaz de colonizar la mucosa gástrica; causa gastritis crónica en el 100 % de las personas infectadas, úlceras gástricas en 10 a 20 %, linfoma de tejido linfoide asociado con la mucosa gástrica (Mucosa-Associated Lymphoid Tissue, MALT) en menos del 1 % y adenocarcinoma gástrico distal en 1 a 2 %.
Helicobacter pylori es el principal factor de riesgo para el desarrollo de cáncer gástrico y ha sido clasificado como carcinógeno de tipo I por la Organización Mundial de la Salud (OMS) 1,2, por lo cual, los expertos mundiales recomiendan que debe erradicarse siempre 3,4. Sin embargo, a nivel mundial existe preocupación porque la eficacia de los tratamientos de primera línea comúnmente empleados es inferior al 80 % 5, lo cual también ocurre en Colombia, como quedó demostrado en un estudio de Trespalacios, et al. (Trespalacios S, Otero W, Mercado M. Efficacy of first-line Helicobacter pylori eradication treatment with two triple regimens with levofloxacin. Gastroenterology. 2014;S-741-2. https://doi.org/10.1016/S0016- 5085(14)62685-8).
Si bien el éxito terapéutico responde a múltiples factores ambientales o relativos al huésped, como el incumplimiento del tratamiento, y a la presencia de polimorfismos en la interleucina 1B (IL-1B) en enzimas involucradas en el metabolismo de los medicamentos (CYP2C19 y CYP3A4) y en el de las bacterias, se ha demostrado que la principal causa del fracaso del tratamiento es la resistencia bacteriana 6. Por ello, en febrero de 2017, la OMS incluyó a H. pylori en la lista de los 16 microorganismos que amenazan a la humanidad y ameritan urgentes medidas de control, clasificándolo en la categoría de más alta prioridad, debido al aumento de la resistencia a los antibióticos de primera línea, metronidazol y claritromicina, especialmente a este último 7, y a la aparición de aislamientos multirresistentes.
A nivel mundial, hay pocos estudios sobre la multirresistencia de H. pylori, es decir, la resistencia a tres o más antibióticos, pero se estima que aproximadamente 15 % de los aislamientos clínicos la presentan; esto implica una disminución de la tasa de erradicación y, por lo tanto, una grave amenaza para la salud humana 8. En el caso de H. pylori, la multirresistencia se da simultáneamente frente a amoxicilina, metronidazol y claritromicina 9,10.
En general, H. pylori se hace resistente a amoxicilina, claritromicina, metronidazol, levofloxacina y tetraciclina, principalmente, mediante mutaciones puntuales en un par de bases del gen que codifica para el sitio de acción del antibiótico, o por acción de las bombas de expulsión bacterianas, que normalmente protegen al microorganismo de la toxicidad de los antibióticos 11-18. Las bombas de expulsión son un complejo estructural de proteínas en la membrana interna de las bacterias, que actúa en sincronía con proteínas adaptadoras del periplasma y canales presentes en la membrana externa, las cuales expulsan eficientemente los medicamentos 19. Dichas proteínas se asocian, especialmente, con el desarrollo de multirresistencia, como en el caso de la codificada por el gen hefA (p=0,003) 20.
En Colombia, la resistencia primaria a claritromicina, metronidazol y levofloxacina es alta, de 13,6, 81 y 27 %, respectivamente 14,21,22; esto limita su utilización en los tratamientos empíricos de primera línea. Sin embargo, son escasos los estudios en pacientes en quienes ya ha fracasado el tratamiento, pues, a menudo, se excluyen de los estudios de sensibilidad 23. Además, el número cada vez mayor de tratamientos de erradicación puede haber resultado en que no se investiguen más pacientes con infecciones resistentes al tratamiento 23. Sin embargo, la vigilancia de la presencia de aislamientos de H. pylori multirresistente es esencial para permitir el uso racional de los antibióticos después de repetidos fracasos terapéuticos, especialmente en lugares donde no se dispone de las pruebas de sensibilidad a los antibióticos 24.
En este contexto, el objetivo de este estudio fue analizar los aislamientos clínicos H. pylori de pacientes con varias fallas terapéuticas previas en Bogotá, Colombia.
Materiales y métodos
Tipo de estudio y pacientes
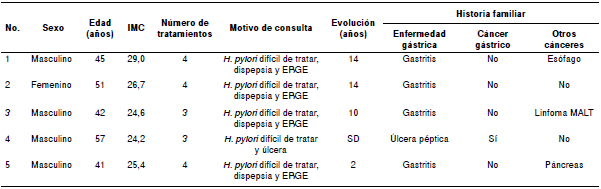
Se llevó a cabo un estudio descriptivo en el que se incluyeron 5 pacientes que fueron previamente excluidos de un ensayo clínico de 355 pacientes 26 debido a que habían recibido tratamiento previo para H. pylori. A los 5 pacientes incluidos en este estudio se les realizó endoscopia de vías digestivas altas en diferentes centros de Bogotá entre febrero y noviembre de 2014. Los motivos de consulta fueron dispepsia y reflujo en cinco pacientes, así como una historia de infección por H. pylori de difícil tratamiento, de 2 a 14 años de evolución.
Después de la explicación médica sobre el procedimiento endoscópico y sobre el estudio, los pacientes firmaron el consentimiento informado. Posteriormente, se hizo la endoscopia de vías digestivas altas después de un ayuno mínimo de seis horas 21. Se tomaron biopsias de rutina para el estudio de la gastritis crónica, según el protocolo propuesto por Rugge, et al. 25, y biopsias adicionales para el cultivo de H. pylori: cuatro biopsias del antro y cuatro del cuerpo del estómago. Una de las biopsias de antro se sometió a la prueba rápida de ureasa.
Transporte de muestras y cultivo
Las biopsias del antro y el cuerpo gástricos destinadas al cultivo de H. pylori, se colocaron en 500 μl de caldo Brucella (Beckton Dickenson) con suplemento de 20 % (p/v) de glicerol (Invitrogen) y se almacenaron a 4 °C hasta su procesamiento. Los aislamientos de H. pylori obtenidos y las biopsias restantes, se mantuvieron a -70 °C 21.
Las biopsias se maceraron en condiciones asépticas y estériles, con un aplicador de madera previamente tratado en solución de carbón activado al 1 % (p/v), hasta que se logró la homogeneidad total 21. Posteriormente, se sembraron en agar Brucella (Beckton Dickenson) con suplemento de IsoVitaleX™ al 4 % (v/v), 7 % (v / v) de sangre de caballo y 2 ml de suplemento selectivo para H. pylori, DENT™ (Thermo Scientific Oxoid, UK).
Los cultivos se incubaron a 37 °C en condiciones microaerofílicas durante 3 a 14 días. Después de la incubación, se observaron pequeñas colonias transparentes que se sometieron a identificación fenotípica mediante coloración de Gram (bacilos curvos Gram negativos), oxidasa (positiva), catalasa (positiva) y prueba rápida de ureasa (positiva) 21. Posteriormente, se obtuvieron cultivos puros, para lo cual se aislaron las colonias con las características apropiadas en agar Brucella y se incubaron a 37 °C en condiciones de microaerofilia durante tres a cinco días.
Pruebas de sensibilidad fenotípica y genotípica
La sensibilidad fenotípica a H. pylori se evaluó a partir de cultivos puros para amoxicilina, claritromicina, levofloxacina y metronidazol mediante la prueba de dilución en agar, según las recomendaciones de los Centers for Disease Control and Prevention (CDC) 26,27. Los puntos de corte utilizados para determinar la resistencia fueron ≥1 μg/ml para amoxicilina, claritromicina y levofloxacina, y 8 μg/ml para metronidazol 27-31. Como control se usó la cepa de H. pylori NCTC 11637.
Además, los aislamientos se analizaron genotípicamente para identificar posibles mutaciones asociadas con la resistencia y confirmar la sensibilidad fenotípica. Se usó DNAzol™ (Invitrogen) para extraer el ADN genómico de las colonias de H. pylori aisladas del antro y del cuerpo del estómago.
Posteriormente, se amplificaron los genes bacterianos relacionados con la resistencia a la amoxicilina (pbp1A), la claritromicina (rna23s), la levofloxacina (gyrA) y el metronidazol (rdxA), mediante reacción en cadena de la polimerasa (PCR) convencional, según protocolos previamente estandarizados 14,22 (cuadro 1).
Análisis bioinformático
La sensibilidad genotípica y las mutaciones relacionadas con la resistencia a los antibióticos, se determinaron con la ayuda de análisis bioinformáticos. Las mutaciones asociadas con la resistencia a la claritromicina se detectaron con la herramienta bioinformática BLAST-N (Basic Local Alignment Search Tool-N). Para analizar las secuencias de detección de mutaciones relacionadas con la resistencia a amoxicilina, levofloxacina y metronidazol se utilizó la BLAST-X. Se empleó la secuencia de referencia proveniente de H. pylori 26695 (taxid: 85962), número de acceso a GenBank CP003904.1.
Resultados
Distribución de los pacientes
Durante el 2014, 5 de los pacientes remitidos a endoscopia de vías digestivas habían recibido varios tratamientos contra H. pylori sin éxito: cuatro hombres y una mujer entre los 41 y los 57 años de edad. Todos informaron tener antecedentes familiares de enfermedades gástricas. Cuatro (4/5) refirieron tener antecedentes familiares de gastritis y, uno (1/5), antecedentes de úlcera péptica. Un paciente (1/5) refirió antecedentes familiares de cáncer gástrico y, tres (3/5), de otros tipos de tumores gastrointestinales, como cáncer de esófago, cáncer de páncreas y linfoma MALT (cuadro 2).
Pruebas de sensibilidad fenotípica y genotípica
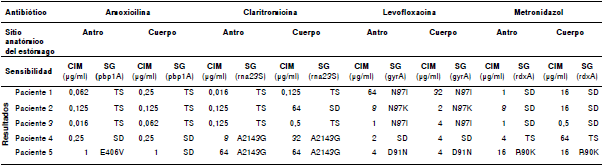
Todos los aislamientos de los pacientes tuvieron resultados positivos en la prueba rápida de ureasa. Se obtuvieron diez aislamientos de antro y de cuerpo del estómago (cinco de antro y cinco de cuerpo). A todos los aislamientos se les hizo antibiograma para amoxicilina, claritromicina, levofloxacina y metronidazol, mediante dilución en agar.
Según el análisis de sensibilidad fenotípica, en un solo paciente se obtuvieron dos aislamientos de H. pylori (uno del cuerpo del estómago y otro del antro) resistentes a la amoxicilina. Los aislamientos de tres de los pacientes fueron resistentes a la claritromicina (tres del cuerpo y dos del antro); en cuatro de los pacientes, se encontraron aislamientos resistentes al metronidazol (cuatro del cuerpo y tres del antro). Por último, los aislamientos de todos los pacientes fueron resistentes a la levofloxacina (cuadro 3).
Cuadro 3 Sensibilidad a los antibióticos
CIM: concentración inhibitoria mínima; SG: sensibilidad genotípica: TS: tipo silvestre; SD: sin datos
Se estudiaron las mutaciones asociadas con resistencia a amoxicilina, claritromicina, levofloxacina y metronidazol, determinando su presencia en los genes pbp1A, rna23s y gyrA, aunque en algunos aislamientos no fue posible analizar las secuencias debido a su mala calidad (cuadro 3).
Las mutaciones relacionadas con resistencia a los antibióticos evaluados, fueron la E406V para la amoxicilina y la A2143G para la claritromicina. Para el metronidazol, se detectó la mutación R90K en la proteína rdxA y, para la levofloxacina, se detectaron tres cambios diferentes en la proteína gyrA en las posiciones 87 y 91, así: N87I, N87K y D91N (cuadro 3).
Se detectó resistencia múltiple en ocho de los diez aislamientos. Dos de ellos (uno de antro y otro de cuerpo provenientes del mismo paciente) presentaron resistencia a dos antibióticos: levofloxacina y metronidazol. Cuatro aislamientos de antro y cuerpo de dos pacientes diferentes, fueron resistentes a tres antibióticos: claritromicina, levofloxacina y metronidazol. Dos aislamientos de otro paciente presentaron resistencia a los cuatro antibióticos: amoxicilina, claritromicina, levofloxacina y metronidazol (cuadro 3). Además, se detectaron diferencias entre los aislamientos de antro y de cuerpo, en los patrones de resistencia en cuatro pacientes, lo que indicaría una infección concomitante con diferentes cepas de H. pylori (cuadro 3).
Discusión
Estos resultados evidencian que en Bogotá se registraron aislamientos de H. pylori con resistencia a múltiples antibióticos después de tres años (2012- 2015) de vigilancia. La aparición de multirresistencia podría ser la consecuencia de tratamientos previos fallidos, o de resistencia ‘secundaria’ a claritromicina, metronidazol y levofloxacina debida al uso extensivo de estos antibióticos en el tratamiento de enfermedades respiratorias, parasitarias o ginecológicas, y de infecciones urinarias, respectivamente 34,35. Estos hallazgos implicarían que dichos antibióticos no deberían utilizarse empíricamente en los tratamientos de primera línea, por lo menos en Bogotá. Resultados similares se han registrado en otros países como Alemania, Bulgaria, Polonia, Brasil, Israel, China y Corea 23,24,36-38, en donde se encontraron resistencias secundarias múltiples a claritromicina, metronidazol y levofloxacina, con una mayor prevalencia de resistencia a claritromicina y metronidazol.
Los aislamientos del cuerpo y del antro presentaron diferentes patrones de sensibilidad, lo cual también se ha reportado previamente en Corea, China y Alemania 39-41. Este hallazgo explicaría el fracaso del tratamiento y no solo indicaría la presencia de más de una cepa de H. pylori, sino que subrayaría la importancia metodológica de hacer la prueba de sensibilidad a partir de muestras de ambos segmentos del estómago, con el fin de orientar mejor la elección del tratamiento y evitar recaídas de infección por H. pylori.
Los resultados del presente estudio resaltan la importancia de implementar pruebas de sensibilidad, ya sea mediante cultivo o biología molecular, antes de un primer tratamiento para H. pylori, con el fin de orientar las terapias de primera línea, lo que concuerda con lo reportado por Park, et al. 38, en cuyo estudio se evidencia que, cuando el tratamiento de primera línea se administra teniendo en cuenta la sensibilidad a los antibióticos, la eficacia es superior a la del tratamiento empírico. Con la prueba previa de sensibilidad, la eficacia fue de 94,7 % (IC95% 88,8-100) y por protocolo fue de 96,4 % (IC95% 91,5-100) y, con el tratamiento empírico fueron de 71,9 % (IC95% 60,2-83,5) y 73,2 % (IC95% 61,5-84,8), respectivamente 38. No obstante, cuando ya han fallado varios tratamientos, la utilidad del cultivo no es la misma debido a la aparición de resistencia, lo cual se suma a que el cultivo y las pruebas moleculares no están disponibles en todas partes y, por lo tanto, lo usual es recurrir a tratamientos empíricos 42, caso en que debería tenerse en cuenta la epidemiología de la resistencia local.
En Colombia, el tratamiento triple de primera línea se utilizó en el pasado, combinando amoxicilina, claritromicina o metronidazol o levofloxacina 43. Sin embargo, en otros estudios, se ha demostrado frecuentemente resistencia primaria a estos antibióticos 22, por lo cual ya no se recomiendan como una primera opción en el país 43.
El tratamiento empírico con estos antibióticos, sin tener en cuenta la epidemiologia local, con frecuencia conduce al fracaso terapéutico 38. Además, cuando hay resistencia a la claritromicina y al metronidazol, los tratamientos híbridos y concomitantes con esos antibióticos fracasan frecuentemente 35. En estos casos, la eficacia de los tratamientos triple, secuencial y concomitante, es de ~50 %, <20 % y 50 %, respectivamente 44. En el informe del consenso Maastricht V/Florence (Maastricht V/Florence Consensus Report) se recomienda no usar tratamientos secuenciales ni concomitantes, sino cuádruples durante 14 días, en casos de resistencia simultánea a la claritromicina y el metronidazol superior al 15 % 4,43; entre estos, están el que combina bismuto y furazolidona, más metronidazol o amoxicilina-tetraciclina, o los tratamientos con rifabutina (tratamiento de rescate), que también son una alternativa 45. La combinación de amoxicilina y tetraciclina tiene, generalmente, muy baja tasa de éxito: 62 a 75 % por intención de tratar y 64 a 80 % por protocolo.
Su escaso éxito podría deberse a la interacción del efecto bacteriostático de la tetraciclina con el efecto bactericida de la amoxicilina 45. La acción bactericida de la amoxicilina implica la inhibición de la formación de la pared celular, que depende de la tasa de proliferación de las bacterias 45. Los antibióticos bacteriostáticos, como la tetraciclina, pueden reducir la efectividad de la penicilina mediante la inhibición de la síntesis de proteínas celulares requerida para la división celular 45.
Se han explorado nuevas opciones terapéuticas para los casos de multirresistencia, entre ellas, el linezolid 46 y los nuevos análogos de las oxazolidionas 47. No obstante, el inconveniente es que el linezolid podría inducir la aparición de Staphylococcus spp. resistentes a su acción, por lo que la decisión sobre su uso requiere de más estudios 46,47.
Los resultados del presente estudio evidenciaron la presencia de H. pylori con resistencia a múltiples antibióticos utilizados en los tratamientos empíricos, principalmente a claritromicina, levofloxacina y metronidazol, en ocho de diez aislamientos. Las pruebas de sensibilidad a los antibióticos deben hacerse, preferiblemente, antes de administrar el primer tratamiento para H. pylori, en cuyo caso se sugiere hacerlas con muestras provenientes del antro y del cuerpo del estómago, para evitar que se pierdan aislamientos resistentes a los antibióticos en ambos sitios. Cuando hay H. pylori con resistencia a múltiples antibióticos, las opciones terapéuticas se restringen a los tratamientos cuádruples de rescate con furazolidona o rifabutina.