Helicobacter pylori es un bacilo Gram negativo que pertenece a la familia Helicobacteraceae y existe en dos formas, espiral y cocoide; tiene múltiples flagelos unipolares y codifica enzimas como catalasa, oxidasa y ureasa, las cuales le permiten adaptarse a las condiciones del epitelio gástrico. Es una bacteria microaerófila y exigente en cuanto a la nutrición, lo que dificulta su cultivo in vitro1,2.
En la mayoría de los casos, H. pylori se adquiere en el entorno familiar por vía oral-oral y fecal-oral durante los primeros cinco años de vida 3. Se estima que el 50 % de la población mundial está infectado con el microorganismo. En los países en desarrollo, la prevalencia de la infección en la población general varía entre 60 y 90 %, mientras que en los países desarrollados está entre el 25 y el 50 %, lo que refleja la importancia de los factores socioeconómicos como determinantes de su prevalencia 4,5.
Helicobacter pylori coloniza de forma selectiva el epitelio gástrico. En la mayoría de las personas la infección es asintomática; sin embargo, puede desencadenar una inflamación crónica y producir una úlcera péptica en cerca del 10 % de las personas. Dos neoplasias se han asociado con la infección por H. pylori, el linfoma del tejido linfoide asociado con la mucosa (Mucosa- Associated Lymphoid Tissue, MALT), que se presenta en el 0,1 % de los casos, y el adenocarcinoma gástrico, presente en 1 a 3 % de los pacientes, y que constituye la quinta neoplasia más frecuente en el mundo 6-8.
El tratamiento para erradicar la infección por H. pylori previene el desarrollo de afecciones gastroduodenales. El esquema recomendado a nivel mundial incluye un inhibidor de la bomba de protones y dos tipos de antimicrobianos. En el tratamiento de primera línea, se utilizan con mayor frecuencia la amoxicilina y la claritromicina, y en el de segunda línea, se emplea la amoxicilina y la levofloxacina. Sin embargo, la gran resistencia a estos antibióticos ha dificultado la erradicación de la infección 9.
El principal mecanismo de resistencia de H. pylori a la claritromicina es la disminución de la afinidad del macrólido con la subunidad 50S del ribosoma bacteriano debida a mutaciones puntuales en nucleótidos adyacentes al dominio V del gen ARNr 23S 10,11. Las mutaciones más comunes se han reportado en las posiciones 2142 y 2143, con sustituciones de adenina por guanina (G) (A2142G y A2143G) o de A por citosina (C) (A2142C); también, se ha registrado la sustitución A2144G, aunque con menor frecuencia 12-15. El desarrollo de la resistencia se ha visto favorecido por el incumplimiento del tratamiento y el uso de macrólidos en el tratamiento de otras infecciones, principalmente de las vías respiratorias 16.
Colombia cuenta con un único estudio que evalúa la prevalencia global de infección por H. pylori. Bravo, et al., informaron de una prevalencia de 69,1 % en 8.652 informes histopatológicos de endoscopias de pacientes con manifestaciones dispépticas en 16 hospitales regionales de las zonas suroccidental, norte, centro, centro-occidental y oriental del país, la cual varió entre las regiones en un rango entre 41,7 y 99,1 % 17.
A nivel nacional, la elevada prevalencia de infección se correlaciona con la incidencia, también elevada, del adenocarcinoma gástrico, que entre 2010 y 2014 fue la primera causa de muerte por cáncer en hombres y la tercera en mujeres, con tasas de mortalidad de 16,84 y 8,73 por 100.000 habitantes, respectivamente 18,19.
La infección por H. pylori tienen un gran impacto en la vida de los individuos afectados y en el sistema de salud, por lo cual son muy importantes su diagnóstico y tratamiento 20. Los esquemas terapéuticos deben sustentarse en estudios de la resistencia a nivel local por la heterogeneidad de su presentación en diferentes poblaciones. No obstante, en Colombia se siguen prescribiendo tratamientos basados en estudios de otros países 21 y, además, esquemas que han perdido eficacia debido a la resistencia bacteriana, aunque los más utilizados sean mencionados en el consenso de Maastricht 9,22.
En un metaanálisis de la resistencia de H. pylori en Latinoamérica, Camargo, et al., señalan una resistencia a la claritromicina del 18 % (rango: entre 2,7 y 60 %) en Colombia, según estudios en Bogotá, Pereira, Armenia, Tumaco, Túquerres y Manizales, lo que sugiere que es uno de los países con mayor prevalencia de resistencia en Latinoamérica 23. Ello implica que debe evaluarse la sensibilidad de la bacteria a otros antibióticos, con el fin de establecer esquemas de primera línea y de rescate, dado que los antibióticos útiles para tratar la infección son pocos 21,24.
La resistencia de H. pylori a la claritromicina se ha comprobado en los estudios de efectividad del tratamiento antibiótico. Castaño, et al., por ejemplo, compararon dos esquemas de tratamiento estándar con claritromicina, uno de 7 días y otro de 10, administrados a pacientes de Medellín; encontraron que las tasas de erradicación en el análisis por intención de tratar, determinadas mediante una prueba de aliento optimizada, fueron de 67,8 y 74,3 %, respectivamente, sin diferencias estadísticamente significativas; estos porcentajes están por debajo de los niveles óptimos de erradicación 25. Posteriormente, los mismos autores compararon el tratamiento estándar con claritromicina y uno con levofloxacina durante 10 días, y determinaron una diferencia significativa en la eficacia de los dos esquemas a favor de la levofloxacina 26.
En Medellín, la prevalencia de infección por H. pylori en pacientes sintomáticos oscila entre 36,4 y 65 %; sin embargo, se desconoce la prevalencia de la resistencia de esta bacteria a la claritromicina 17,27.
El objetivo de este estudio fue determinar la frecuencia de las mutaciones puntuales A2143G y A2142G del gen ARNr 23S asociadas con la resistencia de H. pylori a la claritromicina en biopsias gástricas de pacientes con manifestaciones dispépticas atendidos en una unidad de endoscopia de Medellín.
Materiales y métodos
Población de estudio
Se hizo un estudio descriptivo transversal entre pacientes mayores de 18 años con manifestaciones dispépticas atendidos en una unidad de endoscopia de la IPS SURA (Sede Industriales de Medellín) entre febrero de 2016 y abril de 2017. Esta unidad de atención es de segundo nivel de complejidad y atiende mayoritariamente a pacientes de estratos 4 a 6 que cuentan con plan de medicina prepagada.
Se calculó un tamaño de muestra de 175 pacientes con base en una prevalencia de resistencia estimada del 16 % para la población colombiana, con un intervalo de confianza del 95 % y un error del 5 %.
Los pacientes incluidos en el estudio no habían recibido tratamiento con inhibidores de la bomba de protones o antibióticos en el mes previo a la toma de la muestra. Se excluyeron aquellos con diagnóstico de cáncer, enfermedad terminal o gastrectomías previas.
A cada uno de los pacientes se le explicó el estudio y, en caso de que aceptara participar, se solicitaba la firma del consentimiento informado.
Se tomaron cinco biopsias gástricas para el estudio histopatológico siguiendo el protocolo de Sidney: las curvaturas mayor y menor del antro y del cuerpo, y la incisura angular 28; se tomó una biopsia adicional del antro para el estudio molecular, por ser esta la región del estómago con mayor densidad bacteriana en casos de infección H. pylori29. La muestra del antro se colocó en un tubo plástico estéril de 1,5 ml con caldo de infusión cerebro-corazón (BHI, BD, Sparks, MD, USA) y glicerol al 20 %. Las muestras se refrigeraron a 4 °C y se transportaron hasta el Laboratorio de Gastrohepatología de la Universidad de Antioquia el mismo día del procedimiento endoscópico, donde se conservaron a -70 °C hasta su procesamiento.
Consideraciones éticas
El proyecto fue aprobado por el Comité de Bioética de Investigación en Humanos de la Sede de Investigación Universitaria de la Universidad de Antioquia.
Extracción de ADN y amplificación por PCR
Todas las biopsias obtenidas se sometieron a análisis histopatológico y se tiñeron con la coloración estándar de hematoxilina y eosina. El análisis del 70 % de las muestras, aproximadamente, estuvo a cargo de un patólogo especializado en el tubo digestivo y el 30 % restante de las muestras fue evaluado por otros patólogos.
De las muestras positivas para H. pylori según el informe de histopatología, se obtuvo ADN total utilizando un estuche comercial (QIAamp DNA Mini Kit™, Qiagen, Alemania) y siguiendo las instrucciones del fabricante. El material obtenido se cuantificó (NanoDrop 2000™, Thermo Scientific, USA).
Para determinar la presencia de la bacteria en la muestra, se amplificaron las regiones s y m del gen vacA utilizando los cebadores descritos por Acosta, et al., vacA s: F 5’-ATGGAAATACAACAAACACAC-3’ y R 5’-CTGCTTGAATGCGCCAAAC-3’; vacA m: F ’-CAATCTGTCCAATCAAGCGAG-3’ y R 5’-GCGTCTAAATAATTCCAAGG-3’ 30. En aquellas muestras positivas para las regiones s y m del gen vacA, se amplificó una región del gen ARNr 23S para determinar la presencia de mutaciones, empleando los cebadores descritos previamente por Occhialini, et al.: K1 5’-CCACAGCGATGTGGTCTCAG-3’ y K2 5’-CTCCATAAGAGCCAAAGCCC-3’ 31.
Cada una de las tres reacciones de PCR se llevaron a cabo en un volumen final de 25 μl que contenía 1X de tampón, 1,5 mM de MgCl2, 0,2 mM de dNTP, 0,4 mM de cada cebador, 2,5 U del reactivo Biolasa™ (Bioline, Reino Unido) y 2 μl del ADN extraído.
En un termociclador (C1000 Touch™, Thermal Cycler) se programó un ciclo inicial de desnaturalización a 94 °C durante dos minutos seguido de 35 ciclos de amplificación con variaciones según la región de interés: a 94 °C durante un minuto, a 55 °C durante un minuto y a 72 °C durante un minuto para la región s del vacA; a 94 °C durante un minuto, a 52 °C durante un minuto y a 72 °C durante un minuto para la región m del gen vacA, y a 94 °C durante 30 segundos, a 60 °C durante 30 segundos y a 72 °C durante 30 segundos para la región ARNr 23S, con un ciclo final a 72 °C durante cinco minutos.
Los productos de amplificación se sometieron a electroforesis en geles de agarosa al 2,5 % teñidos con bromuro de etidio. Para la estandarización de las PCR, se utilizó ADN genómico de la cepa NCTC 11637 de H. pylori como control positivo (gentilmente donado por Dra. Alba Alicia Trespalacios, profesora de la Pontificia Universidad Javeriana) y, como control negativo se usó agua destilada estéril. Es de anotar que la cepa NCTC 11637 no presenta mutaciones en los nucleótidos 2142 y 2143 y, por lo tanto, no se incluyó en los ensayos de RFLP.
En la técnica de polimorfismos de longitud de fragmentos de restricción (Restriction Fragment Length Polymorphism, RFLP) se utilizaron las enzimas de restricción BbsI y BsaI (New England Biolabs, USA) y 3,5 μl del producto de PCR del gen ARNr 23S en un volumen final de 20 μl, según las instrucciones del fabricante.
Cuando la mutación A2142G está presente, la digestión de los amplicones de la región de la peptidiltransferasa del gen ARNr 23S con la enzima BbsI genera dos fragmentos de 93 y 332 pares de bases, aproximadamente, en tanto que, con la enzima BsaI (A2143G), produce tres fragmentos de 20, 300 y 105 pares de bases (GenBank U27270).
Como control positivo de los RFLP se incluyeron amplicones del fragmento de lectura abierta (Open Reading Frame, ORF) Core del virus de la hepatitis B (HBV) obtenidos a partir del plásmido pJET-TH24_1,5 (GenBank FJ589065) 32. La amplificación de esta región genera un fragmento de 748 pb, aproximadamente, que fue sometido a digestión con la enzima BbsI para generar dos fragmentos de 608 y 140 pb, y con la enzima BsaI, para producir dos fragmentos de 508 y 240 pb. Esta secuencia fue seleccionada mediante mapeo de restricción utilizando la herramienta BioEdit, versión 7.1.9. Los amplicones del ORF Core del HBV (nucleótidos 1098 a 1846) se incluyeron en todos los ensayos de RFLP (figura 1).
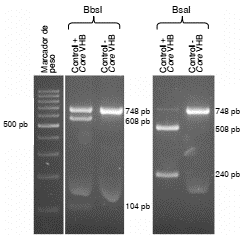
Figura 1 Electroforesis de ADN en gel de agarosa al 2,5 % teñido con bromuro de etidio. Se observan las bandas obtenidas mediante la técnica de RFLP con el control positivo del ORF Core del HBV.
Se seleccionaron al azar 18 (12,5 %) muestras positivas en el estudio de histopatología para evaluar la reproducibilidad de los resultados de PCR de las tres regiones (vacA s y m y ARNr 23S) y del método de RFLP.
Además, 10 biopsias con resultado negativo en el estudio de histopatología se analizaron mediante PCR para verificar la presencia de vacA y del gen ARNr 23S, y aquellas con resultado positivo en dicho estudio, pero que no amplificaron para vacA s y m y ARNr 23S, se analizaron mediante PCR con el fin de verificar la integridad del genoma, utilizando los siguientes cebadores para el gen de la gliceraldehído-3-fosfato deshidrogenasa (GAPDH): 5’-CCTTCATTGACCTCAACTACATGG-3’ y 5’-AGTCTTCTGGGTGGCAGTGATGG-3.
Resultados
Durante el periodo comprendido entre febrero de 2016 y abril de 2017, 588 pacientes remitidos para endoscopia por síntomas dispépticos acudieron a la IPS Sura de Medellín. Se invitó a participar en el estudio a 403 (68,5 %) de estos pacientes, y 396 de ellos aceptaron; siete se excluyeron por presentar antecedentes de cáncer gástrico o consumo de antibióticos, o por no estar de acuerdo con el estudio. El 59 % (234/396) de los participantes en el estudio eran mujeres, el rango de edad fue de 18 a 86 años, con un promedio de 48 (±14) años.
De los 396 casos incluidos en el estudio, 175 fueron positivos para la infección por H. pylori según la evaluación histopatológica con hematoxilina y eosina, es decir, una prevalencia de infección del 44,2 % (175/396). Es de anotar que no se utilizaron otros métodos de diagnóstico, como la prueba rápida de la ureasa y la de aliento con urea, pues no son exámenes de rutina en casos de dispepsia en esta unidad de atención.
De las 175 muestras positivas para H. pylori por histopatología, en 143 (82 %) se obtuvo la amplificación de las regiones s y m del gen vacA y de la región del gen ARNr 23S (figura 2). El gen vacA es un factor de virulencia que se encuentra presente en todas las cepas de H. pylori y su utilidad para demostrar esta bacteria en muestras biológicas es comparable con la de otros genes bacterianos, como ureA, ureC, ARNr 16S, ARN 23S y Hsp60 30,33,34.
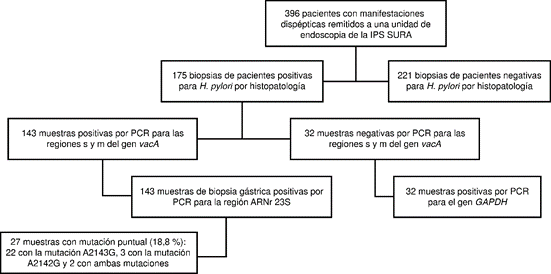
Figura 2 Diagrama de los procedimientos y resultados obtenidos para las biopsias gástricas procesadas en el estudio
En 27 de 143 (18,8%) muestras analizadas, el método de RFLP permitió identificar las mutaciones puntuales A2143G y A2142G en el gen ARNr 23S de H. pylori (figura 2 y 3).
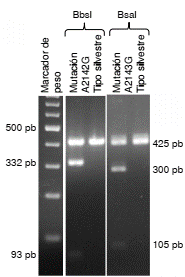
Figura 3 Electroforesis de ADN en gel de agarosa al 2,5 % teñido con bromuro de etidio. Se observan las bandas obtenidas con la técnica de RFLP en presencia de las mutaciones A2143G y A2142G
Las sustituciones más frecuentes fueron la A2143G en 22 de 27 muestras (81,5 %) y la A2142G en 3 de 27 (11 %); en 2 de 27 (7,4 %) de las muestras se encontraron simultáneamente estas dos mutaciones (figura 2). En el cuadro 1 se presentan las variables demográficas de los pacientes con muestras positivas para la infección por H. pylori detectada mediante PCR.
Cuadro 1 Variables demográficas de pacientes con manifestaciones dispépticas y muestras positivas para la infección por H. pylori mediante PCR atendidos en una unidad de endoscopias de Medellín
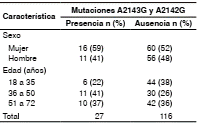
La información de seguimiento de los pacientes no estaba disponible, por lo tanto, no se logró establecer si había correlación entre la presencia de mutaciones y la resistencia al tratamiento.
El 81 % (116/143) de las muestras analizadas en las que se logró la amplificación de las secuencias del genoma bacteriano, no presentaba las mutaciones puntuales asociadas con la resistencia a la claritromicina. Las 18 muestras analizadas por duplicado mediante PCR para las tres regiones del genoma bacteriano, vacA s y m y ARNr 23S, y mediante el método de RFLP, demostraron una reproducibilidad del 100 %. Asimismo, en ninguna de las 10 muestras negativas para infección por H. pylori en el estudio histopatológico, se logró amplificar las regiones de interés del genoma bacteriano (no se incluyen los datos).
Discusión
Según el informe de histopatología, la prevalencia de infección por H. pylori en la población de estudio atendida en la unidad de estudio de Medellín entre el 2016 y el 2017, fue de 44,2 %. Esta prevalencia es similar a la descrita en un estudio retrospectivo en muestras obtenidas de pacientes con síntomas dispépticos atendidos en la Clínica Diagnóstica VID de Medellín entre el 2012 y el 2013 (36,4 %; 986/2708), estudio en que el diagnóstico de infección por H. pylori también se determinó por histopatología 27.
Sin embargo, los resultados de ambos estudios muestran una disminución de la prevalencia de infección por H. pylori, en comparación con la prevalencia de 65 % según los informes de histopatología de 494 pacientes de un hospital público de Medellín en 1997 17. Esta disminución obedecería al mejoramiento de las condiciones sanitarias de la población en las últimas dos décadas, así como al acceso a agua potable y la recolección y el tratamiento de aguas residuales 5.
Dadas las dificultades técnicas que implica el aislamiento de la bacteria, el estudio de histopatología se considera una de las técnicas de referencia para el diagnóstico de la infección por H. pylori1. Para garantizar la menor probabilidad de falsos negativos, se debe hacer un muestreo estandarizado, como el descrito en el protocolo de Sidney; además, se debe restringir el consumo de antibióticos e inhibidores de la bomba de protones por lo menos 30 días antes de la endoscopia 35, con el fin de obtener una mejor sensibilidad y especificidad diagnóstica en comparación con otros métodos directos, como la prueba rápida de la ureasa, la prueba de aliento con urea o la PCR 36,37.
Al considerar las dificultades técnicas para el cultivo de la bacteria, la amplificación mediante PCR a partir de la biopsia gástrica es una metodología que permite la detección de secuencias de H. pylori; la sensibilidad de la PCR para H. pylori se calcula en 20 copias del gen de interés por muestra 38.
Para determinar la resistencia de H. pylori a la claritromicina, se han implementado diversas técnicas, como la difusión en agar, la prueba de epsilometría, la hibridación fluorescente in situ (Fluorescent in situ Hybridization, FISH), la RFLP y la PCR en tiempo real 15. Se ha descrito una correlación entre la detección de las mutaciones puntuales A2142G y A2143G, y la resistencia a la claritrominicina determinada por el aumento de la concentración inhibitoria mínima (CIM) en el cultivo al utilizar la técnica de dilución en agar 12,13,39. El método de RFLP es más rápido y económico que otros métodos cualitativos, como la secuenciación y el FISH, y tiene una especificidad de 88 %, una sensibilidad de 85 %, un valor predictivo positivo de 54 % y un valor predictivo negativo de 97 % comparado con el cultivo 13. Por ello, esta técnica representa una excelente opción frente a la dificultad técnica del aislamiento de H. pylori40-42. Además, la técnica de RFLP puede emplearse en biopsias embebidas en parafina 43.
En el presente estudio, la resistencia genotípica de H. pylori a la claritromicina en la población fue de 18,8 %. Este porcentaje es similar al registrado en pacientes atendidos en instituciones de salud en otras ciudades de Colombia. Trespalacios, et al., reportaron una resistencia de 13,6 % en 256 muestras de pacientes atendidos en un hospital de Bogotá entre el 2009 y el 2011. La resistencia se evaluó utilizando la técnica de dilución en agar y se confirmó por secuenciación de la región del ARNr 23S bacteriano, encontrándose, por lo menos, una de las tres mutaciones (A2143G, A2142G o A2142C) en las muestras que presentaban un aumento de la CIM 14. En estudios previos, se reportó una resistencia de 15 a 17,7 % en muestras de pacientes dispépticos atendidos en instituciones de salud de Bogotá entre el 2007 y el 2009, según los resultados obtenidos mediante la prueba de epsilometría y el método de difusión en disco, respectivamente 16,44.
En un estudio reciente en un hospital en Tumaco (suroccidente de Colombia), se determinó una resistencia de 19,8 % mediante la técnica de dilución en agar en 203 muestras analizadas 45. Sin embargo, en otros dos estudios en Bogotá se han descrito frecuencias más altas de resistencia a la claritromicina. En el primero de estos, en 115 pacientes de una unidad de gastroenterología durante el 2008, se determinó una frecuencia de resistencia primaria de 60 % mediante la técnica de difusión en disco, lo que representa el dato de resistencia más alto de H. pylori a claritromicina descrito hasta el momento en el país 46. En un segundo estudio, se detectó resistencia a claritromicina en el 39,2 % (42/107) de las muestras mediante la técnica de dilución en agar. La resistencia fenotípica fue confirmada por PCR de alelo específico (Allele-Specific Polymerase Chain Reaction) en todos los aislamientos resistentes y se identificaron las mutaciones A2143G o A2142G 12.
Por otra parte, la resistencia a este macrólido ha estado por debajo del 5 % en pacientes de otras ciudades del país. En la región del eje cafetero (Pereira, Armenia y Manizales), se describió una prevalencia de 3,8 % en 106 muestras analizadas por la técnica de la prueba de epsilometría, de las cuales, tres presentaban la mutación A2143G y, una, la sustitución A2142G en el análisis mediante PCR-RFLP 47,48.
Acosta, et al., evaluaron la frecuencia de mutaciones asociadas con la resistencia por secuenciación directa en 162 muestras de pacientes atendidos en un hospital de Popayán (suroccidente de Colombia); el 4,3 % (7/162) de las muestras presentaba mutación y de estas, cuatro correspondían a la sustitución A2143G y tres a A2142G 30. Bustamante, et al., encontraron el porcentaje más bajo de resistencia de H. pylori a la claritromicina reportado en el país, 2,7 %; este estudio se hizo en 149 aislamientos obtenidos de 206 pacientes atendidos en un hospital en Túquerres (suroccidente de Colombia) mediante la técnica de dilución en agar 49. En estos estudios, la mutación puntual más frecuente fue la A2143G, lo que coincide con los hallazgos del presente estudio (81,5 %).
En 116 de las 143 muestras analizadas en otro estudio, no se detectaron mutaciones puntuales; sin embargo, esto no excluye la resistencia bacteriana a este macrólido debida a otras mutaciones u otros mecanismos, como las bombas de eflujo 39.
En 32 de las 175 biopsias gástricas positivas por histopatología en el presente estudio, no se logró amplificar ninguna de las tres regiones del genoma bacteriano (regiones s y m del gen vacA y una región del gen ARNr 23S) mediante PCR y debe anotarse que se descartaron inhibidores de la PCR mediante la amplificación del gen GAPDH (no se incluyen los datos). La discordancia entre el resultado de histopatología y el de la PCR se explicaría por la diferencia entre el número de muestras analizadas por histopatología (cinco biopsias provenientes de diferentes regiones del estómago) y por PCR (una biopsia del antro), lo que aumentó la probabilidad de detectar los bacilos mediante histopatología.
Según el metaanálisis de Camargo, et al., en estudios realizados en Argentina, Cuba, Ecuador, Brasil, Chile, Colombia, Costa Rica, México, Perú y Venezuela, la prevalencia de resistencia de H. pylori a este macrólido presentó un rango de 6 a 18 %, y Colombia fue el país con el porcentaje más alto (18 %) 23. En estudios más recientes, Squarcio, et al., registraron un porcentaje de resistencia del 16,9 % (83/490) mediante métodos moleculares en diferentes regiones de Brasil; la mutación A2143G se encontró en el 90,4 % de los aislamientos resistentes 50. En Argentina, Zerbetto, et al., encontraron una prevalencia de la resistencia del 26,9 %, y la mutación más frecuente fue la A2143G, presente en el 89,4 % de los aislamientos resistentes 51. En México se determinó un porcentaje de resistencia del 17,8 % mediante la técnica de difusión en disco en 45 muestras y se detectó la mutación A2143G en 1 de 8 aislamientos con resistencia fenotípica 52.
Cuando el porcentaje de resistencia de H. pylori a la claritromicina en una población es mayor de 15 %, el tratamiento de segunda línea es el cuádruple con bismuto o el de levofloxacina 9,23. Sin embargo, en un estudio reciente de Trespalacios, et al., en Bogotá, se evidenció un aumento considerable de los aislamientos resistentes a la levofloxacina entre 2009 (11,8 %) el 2014 (27,3 %) 53. Es de gran importancia, entonces, determinar la resistencia de H. pylori a los antibióticos, con el fin de establecer esquemas terapéuticos basados en los resultados en la población colombiana para, así, disminuir la incidencia de enfermedades gastrointestinales asociadas con la infección 54.
Este es el primer estudio en el cual se describe el estado de la resistencia de H. pylori a la claritromicina en pacientes atendidos en Medellín; se encontró una gran prevalencia de mutaciones puntuales asociadas con dicha resistencia. Dado este hallazgo, habría que prever altos niveles de fracaso en la erradicación del microrganismo con el tratamiento triple que incluye este macrólido.
Las limitaciones del presente estudio incluyeron la falta de información sobre el consumo previo de antibióticos para tratar la infección por H. pylori, por lo que no se pudo diferenciar la resistencia primaria de la secundaria, así como de los detalles del informe de histopatología, tales como la carga bacteriana y el estado del epitelio gástrico; además, los participantes del estudio fueron atendidos en una sola unidad de endoscopia de la ciudad.
Se requieren más estudios para determinar la resistencia de la bacteria a los antibióticos en la población colombiana, y establecer los tratamientos de primera línea y de rescate.














