En los estudios previos sobre las alteraciones celulares y tisulares que ocurren en el estómago durante la inflamación crónica de la mucosa gástrica a causa de la infección por Helicobacter pylori, se han podido detectar reacciones fisiológicas diferentes según su localización en el estómago y el riesgo de desarrollar cáncer 1-4. Cuando la gastritis predomina en la región del antro, la producción de ácido gástrico (hiperclorhidria) y el riesgo de presentar úlcera duodenal se incrementan, efecto que protege contra el desarrollo de cáncer. Por el contrario, cuando la gastritis predomina en el cuerpo del estómago, la producción de ácido gástrico disminuye (hipoclorhidria) y puede generar una gastritis atrófica, lo cual aumenta el riesgo de cáncer gástrico 3,4.
Asimismo, este desequilibrio gástrico facilita la colonización sectorizada de bacterias que no crecen en un estómago normal 5. La influencia de las bacterias nitrificantes y de otras especies en la disbacteriosis (alteración de la microbiota bacteriana habitual) contribuye a la producción de especies reactivas del nitrógeno (Reactive Nitrogen Species, RNS) y especies reactivas del oxígeno (Reactive Oxygen Species, ROS), las que, conjuntamente con la infección por H. pylori, podrían ser determinantes en la patogénesis 6-8.
El surgimiento de los métodos moleculares de secuenciación ha abierto el camino para entender la dinámica de la población bacteriana, así como cada evento secuencial en la aparición y en el desarrollo del cáncer gástrico de tipo intestinal descritos en el modelo general de Correa 7-10.
La hipótesis del presente estudio propone que, independientemente del lugar de origen y del riesgo poblacional de cáncer gástrico, los individuos con enfermedad gástrica presentan un patrón diferencial de la microbiota asociada con el sitio anatómico y que determina diferentes reacciones inflamatorias.
En este estudio, se comparó la microbiota del antro y del cuerpo gástrico de individuos con gastritis no atrófica con la de aquellos con gastritis atrófica y metaplasia, todos provenientes de dos poblaciones (Túquerres y Tumaco) del departamento de Nariño (Colombia), en la cuales la prevalencia de la infección por H. pylori es similar pero el riesgo poblacional de cáncer gástrico es diferente.
Túquerres es un municipio ubicado a 3.070 msnm en la cordillera de los Andes, donde el riesgo poblacional de cáncer gástrico es alto, con una tasa de incidencia de 150 por 100.000 habitantes. Tumaco es un municipio ubicado en la Costa Pacífica de Nariño, donde el riesgo poblacional de cáncer gástrico es bajo, con una tasa de incidencia de 6 por 100.000 habitantes 11. Las diferencias geográficas, ecológicas, económicas y culturales entre estas dos zonas, permiten determinar las tendencias generales de la variación de la microbiota gástrica en relación con las lesiones precursoras del cáncer gástrico.
Materiales y métodos
Pacientes y recolección de muestras
Se incluyeron 16 pacientes mayores de 40 años, 8 de Túquerres y 8 de Tumaco, todos con síntomas de dispepsia, a los cuales se les practicó una endoscopia digestiva para detectar lesiones premalignas o malignas de la mucosa gástrica ( cuadro 1).
Cuadro 1 Información demográfica de los individuos según procedencia, tipo de lesión gástrica, sitio anatómico del estómago e infección por Helicobacter pylori
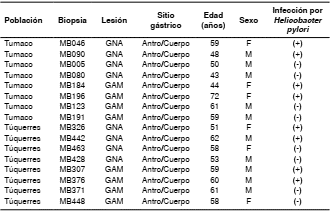
GNA: gastritis no atrófica; GAM: gastritis atrófica con metaplasia; F: femenino; M: masculino
Las biopsias utilizadas en este estudio se obtuvieron de un estudio previo del Registro Poblacional de Cáncer de Cali, dirigido por Luis E. Bravo, del Departamento de Patología de la Universidad del Valle, financiado por Colciencias (contrato RC No1106-408-20549). Se tomaron ocho biopsias de mucosa gástrica. Cuatro (dos del antro y dos del cuerpo) se usaron para el diagnóstico histopatológico de gastritis y su clasificación según el sistema de Sydney 12 y para la detección histológica de H. pylori mediante la tinción modificada de Giemsa; las otras cuatro (dos del antro y dos del cuerpo) se usaron para el aislamiento de H. pylori13 y la extracción de ADN total para la determinación de la microbiota gástrica. Estas últimas, para el estudio de la microbiota, se codificaron como (Bx1) cuando procedían de la curvatura menor del antro y como (Bx10) cuando procedían de la pared anterior media del cuerpo gástrico.
Extracción de ADN
Las biopsias gástricas preservadas a -70 °C se sometieron a un proceso de descongelación según los protocolos de la cadena de frío. Después de la maceración, se siguió el protocolo de extracción de ADN genómico del estuche QIAamp DNA Minikit™ (Qiagen). La calidad y la pureza del ADN se verificaron por espectrofotometría (NanoDrop ND 2000-Thermo Scientific™).
Secuenciación del gen 16S del ARN ribosómico y procesamiento de datos
La región V3-V4 del gen 16S del ARN ribosómico (ARNr) se amplificó por PCR usando los iniciadores 349 F (5´-CCTAGCGGNBGCASCAG-3´) y 805 R (5´-GACTACNVGGGTATCTAATCC-3´) 14. Los productos purificados se secuenciaron usando el estuche de reactivos MiSeq™ (2 x 300 pb) para lecturas emparejadas, según las pautas del fabricante, procedimiento que se hizo en el Laboratorio de Investigación Molecular MR DNA usando un pipeline estándar disponible en www.mrdnalab.com (Shallowater, TX, USA).
En las lecturas de las secuencias se removieron los códigos de barras y los cebadores, luego se eliminaron las secuencias cortas (<200 pb), así como las secuencias con bases ambiguas y las secuencias con ejecuciones de homopolímeros superiores a 6 pb. Las lecturas tamizadas se agruparon en unidades taxonómicas operativas con una divergencia del 3 % (97 % de similitud), seguidas por la eliminación de las secuencias de única instancia (singleton) y las quimeras 15,16.
Las unidades taxonómicas operativas finales se clasificaron taxonómicamente utilizando la herramienta BLASTn y se compararon con una base de datos curada derivada de la de RDPII 17 y el National Center for Biotechnology Information (NCBI) (www.ncbi.nlm.nih.gov).
Los datos se compilaron en los niveles taxonómicos de abundancia bacteriana absoluta (recuentos) y abundancia relativa (porcentajes).
Análisis de la diversidad metagenómica
La riqueza y la abundancia de las poblaciones microbianas se evaluaron estimando la diversidad alfa de las especies observadas, los índices de Shannon y Simpson y el modelo no paramétrico Chao 1, con el programa Qiime™, versión 1.8.0 15,16,18.
La composición de la microbiota gástrica se representó en histogramas con los datos de abundancia relativa a nivel taxonómico de clase. Se determinó la composición general de la microbiota, calculando la abundancia promedio en cada sitio anatómico (cuerpo y antro gástrico) según el diagnóstico histopatológico de la lesión (gastritis no atrófica o gastritis atrófica con metaplasia) y la ausencia o presencia de H. pylori (Hp- o Hp+).
Las diferencias entre las poblaciones microbianas se evaluaron mediante el análisis de varianza multivariado con base en permutaciones (Permutational Multivariate Analysis of Variance, PERMANOVA) con 10.000 permutaciones 19. Las unidades taxonómicas operativas compartidas según sitio anatómico, tipo de lesión gástrica y estatus de infección por Helicobacter sp., se visualizaron mediante diagramas de Venn a nivel de género.
Se generaron diagramas de cajas y bigotes (box plot) para representar la biodiversidad específica evaluada mediante el índice de Shannon, según el tipo de gastritis, la presencia o ausencia de H. pylori y el sitio anatómico de la biopsia, y se usó la prueba U de Mann-Whitney.
La tendencia general de agrupamiento según la composición de la microbiota, se evaluó con el método de pares de grupos no ponderados con la media aritmética (Unweighted Pair Group Method with Arithmetic Mean, UPGMA) y un análisis de los componentes principales 20. Para todos los análisis se consideró significativo un valor de p menor de 0,05. Los análisis estadísticos se hicieron en el programa Past3™ 21.
Accesibilidad de datos
El set de datos de secuenciación del estudio se encuentra disponible en la plataforma BaseSpace de illumina™ con el nombre de SAM1-51 (https:// basespace.illumina.com/s/NiYmU5ZGwDEB).
Consideraciones éticas
Esta investigación contó con la aprobación del Comité de Ética Humana de la Universidad de Nariño. Todos los participantes firmaron un consentimiento informado.
Resultados
Análisis de secuencias de la microbiota gástrica
Se reportaron 32 microbiotas del antro y del cuerpo gástrico de 16 individuos mayores de 40 años provenientes de Túquerres (alto riesgo de cáncer gástrico) y de Tumaco (bajo riesgo de cáncer gástrico), con el set de iniciadores 341F/805R para el análisis de secuencias del dominio V3-V4 del gen 16S del ARN usando el estuche de illumina MiSeq™.
Se analizaron 1.890.135 lecturas y, después del estudio de calidad, se identificaron 1.655.195 secuencias dentro del dominio bacteria. Para el cálculo de los estimadores de diversidad del índice de Shannon, el índice de Simpson y el algoritmo de Chao 1, las muestras se estandarizaron por rarefacción en 15.658 secuencias y se iniciaron en 13.702 secuencias, tomando como punto de corte la muestra más baja (cuadro 2).
Cuadro 2 Datos de secuenciación y estimadores de diversidad
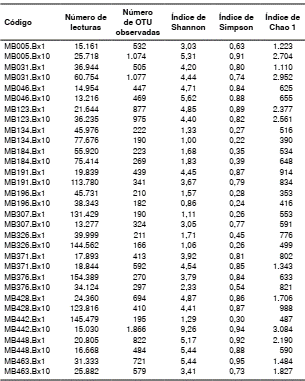
OTU: unidades taxonómicas operacionales
Las unidades taxonómicas operacionales se definieron con una divergencia del 3 % (97 % de similitud). Para calcular los estimadores de diversidad, las muestras se sometieron a rarefacción a 15.658 secuencias y se iniciaron en 13.702 secuencias.
Bx1: biopsia de curvatura menor del antro; Bx10: biopsia de pared anterior media del cuerpo
Diversidad metagenómica de la microbiota gástrica según la población en riesgo de cáncer gástrico
El análisis taxonómico de la microbiota del antro y del cuerpo gástrico de los individuos de Túquerres evidenció una representación de 14 filos, 25 clases, 34 órdenes, 56 familias y 160 géneros, en tanto que el análisis en los individuos de Tumaco arrojó 12 filos, 23 clases, 34 órdenes, 57 familias y 171 géneros. Se reportó un total de 14 filos, 26 clases, 35 órdenes, 59 familias y 182 géneros, de los cuales 36 tuvieron una abundancia mayor de 0,1 %.
En la composición de la microbiota de los individuos positivos para H. pylori, dominó la clase Epsilonproteobacteria en el antro y en el cuerpo gástrico, y en los individuos de Túquerres negativos para H. pylori y con diagnóstico de gastritis no atrófica dominó la clase Gammaproteobacteria. En los individuos de Tumano con diagnóstico de gastritis no atrófica y negativos para H. pylori, la clase Bacilli dominó en los perfiles microbianos del cuerpo gástrico (>60 %), en tanto que en el antro gástrico predominó la clase Betaproteobacteria (>70 %) (figura 1). Se encontraron diferencias significativas en el perfil de la microbiota acompañante de H. pylori según la población en riesgo de cáncer gástrico, independientemente del sitio topográfico del estómago (p<0,0001).
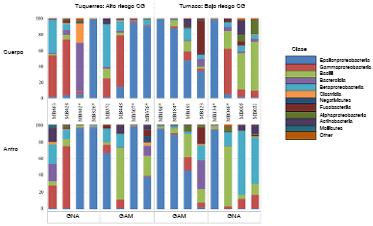
CG: cáncer gástrico; GNA: gastritis no atrófica; GAM: gastritis atrófica con metaplasia
Figura 1 Abundancia relativa de unidades taxonómicas operativas a nivel de clase de la microbiota gástrica según sitio anatómico (cuerpo o antro gástrico) de pacientes con alto y bajo riesgo de cáncer gástrico. Túquerres: alto riesgo de cáncer gástrico. Tumaco: bajo riesgo de cáncer gástrico. Se observó una mayor composición de clases en el cuerpo y el antro gástrico de los individuos libres de la infección por Helicobacter pylori. Por el contrario, la clase Epsilonproteobacteria predominó en las dos regiones gástricas de los individuos infectados (*), dependiendo del tipo de lesión.
Diversidad metagenómica según el diagnóstico histopatológico y el sitio anatómico
Los patrones de composición de los perfiles microbianos generales de las clases halladas en el antro y en el cuerpo gástrico de los individuos con diagnóstico de gastritis no atrófica de las dos poblaciones de estudio variaron. En el cuerpo gástrico de los individuos de Tumaco predominó la clase Bacilli, mientras que en el antro gástrico hubo una mayor proporción de la clase Betaproteobacteria, aunque sin diferencias significativas de composición entre las dos regiones anatómicas (p=0,56). En el cuerpo gástrico de los individuos de Túquerres predominó la clase Gammaproteobacteria, en tanto que en el antro predominó Epsilonproteobacteria; la comparación de los perfiles de la microbiota gástrica no evidenció diferencias significativas (p=0,91). En general, los perfiles microbianos del cuerpo y del antro gástrico en los individuos con gastritis no atrófica no fueron significativamente diferentes (p=0,62), sin embargo, la clase Epsilonproteobacteria, en la cual se clasifica H. pylori, solo estuvo presente en todos los perfiles en una proporción del 20 % (figura 1).
Los patrones de composición de los perfiles microbianos generales en el antro y en el cuerpo gástrico de los individuos con diagnóstico de gastritis no atrófica de las dos poblaciones de estudio fueron similares. La clase Epsilonproteobacteria, que incluye a H. pylori, se observó en ambos sitios anatómicos en una proporción de menos del 50 % en todos los perfiles, y los análisis comparativos de Tumaco, Túquerres y a nivel general no evidenciaron diferencias significativas (PERMANOVA: A p=0,91; B p=0,68; C p=0,66) (figura 1). Con respecto a los resultados globales, según el tipo de gastritis, se encontraron diferencias en la composición de la microbiota acompañante de H. pylori entre los individuos con gastritis no atrófica y aquellos con metaplasia intestinal (p≤0.05).
Diversidad metagenómica de la microbiota según la infección por Helicobacter pylori y el sitio anatómico
Los perfiles del cuerpo y del antro de los individuos sin la infección por H. pylori (H. pylori-) evidenciaron un patrón de composición variable representado por las clases Gammaproteobacteria, Betaproteobacteria y Bacilli; en los análisis no se encontraron diferencias significativas entre los sitios anatómicos en cada población ni a nivel general (PERMANOVA: A p=0,4; B p=0,34; C p=0,84) (figura 1).
Los perfiles microbianos de clase representativos para el antro y el cuerpo gástrico de individuos positivos para H. pylori de las dos poblaciones, presentaron patrones más homogéneos, con una alta proporción de Epsilonproteobacteria (>70 %). La comparación del cuerpo y del antro gástrico no evidenció diferencias significativas entre la composición microbiana del sitio anatómico y la general (PERMANOVA: A p=0,58; B p=0,48; C p=0,75) (figura 1).
Análisis comparativo de biopsias de la mucosa gástrica según la composición microbiana
El análisis de los componentes principales se hizo con los datos de abundancia relativa a nivel de familia. El 80 % de la varianza de los datos se explicó con base en los dos primeros componentes, lo que generó dos grupos principales. La tendencia del agrupamiento A se definió por la presencia de las familias Streptococcaceae y Pseudomonadaceae, con una carga del 70 % (PC2, componente principal 2), en tanto que el grupo B se definió por la presencia de la familia Helicobacteraceae con la infección por H. pylori en un 94 % (PC1, componente principal 1), independientemente del lugar de procedencia, del sitio anatómico y del tipo de lesión gástrica (figura 2).
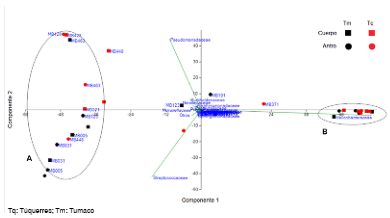
Figura 2 Análisis de componentes principales en función de la abundancia relativa de familias según sitio anatómico y población en riesgo de cáncer gástrico. Túquerres, alto riesgo de cáncer gástrico, en color rojo. Tumaco, bajo riesgo de cáncer gástrico, en color negro. Solamente los individuos negativos para Helicobacter pylori, según la tinción de Giemsa, se etiquetaron con el código de la muestra. En el cuerpo gástrico: microbiota marcada con cuadros; en el antro: microbiota marcada con círculos. En el análisis de componentes principales se diferenciaron dos grupos, independientemente del tipo de lesión gástrica. La variable que influyó para separar el primer grupo correspondió a la familia Helicobacteraceae, con una carga del 94 %, en tanto que la separación del grupo dos se hizo por la presencia de las familias Streptococcaceae y Pseudomonadaceae, presentes, sobre todo, en individuos sin la infección.
Discusión
En este estudio se determinó la diversidad metagenómica de la microbiota gástrica de individuos de dos poblaciones de Colombia ya conocidas por su riesgo opuesto para cáncer gástrico 5: Túquerres, con alto riesgo, y Tumaco, con bajo riesgo. La hipótesis de trabajo propuso que las respuestas fisiológicas frente a la gastritis diferenciada según el sitio anatómico del estómago, se deben a una disbacteriosis particularmente diferente. Se comparó la microbiota gástrica del antro (Bx1) y del cuerpo (Bx10) del estómago para determinar los patrones de composición asociados con el estado de la mucosa gástrica según el diagnóstico histológico y el estatus de infección por H. pylori.
La gastritis no atrófica es una reacción inflamatoria de la mucosa gástrica que se presenta con acumulación de linfocitos y células plasmáticas, y cuyo predominio según la región topográfica del estómago conduce a resultados clínicos y patológicos diferentes 22. Cuando la gastritis se presenta en el antro, la producción de ácido y la predisposición al desarrollo de úlcera duodenal aumenta, reduciéndose, así, el riesgo de cáncer gástrico, en tanto que la gastritis en el cuerpo del estómago disminuye la secreción de ácido y predispone al desarrollo del cáncer gástrico 23-26.
Atendiendo a dicho planteamiento, se compararon estos sitios anatómicos en individuos de Túquerres y de Tumaco. Se observaron patrones de composición microbiana variables y sin diferencias significativas. Se considera que H. pylori es la principal causa de la gastritis no atrófica 22, y en el análisis general la proporción de la clase Epsilonproteobacteria fue similar a la de otras unidades taxonómicas operativas. En Tumaco, Lactococcus lactis fue más representativa en el antro y Burkholderia mana, en el cuerpo gástrico, en tanto que en Túquerres las unidades taxonómicas operativas representativas del antro y del cuerpo correspondieron a Pseudomonas putida (figura 1). Más del 60 % de los géneros identificados se encontró tanto en el antro como en el cuerpo gástrico (figura 3).
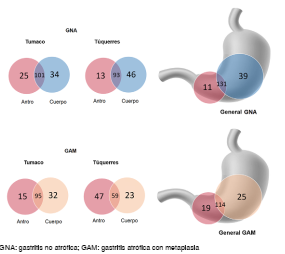
Figura 3 Diagramas de Venn. Unidades taxonómicas operativas a nivel de género y según sitio anatómico y tipo de lesión gástrica en las dos poblaciones opuestas en cuanto al riesgo de cáncer gástrico. Las unidades taxonómicas operativas compartidas se pueden ver en las intersecciones de los círculos. Parte superior: diagrama en la población de Túquerres y en la de Tumaco, y general de unidades taxonómicas operativas del antro y del cuerpo gástrico de individuos con gastritis no atrófica. Parte inferior: diagrama de las poblaciones de Túquerres y de Tumaco y general de las unidades taxonómicas operativas del antro y del cuerpo gástrico de individuos con gastritis atrófica con metaplasia. Los diagramas de Venn se construyeron con las unidades taxonómicas operativas que presentaron 97 % de similitud.
Si bien no se encontraron diferencias en la composición de la microbiota gástrica entre el cuerpo y el antro gástrico de los individuos de las dos poblaciones estudiadas, sí se observaron diferencias significativas en el perfil de la microbiota acompañante de H. pylori según la población en riesgo de cáncer gástrico, independientemente del sitio topográfico del estómago (p<0,0001), resultados que concuerdan con lo encontrado por Yang, et al., en poblaciones similares 9. El efecto de la infección en la composición de la microbiota gástrica según la población en riesgo de cáncer gástrico sugiere que las unidades taxonómicas operativas que conforman los perfiles de la microbiota acompañante se asocian con el riesgo de la enfermedad.
Lactococcus lactis es una bacteria de uso en la industria láctea cuya presencia en individuos con cáncer gástrico incrementa 27 y que se asocia con otros tipos de malignidad en pacientes inmunocomprometidos 28. El papel del lactato potencia la inflamación y su producción aumenta en el metabolismo de las células cancerígenas 29,30. Si bien las bacterias ácido lácticas son beneficiosas para el intestino 31, debe estudiarse más la relación del metabolismo fermentativo y el ácido láctico bacteriano exógeno con el proceso de digestión en los casos de gastritis o lesiones precursoras de cáncer gástrico. No hay reportes de asociación de B. mana y P. putida con las enfermedades gástricas.
La microbiota de los individuos con gastritis atrófica y metaplasia de las dos poblaciones no evidenció diferencias entre el cuerpo y el antro del estómago. En este tipo de lesión precancerosa el reemplazo de las glándulas gástricas por criptas tubulares rectas y células absorbentes y caliciformes 22 es un cambio que homogeniza las condiciones fisiológicas del ambiente gástrico en ambas regiones debido a la atrofia de las glándulas secretoras de ácido, incrementando progresivamente el espectro de bacterias externas que pueden formar colonias 32,33. La alta proporción de Epsilonproteobacteria (>50 %) evidencia su influencia en la malignidad (figura 1). Más del 40 % de los géneros identificados estaban presentes en el cuerpo y en el antro (figura 3).
En Tumaco las unidades taxonómicas operativas representativas halladas en el antro y en el cuerpo gástrico fueron Fusobacterium periodonticum, Haemophilus parainfluenzae y L. lactis, en tanto que en Túquerres, en el antro lo fueron Streptococcus salivaris y Prevotella spp., y en el cuerpo gástrico, Neisseria sicca y H. parainfluenzae. Estas bacterias se han descrito como parte normal de la microbiota oral y de las vías respiratorias con potencial patogénico en pacientes inmunocompetentes e inmunocomprometidos, y su presencia estaría asociada con el aumento del pH como consecuencia de la hipoclorhidria 10,24,32-34.
A pesar de que los métodos de diagnóstico de la infección por H. pylori empleados (tinción de Giemsa y cultivo) no detectaron la bacteria en individuos negativos para H. pylori, mediante secuenciación metagenómica se registraron lecturas en un rango que abarcó desde muy poca abundancia de H. pylori hasta una considerable, especialmente en las muestras MB123 y MB191, lo que concuerda con lo reportado en otras investigaciones 10,35,36.
Debido a este fenómeno, se hicieron los análisis de la microbiota en individuos negativos para H. pylori, pues factores como la baja carga bacteriana, la distribución heterogénea de H. pylori en el estómago y otros asociados con el huésped (gastritis activa, consumo de alcohol, uso de antagonistas de los receptores H2, inhibidores de la bomba de protones, antibióticos, etc.), serían elementos comunes que influyeron en los resultados 37,38. En este caso, se observaron patrones microbianos heterogéneos de composición similar tanto en el antro como en el cuerpo gástrico, lo cual se evidenció por la diversidad metagenómica en ambos sitios anatómicos.
En los perfiles microbianos, la proporción de la clase Epsilonproteobacteria fue de <20 %, pero en Tumaco las unidades taxonómicas operativas representativas del antro y del cuerpo fueron L. lactis y F. periodonticum, mientras que en Túquerres fueron P. putida y B. mana.
El presente análisis sugiere que, aunque la proporción de la infección por H. pylori fue baja, la actividad de la ureasa, que neutraliza el ácido del estómago, puede generar un ambiente más favorable para la colonización progresiva de bacterias externas 5,32,35. El papel de otras bacterias diferentes a H. pylori en el proceso inflamatorio y la enfermedad gástrica es un tema de debate; sin embargo, los subproductos de distintos tipos de metabolismo bacteriano podrían incrementar la producción de compuestos dañinos para el ADN, como los N-nitroso, que influyen en la metilación de las células epiteliales 8,39, y constituir un factor complementario en la malignidad. Más del 50 % de los géneros identificados estaban presentes tanto en el cuerpo como en el antro (figura 4).
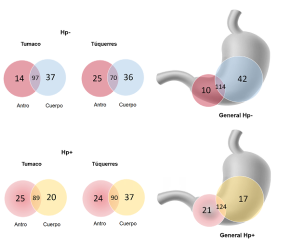
Figura 4 Diagramas de Venn. Unidades taxonómicas operativas a nivel de género y según sitio anatómico y estatus de infección por Helicobacter pylori en las dos poblaciones con riesgo opuesto de cáncer gástrico. Las unidades taxonómicas operativas compartidas se pueden ver en las intersecciones de los círculos. Parte superior: diagrama poblacional de Túquerres y de Tumaco y general de unidades taxonómicas operativas del antro y del cuerpo gástrico de individuos libres de la infección por H. pylori (Hp-). Parte inferior: diagrama poblacional de Túquerres y de Tumaco y general de unidades taxonómicas operativas del antro y del cuerpo gástrico de individuos infectados con H. pylori (Hp+). Los diagramas de Venn se construyeron con las unidades taxonómicas operativas que presentaron un 97 % de similitud.
Los perfiles de microbiota del antro y del cuerpo gástrico de individuos positivos para H. pylori evidenciaron patrones de composición homogénea con un predominio de la clase Epsilon proteobacteria de más del 70 %. Rolig, et al.25, estudiaron en modelos en ratón la capacidad proliferativa de H. pylori y describieron el receptor quimiotáctico TlpD como responsable de que la bacteria prolifere en el antro, así como su capacidad quimiotáctica para guiar la bacteria hacia el cuerpo gástrico. Conjuntamente con la motilidad flagelar, la actividad de la ureasa y el sistema de secreción de tipo IV (T4SS) 26, estos elementos se consideran esenciales para la persistencia inflamatoria en distintas regiones de la mucosa del estómago 39,40.
A pesar de que H. pylori fue la especie más abundante (figura 1), en Tumaco L. lactis se encontró en el antro y en el cuerpo gástrico, en tanto que en Túquerres las unidades taxonómicas operativas representativas en el antro fueron S. salivaris y Prevotella spp., y en el cuerpo gástrico, L. lactis. Además, más del 60 % de los géneros identificados estaban presentes tanto en el cuerpo como en el antro (figura 4).
Con la intención de establecer las tendencias de la microbiota según la presencia de las condiciones estudiadas, se comparó la transición de la gastritis no atrófica a la atrófica metaplásica y el estatus de H. pylori, y se observó que la lesión no influyó en la diversidad de la microbiota en el antro y en el cuerpo, donde unas especies microbianas serían reemplazadas por otras manteniendo la diversidad (p>0,05; no se presentan los datos), en tanto que la infección por H. pylori reduce la diversidad metagenómica en el antro (p=0,005) y tiende a limitarla en el cuerpo, aunque no de forma significativa (figura 5).
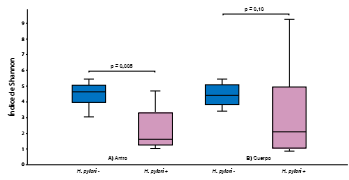
Figura 5 Diversidad metagenómica de la microbiota gástrica: índices de Shannon según infección por Helicobacter pylori y sitio anatómico del estómago. A) Antro gástrico. B) Cuerpo gástrico. En el antro gástrico de individuos positivos para H. pylori (H. pylori +) la diversidad metagenómica disminuyó significativamente con respecto al grupo de individuos libres de la infección (H. pylori -) (p=0,005). Aunque se observó una tendencia a la diferenciación de la microbiota en el cuerpo gástrico, los análisis no revelaron diferencias estadísticas en la transición del estatus de H. pylori (p=0,10; prueba U de Mann-Whitney).
La progresión de la gastritis a lesión precancerosa está asociada con el incremento de la abundancia de H. pylori, y dada la aparición de nuevos grupos bacterianos en la carcinogénesis 27,41-43, el mantenimiento del agente patógeno se vería limitado por la posible competencia interespecífica debida a la escasez de recursos 43, por lo cual también depende del estado focal del tejido tumoral o del no maligno 36.
Este estudio permitió identificar microorganismos taxonómicamente comunes o no, participantes en el desarrollo de la enfermedad gástrica según la región anatómica. Aunque el contar con una muestra de mayor tamaño e incluir pacientes con diagnóstico de cáncer complementarían los análisis, se pudo establecer que H. pylori es un factor común en la malignidad y en el proceso de disbacteriosis gástrica en individuos con alto y bajo riesgo de cáncer gástrico.
Se resalta la importancia de implementar los métodos de alta sensibilidad para la detección de H. pylori, pues independientemente de la carga bacteriana u otros elementos inhibitorios, es necesario tener un diagnóstico certero, con el fin de evitar problemas de responsabilidad médica debido a los falsos negativos 37. Cada vez hay más información que sustenta que la erradicación de H. pylori podría restaurar el equilibrio gástrico 36,41-43, por lo que el diagnóstico temprano y el tratamiento adecuado son elementos muy valiosos en la prevención del cáncer gástrico 44,45.
En conclusión, no se observaron diferencias significativas en cuanto a la microbiota asociada con el sitio anatómico, lo que significa que, independientemente de las respuestas fisiológicas sectorizadas en la inflamación, los grupos bacterianos involucrados en la disbacteriosis pueden colonizar tanto el antro como el cuerpo gástrico.
La microbiota acompañante de H. pylori se asocia con el riesgo poblacional de cáncer gástrico (bajo en Tumaco y alto en Túquerres), independientemente del sitio del estómago afectado. El incremento de la abundancia de H. pylori podría ser un elemento usualmente presente en la progresión de la gastritis no atrófica a la de tipo atrófico con metaplasia intestinal, por lo tanto, es posible establecer que la disbacteriosis de la microbiota gástrica precedida por el agente patógeno favorece dicha progresión. En el presente estudio se evidenció que la infección por H. pylori asociada con la microbiota gástrica se relacionaba con las regiones topográficas del estómago y con el tipo de lesión gástrica, lo que sugiere su importancia en la disbacteriosis microbiana y en la enfermedad gástrica.
Si bien la disminución de la eficacia del tratamiento contra H. pylori es un tema de actual preocupación 13,32,41, la modulación de la microbiota gástrica plantea métodos alternativos de investigación para prevenir la carcinogénesis gástrica en individuos infectados con H. pylori.














