El género Listeria está conformado por bacterias Gram positivas con un bajo contenido de G+C 1. Hasta 1984, se incluían en él seis especies 1: L. monocytogenes, L. innocua, L. seeligeri, L. welshimeri, L. grayi y L. ivanovii2-6. En el 2009, se describieron otras dos especies: L. rocourtiae y L. marthii7-8, y posteriormente, se reconocieron nueve más: L. aquatica, L. cornellensis, L. floridensis, L. grandensis, L. riparia, L. booriae, L. newyorkensis, L. fleischmannii y L. weihenstephanensis9-12. Recientemente, mediante estudios filogenéticos y a partir de la información de la secuencia genómica completa, se identificaron las siguientes nuevas especies: L. costaricencis, L. goaensis, L. thailandesis, L. valentina, L. cossartiae, L. farberi, L. immoviles, L. portnoyi y L. rustica13-17.
Listeria monocytogenes se considera una especie patógena para los humanos y los animales, y L. ivanovii, para los animales 1. No obstante, existen reportes de listeriosis humanas ocasionadas por L. ivanovi y L. innocua1,18,19.-L. monocytogenes es un microorganismo ubicuo, facultativo, intracelular y oportunista, que causa graves infecciones en humanos y animales 1. Su principal ruta de transmisión es el consumo de alimentos contaminados 20. Tiene la capacidad de adaptarse a condiciones extremas, lo que permite su supervivencia en la cadena de producción de una gran variedad de alimentos de origen animal y vegetal 20,21. Puede ocasionar grandes pérdidas económicas en la industria a nivel nacional e internacional, dada la obligación de retirar del mercado los alimentos contaminados con el microorganismo 22,23.
Este patógeno causa infecciones sistémicas graves, como meningitis, meningoencefalitis, encefalitis y septicemia 1,20,24-26. La diseminación bacteriana subclínica puede causar endocarditis, e infecciones cutáneas, osteoarticulares o de las vías biliares 24. Además, las infecciones de la madre y del recién nacido pueden producir aborto espontáneo, meningitis, septicemia neonatal e, incluso, la muerte fetal 1,20,24-27.
Listeria monocytogenes ocupa el tercer lugar como agente etiológico causante de meningitis neonatal, con una letalidad de hasta el 60% 27. Las mujeres embarazadas tienen diez veces más probabilidades de contraer infecciones por listeriosis que otras personas 25.
A pesar de que la listeriosis continúa siendo una enfermedad poco frecuente, con una morbilidad baja comparada con otras enfermedades transmitidas por alimentos, la mortalidad por esta causa está entre el 20 y el 30% 1,23,26,28, y en el 95% de los casos requiere hospitalización 28.
En el mundo se han reportado muchos brotes de listeriosis humana asociados con diferentes tipos de alimentos, como quesos y otros derivados lácteos, frutas (melones), productos cárnicos, mariscos y pescados ahumados 20,29-36.
Para identificar las cepas de L. monocytogenes, se utilizan diferentes métodos fenotípicos como las pruebas bioquímicas de identificación y confirmación de especies 37; y la de tipificación serológica, muy útil para determinar los serotipos mediante el esquema de Paterson. Esta metodología, que fue modificada por Seeliger y Hohne 38,39 y aún está vigente, ha permitido caracterizar 13 serotipos de la especie: 1/2a, 1/2b, 1/2c, 3a, 3b, 3c, 4a, 4ab, 4b, 4c, 4d, 4e y 7 1,23,26,28,38,39.
Para la caracterización genotípica de L. monocytogenes en este estudio, se utilizó la electroforesis en gel de campo pulsado (Pulsed-Field Gel Electrophoresis, PFGE). En el estudio multicéntrico de subtipificación de L. monocytogenes de la Organización Mundial de la Salud (OMS) de 1996, se determinó que la PFGE era un método preciso y reproducible para comparar molecularmente los aislamientos de este agente patógeno, utilizando las enzimas de restricción Ascl y Apal 40.
En Colombia, la listeriosis no es una enfermedad de notificación obligatoria; sin embargo, en el periodo 2010-2018 se notificaron al Sistema de Vigilancia en Salud Pública (Sivigila) 42 brotes de enfermedad transmitida por alimentos ocasionados por L. monocytogenes, los cuales afectaron a 1.255 personas por el consumo de alimentos como quesos frescos, derivados cárnicos, comidas de restaurantes y, en general, alimentos listos para el consumo 41.
Para el estudio, se hizo una búsqueda institucional activa en el Sistema de Información de Apoyo a la Notificación e Investigación de Eventos de Interés en Salud Pública (SIANIESP) del Sivigila en los registros individuales de prestación de servicios de salud (RIPS), para: listeriosis cutánea (A230), otras formas de listeriosis (A328), listeriosis no específica (A329), listeriosis congénita (P372), meningitis y meningoencefalitis (A321), y septicemias (A327). Hubo un total de 696 consultas externas por listeriosis, de las cuales 487 fueron por meningitis y meningoencefalitis, y 209 por las otras formas de listeriosis. Asimismo, en este periodo los servicios de urgencias notificaron al Sivigila 85 casos de listeriosis 42.
En Colombia, la meningitis bacteriana es una enfermedad de notificación obligatoria cuando el agente causal es Haemophillus influenzae, Streptococcus pneumonie o Neisseria meningitidis, pero no cuando es L. monocytogenes, lo cual ocasiona un vacío en la vigilancia de este microorganismo 43. El Instituto Nacional de Vigilancia de Medicamentos y Alimentos (Invima), consciente de la gravedad de la listeriosis y teniendo en cuenta que aún no está contemplada en la vigilancia de los alimentos, ha incluido L. monocytogenes como un indicador obligatorio de inocuidad en la vigilancia de los alimentos que se producen o se consumen en el país. Esta vigilancia es rutinaria y es ejercida por las entidades territoriales; además, el envío de los aislamientos de los laboratorios de salud pública al laboratorio de referencia del Invima para su confirmación, serotipificación y subtipificación molecular, es de obligatorio cumplimiento.
En este contexto, los objetivos de este estudio fueron confirmar el resultado de los aislamientos de L. monocytogenes enviados por los laboratorios de salud pública, y determinar la frecuencia y distribución de los serotipos y subtipos moleculares que circulan en el país procedentes de los alimentos que son motivo de vigilancia, como también, los de aquellos asociados epidemiológicamente con los brotes en el periodo comprendido entre el 2010 y el 2018.
Materiales y métodos
Tipo de estudio
Se hizo un estudio descriptivo y retrospectivo entre el 2010 y el 2018 para confirmar la presencia de L. monocytogenes en los aislamientos recibidos en el Laboratorio de Microbiología de Alimentos del Invima.
Muestra
Durante este periodo, se recibieron 2.420 aislamientos de L. monocytogenes enviados por los laboratorios de alimentos de los departamentos adscritos a los de salud pública (LDSP) de Arauca, Antioquia, Atlántico, Bolívar, Boyacá, Caldas, Caquetá, Casanare, Cauca, Cesar, Córdoba, Cundinamarca, Bogotá, La Guajira, Magdalena, Meta, Nariño, Norte de Santander, Putumayo, Quindío, Risaralda, San Andrés Islas, Santander, Sucre y Valle del Cauca, además de los aislamientos recuperados en el laboratorio de referencia. Los servicios de Amazonas, Guainía, Guaviare, Huila, Tolima, Vaupés y Vichada, no enviaron ningún aislamiento.
En el análisis de las variables, se utilizó el programa Microsoft Office Excel 2013® para calcular las frecuencias y las proporciones, y luego se hicieron un análisis univariado y uno bivariado de los datos del estudio. Se incluyeron las siguientes variables: categoría de alimentos, alimentos, procedencia, serotipos y patrones de PFGE.
Caracterización fenotípica
Para identificar los aislamientos se siguió el procedimiento del manual de bacteriología analítica (Bacteriologycal Analytical Manual): coloración de Gram, determinación de catalasa, pruebas bioquímicas (ramnosa, xilosa y manitol), crecimiento en forma de sombrilla en agar, motilidad, pruebas de hemólisis y prueba de Christie-Atkins-Much-Peterson (CAMP). Asimismo, se utilizó el sistema bioquímico API Listeria de Biomérieux37.
Para la tipificación serológica de Listeria spp., se siguió el protocolo de Seeliger y Hohne 38 desarrollado por el Laboratorio de Listeria del Instituto Pasteur en París, Francia 44. Se incluyeron antisueros de factores somáticos y flagelares producidos por el Laboratorio de Microbiología de Alimentos del Invima, y se utilizaron cepas de referencia del Instituto Pasteur de París mediante la inmunización de conejos Nueva Zelanda; asimismo, se utilizaron los antisueros comerciales Eurobio™ y Denka Seiken™.
El control externo de calidad de la identificación y serotipificación, y su seguimiento, estuvieron a cargo del Laboratorio de Listeria y del Centro Colaborativo de la OMS para la listeriosis de origen alimentario del Instituto Pasteur de París.
Los alimentos de los cuales se aisló L. monocytogenes se clasificaron según su categoría y procedencia.
Caracterización genotípica
Los aislamientos de L. monocytogenes se caracterizaron con la técnica de PFGE ya descrita 45. Se seleccionó la enzima de restricción Apal, teniendo en cuenta que el poder discriminatorio calculado mediante el índice de Simpson no difiere entre las dos enzimas recomendadas, Apal y Ascl, cuando se evalúa L. monocytogenes46; además, se ha demostrado que el número de perfiles únicos observado con Apal es mayor que el obtenido con Ascl 47, y se ha evidenciado que el análisis del serotipo 4b con la enzima Apal tiene mayor poder de discriminación que otros métodos de tipificación, incluida la restricción con Ascl 48. Los dendrogramas se generaron con el programa Bionumérics™, versión 7.5 (Applied Maths, Bélgica) mediante el método UPGMA con una optimización de tolerancia de 1,5%.
La muestra para seleccionar los aislamientos sometidos a PFGE, se calculó utilizando Epi-Info, versión 7.2, de 2018, con una estimación del 50% (por ser desconocida), un error esperado del 5 % y una significación del 95%. Se le atribuyó un peso porcentual a cada categoría de alimento y a la frecuencia de sus serotipos. La selección fue aleatoria y quedó conformada por 368 aislamientos distribuidos así: leche y derivados lácteos (n=188), carne y derivados cárnicos (n=93), alimentos listos para el consumo (n=30), aves (n=15), muestras de plantas procesadoras de alimentos (n=3), y productos de panadería y pastelería (n=1). Además, se analizaron todos los aislamientos (n=38) implicados en casos y brotes de enfermedades transmitidas por alimentos.
Resultados
De los 2.420 aislamientos recibidos, L. monocytogenes se confirmó en 2.326 (96,1%) y se identificaron otras especies del género Listeria en 94 (3,9%). De los 2.326 de L. monocytogenes, 2.288 (98,4%) provenían de alimentos que son objeto de los programas de vigilancia del Invima como parte del control de calidad de alimentos en el país, y los 38 (1,6%) restantes correspondían a casos y brotes de enfermedades transmitidas por alimentos.
Los alimentos remitidos y confirmados como L. monocytogenes por el laboratorio del Invima procedían, con mayor frecuencia, de Bogotá, Antioquia y Valle del Cauca (figura 1).
Las categorías de alimentos más frecuentemente contaminados por L. monocytogenes fueron las leches y los derivados lácteos (n=1.317; 56,6%), las carnes y los derivados cárnicos (n=659; 28,3%) y los alimentos listos para el consumo (n=209; 9%) (cuadro suplementario 1).
Las categorías de alimentos en los que más frecuentemente se aisló L. monocytogenes fueron las leches y los derivados lácteos, con 19,91 % en Antioquia y 17,41% en Bogotá, y las carnes y derivados cárnicos, con 17,37% en Bogotá (cuadro suplementario 2).
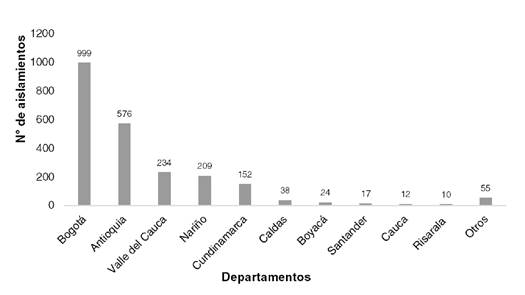
Figura 1 Presencia de Listeria monocytogenes aislada de alimentos según su procedencia, Colombia 2010-2018
Durante el periodo de estudio, se determinaron 12 serotipos. El serotipo más frecuente fue el 4b (n=1.208) (figura 2), y la mayor incidencia de este serotipo se encontró, en primer lugar, en la categoría de leches y derivados lácteos (n=711) y, en segundo lugar, en la de carnes y derivados cárnicos (n=327) (cuadro suplementario 1).
Los quesos campesinos fueron el alimento con mayor frecuencia (31,34%) de aislamientos de L. monocytogenes. El serotipo aislado más veces en el grupo completo de alimentos, fue el 4b (51,93%) (cuadro 1).
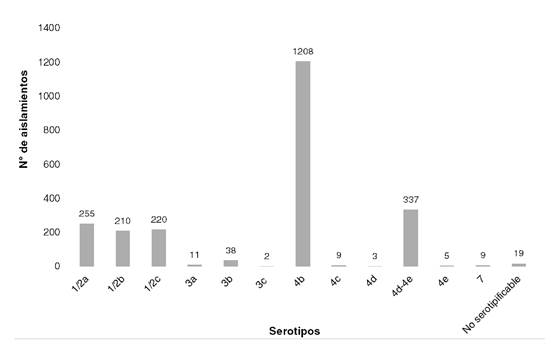
Figura 2 Determinación de serotipos de Listeria monocytogenes aislados de alimentos, Colombia, 2010-2018
Cuadro 1 Frecuencia de Listeria monocytogenes y determinación de serotipos aislados de alimentos, Colombia, 20102018
| Alimentos | 1/2a | 1/2b | 1/2c | 3a | 3b | 3c | 4b | 4c | 4d | 4d-4e | 4e | 7 | NS | Total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| % | % | % | % | % | % | % | % | % | % | % | % | % | % | |
| Queso campesino | 2,24 | 2,19 | 4,56 | 0,39 | 0,77 | 0,04 | 16,17 | 0,00 | 0,04 | 4,69 | 0,00 | 0,04 | 0,21 | 31,34 |
| Jamón cocido de cerdo, cordero, pavo y pollo | 1,03 | 1,76 | 0,95 | 0,00 | 0,13 | 0,00 | 6,15 | 0,09 | 0,04 | 1,29 | 0,04 | 0,04 | 0,09 | 11,61 |
| Cuajada | 0,73 | 0,26 | 1,12 | 0,00 | 0,04 | 0,00 | 2,49 | 0,00 | 0,00 | 0,60 | 0,00 | 0,00 | 0,04 | 5,29 |
| Leches crudas | 0,52 | 0,09 | 0,04 | 0,00 | 0,04 | 0,04 | 2,71 | 0,00 | 0,00 | 0,52 | 0,00 | 0,00 | 0,00 | 3,96 |
| Salchichas cocidas | 0,39 | 0,34 | 0,21 | 0,00 | 0,17 | 0,00 | 1,16 | 0,00 | 0,04 | 0,52 | 0,00 | 0,00 | 0,00 | 2,84 |
| Jamón de cerdo ahumado o madurado | 0,90 | 0,04 | 0,17 | 0,00 | 0,00 | 0,00 | 1,16 | 0,00 | 0,00 | 0,39 | 0,00 | 0,13 | 0,04 | 2,84 |
| Queso mozzarella | 0,21 | 0,26 | 0,00 | 0,00 | 0,00 | 0,00 | 1,72 | 0,04 | 0,00 | 0,30 | 0,00 | 0,04 | 0,09 | 2,67 |
| Sánduche de pollo, jamón, y queso | 0,09 | 0,13 | 0,30 | 0,00 | 0,00 | 0,00 | 1,20 | 0,04 | 0,00 | 0,69 | 0,00 | 0,00 | 0,00 | 2,45 |
| Mortadela | 0,26 | 0,09 | 0,04 | 0,00 | 0,17 | 0,00 | 1,42 | 0,00 | 0,00 | 0,39 | 0,00 | 0,04 | 0,00 | 2,41 |
| Ensalada de verduras | 0,26 | 0,17 | 0,00 | 0,00 | 0,00 | 0,00 | 1,07 | 0,00 | 0,00 | 0,34 | 0,00 | 0,00 | 0,00 | 1,85 |
| Total | 6,62 | 5,33 | 7,39 | 0,39 | 1,33 | 0,09 | 35,25 | 0,17 | 0,13 | 9,72 | 0,04 | 0,30 | 0,47 | 67,24 |
| Otros | 4,34 | 3,70 | 2,06 | 0,09 | 0,30 | 0,00 | 16,68 | 0,21 | 0,00 | 4,77 | 0,17 | 0,09 | 0,34 | 32,76 |
| Total | 10,96 | 9,03 | 9,46 | 0,47 | 1,63 | 0,09 | 51,93 | 0,39 | 0,13 | 14,50 | 0,21 | 0,39 | 0,82 | 100,00 |
NS: no serotipificable
De los 368 aislamientos seleccionados para PFGE, la técnica pudo utilizarse en 333; no fue posible hacerlo en 35 (9,5%), probablemente por la calidad del ADN. Estos aislamientos se agruparon en 167 patrones de PFGE diferentes y se designaron de LM-001 a LM-167 (figura suplementaria 1).
Distribución de patrones frecuentes de PFGE
Para el análisis de los patrones de PFGE obtenidos, se hizo una clasificación de los cuatro patrones más frecuentes, es decir, aquellos con más de 10 aislamientos (cuadro 2) (figura suplementaria 1).
Patrón LM-076. El 81,2% (13/16) de los aislamientos pertenecía al serotipo 4b, el cual se presentó en siete clases diferentes de alimentos y en tres departamentos (cuadro 2) (figura suplementaria 1). Este patrón únicamente se presentó en los cinco primeros años de vigilancia.
Patrón LM-0154. El 87,5% (14/16) de los aislamientos con este patrón correspondió al serotipo 1/2c, el cual fue el más representativo, seguido por el 1/2b y el 4b. El queso campesino fue el alimento en el cual se encontró con mayor frecuencia y las muestras provenían casi exclusivamente del departamento de Antioquia (14/16) (87,5%) (cuadro 2) (figura suplementaria 1). Este patrón únicamente se presentó en los cinco primeros años de vigilancia (cuadro 3).
Cuadro 2 Distribución de perfiles de PFGE más frecuentes según tipo de alimento, Colombia, 2010-2018
| Patrón PFGE | Alimento relacionado | Número de aislamientos por patrón | Serotipos | Procedencia | |||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1/2a | 1/2b | 1/2c | 3a | 3b | 4b | 4d | NS | ||||
| LM 076 | Ensalada de verduras (1), helado (1), jamón (3), leche cruda (1), pollo crudo (1), queso campesino (8) y queso doble crema (1) | 16 | 1 | 0 | 0 | 0 | 0 | 13 | 0 | 2 | Antioquia (9) Bogotá (5) Caldas (2) |
| LM 154 | Cuajada (1), jamón de cerdo (1), queso campesino (14) | 16 | 0 | 1 | 14 | 0 | 0 | 1 | 00 | 0 | Antioquia (14) Bogotá (2) |
| LM 047 | Agua de panela (1), carne cocinada (1), carne cruda (1), crema de leche (1), flan de leche (1), jamón (2), queso campesino (2), salami (1), sánduche (1), tocineta (1) | 12 | 0 | 0 | 0 | 0 | 0 | 6 | 6 | 0 | Bogotá (11) Valle (1) |
| LM 072 | Arroz con pollo (1), ensalada de frutas (1), ensalada de verduras (1), jamón cocido (1), menú (1), pechuga de pollo cocinada (1), queso campesino (4) | 10 | 0 | 0 | 0 | 0 | 0 | 10 | 0 | 0 | Antioquia (6) Bogotá (1) Caldas (1) Cundinamarca (1) Valle (1) |
| Otros patrones n=163 | 67 diferentes tipos de alimentos, el queso campesino concentró el mayor número de muestras: 69 en total | 279 | 34 | 37 | 21 | 2 | 5 | 146 | 32 | 2 | Antioquia (66) Bogotá (108) Nariño (36) Valle (28) Cundinamarca (9) Caldas (7) San Andrés (3) Amazonas, Boyacá, Cauca, Magdalena, Quindío, Risaralda y Putumayo (2 aislamientos cada uno) Arauca, Bolívar, Caquetá, Cesar, Córdoba, Magdalena, Norte de Santander, San Andrés (1 aislamiento cada uno) |
| Total de aislamientos | 333 | 35 | 38 | 35 | 2 | 5 | 176 | 38 | 4 | ||
NS: no serotipificable
Cuadro 3 Distribución de perfiles de PFGE más frecuentes por serotipo y año en aislamientos de Listeria monocytogenes, Colombia, 2010-2018
| Año/patron de PFGE | Serotipos | Número de aislamientos | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1/2a | 1/2b | 1/2c | 3a | 3b | 4b | 4d | NS | |||||||||||
| 76 | O | 154 | O | 154 | O | O | O | 47 | 72 | 76 | 154 | O | 47 | O | 76 | O | ||
| 2010 | 0 | 5 | 1 | 11 | 2 | 1 | 1 | 2 | 0 | 0 | 1 | 1 | 32 | 3 | 7 | 0 | 0 | 67 |
| 2011 | 0 | 8 | 0 | 9 | 2 | 3 | 1 | 1 | 1 | 3 | 1 | 0 | 25 | 2 | 6 | 0 | 0 | 62 |
| 2012 | 0 | 5 | 0 | 3 | 4 | 5 | 0 | 2 | 1 | 0 | 5 | 0 | 20 | 0 | 2 | 1 | 1 | 49 |
| 2013 | 0 | 7 | 0 | 5 | 2 | 3 | 0 | 0 | 0 | 5 | 6 | 0 | 18 | 0 | 8 | 0 | 0 | 54 |
| 2014 | 1 | 4 | 0 | 6 | 4 | 6 | 0 | 0 | 0 | 1 | 0 | 0 | 23 | 1 | 8 | 1 | 1 | 56 |
| 2015 | 0 | 2 | 0 | 1 | 0 | 1 | 0 | 0 | 1 | 1 | 0 | 0 | 11 | 0 | 1 | 0 | 0 | 18 |
| 2016 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 7 | 0 | 0 | 0 | 0 | 9 |
| 2017 | 0 | 2 | 0 | 0 | 0 | 1 | 0 | 0 | 2 | 0 | 0 | 0 | 7 | 0 | 0 | 0 | 0 | 12 |
| 2018 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 6 |
| Total | 1 | 34 | 1 | 37 | 14 | 21 | 2 | 5 | 6 | 10 | 13 | 1 | 146 | 6 | 32 | 2 | 2 | 333 |
NS: no serotipificable; patrón de PFGE: (047/072/076/154); O: otros, corresponde a 163 perfiles que contenían menos de 10 aislamientos cada uno
Patrón LM-047. Se encontraron 12 aislamientos con este patrón, seis del serotipo 4b y seis del 4d, en 10 alimentos diferentes y casi exclusivamente (91,6%) en Bogotá (cuadro 2) (figura suplementaria 1).
Patrón LM-072. Todos los aislamientos de este patrón fueron del serotipo 4b, es decir que, en las muestras evaluadas en el estudio, este patrón se detectó exclusivamente en este serotipo. Se presentó en siete clases de alimentos diferentes y en cinco departamentos, en los años 2011, 2013, 2014 y 2015 (cuadro 2) (figura suplementaria 1).
Otros patrones. Se encontraron 163 patrones diferentes de PFGE en 279 aislamientos distribuidos en 67 diferentes tipos de alimentos, de los cuales el más frecuente fue el queso campesino. Los serotipos más frecuentes en esta categoría fueron el 4b, con 146 (52,4%) aislamientos, el 1/2b, con 37 (13,2%), el 1/2a, con 34 (12,2%), y el 4d, con 32 (11,5%). Los aislamientos de esta categoría procedían de 22 departamentos (cuadro 2) (figura suplementaria 1).
Distribución de patrones de PFGE por serotipo
Serotipo 4b. De los 333 aislamientos evaluados por PFGE, más de la mitad (n=176; 52,9%) correspondió al serotipo 4b, recuperados durante todos los años de vigilancia (cuadro 3).
Serotipo 4d. Se procesaron 38 (11,4%) aislamientos recuperados en el 2010, el 2011 y el 2014 (cuadro 3).
Serotipo 1/2b. Se procesaron 38 (11,4%) aislamientos recuperados en todos los años, excepto en el 2017. Solo uno se relacionó con los patrones frecuentes.
Serotipos 1/2a y 1/2c. Del serotipo 1/2a se analizaron 35 (10,5%) aislamientos encontrados en todos los años de estudio, excepto en el 2016, en tanto que el serotipo 1/2c se evaluó en todos los años de estudio, excepto en el 2018 (cuadro 3).
Otros serotipos. Los 11 aislamientos restantes correspondieron a los serotipos 3b (1,5%) y 3a (0,6%), y cuatro (1,2%) no pudieron ser serotipificados (cuadro 3). Del serotipo 4c no se evaluaron aislamientos con la PFGE.
Patrones de PFGE en aislamientos causantes de enfermedades transmitidas por alimentos
Los 38 aislamientos clasificados en esta categoría fueron relacionados epidemiológicamente con enfermedades transmitidas por alimentos por los laboratorios de salud pública. Se evaluaron 35 aislamientos mediante PFGE, los cuales se distribuyeron en cuatro serotipos y 30 patrones.
Serotipo 1/2a. Se encontraron cuatro aislamientos distribuidos en cuatro patrones de PFGE.
Serotipo 1/2b. Se encontraron cuatro aislamientos distribuidos en cuatro patrones de PFGE.
Serotipo 1/2c. Se encontraron siete aislamientos distribuidos en cinco patrones de PFGE.
Serotipo 4b. Se encontraron 20 aislamientos distribuidos en 18 patrones de PFGE (figura 3). Los serotipos 3a, 3b, 4c y 4d no se relacionaron enfermedades transmitidas por alimentos.
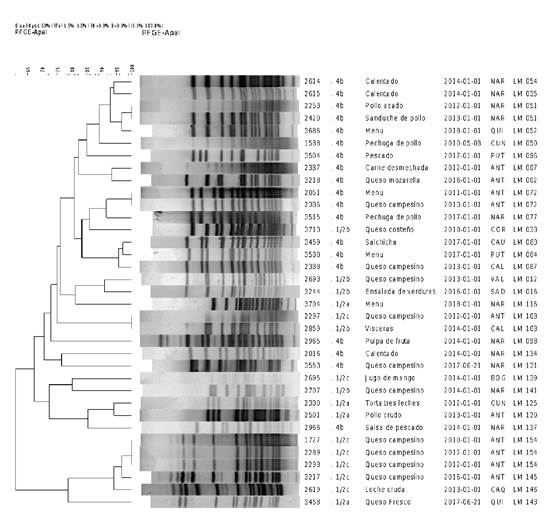
Figura 3 Patrones de PFGE de aislamientos de Listeria monocytogenes relacionados con brotes de enfermedades transmitidas por alimentos, Colombia, 2010-2018
Los brotes de enfermedades transmitidas por alimentos se relacionaron con las categorías de alimentos preparados (arroz con pollo, calentado, carne desmechada, jugo, tortas), derivados lácteos (leche cruda, queso) y derivados cárnicos (pollo) (figura 3). Los departamentos que enviaron mayor cantidad de aislamientos relacionados con este tipo de enfermedades durante este periodo de estudio, fueron Antioquia y Nariño.
Dos aislamientos del serotipo 4b presentaron el patrón LM-51 y ambos provenían del departamento de Nariño; se encontraron en alimentos que contenían pollo y se enviaron en dos años diferentes, lo cual demuestra la persistencia en el tiempo de este patrón y su relación con enfermedades transmitidas por alimentos.
Una situación similar se presentó en Antioquia con el patrón 072, el cual se recuperó en el 2011 y en el 2013 en alimentos relacionados con estas enfermedades; el patrón LM-154 se encontró en tres aislamientos con el serotipo 1/2c, todos procedentes de queso campesino en dos años diferentes.
Discusión
En Colombia, la autoridad sanitaria ha incluido L. monocytogenes en la vigilancia rutinaria de los alimentos como indicador de inocuidad, por ser un microorganismo causante de enfermedades transmitidas por alimentos, aunque aún no se encuentra regulado en las normas nacionales.
Los serotipos encontrados en este estudio, el 4b, el 1/2a y el 1/2b, coinciden con los reportados a nivel mundial relacionados con casos o brotes de listeriosis 1,21,23,24,26,28. Según el ciclo evolutivo de L. monocytogenes, hay cuatro linajes. Los serotipos 4b y 1/2b pertenecen al linaje I y son causantes de la mayoría de las listeriosis humanas transmitidas por alimentos; el serotipo1/2a pertenece al linaje II, implicado en brotes de listeriosis humanas y también aislado del ambiente y de alimentos 49. Aunque no todos los serotipos de L. monocytogenes tienen factores de virulencia similares, para efectos de la salud pública, todos ellos se consideran potencialmente patógenos 1,23,26.
En la vigilancia rutinaria de alimentos, Bogotá fue la entidad territorial que envió el mayor número de aislamientos de L. monocytogenes, seguida por Antioquia, lo que posiblemente se debe a que existe un mejor programa de vigilancia de este agente patógeno en estas dos seccionales.
Los derivados lácteos, principalmente el queso campesino y las cuajadas, fue el grupo de alimentos en el que más se aisló L. monocytogenes, principalmente del serotipo 4b, lo cual también puede deberse a que muchos son de producción artesanal, en la cual es más común el uso de leches no pasteurizadas, así como la ausencia de buenas prácticas de manufactura.
En los derivados cárnicos, las concentraciones de nitritos permitidas como conservantes no inhiben a L. monocytogenes31 y permiten su crecimiento. En el presente estudio se reportaron aislamientos, principalmente del serotipo 4b, en jamones cocidos de cerdo, cordero, pavo y pollo. Es probable que esto se deba a fallas en el mantenimiento de la higiene a lo largo de la cadena de producción, conservación, transporte, fraccionamiento y manipulación en la venta de estos productos, lo cual, unido a características como el gran contenido de nutrientes, la importante actividad del agua (AW) (≥0,92%), un pH de 4,4 o menos, y la conservación prolongada a bajas temperaturas de refrigeración, permite la supervivencia de L. monocytogenes50.
En comparación con un reporte previo de la distribución de serotipos de L. monocytogenes en alimentos en Colombia 51, se encontró que no se han presentado variaciones en el tipo de alimento contaminado con este agente patógeno con respecto a los años anteriores y, tampoco, en los serotipos encontrados, lo cual pone en evidencia la falta de intervención en los establecimientos que son de obligatoria vigilancia y control.
Se ha sugerido que en los aislamientos de alimentos se encuentran principalmente los serotipos 1/2a,1/2b y 1/2c 1,21. En el presente estudio, se observó con mayor frecuencia el serotipo 4b, de gran virulencia y causante de la enfermedad en humanos 1,21,23,26, como ya lo reportaron Montero, et al. 52, quienes evaluaron los serotipos de L. monocytogenes aislados de alimentos listos para el consumo y encontraron una asociación del serotipo 4b con alimentos como el queso y los mariscos congelados. Esto puede deberse a que pertenecen a uno de los clones epidémicos identificados que circulan en el mundo, característica que debería evaluarse en estudios posteriores; además, cobra importancia porque uno de los patrones frecuentes encontrados en este estudio, el LM 072, se asoció exclusivamente con aislamientos de serotipo 4b provenientes de alimentos listos para el consumo, como arroz con pollo, ensalada de frutas y verduras, menú listo para consumir, pechuga de pollo cocinada y queso campesino. Ello evidencia la necesidad de incluir a la ausencia de L. monocytogenes como parámetro de inocuidad obligatorio, principalmente en la categoría de alimentos listos para el consumo.
Se ha reportado que los aislamientos de origen alimentario tienen mayor diversidad genética que los de origen humano 53. Esta característica también se observó en el presente estudio, en el cual se obtuvieron más de 160 perfiles diferentes de electroforesis, lo que sugiere diversas fuentes y condiciones de selección para los genotipos específicos encontrados en alimentos y ambientes de plantas de producción; además, sugiere que solo ciertos grupos de alimentos están relacionados con la enfermedad en humanos 54,55.
Los aislamientos caracterizados mediante PFGE permitieron identificar patrones moleculares que pueden indicar contaminación y persistencia de clones en el tiempo. La persistencia de ciertos genotipos en las instalaciones destinadas a la producción de alimentos, puede ser un signo de fallas en el control del proceso de fabricación o indicar resistencia de los microorganismos a los métodos de limpieza utilizados. Tal persistencia puede deberse a la supervivencia y crecimiento de ciertas cepas en biopelículas o nichos dentro del entorno alimentario, las cuales pueden ser difíciles de limpiar y desinfectar, así como a la reintroducción repetida de tales cepas del ambiente externo en las instalaciones de procesamiento de alimentos a lo largo del tiempo 54.
La cantidad de patrones caracterizados como no persistentes, que en este trabajo se clasificaron como “otros”, indica la diversidad de aislamientos que existen en el entorno del procesamiento de alimentos, así como una probable contaminación esporádica continua, lo que indicaría falta de cumplimiento de los protocolos de higiene y limpieza y, por lo tanto, el riesgo de contaminación de los productos alimenticios 54.
Los patrones de PFGE clasificados como más frecuentes en este estudio, se presentaron por lo menos una vez en la mayoría de los años en varios departamentos y en alimentos listos para consumir, como quesos, carnes, comidas preparadas y sánduches, entre otros. La permanencia de estos patrones de PFGE en el tiempo, sugiere que los aislamientos de L. monocytogenes implicados tienen un reservorio permanente en los intervalos de tiempo, por lo general largos, entre sus apariciones 53,54.
Este estudio brinda información importante sobre la epidemiología de L. monocytogenes y puede ayudar a adoptar decisiones sanitarias en Colombia, así como servir de base para estudios posteriores de identificación de los clones epidémicos. Consideramos que los resultados de la genotipificación se pueden utilizar como información de base para hacer análisis genéticos ampliados, por ejemplo, la secuenciación completa del genoma de L. monocytogenes.
La clasificación de algunos alimentos, como los 'menús', se dificultó porque la información enviada al laboratorio no estaba completa. Los resultados obtenidos indican la necesidad de implementar buenas prácticas de manufactura con procedimientos estandarizados, en las fábricas artesanales del país 20; además, que las autoridades sanitarias del país deben continuar implementando programas eficaces de vigilancia frente a la prohibición de la venta informal de leche cruda, y de la de quesos o cuajadas elaboradas con leche sin pasteurizar.
Se recomienda, al Ministerio de Salud y Protección Social, incluir la listeriosis entre las enfermedades de notificación obligatoria. Asimismo, incluir en la legislación colombiana la ausencia de L. monocytogenes como parámetro de inocuidad en el análisis de los alimentos que, por sus características intrínsecas y extrínsecas, permiten su crecimiento, como es el caso de aquellos listos para el consumo.















