Candida albicans es un complejo de especies crípticas o estrechamente relacionadas que incluye a C. albicans sensu stricto, C. dubliniensis y C. africana. En la actualidad, C. albicans sensu stricto es reconocida como el agente causal más común en las infecciones micóticas 1,2.
Candida dubliniensis y C. africana se consideraron inicialmente como especies "menores" o variedades biológicas de C. albicans, y se reportaron como "cándidas atípicas" por compartir características fenotípicas. Hoy, C. dubliniensis se reconoce como una especie y C. africana se considera una variedad de C. albicans3.
Entre las características estudiadas mediante pruebas fisiológicas para diferenciar las especies del complejo, se encuentran el color de las colonias en los medios cromógenos, el crecimiento a 45 °C, la producción de clamidosporas, la morfología de las colonias en agar Staib o agar tabaco y la inhibición del crecimiento en medio con concentraciones altas de sal, etc. No obstante, ninguna prueba es completamente específica 4,5.
Candida dubliniensis fue descrita por primera vez en 1995 6 y se reconoce como una levadura capaz de producir clamidosporas y tubos germinativos, al igual que C. albicans. C. dubliniensis es un agente patógeno significativamente menos virulento y versátil, que se ha recuperado en ubicaciones geográficas en diversas zonas geográficas 7. En Colombia, este microorganismo fue identificado por primera vez mediante métodos fenotípicos en 2008 8.
Candida africana es la especie emergente más reciente de este complejo, posee tubo germinal, no asimila N-acetilglucosamina y no produce clamidosporas. Se aisló por primera vez a partir de muestras de pacientes de África y Alemania 9 y, desde el 2001, se considera una variedad de C. albicans (C. albicans var. africana)3,10. Se ha identificado en países como Chile, Nigeria, Irán y Argentina 1,3-4,11,12. En Colombia, la identificación molecular de C. africana a partir de cepas caracterizadas previamente como C. albicans atípicas, arrojó resultados sugestivos de C. albicans ( 5. En la actualidad, la identificación definitiva de C. africana requiere pruebas adicionales, como técnicas moleculares para detectar el gen hwp113,14.
Por otra parte, y en relación con los factores determinantes de la patogénesis (producción de hemolisinas, biopelículas y enzimas), con los recientes estudios se demuestra el creciente interés en aislamientos de C. albicans y C. dubliniensis15-17. Se sabe que todos estos factores patogénicos juegan un rol importante en el proceso infeccioso, la colonización y la permanencia del hongo en la superficie corporal, además de la formación de poros y el efecto lítico en las células eucarióticas; no obstante, aún es mucho lo que se desconoce.
El objetivo de este trabajo fue identificar las características fenotípicas y patogénicas de aislamientos vulvovaginales del complejo Candida albicans, mediante su cultivo en diferentes medios enriquecidos, la evaluación de la actividad de las hemolisinas y la formación de biopelículas.
Materiales y métodos
Se estudiaron 300 aislamientos de levaduras, recolectados durante los años 2018 y 2019, y conservados en una colección de cultivos. Las cepas se aislaron de frotis vulvovaginales de igual número de pacientes adultas, con diagnóstico presuntivo de candidiasis vulvovaginal, quienes asistieron a dos laboratorios clínicos de Valledupar.
Para la recuperación de las cepas, las muestras se sembraron en agar Sabouraud y se incubaron a 37 °C durante 48 horas. Los tubos en los que no se observó crecimiento, se resembraron en caldo infusión de cerebro-corazón (Brain Heart Infusión, BHI) con enriquecimiento Fildes.
Identificación morfológica y fenotípica
CHROMagar™ Candida. Las cepas aisladas se sembraron en CHROMagar™ Candida y se incubaron a 35 °C por 48 horas. Se seleccionaron las colonias de color verde por considerarse presuntivas del complejo C. albicans.
Como controles, se emplearon las cepas de C. albicans de la American Type Culture Collection (ATCC), con números de catálogo ATCC 10231 y ATCC 90028; C. africana MYA-2669 y C. dubliniensis - ATCC 3949.
Formación de tubo germinal. La formación del tubo germinal se evaluó con caldo de leche, caldo infusión de cerebro-corazón y suero humano. Para todos los casos, se preparó un inóculo (105 a 106 células por ml) con una colonia aislada y joven -menos de 48 horas- y se homogenizó en tubos que contenían 5 ml de caldo de leche o caldo infusión de cerebro-corazón o 0,5 ml de suero humano, y se incubaron a 37 °C. Después de dos horas, se hicieron los montajes entre lámina y laminilla, y se observaron microscópicamente con ayuda de un objetivo que amplifica 400 veces, en busca de prolongaciones filamentosas 18.
Formación de clamidoconidias. Para la producción de clamidoconidias, se usó el mismo tubo de la prueba para tubo germinal, cuya incubación a 37 °C se extendió hasta por cinco días. Se realizó la lectura por microscopía óptica en busca de estructuras redondas con pared celular gruesa 4.
Crecimiento en agar romero (Rosmarinus officcinalis) y agar tabaco (Nicotiana tabacum). Para la diferenciación presuntiva de C. dubliniensis, cada cepa se sembró por agotamiento en agar romero (Rosmarinus officinalls) y agar tabaco (Nicotiana tabacum), y se incubó por 4 a 5 días a 37 °C.
En el agar romero, se inspeccionó la apariencia de la colonia y se determinó la presencia o ausencia de clamidoconidias 19. Se consideraron aislamientos presuntivos de C. dubliniensis aquellos con colonias rugosas, bordes de hifas periféricas y abundantes clamidosporas; mientras que se presumieron como aislamientos de C. albicans aquellos con colonias lisas y sin clamidosporas.
En el agar tabaco, se analizaron el color y el aspecto de la colonia, y la formación de clamidoconidias. Se consideraron aislamientos presuntivos de C. dubliniensis aquellos con colonias rugosas de color café y con abundantes clamidoconidias 20,21. Se presumió que los aislamientos con colonias lisas blancas o crema y sin clamidoconidias, correspondían a C. albicans.
Evaluación de la patogenicidad
Actividad hemolítica. La actividad hemolítica se evaluó en agar sangre de carnero (Ad-bio- ANNAR™) y agar papa dextrosa (Scharlau™) suplementado con 5 % de sangre fresca de carnero y 3 % de glucosa. Por cada aislamiento se marcó una estría sobre el agar y las placas se incubaron a 37°C, por 48 horas, en atmósfera microaerofílica 22. Las hemólisis beta (total) y la alfa (parcial) se consideraron resultados positivos. Se emplearon aislamientos de S. aureus (ATCC 25923) y C. albicans (ATCC 10231) como controles positivos en agar sangre de carnero y agar PDA con sangre, respectivamente.
Efecto cohemolítico o factor CAMP como indicador de hemólisis de tipo beta en cocultivo con Staphylococcus aureus. Se evaluó la producción de factor CAMP como indicador de hemólisis de tipo beta en cocultivo con S. aureus. Se hizo una siembra perpendicular con una estría de S. aureus ATCC 25923, a una distancia de 10 mm desde el borde de la colonia de la levadura. Las placas se incubaron a 37 °C por 48 horas en atmósfera microaerofílica, en búsqueda de una deformación en punta de flecha en la intersección de las hemólisis 15,17,23. Como controles positivos, se utilizaron las cepas de referencia S. aureus (ATCC 25923) y S. agalactiae (ATCC 12386).
Formación de biopelículas. Para la formación de biopelículas, se prepararon soluciones en tubos de poliestireno cónico, con tapa de rosca, con 5 ml de caldo Sabouraud dextrosa al 8 % inoculado con una colonia de cada aislamiento (105 a 106 células por ml). Los tubos se incubaron a 35 °C en el equipo Shaking Incubator (BeingTM THZ-300) con agitación constante a 75 rpm por 72 horas. Se aspiró el contenido del caldo y se hicieron dos lavados con agua destilada. Para evidenciar la formación de la biopelícula, se agregaron 3 ml de safranina al 1, 5 y 10 % por 10 minutos y se decantó por inversión. Los resultados se interpretaron como positivo (fuerte, moderado, débil) o negativo (ausente), por inspección visual de una capa de biopelícula adherente en la parte inferior y en la pared de los tubos 15,17. Se emplearon C. albicans (ATCC 10231) como control positivo y solución salina fisiológica como control negativo.
Producción de coagulasa. A partir de un subcultivo en agar Sabouraud (incubado a 37 °C durante 48 horas), se inoculó una colonia de los microorganismos en un tubo con 3 ml de plasma de conejo con EDTA (105 a 106 células por ml). Este se incubó por 4 horas a 37 °C. Los tubos se inclinaron cada media hora de forma suave. La prueba de coagulasa se consideró positiva cuando se observó la formación de un coágulo 24. Se usó S. aureus (ATCC 25923) como control positivo.
Todas las pruebas se practicaron por triplicado en tres días diferentes.
Prueba de referencia
Todas las cepas fueron identificadas por espectometría de masas MALDI-TOF (espectometría de masas de tipo desorción-ionización láser, asistida por matriz, acoplada a un detector iónico de tiempo de vuelo) en el equipo Vitek MS™ (bioMérieux), de acuerdo con el protocolo sugerido por el fabricante. La identificación se consideró confiable con un puntaje superior al 98 % 25.
Resultados
Identificación morfológica y fenotípica
En el CHROMagarTM Candida se observaron colonias en diferentes tonalidades de color verde (figura 1a), sin que fuera posible hacer una clara discriminación que permitiera su cuantificación.
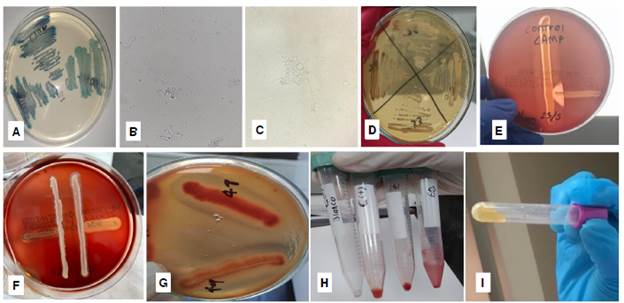
Figura 1 a. Tonalidades de verde en CHROMagar™ Candida, b. Imagen de tubos germinales, 400X. c. Producción de clamidoconidias, 400X. d. Colonia café (izquierda) en agar romero. e. Control positivo de la prueba de Christie, Atkins y Munch-Peterson (CAMP) en medio agar con sangre comercial. f. Prueba CAMP negativa en dos aislamientos de Candida (líneas verticales). g. Hemólisis generada por dos aislamientos de Candida en agar papa dextrosa y sangre, con suplemento de glucosa. h. Controles (negativo y positivo) y resultados positivos de formación de biopelículas (débil/fuerte), con 10% de safranina, en dos aislamientos. i. Prueba positiva para coagulasa.
Todos los 300 aislamientos formaron tubo germinal en medio de suero humano y caldo de leche; mientras que 131 (43,7 %) lo hicieron por el método con caldo infusión de cerebro-corazón (figura 1b y cuadro 1). Se obtuvo producción de clamidoconidias en 141 aislamientos (47 %), luego de cinco días de incubación a 37 °C (figura 1c y cuadro 1).
Cuadro 1 Características fenotípicas y determinantes de la patogenicidad evaluadas en los aislamientos del complejo Candida albicans. Los valores están expresados en porcentaje.
| Tubo germinal | CLAM | Agar romero | Agar tabaco | Hemólisis y CAMP en agar sangre | Hemólisis y CAMP en agar papa dextrosa | Producción de biopelículas | Coag. | Identificación por MALDI-TOF | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Aspecto por MALDI-TOF de la colonia | CLAM | Color y aspecto de la colonia | CLAM | |||||||||||||
| Suero | Caldo leche | Caldo infusión de cerebrocorazón | Pos | Neg | Rugosa | Pos | Café/ rugoso | Blanco/ liso | Pos | Pos | Pos | Débil | Moderado | Fuerte | Pos | Candida albicans |
| 100 | 100 | 43,7 | 47 | 53 | 0 | 0 | 5 | 95 | 0 | 0 | 50 | 24,8 | 28,7 | 28,1 | 100 | 100 |
CLAM: clamidoconidias; CAMP: prueba de Christie, Atkins y Munch-Peterson para detectar Streptococcus agalactiae; AS: agar sangre comercial; PDA: agar papa dextrosa: agar papa dextrosa con suplemento 3 % de glucosa al 3 % y sangre de carnero al 5 %; Coag: prueba de coagulasa; infusión de cerebro-corazón: medio de infusión de cerebro-corazón; Pos: positivo; Neg: negativo; MALDI-TOF: espectometría de masas tipo desorción/ionización láser, asistida por matriz, acoplada a un detector iónico de tiempo de vuelo
En relación con las características de crecimiento en agares artesanales, en el agar romero, se encontró que todas las colonias fueron lisas y sin presencia de hifas periféricas. En el caso del agar tabaco, se encontró que 15 aislamientos (5 %) produjeron colonias de color café; mientras que 285 aislamientos produjeron colonias de color cremablanco. Ningún aislamiento produjo clamidosporas en estos medios (figura 1d y cuadro 1).
Mecanismos de patogenicidad
La prueba de hemólisis se practicó en agar sangre comercial (Ad-bioTM), y agar papa dextrosa (ScharlauTM) suplementado con glucosa y sangre de carnero.
Antes de la selección de los medios, se hicieron pruebas con cepas hemolíticas de S. aureus (ATCC 25923) y C. albicans (ATCC 10231) en agar papa dextrosa suplementado con glucosa y sangre humana, con sangre fresca de carnero y con sangre comercial de carnero, sin que se obtuviera crecimiento del microorganismo bacteriano en ninguno de estos medios.
En agar sangre (Ad-bio™), las colonias de S. aureus crecieron y produjeron hemólisis completa, característica de esta cepa (figura 1e). Sin embargo, para este medio, no se encontraron cepas productoras de hemolisinas y con ningún aislamiento se generó hemólisis o punta de flecha en cocultivo con los aislamientos de S. aureus (figura 1f y cuadro 1).
En todas las diferentes versiones del agar papa dextrosa con sangre y glucosa, hubo crecimiento y hemólisis del microorganismo C. albicans (ATCC 10231). Además, el 50 % de los aislamientos generó hemólisis completa y produjo el factor CAMP (punta de flecha) con el aislamiento de S. aureus (figura 1g).
En relación con la producción de biopelículas, los resultados se apreciaron mejor con 10 % de safranina. De los 300 asilamientos, 246 (82 %) fueron positivos para la formación de biopelículas: 85 aislamientos (28 %) lo hicieron de forma fuerte; 86 (29 %), de forma moderada y 74 (25 %) produjeron una biopelícula débil (figura 1h) (cuadro 1). Todos los aislamientos (300) coagularon el plasma durante las dos primeras horas de incubación (figura 1i y cuadro 1).
Espectometría de masas MALDI-TOF. Todos los aislamientos se identificaron como C. albicans mediante la técnica de MALDI-TOF, utilizando el equipo Vitek MS.
Discusión
Las técnicas de secuenciación de ADN han generado avances en el conocimiento de los microorganismos, lo que ha ocasionado cambios sustanciales en la taxonomía de los hongos. Así, las que antes eran consideradas simples morfoespecies han sido sustituidas por complejos de especies crípticas que difieren en virulencia y en su reacción frente a los antifúngicos, de ahí la importancia de su correcta identificación 26.
En esta investigación, se encontraron aislamientos con distintas tonalidades de color verde en CHROMagar™ Candida. Estos resultados coinciden con los descritos por Vieille et al. en 2022, quien empleó este medio para levaduras del complejo C. albicans y encontró que sus aislamientos mostraban tonalidades verdes con leves diferencias 4.
Igualmente, Ballesté et al. (2005), Alfonso et al. (2010), Odds et al. (2000), Madhavan et al. (2011) y Vecchione et al. (2017), ya habían descrito antes las levaduras de color verde dentro del complejo C. albicans27-31.
En 1998, Sullivan et al. describieron las diferencias entre especies utilizando agar CHROMagar™ Candida: C. albicans produjo colonias de color azul verdoso claro, mientras que C. dubliniensis produjo colonias de color verde oscuro después de 48 horas de crecimiento a 37 °C 32. Estos hallazgos también los describieron Romeo y Criseo, quienes encontraron que C. africana producía colonias con un tono levemente verde 13.
En el presente trabajo, la tonalidad de la colonia no estuvo relacionada con la identificación a nivel de especie. Los hallazgos reportados por otros autores podrían estar relacionados con la preparación del lote del medio, el tiempo de conservación de los aislamientos, etc. 31. Cabe mencionar que tampoco fue posible la diferenciación por tonalidad de las cepas C. albicans, C. africana y C. dubliniensis, empleadas como control.
Por otro lado, la prueba del tubo germinal, conocida también como la de filamentación en suero o filamentación precoz, se consideraba la más sencilla, rápida y económica para la identificación preliminar de los aislamientos del complejo C. albicans18. En el presente trabajo, los resultados de esta prueba variaron según la técnica empleada: los mayores índices de positividad se obtuvieron con suero humano y con caldo de leche. Estos hallazgos son similares a los descritos en 2008 por Pineda et al., quienes obtuvieron una positividad del 93,8 % 21. No obstante, otros estudios, como los de Yazdanpanah y Khaithir (2014), muestran una positividad menor, con variaciones entre el 51 y el 63 % 33.
Los aislamientos de C. albicans no productores de clamidoconidias pueden considerarse infrecuentes y reconocerse como atípicos. En Colombia, Rodríguez-Leguizamón et al. trabajaron con 11 cepas identificadas como C. albicans incapaces de producir clamidosporas y cuyas espectrometrías MALDI-TOF las caracterizaron como C. albicans/C. africana, C. albicans o C. africana. No obstante, todos los aislamientos fueron tipificados molecularmente como C. albicans cuando se amplificó el gen hwp15.
En relación con los medios que se emplean para la formación de clamidoconidias, suelen ser variados e incluyen el caldo con leche diluida 34-36. Aunque la temperatura de incubación para el desarrollo de clamidoconidias suele ser entre 28 y 30 °C, algunos autores han realizado incubaciones hasta los 41 °C 37. Así, el bajo porcentaje de resultados positivos para la formación de clamidoconidias en esta investigación, podría estar relacionado aislamientos.
A propósito del uso del agar tabaco como medio de cultivo para la discriminación rápida entre C. dubliniensis y C. albicans, Pineda et al. (2008) encontraron que el 100 % de los aislamientos de C. dubiniensis produjeron colonias cafés y rugosas, mientras que el 98,5 % de las colonias de C. albicans fueron blancas y lisas 21. A pesar de que la literatura científica indica que las colonias de color marrón amarillento con bordes festoneados son típicas de C. dubliniensis20, nuestros resultados arrojaron 15 aislamientos de color café que fueron identificados como C. albicans por MALDI-TOF. Es importante recordar que ninguno de esos aislamientos había producido clamidoconidias, por lo que, ante la ausencia de estas estructuras, la similitud con C. dubliniensis se limitaba al color de la colonia.
Silveira et al. (2011) también obtuvieron una coloración diferente a la esperada con el agar tabaco: los autores describieron las colonias de C. dubliniensis como de C. albicans, ya que estas presentaron morfotipo y micromorfología típicos de esta última especie 38.
Asimismo, el agar romero fue descrito por De Loreto et al. (2008) para diferenciar entre C. dubliniensis y C. albicans19 y, aunque es escasa la información bibliográfica que documenta su empleo, en esta investigación se encontró que ninguno de los aislamientos produjo colonias rugosas o con clamidoconidias. Curiosamente, 18 aislamientos (6 %) produjeron colonias de color café, pero de contorno liso y sin clamidoconidias, por lo cual se descartó la presencia de aislamientos típicos de C. dubliniensis.
En cuanto a la expresión de mecanismos de patogenicidad, de los 300 aislamientos probados, ninguno fue capaz de producir hemólisis o factor CAMP en el agar sangre preparado y distribuido de forma comercial. Estos resultados son opuestos a los descritos por Pakshir et al., quienes encontraron que todos sus aislamientos (110) produjeron hemólisis completa y casi la mitad de los aislamientos fueron positivos para el factor CAMP con cepas de referencia de S. aureus (43,6 %) y S. agalactiae (49 %) como controles positivos 15.
Por otra parte, en agar papa dextrosa con suplemento de glucosa y sangre de carnero, la mitad de los aislamientos produjo hemólisis completa. Los patrones de hemólisis en las levaduras de este complejo ya se habían descrito anteriormente, con amplia variabilidad en los resultados: Sardi et al. (Brasil), Luo et al. (China) y, recientemente, Nouraei et al. (Irán), encontraron que todos sus aislamientos produjeron hemólisis completa 39-41. Wan et al. (China) informaron que todas sus cepas produjeron hemólisis (sin especificar el tipo de hemólisis), mientras que El Baz et al. (Egipto) y Noori, et al. (Irán), reportaron que la hemólisis fue completa solo en el 79 y el 28 % de los aislamientos estudiados, respectivamente 42-44. Estas variaciones en el patrón de hemólisis podrían estar asociadas con los genotipos circulantes en cada región geográfica.
Todos estos autores emplearon agar Sabouraud dextrosa con 5-7 % de sangre de carnero fresca y 3 % de glucosa. En otro estudio, Maheronnaghsh et al. emplearon sangre humana y obtuvieron hemólisis en más del 70 % de los aislamientos 45.
La ausencia de hemólisis fue descrita en 1994 por Manns et al. al emplear placas de agar Sabouraud o tripticasa de soya con sangre, pero sin enriquecimiento con 3 % de glucosa, o cuando los cultivos se incubaron a temperaturas diferentes a 37 °C 46. No obstante, estas limitaciones se evitaron en esta investigación. Cabe anotar que el papel exacto de las hemolisinas en la infección por hongos no se ha dilucidado por completo 40.
Por otra parte, los resultados en la formación de biopelículas del presente estudio (82 %) fueron similares a los descritos por Pakshir et al.15, quienes encontraron biopelículas en el 62,7 % de los aislamientos; y la producción de biopelículas según su intensidad (débil, moderada fuerte), fue similar a la descrita por Mohammadi et al. en 2021 47.
Las biopelículas han sido descritas como un factor importante de patogenicidad y resistencia en las levaduras 48-49 De hecho, algunos autores consideran que el crecimiento de hifas y la formación de biopelículas son los principales factores que optimizan la progresión de la patogenicidad en Candida spp. 16,41,43. Así, y a pesar de que la formación de biopelículas se asocia más comúnmente con C. albicans, este mecanismo también ha sido descrito en C. glabrata, C. tropicalis, C. parapsilosis y la levadura emergente multirresistente C. auris50-53.
A propósito de la producción de enzima coagulasa como factor de patogénesis, los resultados de esta investigación fueron similares a los descritos por Rodrigues et al.24, quienes no solo describieron la presencia de esta enzima en los aislamientos de C. albicans (88,5 %), sino también, en los de otras especies del género Candida (con excepción de C. krusei), que incluían aislamientos cepas de C. tropicalis (82,6 %) y C. parapsilosis (34,5 %), entre otras. De modo contrario, Seifi et al., en 2015, no encontraron cepas productoras de coagulasa entre más de 100 aislamientos de diferentes especies de Candida (incluyendo 51 de C. albicans), aun con incubaciones de hasta 24 horas 54.
La producción de coagulasa se ha propuesto como una capa de protección frente a los macrófagos y se ha descrito en C. parapsilosis, C. glabrata, C. guilliermondi, entre otras especies del género 24,55.
Finalmente, en esta investigación todos los aislamientos fueron identificados como C. albicans mediante espectrometría de masas MALDI-TOF, lo que sugiere que la circulación y la frecuencia de aislamientos de las especies emergentes C. dubliniensis y C. africana, a partir de colecciones de cultivos o muestras vaginales, son infrecuentes.
Estos resultados coinciden con los obtenidos en 2012 en Nigeria, donde encontraron que solo dos de los 117 aislamientos en una colección de cultivos correspondían a C. africana y no encontraron aislamientos de C. dubliniensis12; o con los descritos por Theil et al., en 2016 en Argentina, quienes determinaron la prevalencia de Candida dubliniensis (1,39 %) y C. africana (0.35 %) en una colección de 287 cepas 1. Asimismo, en 2017, Hazirolan et al. reexaminaron 376 aislamientos identificados como C. albicans y encontraron tres de C. africana y tres de C. dubliniensis, con una prevalencia de 0,8 % 56. Durante ese mismo año, Pakshir et al. realizaron un estudio para discriminar C. africana a partir de 110 cepas identificadas previamente como C. albicans, sin encontrar aislamientos de C. africana15.
Como se mencionó previamente, en el país, Rodríguez-Leguizamón et al. (2015) describieron una colección de aislamientos de C. albicans con variaciones atípicas; no obstante, la identificación molecular reveló correspondencia con C. albicans y los espectros de masas obtenidos por MALDI-TOF (utilizando el equipo MALDI Biotyper - Bruker Daltonics), no fueron concluyentes 5.
En conclusión, la presencia de C. dubliniensis y C. africana es escasa o nula en colecciones de cultivos o aislamientos previamente identificados como C. albicans, y la presencia de mecanismos de patogenicidad en dichos aislamientos es variable.
Debido a que ninguna prueba manual es completamente específica, las pruebas complementarias o comerciales son útiles para discriminar C. albicans y C. dubliniensis. En el caso de C. africana, y por ser una variedad de C. albicans, las pruebas moleculares son la herramienta necesaria para su identificación.














