Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.11 no.2 Bogotá June 2006
Susceptibility/Resistance Genes to Flavivirus, Implications on Disease Severity
JEANETTE PRADAARISMENDY1, JAIME E. CASTELLANOS1,2
1Instituto de Virología, Universidad El Bosque, Bogotá, Colombia.2 Facultad de Odontología, Universidad Nacional de Colombia, Sede Bogotá.
Presentación febrero 28 de 2006, aceptado junio 5 de 2006, correcciones agosto 9 de 2006.
RESUMEN Las infecciones transmitidas por Flavivirus se encuentran entre las enfermedades transmisibles con mayor incidencia en el mundo. La mayoría de ellas se manifiestan clínicamente como un síndrome febril que puede estar o no acompañado de diversos síntomas. La severidad de estas infecciones es variable con casos asintomáticos y otros que pueden llegar a ser letales. La razón de esta variabilidad en la presentación clínica, se desconoce en humanos. En ratones se han identificado cepas susceptibles y cepas resistentes a la infección por algunos Flavivirus. Por clonación posicional se mapeó el gen responsable de la resistencia a virus West Nile en el cromosoma 5 de ratón y se identificó como oligoadenilato sintetasa 1b (Oas1b). Este gen codifica una proteína que sintetiza oligómeros de adenina que activan la RNasaL, que a su vez degrada los RNAs virales. Células provenientes de ratones resistentes a la infección por Flavivirus producen menor cantidad de virus que su contraparte susceptible. Recientemente en humanos, se identificó un polimorfismo asociado con susceptibilidad a infección por virus West Nile en el gen de OasL. Sin embargo, el mecanismo bioquímico y molecular exacto por el cual se produce la susceptibilidad no ha sido completamente dilucidado. Este conocimiento permitiría aclarar aspectos de la fisiopatología de estas enfermedades y enfocar la terapéutica desde un punto de vista más específico.Palabras clave: dengue, oligoadenilato sintetasa, Flavivirus, ratón, susceptibilidad innata.
ABSTRACT
Flavivirus caused infections are among the diseases with the highest incidence in the world. Most of these infections have a wide severity clinical profile, from unspecific fever to lethal hemorrhages and encephalitis. The reason of the clinical variability remains unclear, but it appears to be associated to host genetic features. Susceptible or resistant mouse strains to Flavivirus infection have been identified and the gene responsible for this has been mapped by positional cloning as the West Nile Virus susceptibility mouse gene in chromosome 5, that codifies for Oligoadenylate Synthetase, isoform 1b (OAS1b). OAS produces adenine oligomers that activate endoribonuclease RNAseL to degrade viral RNA. Resistant mice strains produce significantly less virus than susceptible ones. In humans, it has recently been reported a single nucleotide polymorphism associated with susceptibility to West Nile Virus infection in the OASL gene. However, the biochemical or molecular mechanisms that explained this susceptibility are not clear. Further knowledge related with these processes is important for understanding flaviviral pathogenesis and for the design of therapeutic alternatives.
Key words: Dengue, Oligoadenylate Synthetase, Flavivirus, mouse, innate susceptibility.
INTRODUCCIÓN
El dengue y la fiebre amarilla son las principales enfermedades producidas por Flavivirus; ambas causan gran morbilidad y mortalidad en todo el mundo, en particular en países tropicales y subtropicales. Se estima que anualmente ocurren cerca de cien millones de infecciones por dengue y son reportadas más de 20.000 muertes anuales por este virus (WHO, 1997). La fisiopatología de ambas entidades aun presenta muchos vacíos; sin embargo, se han implicado factores tanto virales como del hospedero en el grado de severidad de estas infecciones. En ambas enfermedades ocurre un amplio espectro de manifestaciones clínicas que pueden ir desde infecciones asintomáticas hasta la muerte, pasando por fiebres indiferenciadas, compromiso sistémico severo, encefalitis y síndrome hemorrágico. Las razones por las cuales los Flavivirus causan manifestaciones clínicas solo en un pequeño porcentaje de los individuos infectados no se conocen, pero se sugiere que involucran factores genéticos del hospedero. Por ejemplo, se ha reportado que de los pacientes infectados con el Flavivirus West Nile (WNV, del inglés West Nile virus) solamente cerca del 20% desarrollan un síndrome febril y los pacientes que presentan manifestaciones neurológicas presentan una alta tasa de mortalidad, lo cual sugiere que la susceptibilidad a la infección por WNV podría depender de la constitución genética del paciente u otros factores como la edad, el estado nutricional, el sexo, el genotipo, etc.
LOS FLAVIVIRUS Y SU REPLICACIÓN
La familia Flavivirida es una gran familia de patógenos virales compuesta de tres géneros: Flavivirus, Pestivirus y Hepacivirus (Mukhopadhyay et al., 2005). El género Flavivirus se compone de aproximadamente 73 especies, de los cuales 34 son transmitidos por mosquitos, 17 por garrapatas y 22 no tienen vector conocido. Dentro de este género se encuentran los virus responsables del dengue, la fiebre amarilla, la encefalitis japonesa, encefalitis por virus West Nile y la encefalitis transmitida por garrapatas (Burke y Monath, 2001). Los Flavivirus, de los cuales el virus de fiebre amarilla (VFA) es el prototipo, poseen un ácido ribonucléico de cadena sencilla de polaridad positiva de aproximadamente 11 kb que codifica para diez proteínas, tres estructurales: la proteína de la nucleocápside (C), la proteína asociada a la membrana (prM) y la proteína de la envoltura (E) y siete proteínas no estructurales (NS, non structural): NS1, NS2A, NS2B, NS3, NS4A, NS4B y NS5 (Shu et al., 2003). Una vez los virus se unen a los receptores de membrana en la célula hospedera, ocurre un proceso endocítico. Durante este proceso, la acidificación del medio provoca un cambio conformacional de la proteína E, que pasa de ser un dímero a ser un trímero (Modis et al., 2004). Ésto facilita la fusión de la envoltura viral con la membrana endosómica y se libera así la nucleocápside hacia el citoplasma. Una vez allí, el virus aprovecha la maquinaria de traducción de la célula generando una poliproteína de aproximadamente 3.400 aminoácidos. Esta poliproteína es clivada por proteasas celulares y virales. La traducción de la poliproteína ocurre en el retículo endoplásmico rugoso, lo cual facilita la localización de las proteínas virales dentro y alrededor del retículo para su posterior ensamblaje en viriones maduros. Inicialmente se forman unas partículas virales inmaduras no infecciosas a nivel del retículo endoplásmico, formadas por las proteínas E y prM, además de la nucleocápside y lípidos de membrana. La ruptura proteolítica de prM ocurre en el aparato de Golgi, madurando de esta forma la partícula viral y haciéndola infecciosa. Este virus completo es liberado de la célula por exocitosis para así infectar nuevas células (Diamond, 2003). Algunas proteínas no estructurales forman el complejo de replicasa viral. Este complejo se une a la región 3’ del RNA genómico y copia a partir de la cadena de polaridad positiva, generando cadenas de polaridad negativa, que a su vez generarán copias de RNA de polaridad positiva que tienen tres destinos, pueden servir nuevamente como plantillas transcripcionales, o como plantillas traduccionales o ensamblarse dentro de la nucleocápside y servir como RNA genómico de nuevos viriones. Tras la inoculación de cualquier Flavivirus por un insecto vector, la primera ronda de replicación viral ocurre a nivel de la piel, en las células dendríticas de Langerhans, las cuales una vez activadas por los antígenos virales, migran hacia el nódulo linfático, para infectar otras células, activar linfocitos T e inducir la producción de citoquinas y quimioquinas (Libraty et al., 2001). El virus realiza una segunda ronda de replicación en el tejido linfoide y es llevado hasta la circulación sanguínea, llegando a órganos como el hígado, los riñones, el bazo y aquellos virus neurotrópicos alcanzan el encéfalo. La entrada al sistema nervioso central se da por tres mecanismos principales. El primero es el transporte pasivo a través del endotelio, el segundo es la replicación en las células endoteliales de la barrera hematoencefálica y el tercero es la utilización de células inflamatorias como vehículos (Diamond, 2003).
RESPUESTA ANTIVIRAL INDUCIDA POR LA INFECCIÓN
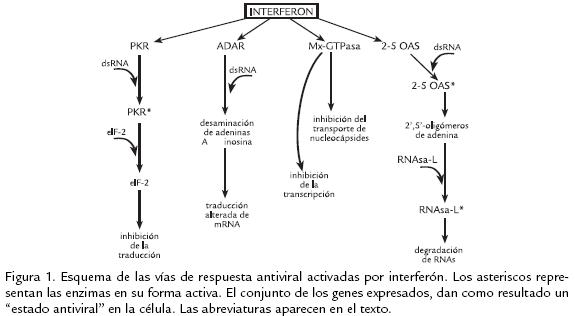
Los virus de dengue y fiebre amarilla infectan monocitos de sangre periférica y macrófagos tisulares. La entrada de los virus a estas células permite la diseminación del virus a diferentes tejidos y la presentación de antígenos virales asociadas a moléculas del complejo mayor de histocompatibilidad. La interacción de estos antígenos con células T de memoria induce su activación y consecuentemente la proliferación y producción de citoquinas, tales como TNF∴(del inglés, tumor necrosis factor), IFN (del inglés, interferon) e IL2 (del inglés, interleukin; Chen y Wang, 2002; ter Meulen et al., 2004). Para el caso del virus de dengue, los complejos anticuerpovirus asociado a estas citoquinas inducen la liberación de factores del complemento como C3a y C5a, los cuales tienen un efecto directo sobre la permeabilidad vascular. Esta secreción masiva de citoquinas y quimioquinas dan lugar al daño endotelial y a la liberación de plasma al espacio extravascular, lo que conlleva a las manifestaciones más severas de la enfermedad. Se ha encontrado efecto citopático de los virus sobre las células hepáticas, lo cual provoca una caída en la síntesis de factores de coagulación, empeorando el cuadro (Lin et al., 2000). Entre todas las citoquinas liberadas durante esta respuesta inmune antiviral, el IFN juega un papel muy importante en el control de la infección por Flavivirus. Existen dos tipos de IFN, los interferones tipo I (IFN ∴, ⊥, θ, ϖ, τ), y tipo II (IFN δ). La señalización activada por el IFN requiere la heterodimerización de las subunidades del receptor para IFN. La unión de los IFN ∴y ⊥a su receptor provoca la fosforilación de las kinasas Jak1 (Janus kinase) y Tyk2 (proteintyrosine kinase), mientras que el IFN δfosforila y activa las kinasas Jak1 y Jak2. La cascada de señalización inducida por IFN ∴/⊥produce la fosforilación de STAT1 y STAT2 (signal transducer and activator of transcription) que se asocian con p48/IRF9 (IFN regulatory factor) formando el complejo ISGF3 (IFN stimulated gene factor3). En el caso de IFN δ, solo se produce la homodimerización de STAT1 fosforilado. Ambos complejos, ISGF3 y el homodímero STAT1, se translocan al núcleo y se unen a elementos de respuesta a IFN en el DNA, denominados ISRE (IFN stimulated response element) en el caso de IFN tipo I, o GAS (gammaIFN activated sites). Estas secuencias (elementos en cis) en el DNA se localizan en los promotores de genes cuya expresión inducirá la inhibición de la replicación viral. Entre estos genes se encuentran la proteína kinasa R (PKR), 2’,5’oligoadenilato sintetasa (OAS), la adenosina deaminasa específica de RNA (ADAR), la proteína de mixoma (MxGTPasa) y proteínas del complejo mayor de histocompatibilidad entre otras (Samuel, 2001). Cada una de las proteínas codificadas por estos genes desencadena un tipo de respuesta antiviral diferente. PKR es una proteína kinasa de RNA, activada por autofosforilación y por unión con RNA de doble cadena; cataliza la fosforilación de eIF2∴(factor de iniciación de la traducción), inhibiendo su actividad y alterando de este modo el intercambio de nucleótidos de guanina catalizado por esta proteína, lo que conlleva a la inhibición de la traducción. Por otro lado, IFN activa transcripcionalmente el gen de la enzima ADAR1, la cual cataliza la desaminación de la adenosina, produciendo inosina. Esto produce una edición del RNA viral generando proteínas no funcionales. Las GTPasas Mx pertenecen a la superfamilia de GTPasas similar a dinamina y tienen dos funciones antivirales hasta ahora descritas, la primera es el bloqueo del transporte de la nucleocápside viral y la segunda es el bloqueo de la síntesis de RNA; las acciones antivirales de Mx han sido descritas claramente en el caso de infección por virus de influenza (familia Orthomyxoviridae; Lindenmann et al., 1963; Fig. 1).
Las enzimas denominadas 2’5’OAS, hacen parte de las enzimas reguladas transcripcionalmente por IFN y su activación depende de un proceso dependiente de RNA de doble cadena, proveniente del proceso replicativo viral o de estructuras secundarias (loops) formadas al interior del RNA genómico viral (Fig. 2). Estas enzimas catalizan la síntesis de 2’,5’oligómeros de adenosina a partir de ATP, que se unen y activan la RNasaL (Ribonuclease L). La RNasaL activada digiere los nucleótidos del extremo 3’ de cualquier RNA en las secuencias UpXp, causando una caída generalizada de RNA tanto virales como celulares. Adicionalmente, se ha demostrado que aparte de las funciones antivirales de la vía 2’,5’OAS, esta enzima está implicada en el control del crecimiento celular, la diferenciación y la apoptosis (Ghosh et al., 2001).
GENÉTICA DE LA RESISTENCIA A Flaviviruse los pacientes infectados por Flavivirus se enferman gravemente, se ha sugerido que existen factores genéticos asociados a la severidad de la patología; sin embargo en humanos no se ha logrado identificar un gen responsable que dé respuesta a este interrogante. En ratones se han descrito cepas resistentes a la infección por Flavivirus, entre ellos la fiebre amarilla y el virus West Nile. La observación de susceptibilidad diferencial a la infección por esta familia de virus viene desde muchos años atrás. El estudio de Lynch y Hughes (1937) identificó a la cepa de ratones Det como resistente a la infección por virus de fiebre amarilla. Simultáneamente Webster (1937) identificó dos cepas de ratón resistentes a la infección con virus de encefalitis de San Luis (otro Flavivirus). Estos trabajos aportaron la primera evidencia experimental de que la constitución genética del hospedero podía determinar la resistencia o susceptibilidad a la infección. En ambos trabajos se pudo establecer que el rasgo de resistencia era un rasgo genéticamente determinado y al parecer mostraba un patrón de herencia autosómico dominante. En 1952, Sabin hizo el reconocimiento de una nueva cepa de ratones resistente a la infección por fiebre amarilla, denominado PRI (Princeton Rockefeller Institute); estos ratones también eran resistentes a la infección por otros Flavivirus (dengue, West Nile, virus de encefalitis japonesa y virus de encefalitis de San Luis). Sabin también hizo experimentos de cruces y retrocruces de prueba que permitieron determinar que el rasgo de resistencia se heredaba con un patrón autosómico dominante. El gen de resistencia se denominó Flvr, mientras que el alelo que confería susceptibilidad se denominó Flvs. En el estudio reportado por Brinton et al. (1982), mediante la utilización de un anticuerpo antiIFN tipo I se observó que el gen Flvr confiere resistencia específica a la infección por Flavivirus mediante un mecanismo aparentemente independiente de la vía de señalización de IFN tipo 1. En este mismo estudio se encontró que las células y los tejidos provenientes de los animales resistentes producen menos partículas virales que su contraparte susceptible. En el trabajo realizado por Sangster et al. (1993) se describieron 2 cepas adicionales llamadas CASA/Rk y CAST/Ei derivadas de Musmusculus castaneus y una cepa de ratones que mostraba resistencia a la cepa vacunal de fiebre amarilla (17D) pero no a una cepa del Flavivirus de la encefalitis del valle de Murray.
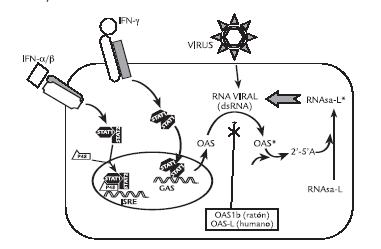
Figura 2. Esquema general del sistema de OAS (Oligoadenilato sintetasa) y su papel en la respuesta antiviral inducida por interferón. Los interferones ∴y ⊥(Tipo 1) se unen a su receptor, fosforilan a STAT1 y STAT2, induciendo su dimerización y la traslocacion al núcleo para, en conjunto con la proteína P48 (complejo ISGF3), reconocer las secuencias en los promotores de los genes de respuesta antiviral (ISRE) y estimular su transcripción. La unión del IFNδcon el respectivo receptor fosforila y causa homodimerización de STAT1, que al traslocarse al núcleo se une a los elementos de respuesta a gammainterferón (GAS) y estimula la transcripción. Una de las proteínas reguladas, es la Oligoadenilato sintetasa (OAS), la cual se activa en presencia de RNAs de doble cadena (dsRNA). La enzima cataliza la polimerización de adeninas, dando como resultado oligonucleótidos de adenina que se unen y activan a la RNAsaL, la cual causa una digestión de RNAs virales y celulares, inhibiendo la replicación viral. Se sugiere, que los genes OAS1b (de ratón) y OASL (de humano), podrían actuar como dominantes negativos en algunos individuos, produciendo una susceptibilidad aumentada a las infecciones por Flavivirus.
El estudio de la localización genómica del gen responsable de la resistencia a la infección por Flavivirus fue iniciado por el grupo de Sangster a principios de los años 90, mediante la realización de análisis de ligamiento de tres puntos utilizando los marcadores genéticos PgmI y rd por un lado, y Guss y EtaI por otro, localizados en el cromosoma 5 de ratón. Utilizando esta estrategia experimental se logró mapear el locus en el cromosoma 5 de ratón (Sangster et al., 1994). Posteriormente en el 2002, dos grupos de investigadores de manera independiente, lograron realizar la clonación posicional del gen responsable del rasgo de susceptibilidad/resistencia en ratones a la infección por WNV. En ambos trabajos se encontró que el rasgo de susceptibilidad a la infección estaba ligado a una mutación en el gen Oas1b. Dicha mutación reemplaza el codón CGA que codifica una arginina, por el codón TGA que codifica un codón de parada. Esta mutación genera una proteína truncada en los ratones susceptibles, en los que no se traduce el dominio Cterminal de la enzima (Mashimo et al., 2002; Perelygin et al., 2002). El grupo de Perelygin, además demostró que la transfección de la forma completa de la proteína en células previamente susceptibles a la infección por WNV, provocaba una disminución en el título viral obtenido comparado con células no transfectadas, además de un retardo en la aparición del efecto citopático. Resultados similares fueron obtenidos por Lucas et al. (2003) utilizando un modelo de células neuronales de ratón.
La familia de 2’,5’OAS tiene un origen remoto, encontrándose en mamíferos, aves, reptiles y en la esponja marina Geodia cydonium. En seres humanos existen cuatro genes de 2’,5’OAS, localizados en la región cromosómica 12q24.2, denominados OAS1, OAS2, OAS3 y OASL1. En el ratón existen ocho genes OAS1, (Oas1aOas1h), Oas2, Oas3 y OasL. Esta familia génica se encuentra organizada en tandem en el cromosoma 5 (Mashimo et al., 2003). En un trabajo reciente realizado por el grupo de Dèspres del Instituto Pasteur (KajasteRudnistki et al., 2006), se logró establecer una línea celular de fibroblastos murinos que expresan o bien la proteína Oas1b completa que se asocia con resistencia a la infección por WNV, o bien la proteína Oas1b truncada que se asocia con susceptibilidad. Se encontró que en el grupo de células que expresan la proteína completa, ocurre una disminución en la acumulación de cadenas positivas de RNA del virus. Además, utilizando un ensayo de RNA de interferencia para silenciar el gen RNasaL, se encontró que esta inhibición de la replicación viral era independiente de la vía clásica OASRNasaL. Esto abre un gran interrogante acerca de cuál es el mecanismo utilizado por la oligoadenilato sintetasa para generar la resistencia a la infección por Flavivirus, específicamente a WNV (Fig. 2)
El grupo de BonnevieNielsen ha venido estudiando la relación que existe entre diabetes mellitus y la actividad de la oligoadenilato sintetasa. Desde finales de los años 70 se ha postulado que la infección viral podría ser un factor desencadenante de enfermedades autoinmunes como la diabetes mellitus, ya que la infección viral desencadena activación de la vía IFN y este grupo de investigadores pensó que OAS podría estar implicado en la patogenia de diabetes mellitus tipo I. En 1989 lograron determinar que linfocitos de sangre periférica de pacientes con diabetes mellitus insulinodependiente (DMID) presentaban una mayor actividad OAS inducida después de la vacunación contra fiebre amarilla (BonnevieNielsen et al., 1989). Posteriormente, encontraron que la oligoadenilato sintetasa, se encontraba persistentemente activada y postularon que esto podría deberse a la estimulación crónica de la enzima o por fallas en el mecanismo de autorregulación. La estimulación podría deberse a un RNA viral que escapa a la acción antiviral innata de la célula y de este modo queda libre para activar enzimas como OAS. Esta incapacidad para eliminar algunos virus puede ser determinada genéticamente, lo que haría que algunos individuos fuesen más susceptibles a la infección y por ende al desarrollo de enfermedades autoinmunes, específicamente DMID (BonnevieNielsen et al., 2000).
Recientemente este mismo grupo (BonnevieNielsen et al., 2005), describió una asociación entre actividad basal de OAS y algunos polimorfismos de esta enzima. El gen OAS1 humano, muestra un polimorfismo en el sitio de empalme del exón 7. La existencia de una guanina (G) genera el sitio de empalme y de esta forma se produce la isoforma p46 de OAS1. Una adenina en este mismo lugar elimina el empalme generando 2 isoformas p52 y p48. p48 tiene un dominio BH3 similar a los encontrados en miembros de la familia Bcl2 y al parecer cumple con funciones proapoptóticas (Ghosh, 2001). En el trabajo de BonnevieNielsen se demostró que el alelo G se encontraba correlacionado con actividad enzimática de OAS alta, mientras que el alelo A con actividad baja. Posteriormente se hizo la correlación con el fenotipo de DMID y encontraron que el alelo G se encuentra en mayor frecuencia en pacientes con DMID, lo que se correlaciona con los datos obtenidos previamente donde estos pacientes tienen mayor actividad de la enzima (Field et al., 2005). Cruzando estos datos con los obtenidos con anterioridad que relacionan la DMID con infección por algunos enterovirus, los autores proponen que esta actividad se encuentra elevada porque la acción antiviral de OAS en estos individuos no es suficiente para eliminar este tipo de virus.
Investigar los polimorfismos genéticos de la región ortóloga humana podría llegar a explicar al menos en algunos casos, la susceptibilidad diferencial a la infección por Flavivirus y las diferentes manifestaciones patológicas en individuos expuestos en regiones endémicas. De hecho, recientemente en humanos se ha investigado la relación entre la infección por Flavivirus y los genes de la familia OAS. En humanos, existen cuatro genes OAS, OAS1, OAS2, OAS3, OASL1, éste último produce una proteína enzimáticamente inactiva, ya que a pesar de poseer los 5 exones de los otros genes OAS, no posee dos de tres residuos de ácido aspártico en algunas cadenas ⊥que son importantes para su actividad catalítica, además posee mutaciones en el asa P que participa en la unión al ATP (Yakub et al., 2005). En el reporte de Yakub et al. (2005), se tomaron pacientes diagnosticados con enfermedad por WNV y se realizó la secuenciación completa de los exones de los cuatro genes que componen la familia OAS y de su efector corriente abajo (RNAsaL). Se logró identificar un polimorfismo de nucleótido único (SNP, single nucleotide polymorphism) en el exón 2 del gen OASL, localizado en un sitio potenciador de empalme (exon splicing enhancer); estos sitios facilitan la actividad del complejo ribonucleoprotéico empalmador, haciendo más eficiente el proceso de empalme del RNA. Los pacientes con formas severas de la infección presentan con mayor frecuencia este SNP, lo que provocaría una mayor tasa de transcripción de este RNA, que se reflejaría en una mayor cantidad de la enzima OASL, que al ser enzimáticamente inactiva, podría actuar como dominante negativo de las isoformas OAS activas. Los autores sugieren que OASL pudiera ser el ortólogo de Oas1b murino, al menos en cuanto a su relación con la susceptibilidad a la infección por WNV.
A pesar de los estudios realizados hasta el momento, no se ha encontrado un gen responsable de la susceptibilidad que algunos individuos tienen frente a la infección por Flavivirus. Sin embargo, los trabajos realizados apuntan hacia genes estimulados por IFN, específicamente de la familia OAS. Es necesario continuar la investigación en este campo para determinar si definitivamente es uno de estos genes el implicado en la susceptibilidad a la infección en humanos, y si es así, cual es la vía molecular y bioquímica utilizada para generar resistencia en los individuos que portan los alelos de resistencia. Este conocimiento permitiría diseñar estrategias terapéuticas novedosas para los individuos que portan los alelos de susceptibilidad y que se infectan con alguno de estos Flavivirus.
AGRADECIMIENTOS
Esta revisión se hizo dentro del marco del Proyecto No. 13080517588 cofinanciado por Colciencias y la División de Investigaciones de la Universidad El Bosque.
BIBLIOGRAFÍA
BONNEVIENIELSEN V, LARSEN ML, FRIFELT JJ, MICHELSEN B, LERNMARK A. Association of IDDM and Attenuated Response of 2’,5’Oligoadenylate Synthetase to Yellow Fever Vaccine. Diabetes. 1989;38:16361642.
BONNEVIENIELSEN V, MARTENSEN PM, JUSTESEN J, KYVIK KO, KRISTENSEN B, LEVIN K, et al. The Antiviral 2’,5’Oligoadenylate Synthetase is Persistently Activated in Type 1 Diabetes. Clin Immunol. 2000;96:1118.
BONNEVIENIELSEN V, FIELD LL, LU S, ZHENG DJ, LI M, MARTENSEN PM, et al. Variation in Antiviral 2’,5’Oligoadenylate Synthetase (2’5’OAS) Enzyme Activity is Controlled by a SingleNucleotide Polymorphism at a SpliceAcceptor Site in the OAS1 Gene. Am J Hum Genet. 2005;76:623633.
BRINTON M, ARNHEITER H, HALLER O. Interferon Independence of Genetically Controlled Resistance to Flaviviruses. Infect Immun. 1982;36:284288.
[ Links ]BURKE DS, MONATH TP. Flaviviruses. En: Knipe DM, Howley PM, Griffin DE, Lamb RA, Martin MA, Roizman B, et al., editors. USA: Fields Virology; 2001.
[ Links ]CHEN YC, WANG SY. Activation of Terminally Differentiated Human Monocytes /Macrophages by Dengue Virus: Productive Infection, Hierarchical Production of Innate Cytokines and Chemokines, and the Synergistic Effect of Lipopolysaccharide. J Virol. 2002;76:98779887.
[ Links ]DIAMOND MS. Evasion of Innate and Adaptive Immunity by Flavivirus. Immunol Cell Biol. 2003;81:196206.
[ Links ]FIELD LL, BONNEVIENIELSEN V, POCIOT F, LU S, NIELSEN TB, BECKNIELSEN H. OAS1 Splice Site Polymorphism Controlling Antiviral Enzyme Activity Influences Susceptibility to Type 1 Diabetes. Diabetes. 2005;54:15881591.
[ Links ]GHOSH A, SARKAR SN, ROWE T, SEN GC. A Specific Isozyme of 2’5’ Oligoadenylate Synthetase Is a Dual Function Proapoptotic Protein of the Bcl2 Family. J Biol Chem. 2001;276:2544725455.
KAJASTERUDNISTKI A, MASHIMO T, FRENKIEL MP, GUENET JL, LUCAS M, DESPRES P. The 2’,5’Oligoadenylate Synthetase 1b is a Potent Inhibitor of West Nile Virus Replication Inside Infected Cells. J Biol Chem. 2006;281:46244637
LIBRATY DH, PICHYANGKUL S, AJARIYAKHAJORN C, ENDY TP, ENNIS FA. Human Dendritic Cells are Activated by Dengue Virus Infection: Enhancement by Gamma Interferon and Implication for Disease Pathogenesis. J Virol. 2001;75:35013508.
[ Links ]LIN YL, LIU CC, LEI HY, YEH TM, LIN YS, CHEN RM, et al. Infection of Five Human Liver Cell Lines by Dengue2 Virus. J Med Virol. 2000;60:425431. [ Links ] LINDENMANN, J, LANE CA, HOBSON D. The Resistance of A2G Mice to Myxoviruses. J Immunol. 1963;90:942951. [ Links ] LUCAS M, MASHIMO T, FRENKIEL MP, MONTAGUTELLI X, SIMONCHAZOTTES D, CECCALDI PE, et al. Infection of Mouse Neurones by West Nile Virus
is Modulated by the InterferonInducible 2’5’Oligoadenylate Synthetase 1b Protein. Immunol Cell Biol. 2003;81:230–236.
LYNCH CJ, HUGHES TP. The Inheritance of Susceptibility to Yellow Fever Encephalitis in Mice. Genetics. 1937;21:104112.
[ Links ]MASHIMO T, LUCAS M, SIMONCHAZOTTES D, FRENKIEL MP, MONTAGUTELLI X, CECCALDI PE, et al. A Nonsense Mutation in the Gene Encoding 2’5’Oligoadenylate Synthetase/L1 Isoform is Associated with West Nile Virus Susceptibility in Laboratory Mice. Proc Natl Acad Sci U S A. 2002;99:1131111316.
MASHIMO T, GLASER P, LUCAS M, SIMONCHAZOTTES D, CECCALDI PE, MONTAGUTELLI X, et al. Structural and Functional Genomics and Evolutionary Relationships in the Cluster of Genes Encoding Murine 2’,5’Oligoadelylate Synthetases. Genomics. 2003;82:537552.
MODIS Y, OGATA S, CLEMENTS D, HARRISON SC. Structure of the Dengue Virus Envelope Protein After Membrane Fusion. Nature. 2004;427:31319.
[ Links ]MUKHOPADHYAY S, KUHN RJ, ROSSMANN MG. A Structural Perspective of the Flavivirus Life Cycle. Nat Rev Microbiol. 2005;3:1322.
[ Links ]PERELYGIN AA, SCHERBIK SV, ZHULIN IB, STOCKMAN BM, LI Y, BRINTON MA. Positional Cloning of the Murine Flavivirus Resistance Gene. Proc Natl Acad Sci U S A. 2002;99:93229327.
[ Links ]SABIN A. Nature of Inherited Resistance to Viruses Affecting the Ner Vous System. Proc Natl Acad Sci U S A. 1952;38:540546.
[ Links ]SAMUEL CE. Antiviral Actions of Interferons. Clin Microbiol Rev. 2001;14:778809.
[ Links ]SANGSTER MY, HELIAMS DB, MACKENZIE JS, SHELLAM GR. Genetic Studies of Flavivirus Resistance in Inbred Strains Derived from Wild Mice: Evidence for a New Resistance Allele at the Flavivirus Resistance Locus (Flv). J Virol. 1993;67:340347
[ Links ]SANGSTER MY, UROSEVIC N, MANSFIELD JP, MACKENZIE JS, SHELLAM GR. Mapping the Flv Locus Controlling Resistance to Flaviviruses on Mouse Chromosome 5. J Virol. 1994;68:448452.
[ Links ]SHU PY, CHANG SF, KUO YC, YUEH YY, CHIEN L, SUE CL, et al. Development of Group and SerotypeSpecific OneStep SYBR Green IBased RealTime Reverse TranscriptionPCR Assay for Dengue Virus. J Clin Microbiol. 2003;41:24082416.
[ Links ]TER MEULEN J, SAKHO M, KOULEMOU K, MAGASSOUBA N, BAH A, PREISER W, et al. Activation of the Cytokine Network and Unfavorable Outcome in Patients with Yellow Fever. J Infect Dis. 2004;190:18211827.
[ Links ]WEBSTER LT. Inheritance of Resistance of Mice to Enteric Bacterial and Neurotropic Virus Infections. J Exp Med. 1937;65:261286.
[ Links ]WORLD HEALTH ORGANIZATION. Dengue Haemorrhagic Fever: Diagnosis, Treatment, Prevention and Control, 2nd Edition. Geneve; 1997.
[ Links ]YAKUB I, LILLIBRIDGE KM, MORAN A, GONZALEZ OY, BELMONT J, GIBBS RA, et al. Single Nucleotide Polymorphisms in Genes for 2’5’Oligoadenylate Synthetase and RNase L in Patients Hospitalized with West Nile Virus infection. J Infect Dis. 2005;192:17411748.





![Ribonucleases: [subtitle]Theurapetical potential on Viral Infections](/img/en/next.gif)