Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.12 no.1 Bogotá Jan./June 2007
UN SISTEMA SENCILLO Y DE BAJO COSTO PARA LA RECONSTRUCCIÓN TRIDIMENSIONAL DE ESTRUCTURAS EMBRIONARIAS A PARTIR DE CORTES SERIADOS
A Simple and Cheap 3D Reconstruction System of Embryonic Structures from Serial Sections
JOHANNA SUÁREZRAIRÁN1, B.Sc., LAURA CÓRDOBAPARRADO1, B.Sc., HERNÁN HURTADOGIRALDO1, Ph. D. 1 Universidad Militar Nueva Granada, Facultad de Ciencias Básicas, Programa de Biología Aplicada. Carrera 11 No.10180, Bogotá, Colombia. hhurtado@umng.edu.co
Presentado 5 de octubre de 2005, aceptado 3 de abril de 2006, correcciones 15 de febrero de 2007.
RESUMEN
En este trabajo se muestra el montaje y utilización de un sistema de reconstrucción tridimensional en estructuras embrionarias, utilizando equipos sencillos, de fácil adquisición y uso, utilizando como ejemplo la inervación del miembro posterior del embrión de rata de 19 días de gestación. Además, se muestra cómo pueden lograrse resultados de buen nivel, en cuanto a la forma, ubicación y distribución de las estructuras de interés, obteniendo adicionalmente información cuantitativa acerca de la forma y tamaño (volumen y área superficial), a partir de la utilización de software gratuitos de libre acceso en internet.
Palabras clave: reconstrucción tridimensional, histología, embrión de rata, inervación, miembro posterior.
ABSTRACT
This work explaines how to setup and use a very cheap and simple 3D reconstruction system, for embryonic structures, using as an example the innervation of E19 hindlimb rat embryo. It also shows how results of good level can be achieved, such as, form, location and distribution of the structures of interest, obtaining additionally quantitative data of the size (volume and superficial area), and form. All the results mentioned above were obtained from the operation of free software available at the Internet.
Key words: 3D reconstruction, histology, rat embryo, innervation, hind limb.
INTRODUCCIÓN
GENERALIDADES
La enseñanza y la investigación en histología, microscopía y embriología, tradicionalmente ha seguido un esquema eminentemente descriptivo. Sin embargo, cada vez es más importante la introducción de componentes cuantitativos en estas disciplinas. Para obtener esta información a partir de imágenes, se debe utilizar sistemas que permitan preparar dichas imágenes para la extracción de esa información. Es así como se define el procesamiento de imágenes como el conjunto de herramientas y procedimientos que permiten la extracción de información cuantitativa a partir de imágenes (Wootton, 1995). En el contexto de la microscopía, se requiere un sistema que contenga los siguientes elementos:
- —
- Sistema para capturar las imágenes.
- —
- Sistema para almacenar las imágenes.
- —
- Sistema para procesar las imágenes.
- —
- Sistema para producir una resultante de imágenes.
Sin embargo, la histología tradicional, aún sumándole herramientas sofisticadas como el análisis de imágenes solo nos da información bidimensional sobre estructuras que son de naturaleza tridimensional. Esto implica necesariamente pasar a un siguiente nivel en el cual se puedan generar imágenes volumétricas de las estructuras bajo estudio, generando una reconstrucción tridimensional, que pueda ser visualizada desde cualquier ángulo y perspectiva, y analizada cuantitativamente para obtener datos como volumen, área superficial, y relaciones espaciales entre los diferentes componentes de la estructura bajo estudio.
RECONSTRUCCIÓN TRIDIMENSIONAL EN MICROSCOPÍA
La reconstrucción tridimensional implica el ensamblaje de la información contenida en planos bidimensionales de orientación conocida en un conjunto coherente y comprensible de imágenes tridimensionales. Ya que se trata de trabajar con objetos que tienen una estructura interna visible solamente a nivel microscópico, una de las alternativas para conocer esta estructura es la producción de cortes seriados o semiseriados. Cuando se lleva a cabo este proceso, debe tenerse en cuenta que se destruye la integridad 3D del objeto; puede ocurrir una distorsión debida a la compresión por el corte y al proceso de montaje. Por lo tanto un buen sistema de reconstrucción tridimensional debe poseer herramientas que permitan la corrección de estos problemas, al menos hasta un nivel aceptable. Adicionalmente, debe permitir la obtención de información cuantitativa de las estructuras reconstruídas, como por ejemplo superficie y volumen.
A nivel de tejidos y órganos el conocimiento de la tercera dimensión es importante para un mejor entendimiento e interpretación de los resultados de una investigación (Verbeek, 2000). Un caso que ilustra perfectamente esta afirmación es el desarrollo embrionario, en el cual ocurren cambios en la organización y relación espacial existente entre las estructuras implicadas en dichos procesos en lapsos de tiempo que pueden ser muy cortos (Whiten et al., 1998).
El objetivo de este trabajo, es mostrar por medio de un ejemplo práctico (la reconstrucción parcial de la inervación de un botón de miembro de embrión de rata), cómo puede lograrse una reconstrucción tridimensional de una estructura compleja, con equipos sencillos, acoplados a una computadora personal de rango medio, y utilizando software gratuito de libre acceso en internet. Esto permite incorporar este tipo de metodologías a la actividad docente e investigativa, sin requerir equipos o software altamente sofisticados y sumamente costosos (Ramm, 1994).
MATERIALES Y MÉTODOSPROCESAMIENTO DE MUESTRAS
Se trabajó con embriones de la cepa Wistar de 13, 14, 17 y 19 de gestación. Para este artículo solo será mencionado como ejemplo el embrión de 19 días de gestación. Los embriones fueron procesados siguiendo las normas éticas utilizadas en el manejo de animales de laboratorio (Beaver et al., 2001). Para el procesamiento de los embriones se siguió el protocolo propuesto por Prophet et al. (1995) y Nakao e Ishizawa (1994). Se realizó una disección para obtener miembros posteriores derechos, que incluyeran una parte de columna vertebral. Se les hizo un lavado en amortiguador fosfato tres veces durante 10 min cada uno. La deshidratación se hizo con concentraciones crecientes de etanol, 50, 70, 90, 95 y 100%, y finalmente la impregnación en parafina que incluye baños en Xilol y tres baños en parafina de una hora cada uno (Córdoba y Suárez, 2005). Se realizaron cortes seriados de 10 µm de espesor cada uno. Los cortes fueron realizados en disposición transversal con un micrótomo rotatorio MICROM Laborgeräte GMBH HM340E. Se utilizó la coloración de hematoxilinaeosina usando el protocolo propuesto por Hurtado (1990). Después de la coloración de los tejidos se realizaron los montajes.
ANÁLISIS DE IMÁGENES
A partir de los cortes obtenidos, se realizó una reconstrucción tridimensional de la distribución de las fibras nerviosas y se estudiaron los tejidos relacionados con los nervios en crecimiento (Bogush y Dierichs, 1995). Para este efecto se realizó la digitalización y reconstrucción tridimensional a partir de un sistema de análisis de imágenes sencillo que comprende los siguientes elementos: Sistema de captura de imágenes. Consta de un microscopio Intel QX3 con una magnificación óptica de 60X y su respectivo software (QX3TM Microscope software; Fig. 1A), el cual permite cambiar las imágenes en escala de grises. El microcopio tiene una resolución de 352x288 pixeles, mientras que el software tiene una resolución de 320x240 pixeles, presentando las imágenes digitalizadas en la ventana del software una resolución final de 512x384 pixeles (Intel Corporation, 2005). Se utilizó un computador Pentium III, con 256 Mbytes de RAM, un disco duro de 20 Gbytes y un monitor de 15 pulgadas. Se digitalizaron cortes seriados y se almacenaron en formato BMP. Cuando los cortes a digitalizar eran muy grandes, fue necesaria la digitalización de varias imágenes para lograr un barrido total del corte. Procesamiento de imágenes. El procesamiento se realizó con el fin de destacar de forma más clara las principales estructuras (cartílago, ganglios sensoriales, médula espinal y nervios). A continuación se crearon los montajes en ventanas de 1.000x1.000 pixeles, se delinearon las estructuras a reconstruir con el programa Scion image (Figs. 1CD), distribuído gratuitamente por la compañía Scion Corporation (www.scioncorp.com). Este software es una versión para PC del NIH Image de Macintosh. Debe anotarse que aunque la detección y marcado de las estructuras previas se hizo manualmente los datos obtenidos por dos personas fueron muy similares con una variación no mayor a 5% en las estructuras de mayor tamaño y alrededor de 10% para pequeños haces de fibras. Para disminuir el error en la delimitación de los haces de fibras se controló su ubicación comparando la imagen digitalizada con la observación directa del micropreparado en un microscopio binocular Zeiss Axiostar básico (Figs. 1BC). Utilizando magnificaciones de 50X, 100X y 400X se dibujaron en la imagen los haces nerviosos y se delimitaron otras estructuras como los primordios cartilaginosos de los huesos, médula espinal, ganglios sensoriales (Fig. 1D). Estas imágenes modificadas se grabaron nuevamente en formato BMP. Para el proceso de reconstrucción se utilizó una serie de programas (Convert, Align, Trace) que hacen parte de un paquete para reconstrucción 3D elaborado por Fiala et al. (1998). Fue necesario convertir las imágenes a un formato BMP reconocido por los programas Align y Trace usando el programa Convert versión 1.11b de distribución gratuita en el sitio http:// synapses.bu.edu/tools/ (Fig. 1E).
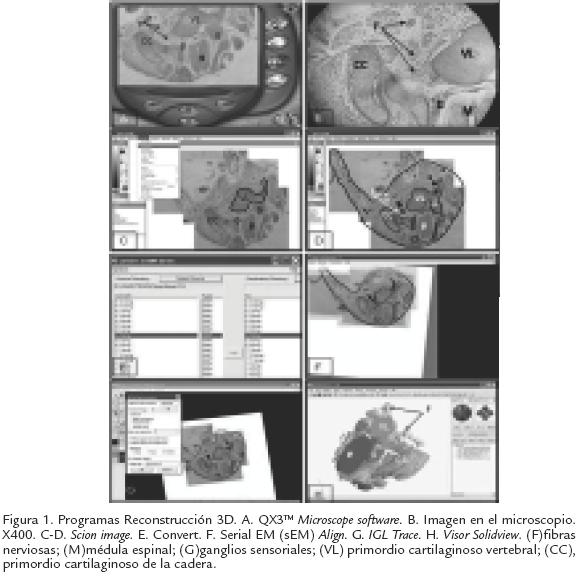
Alineación de imágenes. Utilizando el software serial EM (sEM) Align versión 1.26b (Fig. 1F), se importaron las imágenes, generado una serie. A continuación, y tomando como referencia los haces marcados se procedió a la alineación de cada una de las estructuras bajo estudio: médula espinal, ganglios nerviosos y cartílago que fueron fáciles de identificar, delimitar y seguir en los diferentes cortes. Este programa de distribución gratuita se encuentra en http://synapses.bu.edu/tools/. El programa de alineación también se utilizó para incorporar una imagen digitalizada a 60x con el QX3 de una reglilla milimétrica al inicio de cada serie de imágenes.
Trazado de estructuras. Se usó el software IGL Trace versión 1.26b de libre distribución (Fig. 1G). Permitió delimitar las regiones correspondientes a los nervios y las otras estructuras bajo estudio y generar el archivo de texto que contiene las coordenadas de las estructuras que van a utilizarse para la reconstrucción tridimensional. En este programa también se realizó la calibración de las imágenes y para este efecto se utilizó la imagen de la reglilla milimétrica. Una ventaja adicional de este archivo es que su tamaño es relativamente pequeño comparado con los archivos de imágenes. Visualización de imágenes 3D. Finalmente las reconstrucciones se visualizaron con un programa Visor para realidad virtual (Solidview; Fig. 1H), también de distribución gratuita (ver el mismo sitio). Este programa permite visualizar simultáneamente varias estructuras (series), generadas con el programa Trace. Los programas Trace y Visor Solidview permiten obtener información cuantitativa de la estructura reconstruída, como son el volumen y el área superficial, junto con información acerca de las propiedades espaciales relacionadas a esta.
RESULTADOSA partir de la utilización de los programas ya descritos (Fig. 1AH) se obtuvieron representaciones tridimensionales parciales de la inervación del embrión de 19 días de gestación. Se ilustran reconstrucciones de nervios periféricos, médula espinal y cartílago, obtenidas de cortes seriados (ver materiales y métodos). Cabe resaltar que cuando se realiza una reconstrucción con cortes semiseriados, se presentan demasiados saltos y discontinuidades (datos no mostrados). Por lo tanto es mejor trabajar con cortes seriados para evitar estos tropiezos.
RECONSTRUCCIÓN A PARTIR DE CORTES SERIADOS
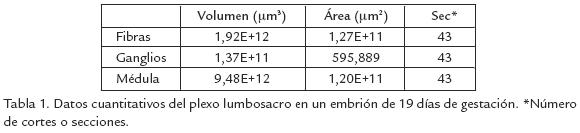
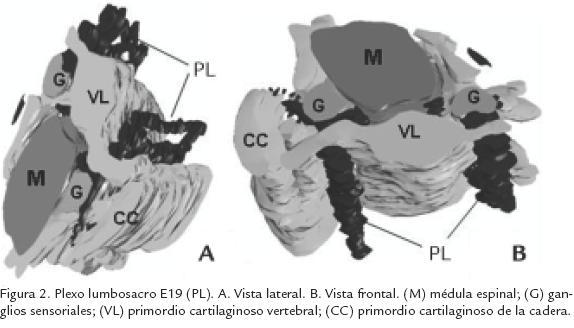
Se tomaron 30 cortes seguidos que correspondían al nivel del tronco y comienzo del miembro posterior. Como se observa en la figura 2A, la reconstrucción de las fibras nerviosas y la médula espinal muestra buena continuidad, sin demasiados “saltos” ni deformaciones, lo que lleva a pensar que el nivel de distorsión introducido por el procesamiento de los tejidos no fue muy grande. Algo similar puede decirse del cartílago, en cuyo caso, se detectó perfectamente la cavidad donde se aloja la médula espinal, al igual que no se presentó un cruce de imágenes entre cartílago y fibras, corroborando así lo que se observa por procedimientos histológicos tradicionales, que muestran que las fibras nerviosas no penetran el cartílago (Fig. 2B). En la tabla 1 se muestran los datos cuantitativos obtenidos a partir del programa IGL Trace.
DISCUSIÓN
En la actualidad la reconstrucción tridimensional de estructuras seccionadas es una herramienta indispensable en la investigación anatómica y embriológica; esto debido a que los organismos en etapa embrionaria experimentan diversas transformaciones en cuanto a su forma y a su desarrollo (Budantsev y Jakovlev, 2000; Fiala y Harris, 2002; Weninger et al., 1998). El potencial de los gráficos 3D radica en la capacidad de permitir la reconstrucción de partículas para ser vistas desde diferentes ángulos y perspectivas. Además, permite obtener datos cuantitativos acerca de la estructura reconstruída, como son el volumen y el área superficial, junto con información sobre las propiedades espaciales relacionadas a esta (Budantsev y Jakovlev, 2000; Fiala y Harris, 2002; Fiala et al., 1998; Lindemann, 2001; Segev y London, 2000; Shepherd et al., 1998; Shum et al., 2003; Teng y Wilkinson, 2000; Ventura y Harris, 1999; Weninger et al., 1998; Whiten et al., 1998). Las partículas que experimentan modificación estructural son las más adecuadas para esta técnica, ya que los cambios morfológicos dinámicos pueden ser claramente apreciados (Budantsev y Jakovlev, 2000). En el ejemplo citado en este artículo, esta herramienta ha sido de gran utilidad ya que permite observar las relaciones topográficas que se presentan a lo largo del desarrollo entre los nervios y las estructuras que lo rodean, aspecto importante en este caso, para comprender la ruta de inervación que se presenta en el miembro posterior del embrión de rata y que solo puede ser conocido en su contexto tridimensional.
Un gran obstáculo para la masificación del uso de los sistemas de reconstrucción tridimensional existentes es que en general requieren programas y equipos sofisticados, cuyos costos tienden a ser muy altos, además de ser muy complejos en su manipulación. En nuestro país, esta combinación de factores impide que muchas entidades como colegios y universidades dispongan de ellos. El método usado para esta reconstrucción esta basado en las imágenes bidimensionales obtenidas de los cortes seriados realizados. Aunque se requirió un proceso de digitalización, orientación, alineación y rectificación, necesarias en las técnicas de reconstrucción no episcópicas (Budantsev y Jakovlev, 2000; Fiala y Harris, 2002; Weninger et al., 1998), esto no lo hace menos útil, ya que el tiempo utilizado en el procesamiento, el bajo costo y la reconstrucción de las estructuras estudiadas (como son los nervios, la médula espinal, el cartílago y los ganglios sensoriales) hace de ésta, una técnica adecuada y accesible para estudios de investigación morfológica y embrionaria y para crear modelos de enseñanza.
Nuestra contribución, adicionalmente al mostrar y divulgar la existencia de dichos programas, es la de ilustrar, que aunque con limitaciones, pueden servir para trabajos que se realicen con técnicas de amplia difusión tales como la histotécnica clásica en parafina, y a partir de equipos de digitalización muy sencillos, de fácil manejo y bajo costo como el microscopio digital Intel QX3. La microtomía de bloques impregnados en parafina es sencilla, su costo es relativamente bajo y permite la obtención de cortes seriados de forma muy rápida. Adicionalmente, las coloraciones clásicas de hematoxilina y eosina, producen una buena tinción general que permite identificar estructuras importantes como cartílago, tejido nervioso, músculo, piel, y vasos sanguíneos. No puede negarse que aún con estas ventajas, este sistema de parafina tiene sus inconvenientes, ya que puede llevar a distorsión de los tejidos, como en el caso de una mala deshidratación de los tejidos, o un exceso de extensión de los cortes. Sin embargo, los programas utilizados tienen herramientas que permiten resolver de forma bastante adecuada esos problemas.
En cuanto a los equipos, es particularmente interesante subrayar el uso del microscopio digital Intel QX3. Este equipo, producto de una asociación temporal entre el gigante de la electrónica Intel y el gigante de los juguetes Mattel, fue comercializado (aparentemente sin éxito) como un juguete tecnológico. Sin embargo, su desempeño, y su costo ($150.000200.000 en el mercado), lo convirtieron en un equipo de laboratorio muy interesante. Es cierto, una cámara digital con los aditamentos para digitalización de imágenes al microscopio, genera imágenes de mejor calidad. Sin embargo, su costo es mucho mayor, y tal inversión no siempre es posible o justificable para cierto tipo de usos que no requieren de un gran detalle. Este es el caso del ejemplo que se cita, ya que tanto los grandes haces nerviosos, como la médula, y el cartílago, son perfectamente distinguibles con este equipo. Adicionalmente, es de muy fácil uso y requiere muy poco entrenamiento. Aunque en la actualidad este microscopio está descontinuado, en su reemplazo se puede encontrar el microscopio digital QX5, el cual posee las mismas características del QX3 pero mejoradas, como es el caso de la resolución (640x480), entre otras, y cuyo costo es también bastante bajo.
En la actualidad, el sistema de reconstrucción tridimensional expuesto en este artículo, está siendo usado en la Universidad Militar Nueva Granada, en investigaciones como: cuantificación del tamaño de los oocitos en varias especies de vertebrados (peces); reconstrucción de testículos de poecilidos para determinar los paquetes de espermatozoides que se encuentran; mediciones de cambio de volumen de la hipófisis de cachama blanca a diferentes momentos de la curva de crecimiento comenzando desde alevinos; obtención de volúmenes de ganglios dorsales para la relación de tamaño y sitios de ubicación, y observar los diferentes sitios de ubicación en diferentes especies de peces; entre otros.
En cuanto a la selección del uso de cortes seriados o semiseriados, esta decisión dependerá del tipo de estructura bajo estudio. En el curso de este trabajo se observó que solamente al utilizar cortes seriados se evitaba la introducción de discontinuidades que deforman de manera importante la estructura reconstruida. Por lo tanto, puede afirmarse que en casos en los cuales las estructuras muestren variaciones significativas en distancias pequeñas, es mejor utilizar cortes seriados, de lo contrario puede pensarse en trabajar con cortes semiseriados.
Hablando específicamente de la calidad de los resultados de reconstrucción, es importante señalar que en general no se presentan discontinuidades importantes cuando se trabaja con cortes seriados. En algunos casos en los haces nerviosos más pequeños pueden aparecer discontinuidades, y probablemente esto se deba, no a defectos del programa, sino a fallas en la detección de los haces en el corte, o a una deformación excesiva de éste.
En lo referente al análisis de imágenes, las funciones que provee cada programa tienen una gran variedad de herramientas que permiten procesar las imágenes digitalizadas mejorando su calidad para así evitar posibles distorsiones en el procedimiento manual de las estructuras bajo estudio. Tal es el caso del programa Scion Image, el cual brinda una serie de herramientas que ayudan a mejorar la imagen (nitidez, contrate, entre otras) haciendo relativamente fácil el marcaje de las estructuras de interés, que es uno de los procedimientos más importantes, y de cuya exactitud depende la buena calidad de la imagen 3D que se vaya a generar. Adicionalmente, el formato con el cual se trabajó (.bmpWindows Bitmap formats, siendo este un formato de archivo de imagen originado en Windows), puede ser expuesto en cualquier dispositivo relacionado con Windows, guardando así todas las propiedades de la imagen independiente del dispositivo en donde se observe (Ying et al., 1999).
En conclusión, con las reconstrucciones 3D, se logra no solo corroborar sino observar de forma más precisa, lo descrito en la parte histológica, en cuanto a la forma, ubicación y distribución de las estructuras de interés, obteniendo adicionalmente información cuantitativa acerca de la forma y tamaño (volumen y área superficial) de cada una de las estructuras, al igual que permite demostrar la relación espacial en este ejemplo, entre las fibras nerviosas y las estructuras relacionadas y mostrar los cambios morfológicos y la interacción que se presenta entre estos a lo largo del desarrollo embrionario. Conjuntamente, este tipo de montaje, de fácil adquisición, permitirá en muchas entidades educativas de nuestro país (como colegios y universidades), acceder a la tecnología de la reconstrucción tridimensional a partir de muestras microscópicas a un bajo costo, empleando programas de computador que son relativamente fáciles de aprender a usar, y de libre adquisición, y a un nivel bastante aceptable, tanto para la docencia como para ciertos tipos de investigación.
AGRADECIMIENTOS
Agradecemos a la Universidad Militar Nueva Granada por el apoyo financiero y tecnológico proporcionado a este proyecto (código del proyecto: CIAS 2002008) y cuyos resultados pertenecen.
BIBLIOGRAFÍA
BEAVER B, REED W, LEARY S, MCKIERNAN B, BAIN F, et al. Report of the AVMA Panel on Euthanasia J Am Vet Med Assoc. 2001;218(5):669696.
[ Links ]BOGUSH G, DIERICHS R. Outgrowing Nerves in the Foreleg of a Mouse Embryo Viewed by ThreeDimensional Reconstruction from Electron Micrographs. Cell Tissue Res. 1995;280(1):197199.
[ Links ]BUDANTSEV A, JAKOVLEV Y. 3D Reconstruction of Biological Objects: The Potential of Standard Computer Programs. Microsc Microanal. 2000;1719.
[ Links ]CÓRDOBA L, SUÁREZ J. Reconstrucción espacial y temporal del desarrollo de la inervación del miembro posterior del embrión de rata blanca [tesis de Pregrado]. Bogotá: Universidad Militar Nueva Granada; 2005.
[ Links ]FIALA J, FEINBERG M, POPOV V, HARRIS K. Synaptogenesis Via Dendritic Filopodia in Developing Hippocampal Area CA1. J Neurosci.1998;18(21):89008911.
[ Links ]FIALA J, HARRIS K. ComputerBased Alignment and Reconstruction of Serial Sections. Microsc Microanal. 2002;57.
[ Links ]HURTADO H. Peripheral Nervous System Regeneration in the Adult Rat: The Regeneration Chamber Model [tesis de doctorado]. Lovaina: Universidad Católica de Lovaina. Bélgica. 1990.
[ Links ]INTEL CORPORATION. Intel® Play™ QX3™ Computer Microscope Answers to Top Questions. http://support.intel.com/support/intelplay/qx3/sb/CS016013.htm 2005.
[ Links ]LINDEMANN B. Receptors and Transduction in Taste. Nature. 2001;413(6852):219225.
[ Links ]NAKAO T, ISHIZAWA A. Development of the Spinal Nerves in the Mouse with Special Reference to Innervation of the Axial Musculature. Anat Embryol. 1994;189(2):115138.
[ Links ]PROPHET E, MILL B, ARRINGTON J, SOBIN L. (editores). Métodos histoembriológicos. Registro de patología de los Estados Unidos de América (ARP). Washington, D.C. 1995.
[ Links ]RAMM P. Advanced Image Analysis Systems in Cell, Molecular and Neurobiology Applications. J Neurosci Methods.1994;54(2):131149.
[ Links ]SHUM D, LUI H, MARTINKA M, BERNARDO O, SHAPIRO J. Computerized Morphometry and ThreeDimensional Image Reconstruction in the Evaluation of Scalp Biopsy from Patients with NonCicatricial Alopecias. Br J Dermatol. 2003;148(2):272278.
[ Links ]SEGEV I, LONDON M. Untangling Dendrites with Quantitative Models. Science. 2000;290(5492):744750.
[ Links ]SHEPHERD G, MIRSKY J, HEALY M, SINGER M, SKOUFOS E, HINES M, NADKARNI P, MILLER P. The Human Brain Project: Neuroinformatics Tools for Integrating Searching and Modeling Multidisciplinary Neuroscience Data. Trends Neurosci. 1998;(21):460468.
[ Links ]TENG H, WILKINSON R. ClathrinMediated Endocytosis Near Active Zones in Snake Motor Boutons. J Neurosci. 2000;20(21):79867993.
[ Links ]VENTURA J, HARRIS K. ThreeDimensional Relationships Between Hippocampal Synapses and Astrocytes. J Neurosci. 1999;19(16):68976906.
[ Links ]VERBEEK F. ThreeDimensional (3D) Reconstruction from Serial Sections. En “Image Processing and Analysis. A Practical Approach”. Baldock R, Graham J (Editores). Oxford University Press; 2000.
WENINGER W, MENG S, STREICHER J, MÜLLER G. A New Episcopic Method for Rapid 3D Reconstruction: Applications in Anatomy and Embryology. Anat Embryol. 1998;197:341348.
[ Links ]WHITEN S, SMART S, MCLACHLAN J, AITON J. Computer-Aided Interactive Three Dimensional Reconstruction of the Embryonic Human Heart. J Anat. 1998;(193):337345.
[ Links ]WOOTTON R. Introduction to Histological Image Processing. En “Image Analysis in Histology. Conventional and Confocal Microscopy”. Wootton R, Springall D, Polak J (Editores). Cambridge University Press; 1995.
YING X, CHEUNG L. Image File Format for Digital Microscopy. Microsc Microanal. 1999;1517.
[ Links ]