Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.13 no.1 Bogotá Jan./Apr. 2008
NELSON M. LOPERA-BARRERO1*, MVZ, M. Sc., Ph. D.; RICARDO PEREIRA RIBEIRO1, Z.Sc., M.Sc., Ph. D., RODOLFO NARDEZ SIROL2, Oc.Sc., M.Sc., Ph. D.; JAYME A. POVH1, Z.Sc., M.Sc., Ph. D.; PATRICIA C. GOMES1, B.Sc., M.Sc.; LAURO VARGAS1, MV.Sc., M.Sc., Ph. D.; CLAUDETE A. MANGOLIN3, B.Sc., M.Sc., Ph. D.
1Programa de Pós-Graduação em Zootecnia, Universidade Estadual de Maringá, Grupo de Pesquisa Peixegen, Av. Colombo, 5790, CEP 87020 900, Maringá, PR, Brasil.
2Estação de Aqüicultura e Hidrologia da Duke Energy International, Chavantes, SP, Brasil.
3Departamento de Biologia Celular e Genética, Universidade Estadual de Maringá, Av. Colombo, 5790, CEP 87020-900, Maringá, PR, Brasil. *Autor responsable de la correspondencia: Nelson Mauricio Lopera-Barrero, nelson.peixegen@gmail.com
Presentado 1 de septiembre de 2007, aceptado 13 de diciembre de 2007, correcciones 9 de febrero de 2008.
RESUMEN
Alteraciones ambientales causadas por el calentamiento global y principalmente causa-das por la acción del hombre, han reducido poblaciones naturales de peces. Como forma de conservación, programas de repoblamiento han sido utilizados; sin embargo, sin una debida orientación científica, estas medidas pueden generar disturbios genéticos sobre la diversidad genética de poblaciones de peces naturales y sobre el ecosistema. El objetivo de este estudio fue estimar y analizar la variabilidad genética de dos lotes y una progenie de Brycon orbignyanus utilizados en programas de repoblamiento, utilizando el marcador molecular RAPD (Random Amplified Polymorphic DNA). Cincuenta y ocho reproductores de dos lotes (A y C) y 30 larvas de la progenie del lote A (B) pertenecientes a la Estação de Aqüicultura e Hidrologia da Duke Energy Internacional (Geração Parana-panema; São Paulo, Brasil) fueron analizados. Los resultados de variabilidad genética estimados por el índice de diversidad de Shannon (A: 0,3184; B: 0,3433 y C: 0,3687) y por el porcentaje de fragmentos polimórficos (A: 54,02%; B: 57,47% y C: 58,62%) mostraron que la variabilidad genética fue mantenida en la progenie, debido posiblemente al adecuado manejo reproductivo y al efecto fundador. Por el contrario, la variabilidad encontrada entre los dos lotes de reproductores indica una similaridad genética, a pesar de ser originarios de diferentes pisciculturas. Este resultado es comprobado en el valor moderado de diferenciación genética encontrado (0,0968), en el alto Nm (4,67) y en el dendrograma, que sugieren que los lotes poseen un pool genético similar.
Palabras clave: Brasil, conservación, peces, variabilidad genética, RAPD-PCR.
ABSTRACT
Environmental alterations caused by the global heating and mainly caused by man's action, have reduced natural fish populations. As a conservation measure, stocking programs have been used; however, without scientific orientation these measures can generate genetic disturbances on the genetic diversity of natural fish populations and the ecosystem. The objective of this study was to estimate and analyze the genetic variability of Brycon orbignyanus stocks and offspring used in stocking programs, using the molecular marker RAPD (Random Amplified Polymorphic DNA). Fifty eight broodstocks of two stocks (A and C) and thirty offspring larvae of the stock A (B) belonging to the Estação de Aqüiicultura e Hidrologia da Duke Energy International (Geração Paranapanema; São Paulo, Brazil) were analyzed. The results of genetic variability estimated by the Shannon Index diversity (A: 0.3184; B: 0.3433 and C: 0.3687) and for the percentage of polymorphic fragments (A: 54.02%; B: 57.47% and C: 58.62%) showed that the genetic variability was maintained in the offspring, due possibly to the appropriate reproductive management and the founder effect. On the contrary, the variability found among the two broodstocks indicates a genetic similarity, in spite of being natives of different fish farming's. This result is verified in the moderate value of genetic differentiation found (0.0968), in the high Nm (4.67) and the dendrogram, which suggests that the stocks hold a similar genetic pool.
Key words: Brazil, conservation, fishes, genetic variability, RAPD-PCR.
INTRODUCCIÓN
Mudanzas ambientales como la deforestación, la contaminación de los ríos, la construcción de hidroeléctricas (Monteiro et al., 2006; Hori et al., 2006), el ecoturismo mal planeado (Metzger y Casatti, 2006) y la falta de conocimiento taxonómico (Agostinho et al., 2005), asociados a los contundentes cambios climáticos causados por el calentamiento global, han provocado la desaparición de poblaciones de peces en los últimos años. Brycon orbignyanus (Valenciennes, 1849), conocido regionalmente en el Brasil como piracanjuba o bracanjuba (orden Characiformes, familia Characidae, subfamilia Bryconinae), es una especie migratoria nativa de las cuencas hidrográficas forma-das por los ríos Uruguay y Paraná (Zaniboni-Filho et al., 2006) muy apreciada por sus características zootécnicas (Borba et al., 2006). Este pez ha despertado un gran interés por investigadores y productores ya que en los últimos años una drástica reducción de sus poblaciones naturales ha sido observada (Zaniboni-Filho et al., 2006) siendo catalogada como especie en vía de extinción (Machado, 2005). Con el objetivo de aumentar la supervivencia y conservación de esta especie, programas de repoblamiento vienen siendo usados con más intensidad en el Brasil, gracias al apoyo de organismos gubernamentales y de la sociedad (Sirol y Britto, 2006). Sin embargo, su utilización es limitada cuando no existe una orientación multidisciplinar que permita objetividad de las estrategias y manejos utilizados, ya que su ineficiente utilización puede acarrear la pérdida de diversidad genética de los peces que serán liberados, siendo igualmente una amenaza para los ecosistemas ya impactados y para las poblaciones naturales de peces (Povh, 2007). De esta forma, estudios biológicos y de comportamiento del B. orbignyanus, en consorcio con el monitoreo genético de las poblaciones naturales y de lotes de piscícolas con marcadores moleculares, como por ejemplo, el RAPD (Random Amplified Polimorphic DNA), representarían informaciones de gran importancia para conseguir éxito en la producción y en la conservación de las especies (Gomes, 2007) especialmente en aquellas amenazadas de extinción. La disminución de la variabilidad genética en las piscícolas es común debido al inadecuado manejo reproductivo (Aho et al., 2006), intensificando su efecto de forma que el componente genético se va homogenizando (Pineda-Santis, 2004), siendo posible encontrar problemas de adaptabilidad y supervivencia en progenies usadas en programas de repoblamiento. Por lo anterior, el objetivo del siguiente estudio fue estimar y analizar la variabilidad genética de dos lotes y una progenie de B. orbignyanus utilizados en programas de repoblamiento del río Paranapanema, con la utilización del marcador molecular RAPD.
MATERIALES Y MÉTODOS
MATERIAL BIOLÓGICO
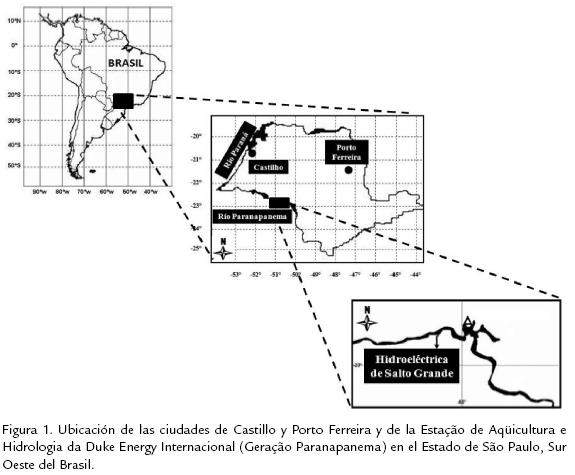
Muestras de aleta fueron recolectadas de dos lotes de reproductores de B. orbignyanus (lote A: 30 individuos; lote C: 28 individuos) mantenidos en cautiverio hace seis años en las instalaciones de la Estação de Aqüicultura e Hidrologia da Duke Energy Internacional, Geração Paranapanema (São Paulo, Brasil). Los lotes fueron obtenidos de dos piscícolas ubicadas en las ciudades de Castillo (Lat: 20° 52' 20" S; Lon 51° 29' 15" O) y Porto Ferreira (Lat: 21° 51' 14" S; Lon 47° 28' 45" O), São Paulo (Figura 1). Después de la inducción hormonal (extracto de hipófisis de carpa) de los 30 reproductores del lote A, utilizando el sistema reproductivo por extrusión descrito por Zaniboni-Filho y Nuñer (2004), fueron colectadas 30 larvas de tres días de nacidas. Esa F1 se utilizó para programas de repoblamiento del río Paranapanema.
EXTRACCIÓN, CUANTIFICACIÓN E INTEGRIDAD DEL ADN
Para la extracción de ADN fue utilizada la metodología descrita por Bardakci y Skibinski (1994), modificada por Povh et al. (2005). Fragmentos de aleta caudal de aproximadamente 0,5 cm2 y larvas enteras, preservadas a -20 ºC con etanol 70%, se colocaron separadamente en microtubos con 550 mL de tampón de lisis (50 mM de Tris-HCl pH 8,0, 50 mM de EDTA, 100 mM de NaCl y 1% de SDS) y 200 mg/mL de proteinasa K, y se incubaron en baño-maría a 50 ºC por 12 horas. El ADN se purificó con dos extracciones con fenol y tres de cloroformo, se precipitó con dos veces y media de volumen de etanol absoluto y un décimo de volumen de acetato de sodio en relación al volumen recuperado y se incubó por dos horas a -20 ºC. El ADN fue centrifugado, lavado con 2 mL de etanol 70%, suspendido en 60 mL de tampón TE (10 mM de Tris pH 8,0 y 1 mM de EDTA) y tratado con 30 mg/mL de RNAsa. Las muestras se incubaron por 40 minutos en baño-maría a 37 ºC y se conservaron a -20 ºC. El ADN se cuantificó en espectrofotómetro Shimadzu (UV 1601 - EE.UU.), en la amplitud de onda de 260 nm, y se diluyó en TE para una concentración de 10-5 ng/mL (aletas y larvas respectivamente). La integridad del ADN se verificó por electroforesis horizontal utilizando un gel de agarosa 1%, con 70 voltios por 60 minutos, en tampón 1X TBE (500 mM de Tris-HC1, 60 mM de ácido bórico y 83 mM de EDTA). El gel se marcó con bromuro de etidio (0,5 µg/mL) por 30 minutos y la imagen fue capturada por el sistema fotográfico EDAS (Kodak 1D Image Analysis 3.5, EE.UU.).
AMPLIACIÓN Y ELECTROFORESIS
Las condiciones de amplificación utilizadas fueron según la metodología descrita por Williams et al. (1990), con algunas modificaciones. El ADN se amplificó en un volumen de reacción de 25 mL, en el cual se utilizó tampón Tris-KCl 1X (Tris-HCl 20 mM pH 8,4 y KCl 50 mM), 2 mM MgCl2, 100 ng primer, 0,2 mM de cada dNTP, una unidad de Taq ADN Polimerasa (Invitrogen®, EE.UU.), y 20 ng de ADN molde. Las reacciones de RAPD fueron amplificadas en un ciclador térmico de PCR Eppendorf Mastercycler” Gradient (EE.UU.), programado para 40 ciclos, con un paso inicial de desnaturación a 94 ºC por cuatro minutos y un paso final de extensión a 72 ºC por cinco minutos. Cada ciclo consistió de un minuto a 94 ºC, un minuto y treinta segundos a 40 ºC y dos minutos a 72 ºC. Se evaluaron 19 primers del Kit Operon (Operon Technologies Inc. Alameda, CA, EE.UU.).
Para evaluar los lotes se seleccionaron aquellos que presentaron características reproducibles y de consistencia, adecuadas. Los productos de amplificación se separaron en gel de agarosa 1,5%. Se usaron 15 mL del producto amplificado y 2 mL de tampón de muestra (40% de sacarosa y 0,25% de azul de bromofenol) en electroforesis horizontal. La electroforesis fue conducida en 70 voltios por cuatro horas en una cuba horizontal usando tampón TBE 1X (500 mM de Tris-HCl, 60 mM de ácido bórico y 83 mM de EDTA). Para verificar la existencia de contaminación, se utilizó un control negativo para cada reacción, donde su amplificación fue ejecutada adicionándose todos los componentes citados anteriormente, excepto el ADN. Para el revelado del gel, se utilizó un baño en solución de 0,5 mg/mL de bromuro de etídio por 30 minutos. Posteriormente, cada gel se fotografió usando el sistema EDAS (Kodak 1D Image Analysis 3.5).
ANÁLISIS ESTADÍSTICO
El tamaño de los fragmentos se estimó por comparación con el padrón ADN Ladder de 100 pb (15 bandas con tamaño entre 100 y 2072 pb-Invitrogen® EE.UU.). La presencia o ausencia de fragmentos de tamaños moleculares idénticos se usó para la construcción de una matriz de similaridad con base en el calculo del coeficiente de similaridad de Jaccard, codificando 1 como presencia de fragmento y 0 como su ausencia. La variabilidad genética se determinó por el Índice de diversidad de Shannon y por el porcentaje de fragmentos polimórficos. La diferenciación genética entre los individuos de los dos lotes fue determinada por la diferenciación genética de Nei (1973; Gst). Para la determinación del nivel de diferenciación se estableció la definición propuesta por Wright (1978), donde valores entre 0,00 a 0,05; 0,05 a 0,15; 0,15 a 0,25 y > 0,25 indican pequeña, moderada, alta y elevada diferenciación genética, respectivamente. Estos análisis estatíticos junto con la determinación del número de migrantes por generación (Nm) se determinaron a través del programa PopGene 1.31 (Yeh et al., 1999). La matriz del coeficiente de similaridad de Jaccard se utilizó para elaborar el dendrograma, con el programa estadistico NTSYS 1.7 (Rohlf, 1989).
RESULTADOS
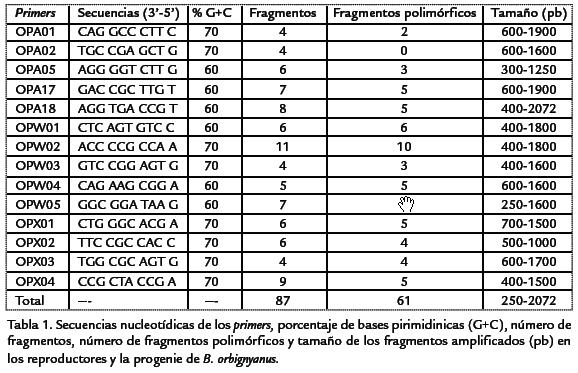
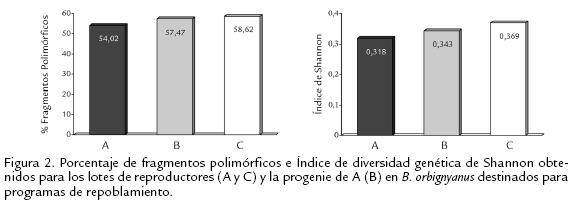
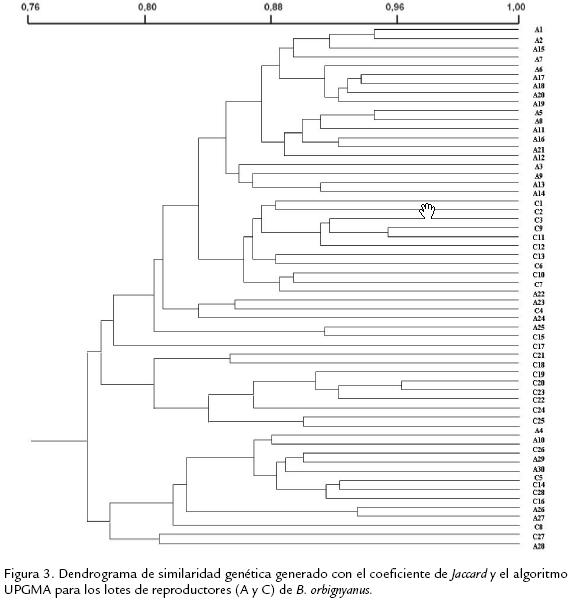
En la Tabla 1 son mostrados los 14 primers seleccionados, el porcentaje de bases pirimidínicas (G+C), los fragmentos, el número de fragmentos polimórficos y el tamaño de los fragmentos amplificados. Con la utilización del marcador molecular RAPD, se amplificaron un total de 87 fragmentos con tamaño entre 2072 pb (obtenido por la amplificación del primer OPA18) y 250 pb (obtenido con la amplificación del primer OPW05), de los cuales 61 (70,11%) fueron polimórficos y 26 (29,89%) no presentaron polimorfismo. Esos fragmentos variaron de cuatro (obtenidos con los primers OPA01, OPA02, OPW03 y OPX03) a 11 (obtenidos con el primer OPW02). Las amplificaciones con el primer OPA02 no mostraron polimorfismo. Los valores de variabilidad genética encontrados en los lotes (A y C) y la progenie (B) son mostrados en la Figura 2. La diversidad genética (Gst) entre los dos lotes de reproductores fue 0,0968, valor que sugiere moderada diferenciación genética según la definición de Wright (1978). El número de emigrantes por generación (Nm) fue de 4,67. El dendrograma de similaridad genética para los dos lotes, generado con el coeficiente de Jaccard es pre-sentado en la Figura 3.
DISCUSIÓN
Objetivando la estimación y análisis de la variabilidad genética de dos lotes de repro-ductores y una progenie de B. orbignyanususados en programas de repoblamiento delrío Paranapanema, marcadores moleculares RAPD fueron utilizados. La variabilidadgenética estimada por el Índice de diversidad de Shannon (IS) y por el porcentaje defragmentos polimórficos (%FP; Figura 2) para el lote de reproductores A y su progenie(B) mostró que la variabilidad genética fue preservada. Al analizar los lotes de repro-ductores (A y C) fue determinada una similaridad genética entre si, evidenciada en losvalores de IS, %FP, diversidad genética (Gst= 0,0968), en el número de emigrantes porgeneración (4,67) y en el dendrograma (Figura 3). La preservación de la variabilidadgenética en la progenie del lote A pudo ser debida al manejo reproductivo eficienteutilizado en la piscícola. Según Moreira et al.(2007), una pérdida de variabilidadgenética siempre es esperada cuando no hay un buen manejo genético de los lotes dereproductores debido al cruzamiento de individuos emparentados, lo que consecuente-mente puede aumentar el coeficiente de endogamia (Kang et al., 2006; Ortega-VillaízanRomo et al., 2006) y reducir el número efectivo de reproductores (Ne) (Melo et al.,2006), afectando la variabilidad genética (Povh et al., 2005).Los resultados encontra-dos en este estudio demuestran que hubo un buen manejo reproductivo, lo que segúnMiller y Kapuscinski (2003) resulta en la preservación de la variabilidad genética entrelos reproductores y sus progenies y en la disminución de la correlación negativa entrela similaridad genética y el número de generaciones (Freitas y Galetti Jr, 2005), que en estaciones de piscicultura es normalmente irreversible (Yokota et al., 2003; Sekino et al., 2004), siendo controlado con la introducción de nuevos reproductores a los lotes, incrementando el pool genético (Freitas y Galetti Jr, 2005). El efecto fundador (diversidad genética con que el lote de reproductores fue constituido) pudo también influenciar en la variabilidad genética de la progenie, ya entre los reproductores del lote A existe una alta variabilidad genética (%FP= 54,02% y IS= 0,3184), maximizando su efecto en la progenie y consecuentemente, preservando la variabilidad genética. Por esta razón, la primera providencia a ser tomada en la implantación de una piscícola o de programas de repoblamiento es verificar la diversidad genética de los lotes y posteriormente de las poblaciones naturales, ya que el cruzamiento entre individuos emparentados puede conducir a un aumento de la endogamia y a una perdida de la variabilidad genética, provocando una disminución de la supervivencia de las progenies en la piscícola (Melo et al., 2006) y de aquellas usadas en programas de repoblamiento, lo que según Freitas y Galetti Jr (2005) puede ser disminuido al incrementar el número efectivo de reproductores o mediante el cruzamiento entre individuos de diferentes orígenes.
Por otro lado, al analizar la variabilidad genética de los lotes de reproductores (A y C), fue encontrada una similaridad genética entre ellos, representada en los resultados de IS y %FP (Figura 2), de diversidad genética y en el número de emigrantes por generación, lo que hace presumir que presentan un mismo origen, formando un grupo homogéneo. Estos resultados coinciden con el dendrograma de similaridad genética (Figura 3), donde de forma general no hubo formación de agrupamientos que evidenciaran una separación de los lotes de reproductores. Algunos individuos presentaron una tendencia al agrupamiento (A10, C26, A29, A30, C5, C14, C28, C16, A26, A27, C8, C27 Y C28) posiblemente debido a características inherentes de cada uno de ellos, sin embargo no hubo una separación genética entre los dos lotes. Al analizar el origen de los dos lotes de reproductores mantenidos en cautiverio en las instalaciones de la Duke Energy Internacional (Geração Paranapanema), estos resultados fueron validados. Los lotes de reproductores son originarios de la primera generación (F1) de dos piscícolas localizadas en las ciudades de Castilho (A) y Porto Ferreira (C) en el estado de São Paulo. La formación de esos lotes fue efectuada a partir de individuos adultos capturados directamente en el río Paraná. A pesar de la separación geográfica de ambas piscícolas y posiblemente de los diferentes sitios de captura de los reproductores, puede ser viable que entre los individuos de cada lote existiese un flujo génico en el ambiente natural, lo que explicaría su similaridad genética.
La existencia de ese flujo génico fue evidenciada con el alto valor de número de emigrantes por generación (4,67) y por la moderada diversidad genética (Gst= 0,0968) encontrada entre los lotes, lo que puede indicar la existencia de relación genética entre sus individuos, sugiriendo que estos presentan un pool genético similar. Esta situación es común en poblaciones naturales, como fue verificado por Leuzzi et al. (2004), quienes al analizar poblaciones de Astyanax altiparanae de dos reservorios del río Paranapanema encontraron que hubo flujo génico entre poblaciones naturales separadas geográficamente. Otra explicación a estos resultados puede ser la pérdida de variabilidad genética que para algunos autores es normalmente irreversible (Wasko et al., 2004; Sekino et al., 2004), lo que consecuentemente aumentó la similaridad genética entre los individuos de cada lote. Estos resultados son de gran importancia, ya que a partir de ellos, el manejo reproductivo de los lotes de esta especie amenazada de extinción, utilizados en programas de conservación (repoblamiento) pueden ser orientados correctamente y con objetividad. Por esta razón, se puede sugerir que el manejo reproductivo, genético y de mejoramiento de los dos lotes de B. orbignyanus analizados en este estudio, debe ser realizado de manera homogénea y no como lotes separados (lo que estaba siendo realizado por la piscícola de la hidroeléctrica), especialmente en programas de conservación de poblaciones naturales a través del repoblamiento. Otros estudios evaluando lotes de peces destinados para programas de repoblamiento han sido realizados. Wasko et al. (2004), analizando progenies de Brycon cephalus obtenidas a partir de lotes mantenidos en piscícolas, encontraron que el cruzamiento de individuos emparentados provocó la disminución de la variabilidad genética en el transcurso de tres generaciones. Povh (2007) comparando un lote de reproductores y una población natural de Piaractus mesopotamicus, encontró una mayor variabilidad genética en la población (IS= 0,345 y 0,289, respectivamente), debido posiblemente a un deficiente manejo reproductivo en la piscícola. En nuestro estudio, la variabilidad genética del lote A fue preservada en la primera generación (B) demostrando, como con una orientación genética objetiva es posible mantener un manejo eficiente de los reproductores, evitando la disminución de la variabilidad genética. Por otro lado, Gomes (2007) analizando tres lotes de Leporinus elongatus, encontró valores de variabilidad genética (IS= 0,4470, 0,4394 y 0,4086) que demostraron que los tres lotes eran genéticamente similares, situación parecida a la encontrada en este estudio para los lotes A y C, demostrando que el monitoramiento continuo de los reproductores de las estaciones de piscicultura es necesario e importante para mantener baja la endogamia de los lotes utilizados en programas de conservación. Un factor importante en programas de repoblamiento es la influencia que tiene el cruzamiento de individuos genéticamente distintos a aquellos encontrados en una población natural, ya que puede promover la pérdida de genes importantes de adaptabilidad al ambiente (Sønstebø et al., 2007), influenciando en la supervivencia de progenies repobladas en el ambiente natural (Leuzzi et al., 2004) y en el equilibrio ecológico de un ecosistema. Por esta razón, en la implantación de programas de repoblamiento es necesario el monitoramiento genético de los lotes de reproductores que serán usados, de sus progenies que serán liberadas en los ríos y de las poblaciones naturales en todos los periodos del año y así verificar la eficacia de este tipo de pro-grama de conservación y su influencia en el ambiente biológico del ecosistema. En conjunto, actividades de reforestación y conservación de la mata ciliar, control de la contaminación de los ríos, campañas de preservación y educación ambiental y la correcta administración y fiscalización de los recursos hídricos permitirán la diversificación de los nichos, permitiendo la revitalización de la cadena alimentar y así la sustentación de la vida y de las poblaciones animales y humanas. Los resultados obtenidos en el presente estudio demostraron que el marcador molecular RAPD puede ser usado con éxito para estimar el nivel de variabilidad genética de lotes de reproductores y progenies, informaciones importantes para su correcta y objetiva orientación y monitoreo de programas de piscicultura y de repoblamiento de peces.
AGRADECIMIENTOS
Los autores agradecen a la empresa de generación de energía eléctrica Duke Energy International (Geração Paranapanema) y al Conselho Nacional de Desenvolvimento Científico e Técnológico (CNPq).
BIBLIOGRAFÍA
AGOSTINHO AA, THOMAZ SM, GOMES LC. Conservation of the Biodiversity of Brazil's Inland Waters. Conserv Biol. 2005;19(3):646-652. [ Links ]
AHO T, RÖNN J, PIIRONEN J, BJÖRKLUND M. Impacts of Effective Population Size on Genetic Diversity in Hatchery Reared Brown trout (Salmo trutta L.) Populations. Aquaculture. 2006;253(1/4):244-248. [ Links ]
BARDAKCI F, SKIBINSKI DOF. Application of the RAPD Technique in Tilapia Fish: Species and Subspecies Identification. Heredity. 1994;73(2):117-123. [ Links ]
BORBA MR, FRACALOSSI DM, PEZZATO LE. Dietary Energy Requirement of Piracanjuba Fingerlings, Brycon orbignyanus, and Relative Utilization of Dietary Carbohydrate and Lipid. Aquac Nut. 2006;12(3):183-191. [ Links ]
FREITAS PD, GALETTI JR PM. Assessment of the Genetic Diversity in Five Generations of Commercial Broodstock Line of Litopenaeus vannamei shrimp. Afr J Biotechnol. 2005;4(12):1362-1367. [ Links ]
GOMES PC. Diversidade genética de três populações de piapara (Leporinus elongatus), utilizando marcadores moleculares [Dissertação Mestrado em Zootecnia]. Maringá: Programa de Pós-Graduação em Zootecnia, Universidade Estadual de Maringá; 2007. [ Links ]
HORI TSF, AVILEZ IM, INOUE LK, MORAES G. Metabolical Changes Induced by Chronic Phenol Exposure in Matrinxã Brycon cephalus (Teleostei: Characidae) Juveniles. Comp Biochem Physiol. 2006;143(1):67-72. [ Links ]
KANG JH, NOH JK, KIM JH, LEE JH, KIM HC, KIM KK, et al. Genetic Relationship Between Broodstocks of Olive Flounder, Paralichthys olivaceus (Temminck and Schlegel) Using Microsatellite Markers. Aquac Res. 2006;37(7):701-707. [ Links ]
LEUZZI MSP, ALMEIDA FS, ORSI ML, SODRÉ LMK. Analysis by RAPD of the Genetic Structure of Astyanax altiparanae (Pisces, Characiformes) in Reservoirs on the Paranapanema River, Brazil. Genet Mol Biol. 2004;27(3):355-362. [ Links ]
MACHADO ABM. Lista da fauna brasileira ameaçada de extinção: incluindo as espécies quase ameaçadas e deficientes em dados. En: Machado ABM, Martins CS, Drummond GM, editors. Lista da fauna brasileira ameaçada de extinção. Belo Horizonte: Fundação Biodiversitas; 2005. [ Links ]
MELO DC, OLIVEIRA DAA, RIBEIRO LP, TEIXEIRA CS, SOUSA AB, COELHO EGA, et al. Caracterização genética de seis plantéis comerciais de tilápia (Oreochromis) utilizando marcadores microssatélites. Arq Bras Med Vet Zootec. 2006;58(1):87-93. [ Links ]
METZGER JP, CASATTI L. Do diagnóstico à conservação da biodiversidade: o estado da arte do programa BIOTA/FAPESP. Biota Neotrop. 2006;6(2):1-23. [ Links ]
MILLER LM, KAPUSCINSKI AR. Genetic Guidelines for Hatchery Supplementation Programs. In: Hallerman E.M, Editor. Population Genetics: Principles and Applications for Fisheries Scientists. Bethesda: American Fisheries Society; 2003. p. 329-355. [ Links ]
MONTEIRO DA, ALMEIDA JA, RANTIN FT, KALININ AL. Oxidative Stress Biomarkers in the Freshwater Characid Fish, Brycon cephalus, Exposed to Organophosphorus Insecticide Folisuper 600 (methyl parathion). Comp Biochem Physiol. 2006;143(2):141-149. [ Links ]
MOREIRA AA, HILSDORF AWS, SILVA JV, SOUZA VR DA. Variabilidade genética de duas variedades de tilápia nilótica por meio de marcadores microssatélites. Pesqui Agropecu Brás. 2007;42(4):521-526. [ Links ]
NEI M. Analysis of Gene Diversity in Subdivided Populations. Proc Nat Acad Sci USA. 1973;70(12):3321-3323. [ Links ]
ORTEGA-VILLAIZÁN ROMO MM, ARITAKI M, TANIGUCHI N. Pedigree Analysis of Recaptured Fish in the Stock Enhancement Program of Spotted Halibut Verasper Variegates. Fish Sci. 2006;72(1):48-52. [ Links ]
PINEDA-SANTIS HR. Estudio genético de las cachamas (subfamilia Serrasalminae) en poblaciones naturales y en cautiverio en Colombia. Rev Col Cienc Pec. 2004;17(S):62-63. [ Links ]
POVH JA. Avaliação da diversidade genética e do manejo reprodutivo do pacu, Piaractus mesopotamicus [Tese Doutorado em Zootecnia]. Maringá: Programa de Pós-Graduação em Zootecnia, Universidade Estadual de Maringá; 2007. [ Links ]
POVH JA, MOREIRA HLM, RIBEIRO RP, PRIOLI AP, VARGAS L, BLANCK DV, et al. Estimativa da variabilidade genética em tilápia do Nilo (Oreochromis niloticus) com a técnica de RAPD. Acta Sci An Sci. 2005;27(1):1-10. [ Links ]
ROHLF FJ. NTSYS-Pc: Numerical Taxonomy and Multivariate Analysis System. New York: Exeter Publishers; 1989. [ Links ]
SEKINO M, SUGAYA T, HARA M, TANIGUCHI N. Relatedness inferred from microsatellite genotypes as a tool for broodstock management of Japanese flounder Paralichthys olivaceus. Aquaculture. 2004;233(1/4):163-172. [ Links ]
SIROL RN, BRITTO SG. Conservação e manejo da ictiofauna: repovoamento. En: Nogueira MG, Henry R, Jorcin A, editors. Ecologia de reservatórios: impactos potenciais, ações de manejo e sistemas em cascatas. São Carlos: RiMA; 2006. p. 275-284. [ Links ]
SØNSTEBØ JH, BORGSTRØM R, HEUN M. Genetic Structure of Brown Trout (Salmo trutta L.) from the Hardangervidda Mountain Plateau (Norway) Analyzed by Microsatellite ADN: A Basis for Conservation Guidelines. Conserv Genet. 2007;8(1):33-44. [ Links ]
WASKO AP, MARTINS C, OLIVEIRA C, SENHORINI JA, FORESTI F. Genetic Monitoring of the Amazonian fish Matrinchã (Brycon cephalus) Using RAPD Markers: Insights Into Supportive Breeding and Conservation Programmers. J Appl Ichthyol. 2004;20(1):48-52. [ Links ]
WILLIAMS JG, KUBELIK AR, LIVAK J, RAFALSKI JA, TINGEY SV. ADN Polymorphisms Amplified by Arbitrary Primers are Useful as Genetic Markers. Nucleic Acids Res. 1990;18(22):6531-6535. [ Links ]
WRIGHT S. Evolution and Genetics of Population. Chicago: University of Chicago Press; 1978. [ Links ]
YEH FC, BOYLE TYZ, XIYAN JM. POPGENE Version 131: Microsoft Window-Based Freeware for Population Genetic Analysis. Alberta: University of Alberta and Center for International Forestry Research; 1999. [ Links ]
YOKOTA M, HARADA Y, IIZUKA M. Genetic Drift in a Hatchery and the Maintenance of Genetic Diversity in Hatchery-Wild Systems. Fish Sci. 2003;69(1):101-109. [ Links ]
ZANIBONI-FILHO E, REYNALTE-TATAJE D, WEINGARTNER M. Potencialidad del género Brycon en la piscicultura brasileña. Rev Col Cienc Pec. 2006;19(2):233-240. [ Links ]
ZANIBONI-FILHO E, NUÑER APO. Fisiologia da reprodução e propagação artificial dos peixes. En: Cyrino JEP, Urbinati EC, Fracalossi DM, Castagnolli N, editors. Tópicos Especiais em Piscicultura de Água Doce Tropical Intensiva. São Paulo: TecArt; 2004. p. 45-74. [ Links ]














