Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Acta Biológica Colombiana
versão impressa ISSN 0120-548X
Acta biol.Colomb. v.13 n.2 Bogotá maio/ago. 2008
CONSTRUCCIÓN DE UNA LIBRERÍA DE ADNc EN YUCA: UNA HERRAMIENTA PARA EL DESARROLLO BIOTECNOLÓGICO DEL CULTIVO
Cassava cDNA Library Construction: One Tool for Biotechnological Development of the Crop
CAROLINA GONZÁLEZ ALMARIO1, Ph. D.; CAMILO E. LÓPEZ CARRASCAL2, Ph. D.
Departamento de Biología, Facultad de Ciencias, Universidad Nacional de Colombia, Sede Bogotá, AA 14490, Bogotá, Colombia. 1 cgonzalezal@unal.edu.co - 2 celopezc@unal.edu.co
Presentado 22 de octubre de 2007, aceptado 6 de mayo de 2008, correcciones 6 de junio de 2008.
RESUMEN
La yuca es un cultivo de importancia en la seguridad alimentaria mundial ya que constituye la base de la alimentación de más de 600 millones de personas en el mundo. También es un alto productor de almidón con niveles que oscilan entre 73,7 y 84,9% de su peso seco total en raíces (FAO, 2007). El almidón de yuca puede utilizarse en una gama amplia de industrias (textil, cosmética, alimentaria, etc). Además, es empleado en la producción de biocombustibles. Una de las principales limitantes en la producción de yuca es la bacteriosis vascular producida por la bacteria Xanthomonas axonopodis pv. manihotis (Xam). Esta enfermedad puede comprometer no solo el suministro de almidón para las plantas industriales productoras de bioetanol sino que también puede amenazar la seguridad alimentaria. El mejoramiento genético convencional de yuca es complicado dado su largo ciclo reproductivo, su alta heterocigocidad y su naturaleza tetraploide. Por estas razones se deben buscar alternativas que involucren desarrollos en biotecnología que permitan un mejoramiento eficaz y rápido. En la actual era genómica y postgenómica muchos de los experimentos dependen de la posibilidad de contar con la secuencia de transcritos clonados. Dado que estos clones provienen de librerías de ADNc contar con este tipo de recursos de excelente calidad es un paso esencial. En este artículo reportamos la construcción de una librería de ADNc empleando el sistema Gateway® a partir de plantas de yuca que han sido inoculadas con la cepa CIO151 de Xam. La librería presentó un título de 1 x 107 unidades formadoras de colonias (ufc)/ml y el tamaño de los insertos osciló entre 600-1.500 pb. Los análisis de secuencia de 14 clones al azar confirmaron que se trata de genes expresados y mostraron similitud con genes previamente reportados en especies estrechamente relacionadas a yuca. Esta librería se convierte en un excelente recurso para identificar novedosos genes y para el estudio de su función a través de la identificación y la interacción entre proteínas.
Palabras clave: yuca, bacteriosis vascular, genes expresados, librería de ADNc, Gateway®.
ABSTRACT
Cassava is one of the most important crops for global food security and provides food and livelihood for 600 million people in the developing world. It is also good source of starch, with levels between 73.7 y 84.9% of total dry weight in roots (FAO, 2007). Cassava starch can be used in a wide range of industries (textile, cosmetic, nourishing, etc) and it has a high potential for the production of biofuel. Cassava bacterial blight, caused by Xanthomonas axonopodis pv. manihotis (Xam), is one of the most important diseases that affects cassava. This disease can compromise the starch supply not only for bioetanol production but also affect global food security. The long reproductive cycle, high heterozigosity and tetraploid character of cassava are characteristics that have complicated the genetic breeding for this crop. For these reasons new alternatives based on biotechnology are necessary to accelerate its improvement. In the postgenomic era many experiments rely on the availability of transcript sequences for cloning. As these clones usually originate from cDNA libraries, the quality of these libraries is crucial. In this article we report the construction of the first cassava cDNA library employing the Gateway® system. For this, in vitro grown plants were inoculated with the Xam strain CIO151. The expression library shows a high titer of 1 x 107 cfu/ml, with inserts ranging between 600 and 1500 bp. The sequence analyses from 14 random clones confirmed that these are expressed genes and showed similarity with previously cloned genes from species related to cassava. This library is an excellent resource for the identification of novel genes and for functional studies through the identification of their interactions with other proteins.
Key words: Cassava, cassava bacterial blight, expressed genes, cDNA library, Gateway®.
INTRODUCCIÓN
La yuca (Manihot esculenta) es un cultivo que se originó en la cuenca amazónica y que se ha cultivado en la región suramericana desde hace más de 10.000 años (Olsen y Schaal, 1999). Actualmente la mitad de los 16 millones de hectáreas dedicadas al cultivo se encuentran en África, el 30% en Asia y el 20% en América Latina (FAO, 1998). La producción mundial total de yuca alcanzó los 272 millones de toneladas en el 2006 (FAO, 2007). Dentro de América Latina Colombia ocupa el tercer puesto en producción después de Brasil y Paraguay (FAO, 2007). Sobre condiciones óptimas y experimentales la producción de yuca puede alcanzar las 90 toneladas por hectárea, sin embargo en condiciones naturales de cultivo el rendimiento solo alcanza en el mejor de los casos 20-30 toneladas por hectárea.
Después del arroz, el trigo y el maíz, la yuca representa la fuente más importante de calorías para la población mundial (FAO, 1998) y constituye la base de la alimentación para más de 600 millones de personas en el mundo, especialmente en los países tropicales de América Latina y África. La yuca también es utilizada en alimentación animal y como materia prima para el procesamiento industrial de productos basados en almidón. Recientemente y como consecuencia del aumento en el precio del petróleo crudo, el cultivo de yuca ha cobrado un fuerte interés por su potencial para producir bioetanol, una de las alternativas bioenergéticas de mayor desarrollo en la actualidad (Lopez et al., en prensa). Alcanzar mayores rendimientos en este cultivo requiere importantes esfuerzos en desarrollo científico y tecnológico. El mejoramiento genético convencional en yuca es complicado ya que presenta un ciclo de crecimiento largo, es una planta tetraploide y con un alto índice de heterocigocidad. Otro de los factores limitantes para el desarrollo de estrategias de mejoramiento es el desconocimiento de su compleja estructura genética. Recientemente se han desarrollado importantes esfuerzos con miras a conocer mejor la estructura genómica y funcional de la yuca a través de la generación de librerías BACs, de una colección de ESTs y desarrollo de mapas genéticos (López et al., 2007). Estos desarrollos biotecnológicos contribuirán al desarrollo de la biotecnología de yuca, lo que permitirá a futuro generar variedades mejoradas.
Dentro de las enfermedades más importantes de la yuca se encuentra la bacteriosis vascular, la cual es causada por la bacteria gram-negativa Xanthomonas axonopodis pv manihotis (Xam). Las pérdidas ocasionadas por esta enfermedad pueden llegar al 100% después de tres ciclos de producción (Lozano, 1986). En los últimos años se han realizado importantes avances hacia una mejor comprensión de los mecanismos moleculares de la yuca que tienen lugar como respuesta a la bacteriosis vascular. Dentro de estos avances cabe destacar la identificación de regiones genómicas asociadas a la resistencia o QTLs (Jorge et al., 2000), de genes candidatos de resistencia (López et al., 2003), la generación de una colección de secuencias expresadas (del inglés Expressed Sequence Tags ESTs) (López et al., 2004) y la construcción de un microarreglo que permitió el estudio de la cinética de expresión de más de 5.700 genes de yuca en respuesta a la infección por Xam (López et al., 2005). Todos estos avances han sido logrados gracias al desarrollo de herramientas moleculares. Una de las principales herramientas para dicho desarrollo biotecnológico es la disposición de librerías de ADNc. La construcción de librerías de ADNc en yuca ha sido particularmente difícil por la baja eficiencia en la obtención de ARN total y en la purificación de ARNm posiblemente por la presencia de compuestos orgánicos (polifenoles, carbohidratos, etc) que dificultan su extracción y purificación. Recientemente la tecnología Gateway® se ha desarrollado de tal manera que permite la clonación en vectores sin necesidad de digestión y ligación, además de que permite la transferencia de los insertos generados a diferente tipo de vectores dependiendo de los intereses del investigador. En la actual era genómica y postgenómica muchos de los experimentos dependen de la posibilidad de contar con la secuencia de transcritos clonados. Dado que estos clones provienen de librerías de ADNc contar con este tipo de recursos de excelente calidad es un paso esencial. La construcción de librerías de ADNc ha permitido la generación de colecciones de ESTs en cultivos como papa (Ronning et al., 2003), tomate (Van der Hoeven et al., 2002), arroz (Zhou et al., 2003) entre otros, lo cual es una vía rápida para la identificación de genes de interés agronómico. Las librerías de ADNc han permitido de igual manera la identificación y clonación completa de genes de interés en tejidos específicos (Demura et al., 2002). En este trabajo reportamos la construcción de la primera librería de ADNc de yuca elaborada a partir de la tecnología Gateway® y una de las pocas que existen empleando esta estrategia.
MATERIALES Y MÉTODOS
MATERIAL VEGETAL
Plantas propagadas in vitro de yuca de la variedad SG107-35 de seis semanas de edad (3-4 hojas desarrolladas) fueron utilizadas para este estudio. Dicha variedad fue obtenida de la colección de germoplasma del Centro Internacional de Agricultura Tropical (CIAT) y propagadas en el Laboratorio de Cultivo de Tejidos del Departamento de Biología de la Universidad Nacional de Colombia, sede Bogotá.
PROPAGACIÓN DE PLANTAS INVITRO
La regeneración de plantas in vitro se realizó a partir de meristemos de 2 a 3 mm de longitud en medio Murashige et Skoog, (complementado con sacarosa 30 g/l, timina 3 ml/l, vitamina Gamborg 1 ml/l, piridoxina 1 ml/l, agar-agar 8 g/l; pH 5,8) a condiciones de 12 horas luz/12 horas oscuridad y a una temperatura de 22 a 25 ºC.
INOCULACIÓN CON XAMCIO151 Y RECOLECCIÓN DEL MATERIAL VEGETAL
Veinte plantas propagadas in vitro de SG107-35 fueron inoculadas con la cepa de Xam CIO151 utilizando el método de punción con aguja reportado por Kpémoua (1.995) y estandarizado en el laboratorio de Fitopatología Molecular de la Universidad Nacional. Dicho método consistió en sumergir una aguja de insulina en una suspensión bacteriana a 108 ufc/ml (D.O.600 nm=0,2) con la que se realizó una punción a cada lado de la nervadura principal de cada foliolo. Las plantas inoculadas fueron mantenidas en incubadora a 28 ºC. Plántulas completas, sin raíz, fueron colectadas a seis tiempos diferentes (0 a 5 días post inoculación) y almacenadas a -80 ºC hasta la posterior extracción de ARN.
AISLAMIENTO DE ARN TOTAL Y PURIFICACIÓN DE ARNM
Dos plántulas completas (aproximadamente 100 mg) por tiempo y dos réplicas por tiempo fueron maceradas en nitrógeno líquido y conservadas a -80 ºC. La extracción total de ARN fue realizada a partir de este tejido empleando el kit SV Total RNA Isolation System® (Promega), el cual incluye un tratamiento con DNAsa (Promega, Madison, WI, USA). El ARN obtenido se cuantificó en el espectrofotómetro Smart-Spec 3000 BIO-RAD a 260 nm y la calidad fue estimada en gel de agarosa al 1,2%. A partir de 580 mg de ARN total se realizó la purificación de ARNm empleando el kit PolyATtract de Promega, según las recomendaciones de la casa comercial. El ARNm obtenido fue precipitado con 1/10 Vol de Acetato de sodio 2,5 M y 2,5 vol de EtOH al 96%. El ARNm purificado fue resuspendido en un volumen final de 20 µl de agua libre de RNAsas.
SÍNTESIS DE ADNC Y LIGACIÓN DE ADAPTADORES
Para la síntesis de ADNc, se empleó el kit CloneMiner™ cDNA Library Construction Kit (Invitrogen). Se tomaron 18 µl de ARNm lo que corresponde a una cantidad de ARNm aproximada de 3,5 µg. Con el fin de utilizar todo el volumen de ARNm, los volúmenes de Biotin-attB2-Oligo(dT) primer (30 pmol/µl) y dNTPs (10 mM de cada uno) fueron duplicados a 2 µl. La mezcla fue incubada a 65 ºC por cinco minutos y enfriada a 45 ºC por dos minutos. La síntesis de primera y segunda cadena se realizó empleando las recomendaciones sugeridas por el fabricante. Al ADNc de doble cadena y de extremos romos obtenido se le ligó el adaptador attB1 lo que permite que el ADNc contenga los sitios de recombinación attB y se pueda entrar de esta forma en el sistema Gateway®.
FRACCIONAMIENTO DEL ADNC
El fraccionamiento del ADNc fue realizado mediante el uso de una columna de Sephacryl® S-500 HR suministrada con el kit CloneMiner cDNA library construction de Invitrogen. Después de cuatro lavados con buffer TEN (10 mM Tris-HCl, pH 7,5; =0,1 mM EDTA; 25 mM Nacl) se pasó la muestra que contenía 50 µl del ADNc con los adaptadores ligados más 100 µl de buffer TEN, y se colectaron fracciones en 20 tubos independientes según las recomendaciones del kit. La concentración de cada una de las fracciones obtenidas de ADNc se determinó con ayuda de un control de ADN (pEXP7) (Invitrogen), preparado a concentraciones estándar de: 25 ng/µl, 10 ng/µl, 5 ng/µl y 1 ng/µl. Un microlitro de cada uno de los estándares y de las fracciones fue visualizado en una caja de petri que contenía agarosa preparado al 1% en TAE 1X y teñido con bromuro de etidio a una concentración final de 1 µg/ml. A partir de las fracciones seleccionadas se realizó la reacción de recombinación BP entre el ADNc y el plásmido pDONR™222 (Invitrogen) empleando la enzima BP Clonasa (Invitrogen) y según las recomendaciones de la casa comercial. La reacción fue incubada toda la noche a 25 °C. Cada reacción BP fue detenida con la enzima proteinasa K después de una corta incubación a 37 °C por 15 min, seguida de una inactivación a 75 °C por 10 min. Posteriormente cada una de las reacciones BP se precipitó con Glicógeno (20 µg/µl), NH4OAc (7,5 M) y etanol absoluto según las indicaciones del kit. Cada pellet de ADNc fue resuspendido en 9 µl con H2O destilada estéril y se transformaron células competentes de E. coli. ElectroMAX™ DH10B™ T1 Phage Resistant Cells suministradas con el kit. Las transformaciones fueron realizadas por electroporación con el electroporador BioRad Gene Pulser® siguiendo las condiciones de voltaje, resistencia y capacitancia recomendadas por el kit. Después de una hora de incubación a 37 °C en medio S.O.C. y en agitación constante, el pool de células que representa cada una de las librerías fue alicuotado en tubos de crío preservación de 2 ml en un volumen igual de medio de conservación estéril (freezing media) (60% S.O.C.: 40% glicerol) y conservados a -80 °C. Antes de ser conservados, 200 µl de cada librería fueron apartados y guardados en hielo para estimar el título de cada una de ellas.
ESTIMACIÓN DEL TÍTULO DE LA LIBRERÍA
Diluciones seriadas de 1:10 (100 µl de la muestra en 900 µl de S.O.C.), comprendidas desde 10-1 hasta 10-3, fueron realizadas y sembradas en medio LB con Kanamicina (50 ng/µl). Los platos correspondientes a cada dilución fueron incubados toda la noche a 37 °C. El número de ufc/ml a partir de 100 µl plateados se estimó con la siguiente fórmula:
Cfu/ml = colon x factor de diluc / Vol plateado (0,1 ml).
ESTIMACIÓN DE LA CALIDAD DE LA LIBRERÍA
Se tomaron al azar colonias de cada uno de los eventos de transformación y se procedió a realizar un PCR directo sobre estas colonias. Las reacciones de PCR se llevaron a cabo en un volumen final de 25 µl que contenía: 1X de Buffer de PCR (50 mM de KCl, 100 mM de Tris-HCl, 0,1% de TritónX-100), 2,5 mM de MgCl2, 0,2 mM de dNTPs, 0,1 µM de los primers M13F y M13R y 1 U de Taq DNA polimerasa (Invitrogen Life Technologies, Carlsbad, Calif.). Con un palillo de dientes estéril se picó cada una de las colonias y este palillo se sumergió en la mezcla de PCR. Las condiciones de amplificación por PCR fueron las siguientes: denaturación inicial a 94 °C por 5 min seguido de 35 ciclos de amplificación que incluía una denaturación por 30 s a 94 °C, un anillaje por 30 s a 52 °C y una extensión por 2 min a 72 °C. Los productos de PCR se corrieron en geles de agarosa al 1,2% en buffer TAE 0,5X. Los geles se observaron en un transiluminador con luz ultravioleta y el tamaño de los fragmentos amplificados se determinó empleando el marcador de peso molecular 1 Kb Plus Ladder ® (Invitrogen, Carlsbad, CA, USA). Para las reacciones de secuenciación se tomaron al azar 14 colo-nias y se pusieron a crecer en medio líquido LB con ampicilina (100 mg/ml) toda la noche a 37 °C. A partir de este cultivo se realizaron minipreps empleando el kit de Fermentas y a partir del ADN plasmídico se secuenciaron los insertos mediante secuenciación automática.
RESULTADOS Y DISCUSIÓN
AISLAMIENTO DE ARN
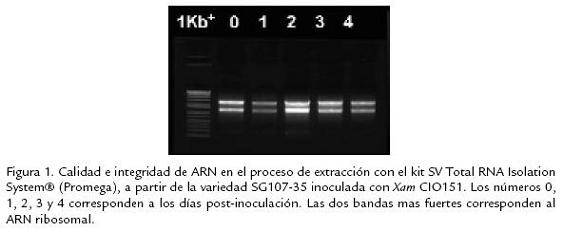
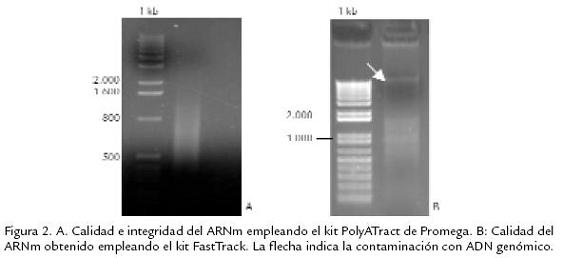
ODado que la construcción de una librería requiere cantidades importantes de ARNm (5 µg) y considerando que el ARNm corresponde a aproximadamente a 10% del ARN total, es necesario contar con mínimo 0,5 g de ARN total. Partiendo del material vegetal propagado in vitro fue necesario realizar varias extracciones de ARN total para contar con el material suficiente requerido. Las extracciones de ARN fueron exitosas (Fig. 1 ) con un promedio de eficiencia de 70 µg de ARN total obtenido a partir de 100 mg de tejido. Se evaluaron dos métodos diferentes para la purificación del ARNm empleando tanto el kit FastTrack (Invitrogen) como el PolyATract (Promega), pero solo se obtuvieron resultados óptimos con el PolyATract. El kit FastTrack es más rápido y permite realizar la purificación del ARNm directamente a partir de tejidos vegetales, sin embargo para el caso de yuca, solo se logró obtener cantidades muy bajas de ARNm y en algunos casos se presentó contaminación con ADN genómico (Fig. 2B ). Paralelamente se realizó el mismo procedimiento para purificar ARNm de papa con resultados positivos (datos no mostrados). Al emplear el kit PolyATract se logró obtener suficiente cantidad de ARNm para iniciar la síntesis de ADNc (Fig. 2A ). El ARNm se debe observar como un barrido que corresponde a los diferentes fragmentos de los genes que se están expresando los cuales poseen tamaños diferentes. Un barrido que se encuentre en el rango de 500 a 1.500 pb es indicativo de un ARNm de buena calidad. El ARNm no debe presentar las dos bandas ribosomales que se presentan en el ARN total. Como se muestra en la Fig. 2B , el ARNm obtenido presenta estas características indicando que se trata de un ARNm de excelente calidad para proceder con la síntesis de ADNc y construcción de la librería.
SÍNTESIS DE ADNC Y CONSTRUCCIÓN DE LA LIBRERÍA
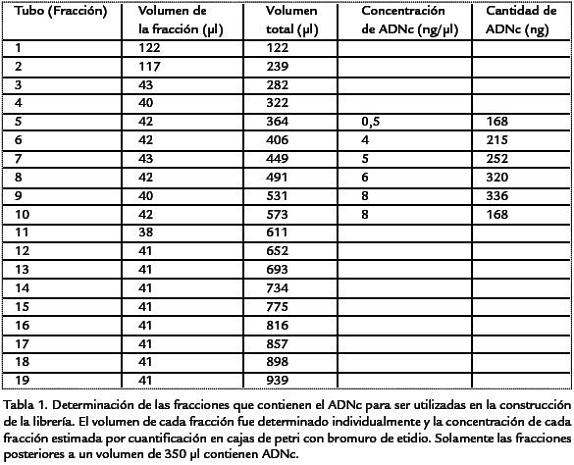
A partir del ARNm purificado se procedió a realizar la síntesis de primera y segunda cadena de ADNc siguiendo las condiciones definidas en el manual Clone Miner cDNA Library (Invitrogen). Para poder entrar al sistema Gateway®, al ADNc obtenido se le ligaron los adaptadores attB que permiten ser reconocidos por las recombinasas y poder posteriormente ser clonados en el vector de entrada. El ADNc así obtenido fue fraccionado a través de una columna con el fin de seleccionar los fragmentos de mayor tamaño y eliminar las fragmentos de bajo tamaño y el exceso de adaptador no ligado, las cuales se clonarían preferencialmente. Cada una de las fracciones colectadas y su volumen fue registrado y se procedió a realizar la cuantificación de las diferentes fracciones (Tabla.1 y Fig. 3 ). Los fragmentos más grandes se encontrarán en las primeras fracciones, mientras que los fragmentos de ADNc pequeños y el exceso de adaptador aparecerán en las últimas fracciones. Como se observa en la Fig. 3 y en la Tabla.1, se alcanzó a visualizar ADNc a partir de la quinta fracción y se observó una buena cantidad de ADNc hasta la fracción 10 (Fig. 3 ). A partir de las fracciones 1314 se observó una cantidad importante de ADN que corresponde muy seguramente al exceso de adaptadores. Las primeras fracciones que contienen ADNc (cinco a nueve) son las más importantes por cuanto contienen los fragmentos más grandes ypueden constituir genes completos. Dado que es necesario obtener 75 ng de ADNcpara la reacción de recombinación BP, y teniendo en cuenta que aproximadamente50% de la muestra puede ser perdida en el proceso de precitación con etanol, se re-quiere un mínimo 150 ng de ADNc a partir de las fracciones colectadas. Dos poolesfueron originados, el pool 1 contenía ADNc de las fracciones 5-7 y el pool 2 conteníalas fracciones 8-10. Sin embargo, antes de mezclar los ADNc de estas fracciones sesepararon 10 µl de las fracciones 8, 9 y 10. El ADNc de los pooles 1 y 2 y de la fracción8 fue utilizado en las transformaciones para constituir tres tipos de librerías diferentes.

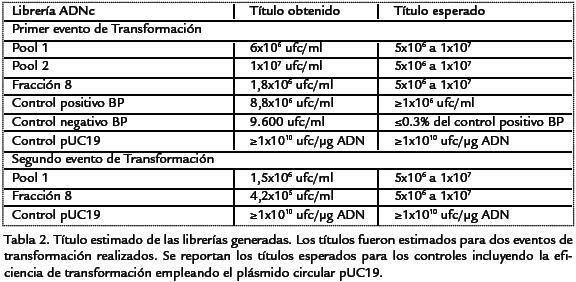
Dado que los fragmentos más pequeños son clonados más fácilmente, se decidiótomar una alícuota de la fracción 8 para tratar de obtener una librería con fragmentosmás grandes. Se realizaron dos eventos de transformación, los cuales permitierondeterminar la eficiencia de transformación y el titulo de cada una de las librerías ge-neradas pudo de esta manera ser determinado (Tabla.2). Los títulos de las libreríasvariaron entre 4,2x105y 1x107unidades formadoras de colonias (ufc)/ml, lo que co-rresponde a los títulos esperados para librerías de buena calidad y en consecuenciaconstituye un abundante número de clones lo que significa que la mayoría de genesexpresados en estas condiciones se encuentran representados en estas librerías.
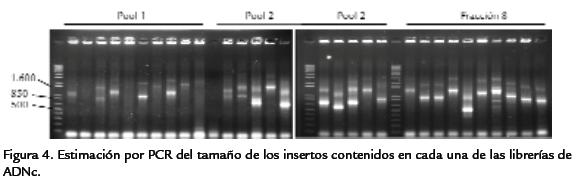
ESTIMACIÓNDELACALIDADDELALIBRERÍA
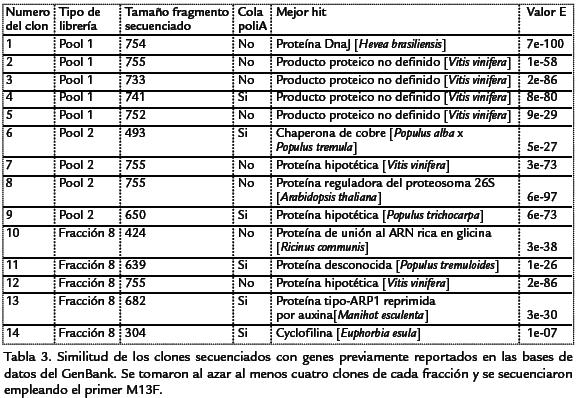
Para determinar la calidad de las librerías se procedió a tomar al azar 10 colonias decada librería y mediante PCR sobre colonias amplificar los insertos empleando losprimers M13 F y M13 R. Como se observa en la Fig. 4 el tamaño de los insertosobtenidos a partir de los clones de la librería generada a partir del pool 1 varió entre850-1.500 pb con un promedio de 1.170 pb. Para la librería generada a partir delpool 2 el tamaño de los insertos osciló entre 600 y 1.500 pb, siendo el tamaño pro-medio de los insertos de 870 pb y para la librería obtenida empleando la fracción 8los insertos presentaron un tamaño promedio de 1.090 pb. Estos tamaños corresponden posiblemente, en la mayoría de los casos, a genes completos o a una buena por-ción de ellos, confirmando la buena calidad de las librerías. Adicionalmente se toma-ron 14 colonias para realizar extracciones de ADN plasmídico a partir del cual se ge-neraron secuencias para confirmar que se tratase de secuencias expresadas de yucay no constituyeran contaminación con fragmentos de ADN genómico o de ADN con-taminante. La reacciones de secuenciación se realizaron desde el extremo 5’ y en algu-nos casos se logró detectar la cola poliA, característica de los ARNm de eucariotes,confirmando que se trata efectivamente de genes expresados. En la Tabla.3 se muestrala similitud que presentaron las secuencias cuando se compararon con las secuenciaspresentes en las bases de datos empleando el algoritmo BLASTX (Altschul et al.,1997). La mayoría de secuencias presentaron similitud con genes de uva (Vitis vinifera)y en algunos casos también con genes previamente identificados en especies estrecha-mente relacionadas a yuca, tales como Euphorbiasp. o Populusspp. (Tabla.3). El núme-ro de secuencias reportadas en yuca no es muy alto a pesar de los esfuerzos que sehan realizado en los últimos años, razón por la cual las secuencias por nosotros gene-radas a partir de la librería de ADNc no presentan similitud con secuencias previa-mente reportadas en yuca. La mayoría de las secuencias presentan similitud con Vitisdebido posiblemente a que a la hora actual es una de las especies para las cuales mássecuencias se han generado y por su relativa cercanía evolutiva con yuca. El hecho deque las secuencias obtenidas a partir de la librería de ADNc no presenten similitudcon secuencias previamente reportadas en yuca sugiere que esta librería es una fuentepara la búsqueda de nuevos genes en este cultivo.
En este trabajo reportamos la construcción de la primera librería de ADNc de yuca empleando el sistema Gateway®. Si bien existen reportes de construcción y empleo de librerías de ADNc en yuca para la identificación de ESTs (Expressed Sequence Tags), la obtención de suficiente cantidad de ARNm y de buen tamaño a partir de material vegetal de yuca ha sido siempre un factor limitante para la construcción de un amplio numero de librerías de ADNc (López et al., 2005; Sakurai et al., 2007). En nuestro caso también se presentaron numerosos inconvenientes para obtener suficiente ARNm y de buena calidad. Sin embargo al utilizar material vegetal propagado in vitro se solucionaron al menos parcialmente algunos de los inconvenientes presentados y la eficiencia en la extracción de ARN fue mejor que empleando tejido vegetal de plantas adultas. La tecnología Gateway® se ha desarrollado recientemente y permite la fácil clonación de insertos en vectores sin necesidad de reacciones de restricción y ligación, sino que emplea la recombinación para introducir los insertos en un vector de entrada. Con esta estrategia es posible transferir fácilmente los insertos en diferentes tipos de vectores empleando recombinasas. La metodología Gateway® se ha aplicado en numerosos casos para la clonación de genes particulares pero solo recientemente se desarrolló la metodología para la construcción directa de librerías empleando este sistema (Invitrogen). La librería de yuca que generamos utilizó el sistema Gateway® y constituye el primer tipo de librería de este tipo en yuca y una de las pocas que se han desarrollado empleando este sistema. El tamaño de insertos de nuestra librería osciló entre 600 y 1.500 pb y el título estuvo cercano a las 1x107 ufc/ml lo que indica que estas librerías son de alta calidad y permitirán la búsqueda de genes en diferente tipo de proyectos. En particular esta librería fue construida a partir de material vegetal que ha sido inoculado con la cepa CIO151 de Xam, y colectado a diferentes tiempos postinoculación lo que aumenta la probabilidad de encontrar genes que se expresan a lo largo de la respuesta de defensa. El hecho de clonar esta librería empleando el sistema Gateway® permitirá transferir los insertos a otro tipo de vectores dependiendo de las aplicaciones posteriores que se requieran, tales como pasarlos a vectores de expresión
o para la construcción de vectores con proteínas de fusión. En particular la librería actualmente está siendo transferida al vector pDEST22 para ser utilizada en estudios de doble híbrido para encontrar las proteínas de yuca que interactúan con proteínas del patógeno o con otras proteínas de la planta. De esta manera la librería que se ha generado se constituye en una herramienta fundamental para posteriores estudios encaminados a la búsqueda y función de genes en yuca lo que contribuirá de manera importante a desarrollar metodologías biotecnológicas que contribuyan al mejoramiento del cultivo. De la misma manera la librería de ADNc puede ser una herramienta para desarrollar una nueva colección de ESTs. Las librerías de ADNc en otros cultivos han permitido la generación de colecciones importantes de ESTs (Ronning et al., 2003, Van der Hoeven et al., 2002) de tal manera que la librería de ADNc reportada en este estudio permitiría ampliar la colección de ESTs en yuca con un potencial importante de encontrar nuevas secuencias ya que proviene de plantas propagadas in vitro para lo cual no existen reportes de secuencias. La librería también podrá ser organizada en filtros para posteriormente, empleando sondas de genes específicos, poder aislar las secuencias completas de genes.
AGRADECIMIENTOS
Agradecemos a la profesora Margarita Perea, directora del Laboratorio de Cultivos de Tejidos Vegetales del Departamento de Biología, por permitirnos acceder a su laboratorio y por su apoyo y asesoría prestada para la propagación del cultivo de yuca in vitro. A Laura Catalina Bohórquez por ocuparse de la multiplicación y del mantenimiento de todo el material de yuca in vitro. Al Dr. Jairo Cerón por su amabilidad al facilitarnos el uso del electroporador. A la facultad de Ciencias de la Universidad Nacional de Colombia por permitir a la Dra. Carolina González Almario desempeñarse como post doc en el grupo de Fitopatología Molecular del Departamento de Biología.
BIBLIOGRAFÍA
ALTSCHUL S, MADDEN T, SCHAFFER A, ZHANG J, ZHANG Z, MILLER W, LIPMAN D. Gapped BLAST and PSI BLAST: a new generation of protein database search programs. Nucl. Acids Res. 1997;25:3389-3402. [ Links ]
DEMURA T, TASHIRO G, HORIGUCHI G, KISHIMOTO N, KUBO M, MATSUOKA N, et al. Visualization by comprehensive microarray analysis of gene expression programs during transdifferentiation of mesophyll cells into xylem cells. Proc Natl Acad Sci USA. 2002;99:15794-9. [ Links ]
FAO. Food and agriculture Organization of the United Nations (FAO), Agricultural commodity Projections, FAO. 1998 Rome, Italy. [ Links ]
FAO. Perspectivas alimentarias. Análisis de los mercados mundiales. 2007. http://www.fao.org/docrep/010/ah864s/ah864s06.htm [ Links ]
JORGE V, FREGENE MA, DUQUE MC, BONIERBALE MW, TOHME J, VERDIER V. Genetic mapping of resistance to bacterial blight disease in cassava (Manihot esculenta Crantz). Theor. Appl. Genet. 2000;101:865-872. [ Links ]
KPÉMOUA KE. Etude comparative du développement de Xanthomonas campestris pv. manihotis chez des variétés de manioc sensibles et résistantes. Approches histologique, ultratructurale et cytochimique des mécanismes de la pathogènese. Thesis Faculté des sciences et techniques. 1995. Université de Nantes, pp. 95. [ Links ]
LOPEZ C, ZULUAGA A, COOKE R, DELSENY M, TOHME J, VERDIER V. Isolation of resistance gene candidates (RGCs) and characterization of an RGC cluster in cassava. Mol. Genet. Genom. 2003;269:658-671. [ Links ]
LOPEZ C, JORGE V, PIEGU B, MBA C, CORTES D, RESTREPO S, et al. A unigene catalogue of 5700 expressed genes in cassava. Plant Mol. Biol. 2004;56:541-554. [ Links ]
LOPEZ C, SOTO M, RESTREPO S, PIEGU B, COOKE R, DELSENY M, et al. Gene expression profile in response to Xanthomonas axonopodis pv. manihotis infection in cassava using a cDNA microarray. Plant Mol Biol 2005;57:393-410. [ Links ]
LOPEZ C, RESTREPO S, VERDIER V. Limitaciones de la bacteriosis vascular de yuca: Nuevos avances. Acta Biol. Colomb. 2005;11:21-45. [ Links ]
LÓPEZ C, QUESADA L, BOHÓRQUEZ A, DUQUE MC, VARGAS J, et al. Mapping EST-derived SSRs and ESTs involved in resistance to bacterial blight in Manihot esculenta. Genome. 2007;50:1078-1088. [ Links ]
LOZANO JC. Cassava bacterial blight: a manageable disease. Plant Dis. 1986;70:1089-1093. [ Links ]
OLSEN KM, SCHAAL BA. Evidence on the origin of cassava: Phylogeography of Manihot esculenta. Proc. Natl. Acad. Sci. 1999;96:5586-5591. [ Links ]
RONNING CM, STEGALKINA SS, ASCENZI RA, BOUGRI O, HART AL, et al. Comparative analyses of potato expressed sequence tag libraries. Plant Physiol. 2003;131:419-29. [ Links ]
SAKURAI T, PLATA G, RODRÍGUEZ-ZAPATA F, SEKI M, et al. Sequencing analysis of 20,000 full-length cDNA clones from cassava reveals lineage specific expansions in gene families related to stress response BMC Plant biol. 2007;7:66. [ Links ]
VAN DER HOEVEN R, RONNING C, GIOVANNONI J, MARTIN G, TANKSLEY S. Deductions about the number, organization, and evolution of genes in the tomato genome based on analysis of a large expressed sequence tag collection and selective genomic sequencing. Plant Cell. 2002;14:1441-56. [ Links ]
ZHOU Y, TANG J, WALKER MG, ZHANG X, WANG J, HU S, et al. Gene identification and expression analysis of 86,136 Expressed Sequence Tags (EST) from the rice genome. Gen Prot Bioinformatics. 2003;1:26-42. [ Links ]














