Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.13 no.3 Bogotá Sep./Dec. 2008
ESTRUCTURA MOLECULAR Y ANTIGÉNICA DE LA VACUNA CONTRA EL VIRUS DEL PAPILOMA HUMANO 16 (VPH 16)
Antigenic and Molecular Structure of Human Papillomavirus (HPV) 16 Vaccine
VÍCTOR ANDRÉS VANEGAS1, Bact.; ARLETH IVONNE RUBIO1,2, MSc.; ASTRID MILENA BEDOYA1,3, MSc.; GLORIA INÉS SÁNCHEZ1,4, Ph. D.
1 Grupo Infección y Cáncer, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia.
2 Grupo Estrategias de vacunación específicas para tumorviruses, Centro Alemán de Investigación en Cáncer, Heidelberg, Alemania.
3 Escuela de Microbiología, Universidad de Antioquia, Medellín, Colombia.
4 Departamento de Microbiología y Parasitología, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia. Correspondencia: Dra. Gloria Inés Sánchez. Carrera 51 D No. 62-29, Laboratorio 283B. Facultad de Medicina. Universidad de Antioquia. Teléfono-fax: 219 60 66. Medellín, Colombia. sanchezg@une.net.co
Presentado 2 de mayo de 2008, aceptado 11 de junio de 2008, correcciones 14 de agosto de 2008.
RESUMEN
La proteína L1 del Virus del Papiloma Humano (VPH) constituye el 80% de la cápside viral. Las vacunas profilácticas contra el VPH son sintetizadas a partir de la proteína L1 ensamblada en Partículas similares al Virus (del inglés VLP), las cuales son altamente inmunogénicas generando anticuerpos específicos de tipo y en algunos casos pueden presentar reacción cruzada entre tipos de VPH filogenéticamente próximos. La estructura de la proteína L1 del VPH es importante porque confiere estabilidad a la cápside mediante el establecimiento de interacciones intra e intercapsoméricas lo que asegura la integridad viral y antigénicamente porque contiene los epítopes que inducen la respuesta inmune protectora. En estudios en los que se evaluó la antigenicidad de la proteína L1 se determinó que los epítopes inmunodominantes de la cápside viral se encuentran en los bucles B-C, D-E, F-G, H-I y en el extremo C-terminal. Estos bucles son poco conservados entre los diferentes genotipos y se encuentran en segmentos de la proteína expuestos en la superficie de la cápside. Los aminoácidos situados en los bucles B-C, F-G y H-I son primordiales para el reconocimiento por los anticuerpos neutralizantes. Los diferentes subtipos y variantes presentan cambios en estos aminoácidos o en residuos que conforman otros epítopes. En esta revisión se presentará un estado del arte de la proteína L1 del VPH genotipo 16, la estructura y su importancia en el desarrollo de vacunas contra la infección producida por este virus.
Palabras clave: Virus del Papiloma Humano 16, cápside, vacunas contra el Papilomavirus, epítopes, anticuerpos.
ABSTRACT
Human Papillomavirus L1 protein makes up 80% of the viral capsid and self assembles in Virus-like Particles (VLP); these particles are immunogenic, generate type-specific antibodies and can induce very limited cross-reactivity among highly homologous HPV types. In addition to its structural function, it confers the stability to the capsid by establishing disulfide bonds and other intra and intercapsomeric interactions, and also contains the epitopes that induce the protective immune response of prophylactic vaccines. Immunological studies of this protein have concluded that the main epitopes of the HPV viral capsid are found in the loops B-C, D-E, F-G, H-I and the C-terminal arm. These loops are exposed on the surface of the viral capsid and have a low degree of conservation among the different genotypes. Specifically, amino acid 50 located on loop B-C and aminoacids 266, 271 and 288 located on loop F-G are important for the recognition on neutralizing antibodies. The different genotypes and variants exhibit mutations on these residues.
Key words: Human papillomavirus 16, capsid, Papillomavirus Vaccines, epitopes, antibodies.
INTRODUCCIÓN
El Virus del Papiloma Humano (VPH) es un virus ADN, desnudo y con una cápside de simetría icosahédrica asociado a varios tipos de cáncer, de los cuales el más común es el cáncer de cuello uterino. Entre las mujeres, este cáncer es el segundo en frecuencia a nivel mundial pero es el primer cáncer más común entre las mujeres en países en desarrollo seguido por el cáncer de mama. En el mundo durante el año 2002 se diagnosticaron 500.000 nuevos casos y 274.000 muertes por esta enfermedad, de las cuales el 80% ocurrieron en países en vía de desarrollo (Parkin et al., 2005).
Las vacunas profilácticas contra el VPH utilizan la proteína L1 de este virus como antígeno. Cuando la proteína L1 es expresada en sistemas in vitro, tiene la capacidad de autoensamblarse espontáneamente, adquiriendo una conformación estructural y antigénica similar al virus nativo pero sin ser infecciosa. Tales estructuras se conocen con el nombre de VLPs y hasta el momento los ensayos clínicos en humanos muestran que son inmunógenas, bien toleradas y proporcionan protección (Lowy y Schiller, 2006). La protección que ofrecen las VLPs es mediada por la producción de anticuerpos neutralizantes que reconocen epítopes específicos de genotipo (Pastrana et al., 2001). Sin embargo evidencias preliminares de algunos ensayos clínicos llevados a cabo para evaluar la eficacia de esta vacuna en humanos, sugieren que esta respuesta protectora puede tener efecto contra genotipos filogenéticamente relacionados (Paavonen et al., 2007). El objetivo de la presente revisión es describir las propiedades moleculares, antigénicas, funcionales de la proteína L1 del VPH 16, así como sus variantes genéticas.
GENERALIDADES DEL VIRUS DEL PAPILOMA HUMANO
Los VPH tienen la capacidad de infectar todo tipo de epitelio, causando diferentes manifestaciones clínicas que van desde lesiones benignas como las verrugas hasta malignas asociadas con el desarrollo de cáncer cervical y otros tipos de cáncer. Aislados o cepas de VPH se clasifican en categorías atendiendo a la homología de sus secuencias. Hasta el momento se conocen más de 120 genotipos que infectan al hombre, de los cuales VPH 16, 18, 31, 33, 35, 39, 45, 51, 52, 56, 58 y 59 están clasificados como de alto riesgo en función de su asociación causal con el cáncer cervical (Munoz et al., 2006).
El VPH posee un genoma de ADN circular de doble cadena con un tamaño de 8 kb. Funcionalmente el genoma del VPH se puede dividir en 4 regiones diferentes: la conformada por los genes E1 y E2 que regulan la replicación y la transcripción viral, la región constituída por los genes E5, E6 y E7 que codifican proteínas con alto poder oncogénico, la región LCR o Long Control Region en donde se localizan las secuencias de ADN que contienen los promotores y sitios de iniciación de replicación del genoma viral y la región conformada por los genes estructurales L1 y L2 que codifican las proteínas que forman la cápside (Lowy y Howley, 2001).
La cápside viral es de simetría icosahédrica sin envoltura y está formada por dos proteínas: La proteína L1 o mayor, de 55 kDa y que representa el 80% de la cápside, y la proteína L2 o menor, de 74 kDa (Lowy y Howley, 2001). El diámetro de la cápside es de aproximadamente 60 nm y está constituída por 72 capsómeros pentaméricos, cada uno constituído por 5 monómeros de la proteína L1. De los 72 capsómeros, 60 interactúan en simetría seis, mientras que los 12 restantes lo hacen en simetría cinco (Chen et al., 2000; Modis et al., 2002; Casini et al., 2004).
Estudios de reconstrucción en tres dimensiones muestran una estequiometría 30:1 en la composición de L1:L2, ubicando las unidades L2 en el espacio cónico central de los capsómeros pentavalentes (Griffith et al., 1992; Ishii et al., 2005; Yang et al., 2003). Estudios realizados con el VPH genotipo 11 muestran que la interacción entre L1 y L2 es de tipo hidrofóbico y que en ella participan las prolinas de las posiciones 413 y 419 de L2. La proteína menor L2 tiene dos funciones importantes en la infectividad del virus. Por un lado, participa en el proceso de encapsidación (Finnen et al., 2003) mediante el reclutamiento de pentámeros de L1 y del genoma viral (Florin et al., 2002; Day et al., 1998). Específicamente, L2 se une al ADN viral a través de islas de aminoácidos cargados positivamente en los extremos amino y carboxilo (Chen et al., 2000).
De otro lado, se demostró in vitro que anticuerpos contra epítopes de la proteína L2 son capaces de bloquear la infectividad del virus, aunque estos anticuerpos no necesariamente reconocen aminoácidos implicados en la entrada del virus a la célula huésped, lo que permite concluir que la proteína L2 participa en el proceso de adhesión celular como un ligando secundario. En este sentido, los residuos de los aminoácidos 13 a 31 y 108 a 126 en L2 juegan un papel importante en la interacción ligando-receptor (Yang et al., 2003).
CARACTERÍSTICAS MOLECULARES DE LA PROTEÍNA L1
L1 es la principal proteína estructural de la cápside debido a su participación en la entrada del virus a la célula hospedadora (Joyce et al., 1999; Culp et al., 2006) y además la proteína L1 induce una respuesta inmune protectora. En la estructura tridimensional de esta proteína se distinguen tres regiones bien definidas: el núcleo y los extremos amino y carboxilo (Chen et al., 2000; Fig. 1).
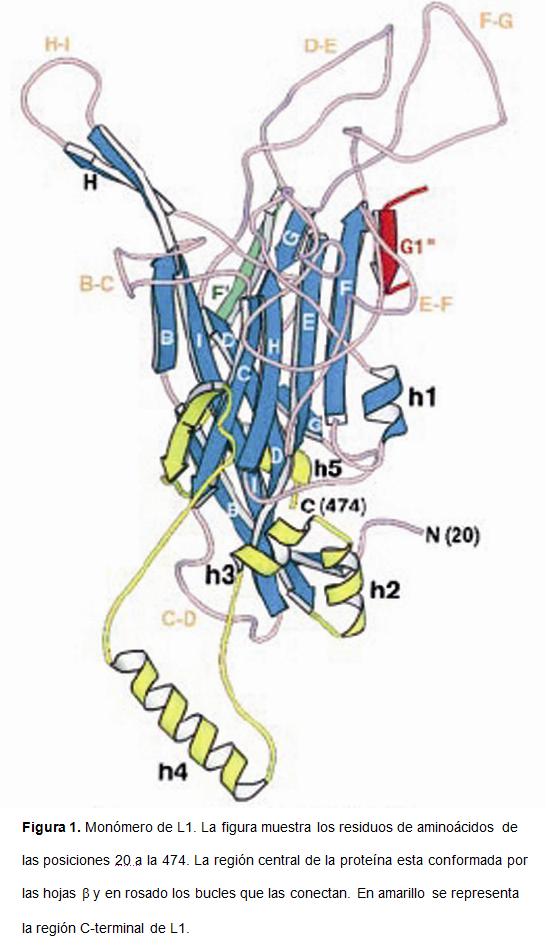
La proteína L1 está compuesta por 504 aminoácidos. El extremo amino lo componen los primeros 19 aminoácidos que son altamente conservados entre los diferentes genotipos sugiriendo que cumplen un papel fundamental en el ensamblaje. Hasta ahora no se conoce su estructura tridimensional, pero se sugiere que probablemente es una horquilla β (Modis et al., 2002). En la región central o el núcleo de la proteína se encuentran ubicados los aminoácidos 20 al 382 cuya estructura tiene forma de barril compuesto por 11 láminas ß plegadas (ß-B1, ß-B2, ß-C, ß-D, ß-E, ß-F, ß-G1, ßG2, ß-H1, ß-H2, ß-I) las cuales están conectadas entre sí por lazos o bucles que están expuestos en la superficie del capsómero (B-C, C-D, D-E, E-F, F-G, H-I). Entre los lazos F-G y H-I y las láminas ß-F y ß-G1 respectivamente se establecen puentes de hidrógeno que mantienen los monómeros de L1 juntos y confieren estabilidad a la estructura terciaria del barril (Chen et al., 2000).
Los aminoácidos 383 a 504 están en el extremo carboxilo y en él se ubican 4 de las 5 hélices α (h2, h3, h4 y h5) que tiene esta proteína. La hélice α h1 interacciona con las láminas ß-E y ß-F en la parte central o núcleo de la proteína. Las hélices α h4 y α h5, albergan la lámina ß-J y ésta ancla el brazo C-terminal a la región central o núcleo de la proteína. El brazo C-terminal es muy flexible gracias a que las posiciones 403 a 413 son ricas en glicinas y prolinas. Se postula que este segmento actúa como una bisagra que facilita los re-ordenamientos espaciales que debe hacer el extremo carboxilo para asegurar la estabilidad de la cápside viral (Chen et al., 2000). El extremo carboxilo, también es importante para mantener la integridad de L1; ya que cuando se introducen mutaciones en este extremo, la cápside es más susceptible a la digestión proteolítica por tripsina (Modis et al., 2002).
Para explicar cómo los monómeros de la proteína L1 se mantienen unidos, Modis et al. propusieron el modelo del brazo invasor. Este modelo plantea que cada capsómero dona y recibe al mismo tiempo cinco brazos terminales de los pentámeros vecinos; de manera similar a como se observa en el virus SV40 y Poliomavirus murino. La orientación del brazo del extremo carboxilo hacia cada subunidad L1 vecina tiene como finalidad formar puentes disulfuro, entre el residuo de cisteína en la posición 175 y la cisteína de la posición 428 ubicado en el brazo C-terminal del monómero adyacente, brindándole una mayor estabilidad a la cápside viral (Modis et al., 2002).
El conocimiento acerca de la estructura de la proteína L1 es importante porque permite determinar las regiones expuestas en la superficie de esta proteína lo cual tie-ne implicaciones en la ubicación de epítopes neutralizantes que pueden ser determinantes importantes para la protección inmune mediada por anticuerpos, información esencial para el desarrollo e implementación de vacunas profilácticas.
DISTRIBUCIÓN DE EPÍTOPES DE LA PROTEÍNA L1 DEL VPH 16
Mucho del conocimiento acumulado acerca de la estructura antigénica de esta proteína y por ende de la cápside del VPH 16, se logró mediante el uso de anticuerpos monoclonales y la construcción de VLPs híbridas (intercambio de aminoácidos de la proteína L1 provenientes del VPH 16 y de otros genotipos en el esqueleto de la proteína L1, tales como VPH 52, VPH 31, Papilomavirus bovino y de conejos; Carter et al., 2003). La creación de estas quimeras o híbridos es posible gracias a que las cápsides del VPH de estos papilomavirus muestran una gran similitud estructural, aunque la semejanza de sus secuencias de aminoácidos es solo del 50% (Modis et al., 2002). La secuencia de aminoácidos de la proteína L1 presenta regiones conservadas 41y regiones hipervariables. Estas últimas están preferentemente ubicadas en zonasexpuestas en la superficie del capsómero. Estas regiones variables son las queconforman los bucles o asas B-C, D-E, F-G y H-I (Chen et al., 2000; Roden et al., 1997;Carter et al., 2006; Tabla.1).

Carter et al. demostraron que la fenilalanina en la posición 50 de la proteína L1 delVPH 16, localizada en el asa B-C juega un papel crucial en el reconocimiento pordeterminados anticuerpos neutralizantes. Esto fue demostrado al observar que la sus-titución de fenilalanina por una leucina conlleva la pérdida de la unión y reconoci-miento de la cápside por los anticuerpos monoclonales H16.V5 y H16.E70. A pesarde esto, modelos tridimensionales de la cápside demostraron que este aminoácido noestá expuesto en la superficie, y tampoco es directamente reconocido por ningúnanticuerpo; sugiriendo que la sustitución de este residuo induce cambios conforma-cionales en la proteína los cuales modifican la unión antígeno-anticuerpo (Carter etal., 2003). Por otro lado, Chen et al.proponen que este aminoácido se localiza debajodel bucle F-G y que mutaciones en la posición 50 interfieren con esta asa alterandola estructura de la proteína (Chen et al., 2000). En otros estudios utilizando VLPsmutantes para el residuo de fenilalanina en la posición 50 se demostró que los mu-tantes con cambios en este aminoácido son más sensibles a la degradación portripsina que el tipo silvestre. Ello sugiere que la sustitución de Phe50 da lugar a unplegamiento alternativo de la proteína (Carter et al., 2003).
Otro anticuerpo utilizado para la identificación de la estructura antigénica del VPH esel anticuerpo monoclonal H16.V5 cuyo epítope fue descrito por Christensen et al.(2001). En los estudios realizados por Christensen et al. y luego por Carter et al. seencontró que este anticuerpo reconoce un epítope conformacional formado por lainteracción de los bucles F-G y H-I de la proteína L1 de VPH 16 y que reaccionaespecíficamente con los aminoácidos 266 al 297 y 339 al 365 según Christensen et al.,mientras que en el trabajo de Carter et al. este mismo anticuerpo reconoce losaminoácidos 260 al 290 y 349 al 358 (Carter et al., 2003).
Al igual que para el bucle B-C, la reactividad del anticuerpo H16.E70 se ve afectadapor cambios en el bucle D-E, en donde no se demostró unión directa del anticuerpocon este bucle. Esta observación permite sugerir que, tal como sucede con el bucle B-C, cambios en el bucle D-E, afectan la estructura terciaria de la cápside viral alterando su estructura antigénica (Carter et al., 2003). Por otro lado, se reportó que el anticuerpo monoclonal H16.E70 muestra una reducción en su reactividad, cuando son substituídos los aminoácidos 266, 285 y 288 en el bucle F-G de la proteína L1 aunque su epítope es aún desconocido (Carter et al., 2003). Aunque las observaciones hechas por Chen et al. a nivel del bucle F-G sugieren que los residuos en las posiciones 266, 271 y 282 son antigénicamente importantes, debido a que mutaciones en estos residuos afectan el reconocimiento por anticuerpos neutralizantes (Chen et al., 2000; White et al., 1999). Carter et al., cuyos resultados difieren de lo encontrado por Chen et al., proponen que es la posición 270 la de mayor importancia ya que al reemplazar asparagina por alanina en esta posición se induce una reducción en el reconocimiento por los anticuerpos H16.V5 y H16.E70 (Carter et al., 2003).
Aparte de su importante función en la estabilidad interpentamérica, el brazo C-terminal posee una región poco conservada entre los diferentes genotipos de VPH la cual está expuesta en la superficie de la cápside (Modis et al., 2002) y dicha variabilidad sugiere que esta región probablemente participa en la evasión del reconocimiento por anticuerpos neutralizantes. Carter et al. encontraron que el anticuerpo monoclonal H16.U4 se une a aminoácidos cercanos al residuo en la posición 430 de la región C-terminal de la proteína L1 (Carter et al., 2003), aunque esta interacción es muy débil (Roden et al., 1997). Sin embargo cabe anotar que no se ha logrado la cristalización del brazo C-terminal, y por tanto no se ha podido dilucidar su estructura terciaria, impidiendo el conocimiento de la estructura antigénica de esta región (Chen et al., 2000).
VARIANTES FILOGENÉTICAS DEL GEN L1
El Comité Internacional para la Taxonomía Viral, clasifica a los papilomavirus en genotipos, subtipos y variantes según el grado de identidad de su secuencia de nucleótidos en el gen L1. Para tal fin se compararon el genoma, o los genes de diferentes aislados para generar árboles filogenéticos. Actualmente los tipos de VPH presentan menos del 90% de identidad en el gen L1. De la misma forma los subtipos corresponden a aislamientos que tienen porcentajes de identidad en el gen L1 entre 90% y 98%, mientras que las variantes tienen valores superiores al 98% de identidad en L1 (de Villiers et al., 2001). Se han identificado variantes moleculares para casi todos los genotipos de VPH, y estos en su mayoría se agrupan en árboles filogenéticos cuyas ramas o grupos tienen una distribución geográfica específica para algunas de estas variantes (Calleja-Macias et al., 2005; Prado et al., 2005). Independientemente de la región del genoma que se estudie, las variantes del VPH 16 se agrupan en las ramas filogenéticas: Europea, Africana 1 y 2, Asiático-Americana y Norteamericana cuya frecuencia es más alta en los mismos grupos étnicos (Yamada et al., 1997), por ejemplo cerca del 80% de las mujeres caucásicas están infectadas con variantes europeas mientras que las mujeres africanas presentan con mayor frecuencia variantes africanas. Adicionalmente la variante Norteamericana se presenta exclusivamente en las tribus de indígenas norteamericanos, pero la utilidad de estas variantes no ha sido completamente estudiada (Yamada et al., 1997).
Las variaciones menores de aminoácidos que tienen las variantes pueden generar diferencias respecto a (i) la patogenicidad de las diferentes variantes y (ii) el reconocimiento antigénico por parte del sistema inmune del hospedador especialmente de los anticuerpos neutralizantes. Con respecto a las diferencias en la patogenicidad (i), estudios realizados en Brasil (Villa et al., 2000), Costa Rica (Hildesheim et al., 2001), México (Berumen et al., 2001) y Estados Unidos (Xi et al., 1997), encuentran que la variante Europea está más asociada a la presencia de infecciones persistentes mientras que las variantes Africanas y Asiático-Americanas están más asociadas al desarrollo de cáncer de cérvix. Se postuló que estas variaciones se deben principalmente a cambios en la secuencias de aminoácidos a nivel de los genes que codifican para E6 y E7, y con la tasa de actividad transcripcional del promotor de E6 (Kammer et al., 2002), permitiéndole al virus alterar los niveles de p53 y otras proteínas celulares reguladas por p53 como Bax (De la Cruz-Hernandez et al., 2005).
Los cambios en la antigenicidad de L1 (ii) han sido estudiados y aunque los subtipos y variantes de los VPH incluido el genotipo 16 tienen mapas antigénicos diferentes, (de hecho en 85 variantes de VPH 16 se observan residuos de aminoácidos diferentes cerca de los epítopes principales) estos cambios no parecen afectar el reconocimiento por los anticuerpos neutralizantes, debido a que la respuesta inmune protectora es polivalente, es decir, está dirigida a varios epítopes presentes en diferentes regiones de la proteína L1, y esto se demuestra porque a pesar de estas variaciones las VLPs son capaces de inducir una protección completa. No obstante la respuesta inmune contra cada uno de los genotipos es específica y de hecho anticuerpos dirigidos contra VPH 16 no neutralizan VPH 18 (Gasparic et al., 2007). En estudios realizados en nuestro laboratorio observamos inclusive, que sueros polivalentes con capacidad de reconocer la variantes L1 Europeas, tienen una pobre reacción contra variantes Africanas (resultados sin publicar) y Roden et al. demostraron que estas variaciones podrían dar lugar a epítopes que no son reconocidos por los anticuerpos monoclonales neutralizantes, principalmente el H16.E70 (Roden et al., 1997; White et al.,1999) y podrían afectar el ensamblaje de las VLPs in vitro interfiriendo negativamente con su inmunogenicidad en ratones (Yang et al., 2005).
El conocimiento de las variantes de la proteína L1 es importante porque permite determinar la relación entre la variación intratipo y las diferencias en el reconocimiento por los anticuerpos neutralizantes y predecir si en el futuro la presencia de variantes en una región puede disminuir la capacidad de protección de las vacunas profilácticas.
CONCLUSIONES
En ensayos clínicos aleatorizados, las vacunas profilácticas Cervarix® (GlaxoSmithKline Biological Inc) y Gardasil® (Merck Sharp & Dhome) que contienen los genotipos VPH 16 y 18 que causan el 50-70% de casos de cáncer de cérvix, han mostrado una eficacia cercana al 100% contra lesiones neoplásicas intraepiteliales de alto grado causadas por estos genotipos, en mujeres sin previa exposición viral (Paavonen et al., 2007; Ault, 2007). Debido a que las vacunas no son terapéuticas (Hildesheim et al., 2007; Markowitz, 2007), esta eficacia disminuye cuando las mujeres son vacunadas con previa exposición viral, o cuando se tiene en cuenta las lesiones causadas por genotipos no incluidos en las vacunas. Adicionalmente, la vacuna Gardasil® que también contiene los genotipos VPH 6 y 11, ha mostrado una eficacia del 90% contra lesiones neoplásicas intraepiteliales de alto grado de vagina y vulva y contra las verrugas genitales (Joura et al., 2007). Estos hallazgos claramente ofrecen la mayor esperanza para la prevención primaria de cáncer de cérvix, vagina, vulva, oral y de las verrugas genitales.
El gran avance en el desarrollo de vacunas profilácticas contra la infección por VPH se debe al descubrimiento hace aproximadamente una década de la capacidad de L1 de autoensamblarse en VLPs (Lowy y Schiller, 2006). Desde entonces se adquirió más conocimientos sobre la conformación tridimensional y el mapa antigénico de la cápside de los Papilomavirus, conocimiento que había estado limitado ya que el ciclo de vida de dicho virus requiere células epiteliales en los distintos estadios de diferenciación (Lowy y Howley, 2001). Estas VLPs son similares a las partículas virales nativas, con la capacidad de proveer epítopes que son reconocidos por anticuerpos neutralizantes en los h umanos, pero que al no poseer el ADN viral, no son infecciosas. En teoría, las VLPs pueden producirse a partir de cualquier genotipo de VPH (Lowy y Schiller, 2006), y generar una respuesta específica contra el genotipo original y en ciertos casos la respuesta generada muestra ser efectiva contra sus variantes en ciertas preparaciones de vacunas (Pastrana et al., 2001).
Las VLPs del VPH 16 son reconocidos principalmente por tres anticuerpos monoclonales neutralizantes: H16.V5, H16.U70 y H16.U4, los cuales son dependientes de la configuración tridimensional de la misma (Carter et al., 2003; Christensen et al., 2001). Los anticuerpos H16.V5 y H16.U70 reconocen epítopes ubicados en el bucle F-G donde los residuos de las posiciones 266, 271 y 282 o la 270, son cruciales para el reconocimiento por estos dos anticuerpos (Carter et al., 2006). Indirectamente, sustituciones en la posición 50 también afecta la unión de estos anticuerpos en el asa F-G al cambiar su conformación espacial. El anticuerpo H16.U4 se une al extremo C terminal de L1 y aunque este tiene la menor capacidad neutralizante es el que produce mayor título de anticuerpos (Roden et al., 1997). Los subtipos y variantes pueden alterar el reconocimiento por los anticuerpos neutralizantes, al presentar cambios en los residuos de aminoácidos que forman los epítopes neutralizantes. Las implicaciones de las variaciones de la proteína L1 de cada genotipo en la eficacia de vacunas profilácticas que usan VLPs no han sido extensamente evaluadas.
AGRADECIMIENTOS
Al Doctor Ignacio González Bravo por sus aportes para mejorar este artículo. La figura presentada fue tomada de Molecular Cell, Volumen 5, autores Chen XS, Garcea RL, Goldberg I, Casini G, Harrison SC, titulo Structure of small virus-like particles assembled from the L1 protein of human papillomavirus 16, página 560, Copyright Elsevier 2000.
BIBLIOGRAFÍA
AULT KA. Effect of prophylactic human papillomavirus L1 virus-like-particle vaccine on risk of cervical intraepithelial neoplasia grade 2, grade 3, and adenocarcinoma in situ: a combined analysis of four randomised clinical trials. Lancet. 2007;369:1861-8.
[ Links ]BERUMEN J, ORDONEZ RM, LAZCANO E, SALMERON J, GALVAN SC, ESTRADA RA, et al. Asian-American variants of human papillomavirus 16 and risk for cervical cancer: a case-control study. J Natl Cancer Inst. 2001;93:1325-30.
[ Links ]CALLEJA-MACIAS IE, VILLA LL, PRADO JC, KALANTARI M, ALLAN B, WILLIAMSON AL, et al. Worldwide genomic diversity of the high-risk human papillomavirus types 31, 35, 52, and 58, four close relatives of human papillomavirus type 16. J Virol. 2005;79:13630-40.
[ Links ]CARTER JJ, WIPF GC, BENKI SF, CHRISTENSEN ND, GALLOWAY DA. Identification of a human papillomavirus type 16-specific epitope on the C-terminal arm of the major capsid protein L1. J Virol. 2003;77:11625-32.
[ Links ]CARTER JJ, WIPF GC, MADELEINE MM, SCHWARTZ SM, KOUTSKY LA, GALLOWAY DA. Identification of human papillomavirus type 16 L1 surface loops required for neutralization by human sera. J Virol. 2006;80:4664-72.
[ Links ]CASINI GL, GRAHAM D, HEINE D, GARCEA RL, WU DT. in vitro papillomavirus capsid assembly analyzed by light scattering. Virology. 2004;325:320-7.
[ Links ]CHEN XS, GARCEA RL, GOLDBERG I, CASINI G, HARRISON SC. Structure of small virus-like particles assembled from the L1 protein of human papillomavirus 16. Mol Cell. 2000;5:557-67.
[ Links ]CHRISTENSEN ND, CLADEL NM, REED CA, BUDGEON LR, EMBERS ME, SKULSKY DM, et al. Hybrid papillomavirus L1 molecules assemble into virus-like particles that reconstitute conformational epitopes and induce neutralizing antibodies to distinct HPV types. Virology. 2001;291:324-34.
[ Links ]CULP TD, BUDGEON LR, MARINKOVICH MP, MENEGUZZI G, CHRISTENSEN ND. Keratinocyte-secreted laminin 5 can function as a transient receptor for human papillomaviruses by binding virions and transferring them to adjacent cells. J Virol. 2006;80:8940-50.
[ Links ]DAY PM, RODEN RB, LOWY DR, SCHILLER JT. The papillomavirus minor capsid protein, L2, induces localization of the major capsid protein, L1, and the viral transcription/replication protein, E2, to PML oncogenic domains. J Virol. 1998;72:142-50.
[ Links ]DE LA CRUZ-HERNANDEZ E, GARCIA-CARRANCA A, MOHAR-BETANCOURT A, DUENAS-GONZALEZ A, CONTRERAS-PAREDES A, PEREZ-CARDENAS E, et al. Differential splicing of E6 within human papillomavirus type 18 variants and functional consequences. J Gen Virol. 2005;86:2459-68.
[ Links ]DE VILLIERS EM, FAUQUET C, BROKER TR, BERNARD HU, ZUR HAUSEN H. Classification of papillomaviruses. Virology 2004;324:17-27.
[ Links ]FINNEN RL, ERICKSON KD, CHEN XS, GARCEA RL. Interactions between papillomavirus L1 and L2 capsid proteins. J Virol. 2003;77:4818-26.
[ Links ]FLORIN L, SAPP C, STREECK RE, SAPP M. Assembly and translocation of papillomavirus capsid proteins. J Virol. 2002;76:10009-14.
[ Links ]GASPARIC M, RUBIO I, THONES N, GISSMANN L, MULLER M. Prophylactic DNA immunization against multiple papillomavirus types. Vaccine. 2007;25:4540-53.
[ Links ]GRIFFITH JP, GRIFFITH DL, RAYMENT I, MURAKAMI WT, CASPAR DL. Inside polyomavirus at 25-A resolution. Nature. 1992;355:652-4.
[ Links ]HILDESHEIM A, HERRERO R, CASTLE PE, WACHOLDER S, BRATTI MC, SHERMAN ME, et al. HPV Co-factors Related to de Development of Cervical Cancer: Results from a Population Based Study in Costa Rica. Br J Cancer. 2001;4(9),1219-1226.
[ Links ]HILDESHEIM A, HERRERO R, WACHOLDER S, RODRIGUEZ AC, SOLOMON D, BRATTI MC, et al. Effect of human papillomavirus 16/18 L1 virus like particle vaccine among young women with preexisting infection: a randomized trial. JAMA. 2007;298:743-53.
[ Links ]ISHII Y, OZAKI S, TANAKA K, KANDA T. Human papillomavirus 16 minor capsid protein L2 helps capsomeres assemble independently of intercapsomeric disulfide bonding. Virus Genes. 2005;31:321-8.
[ Links ]JOURA EA, LEODOLTER S, HERNANDEZ-AVILA M, WHEELER CM, PEREZ G, KOUTSKY LA, et al. Efficacy of a quadrivalent prophylactic human papillomavirus (types 6, 11, 16, and 18) L1 virus-like-particle vaccine against high-grade vulval and vaginal lesions: a combined analysis of three randomised clinical trials. Lancet. 2007;369:1693-702.
[ Links ]JOYCE JG, TUNG JS, PRZYSIECKI CT, COOK JC, LEHMAN ED, SANDS JA, et al. The L1 major capsid protein of human papillomavirus type 11 recombinant virus-like particles interacts with heparin and cell-surface glycosaminoglycans on human keratinocytes. J Biol Chem. 1999;274:5810-22.
[ Links ]KAMMER C, TOMMASINO M, SYRJANEN S, DELIUS H, HEBLING U, WARTHORST U, et al. Variants of the long control region and the E6 oncogene in European human papillomavirus type 16 isolates: implications for cervical disease. Br J Cancer. 2002;86:269-73.
[ Links ]LOWY D, HOWLEY PM. Papillomaviruses. En: Howley PM, Knipe DM, editors. Fields Virology. Philadelphia: Lippincott Williams & Wilkins, 2001. p. 2231-2263.
[ Links ]LOWY DR, SCHILLER JT. Prophylactic human papillomavirus vaccines. J Clin Invest. 2006;116:1167-73.
[ Links ]MARKOWITZ LE. HPV vaccines prophylactic, not therapeutic. JAMA. 2007;298:805-6.
[ Links ]MODIS Y, TRUS BL, HARRISON SC. Atomic model of the papillomavirus capsid. EMBO J. 2002;21:4754-62.
[ Links ]MUNOZ N, CASTELLSAGUE X, DE GONZALEZ AB, GISSMANN L. Chapter 1: HPV in the etiology of human cancer. Vaccine. 2006;24S3:S1-S10.
[ Links ]PAAVONEN J, JENKINS D, BOSCH FX, NAUD P, SALMERON J, WHEELER CM, et al. Efficacy of a prophylactic adjuvanted bivalent L1 virus-like-particle vaccine against infection with human papillomavirus types 16 and 18 in young women: an interim analysis of a phase III double-blind, randomised controlled trial. Lancet. 2007;369:2161-70.
[ Links ]PARKIN DM, BRAY F, FERLAY J, PISANI P. Global cancer statistics, 2002. CA Cancer J Clin. 2005;55:74-108.
[ Links ]PASTRANA D, VASS W, LOWY DR, SCHILLER T. NHPV 16 VLP Vaccine Incudes Human Antibodies that Neutralize Divergent Variants of 16 HPV. Virology 2001;279:361-369.
[ Links ]PRADO JC, CALLEJA-MACIAS IE, BERNARD HU, KALANTARI M, MACAY SA, ALLAN B, et al. Worldwide genomic diversity of the human papillomaviruses-53, 56, and 66, a group of high-risk HPVs unrelated to HPV-16 and HPV-18. Virology. 2005;340:95-104.
[ Links ]RODEN R, ARMSTRONG A, HADERER P, CHISTENSEN N, HUBBERT NL, LOWY D, et al. Characterization of a Human Papillomavirus Type 16 Variant-Dependent Neutralizing Epitope. J Virol. 1997;78:6247-6252.
[ Links ]VILLA L, SICHERO L, RAHAL P, CABALLERO O, FERENCZY A, ROHAN T, et al. Molecular Variants of Human Papillomavirus Types 16 and 18 Preferentially Associated with Cervical Neoplasia. J Gen Virol. 2000;81:2959-2968.
[ Links ]WHITE W, WILSON S, PALMER-HILL FJ, WOODS RM, GHIM S-J, HEWITT LA, et al. Characterization of a Major Neutralizing Epitope of Human Papillomavirus Type 16 L1. J Virol. 1999;73:4882-4889.
[ Links ]XI LF, KOUTSKY LA, GALLOWAY DA, KUYPERS J, HUGHES JP, WHEELER CM, et al. Genomic variation of human papillomavirus type 16 and risk for high grade cervical intraepithelial neoplasia. J Natl Cancer Inst. 1997;89:796-802.
[ Links ]YAMADA T, MANOS MM, PETO J, GREER CE, MUNOZ N, BOSCH FX, et al. Human papillomavirus type 16 sequence variation in cervical cancers: a worldwide perspective. J Virol. 1997;71:2463-72.
[ Links ]YANG R, DAY PM, YUTZY WH, LIN KY, HUNG CF, RODEN RB. Cell surface-binding motifs of L2 that facilitate papillomavirus infection. J Virol. 2003;77:3531-41.
[ Links ]YANG R, WHEELER CM, CHEN X, UEMATSU S, TAKEDA K, AKIRA S, et al. Papillomavirus capsid mutation to escape dendritic cell-dependent innate immunity in cervical cancer. J Virol. 2005;79:6741-50.
[ Links ]













