Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.15 no.1 Bogotá Jan./Apr. 2010
POLIMORFISMO DE MICROSATELITES EN INDIVIDUOS DE RAZAS DE BOVINO CRIOLLO COLOMBIANO
Microsatellites polymorphism in individuals of Colombian Creole Cattle
JANETH ORTEGA TORRES1, M. Sc.;LUIS FERNANDO GARCÍA2, Ph. D. 1Facultad de Medicina Veterinaria y Zootécnica. Universidad Nacional de Colombia. Sede Bogotá. Bogotá-Colombia. yanethortega2001@yahoo.com Correspondencia:Carrera 50 C No.39-07 Bogotá-Colombia. 2 Facultad de Ciencias. Departamento de Biología. Universidad Nacional de Colombia. Sede Bogotá. Bogotá-Colombia. lfgarciap@unal.edu.co
Presentado 24 de noviembre de 2008, aceptado 15 de julio de 2009, correcciones 18 de noviembre de 2009.
RESUMEN
Se evaluó el polimorfismo de tres sistemas microsatelitales (BMS 527, BMS 4440 y BMS 2113) en 5 razas de ganado bovino criollo colombianas: ROM (Romosinuano), BON (Blanco Orejinegro), CAS (Casanareño), SM (San Martinero) CCC (Costeño con cuernos) y dos razas foráneas: Cebú y Holstein. Se encontraron 38 alelos en 105 individuos estudiados y se reportan alelos únicos para BON, ROM, CCC y CAS. La heterocigosidad esperada total incluidas las dos razas foráneas fue de 0.7228, mientras que la observada osciló entre 0.3511 y 0.7787. Se evidenció desequilibrio de Hardy- Weinberg para algunas razas en dos sistemas microsatélitales, resultado probable de efectos de selección a los cuales las razas criollas y foráneas han estado sujetas. El análisis de diversas distancias genéticas mostró que la raza más distante fue la Cebú en relación a las colombianas y a Holstein y un cluster consistente que agrupa las razas HOL-ROM-CAS. Los análisis de mezcla sugieren que las razas criollas colombianas tienen mayor aporte genético de la raza Holstein (entre un 76 y un 88%) que de Cebú (entre un 8 y un 22 %). Finalmente, se sugiere que el sistema BMS2113, podría ser de utilidad para realizar análisis de paternidad en razas criollas colombianas.
Palabras clave: microsatélites, razas criollas, polimorfismo.
ABSTRACT
The polymorphism of three microsatellite systems (BMS527, BMS4440 y BMS 2113) was evaluated in 5 Colombian creole breeds: ROM (romosinuano), BON (Blanco orejinegro), CAS (casanareño), SM (San Martinero), CCC (Costeño con cuernos) and two foreign breeds: Cebu and Holstein. Thirty eight alleles in 105 studied individuals were fouund and unique alleles are reported for BON, ROM, CCC and CAS. Total expected heterocigocity which includes the two foreign breeds was 0.7228, where as observed heterocigocity varied between 0.3511 and 0.7787.
Hardy/Weinberg desequilibrium was evident in some breeds in two microsatellite systems, as a result, probably of selection effects in creole and foreign breeds. The analysis of several genetic distances showed that the most distant breed was Cebu compared to the Colombian ones and to Holstein, and a cluster that shows HOL-ROM-CAS together. Mixture analysis suggests that Colombian creole breeds have a large genetic contribution from Holstein (between 8 and 22%). Finally we suggest that system BMS2113, could be useful for paternity tests in Colombian creole breeds.
Key words: Microsatellites, Creole breeds, polymorphism
INTRODUCCIÓN
La ganadería bovina en Colombia es una fuente importante de aporte al Producto Interno Bruto (PIB) Nacional con una participación total de 3.5%, equivalente al 26% del PIB agropecuario ya 56% del pecuario (DANE, 2001). De acuerdo con la FAO, Colombia ocupa el noveno lugar en producción ganadera a nivel mundial y el tercero en America Latina, después de Brasil y Argentina (FAO, 2003). Se reconocen en el país siete razas criollas colombianas ROM (Romosinuana), CCC (Costeño con cuernos), CAS (Casanareño), SM (San Martinera), HV (Hartón del Valle), CH (Chino Santandereano) y BON (Blanco Orejinegro) (Hernández et ál, 1996) adaptadas a diferentes condiciones medio ambientales y derivadas de ganado originario de las Islas Gomera en España, las cuales fueron traídas al continente en el segundo viaje realizado por Cristóbal Colón hacia 1493 (Beteta, 1999).
El análisis de parámetros genéticos en las poblaciones de ganado criollo es importante pues a partir de estos, es posible proponer políticas de manejo de estos recursos. El objetivo principal en todos los programas de conservación es mantener altos índices de diversidad genética, evitando los cruces consanguíneos, de tal manera que parámetros tales como Fis y heterocigosidad son importantes para determinar los niveles de consanguinidad que dichas poblaciones puedan tener (FAO, 2003).
La utilización de marcadores moleculares tipo microsatélite para analizar parámetros de diversidad genética, relaciones de parentesco, relaciones filogenéticas e identificaciones forenses, ha sido ampliamente reportada en estudios de poblaciones, básicamente debido a su alto polimorfismo y reproducibilidad (Edin y Meuwissen, 2001).
Estudios de razas de ganado criollo a nivel mundial muestran que las razas con más alta heterocigosidad son las españolas (Arranz et ál,1997), seguidas por las Africanas y Asiáticas y las de menor diversidad genética las inglesas y francesas (Machugh et ál, 1997, Moazami-Goudarzi et ál, 1997). De acuerdo a estudios previos reportados para razas criollas colombianas (Moreno et ál, 1999, Bedoya et ál, 2001, Sastre 2003y Barrera et ál, 2006) la diversidad genética de estas es relativamente alta al igual que las obtenidas para razas españolas, argentinas y bolivianas (Liron et ál, 2006), lo cual demuestra el buen manejo zootécnico que se le ha dado al ganado criollo colombiano. Sin embargo, en algunos de estos núcleos de conservación el tamaño de la población podría tener un efecto inmediato para la conservación de las mismas. Este trabajo evaluó el polimorfismo en tres sistemas microsatelitales en cinco de siete razas criollas colombianas, y dos foráneas, Holstein y Cebú.
MATERIALES Y METODOS
Se analizaron 105 individuos no emparentados, pertenecientes a cinco razas de bovinos criollos Colombianos: BON, CAS, ROM, CCC, SM y dos razas externas comerciales Freisian Holstein y Brahman. Los núcleos fueron derivados de los hatos de Corpoica e ICA y de la Hacienda Q-May (Departamento del Casanare).
Extracción de ADN y Reacción de PCR
El ADN fue extraído a partir de PBL (Pellet Bovine linfocites - por sus siglas en inglés) según el protocolo de Sambrook J, Fritsch EF, Maniatis T. (1989), o a partir de semen en pajillas congeladas en nitrógeno líquido en el laboratorio de Reproducción Animal de Corpoica. El ADN se visualizó bajo luz ultravioleta mediante geles de agarosa teñidos con bromuro de etidio. La reacción de PCR fue realizada en un termociclador MJ Research ® utilizando entre 40 y 60 µg de ADN para un volumen total final de 25 µl. Los primers utilizados fueron cedidos por la Universidad de Texas y las secuencias se encuentran disponibles con los autores. Las concentraciones de MgCl2, al igual que el número de ciclos y la temperatura de anillamiento fueron ajustadas para cada par de primers. Los primers fueron marcados en su extremo 5` con 6-Fam, Rox, o Hex. Los productos de la amplificación se visualizaron bajo luz UV en geles de agarosa al 2%.
Genotipificación
Las muestras se corrieron por electroforesis capilar en un equipo ABI 310 junto con un marcador ROX (75 y 450 pb). La asignación de alelos en pares de bases se realizó con el programa GeneScan de Applied Biosystems ®
Análisis genéticos
Las frecuencias de alelos se determinaron por conteo directo mediante MSA (Dieringer et ál, 2002). El número promedio de alelos por locus polimorfico yalelos únicos por sistema fueron determinados utilizando el programa GDA (Lewis y Zaykin, 2001). Se estableció el desequilibrio de Hardy-Weinberg por microsatelite y por raza. Se realizaron pruebas no parámetricas Mann y Witney (1947), Kolmogorov (1933) y Smirnov (1939) para muestras independientes con el fin de probar por pares la relación que existe entre los valores observados y los modelos mutacionales de microsatélites SMM (Step-wise mutational model) e IAM (Infinite allele model). Se determinaron estas pruebas como las más convenientes ya que su análoga paramétrica (prueba t), requiere supuestos de normalidad que no fueron el caso en estos datos. Las mismas pruebas evaluaron posibles diferencias para heterocigosidades observadas por raza en sistemas individuales o en combinaciones de ellos.
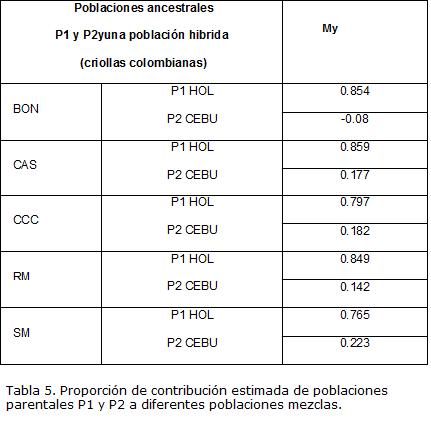
Se calculó la heterocigosidad genética esperada (He), y el contenido de información polimorfica (PIC) utilizando el programa MSA, además se calculó el poder de exclusión a priori utilizando el programa Cervus (Marshall, et ál, 1998), para cada uno de los marcadores. Mediante el programa ADMIX v 2.0 (Dupanloup y Bertorelle, 2001) se estimó el porcentaje de mezcla de cada una de las razas criollas, con respecto a las razas Cebú y Holstein, bajo el supuesto teórico del aporte genético de dos poblaciones ancestrales a una población híbrida (criollos colombianos).
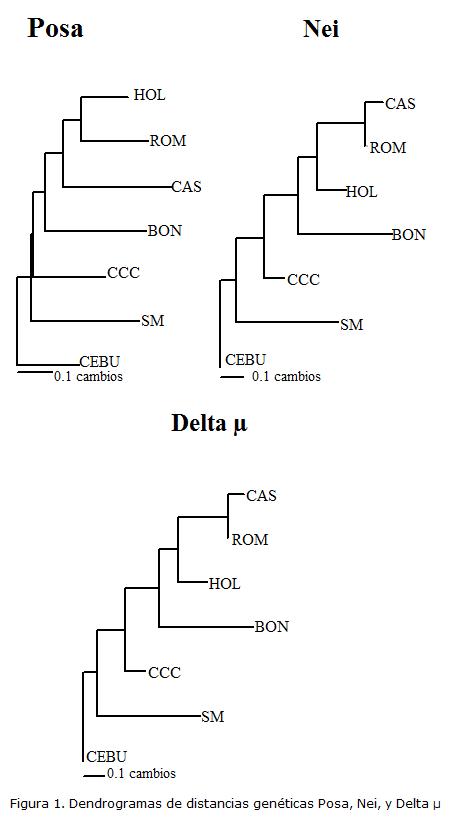
Se calcularon las distancias genéticasde proporción de alelos compartidos (POSA), Nei 83 y Delta µ(∂µ)2, utilizando el programa MSA. Los dendrogramas fueron construidos mediante PAUP (Swofford, 1999).
RESULTADOS
Frecuencias alélicas y NPA
Se detectaron 38 alelos en los tres sistemas microsatelitales estudiados con un promedio de 10.3 alelos por sistema valor cercano a los reportados por otros autores: Barrera et ál. 2006 (11.58), Moreno et ál. 2001 (8.9), Bedoya et ál. 2001 (8.8). No se encontró una distribución homogénea de frecuencias en ninguna de las razas, y en cada una se presentaron alelos con frecuencias altas vs. alelos con frecuencias bajas. El número promedio de alelos más alto fue para la raza ROM (7.3), considerando el total de los tres loci y el más bajo fue para CAS (4).
Para el sistema BMS 527, el alelo con mayor frecuencia en la población corresponde al 176 con porcentajes superiores al 30%, excepto en SM con 5%. Considerando el número total de alelos encontrados para este microsatélite (12), las razas CAS y SM presentaron únicamente 6 de ellos, mientras que ROM presentó 11. Para este mismo sistema, se encontró un alelo único para la raza BON (184) y se reportó un alelo fuera del rango establecido para este sistema en las razas HOL y ROM (118). Se encontró un alelo compartido (160) para las razas CEBU, ROM y CCC, mientras que tres alelos (170, 172 y 176) estaban presentes en todas las razas.
Para el sistema BMS4440 se encontraron un total de 10 alelos, frente a 11 reportados. La raza con menor número de alelos fue HOL con dos, y las razas con mayor número de alelos fueron BON y ROM con cinco cada una. El alelo 122 está presente en todas las razas con porcentajes superiores a 25%, excepto en SM con 5%. Para este sistema se encontraron alelos compartidos en CEBU y BON (alelo 140) mientras que para HOL y ROM el 126. Los alelos privados por raza son el 148 presente en BON, el 146 en ROM y el 134 en la raza CCC.
Para el sistema BMS2113 se encontraron 9 alelos de 11 reportados. La raza con mayor número de alelos fue ROM que presentó 6 alelos y la raza con menor fue BON con 1. Se presentaron alelos privados para ROM (alelo 144) y CAS (con el alelo 138).
Equilibrio de Hardy-Weinberg
Al examinar todos los microsatélites en conjunto entre razas, solo ROM presentó una desviación significativa del equilibrio de Hardy –Weinberg. Cuando las razas fueron examinadas individualmente por marcador, se observaron desviaciones significativas (Tabla 1). CAS fue la única raza que no presentó desequilibrio en ninguno de los loci.
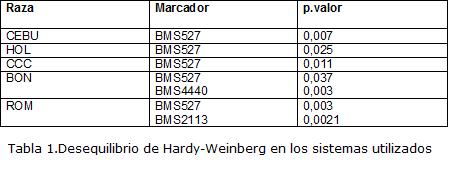
Modelos de Evolución de microsatélites
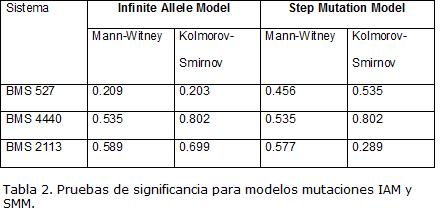
La evaluación de si existen diferencias significativas en el número de alelos observados en cada sistema y el número de alelos esperados bajo dos modelos de evolución de microsatélites (stepwise vs infinito) de acuerdo a las pruebas no paramétricas de Mann-Witney y Kolmogorov Smirnov para dos muestras independientes, mostraron que no existen diferencias significativas respecto al número de alelos obtenidos y los esperados bajo los dos supuestos mutacionales (Tabla 2).
Heterocigosidades observadas por raza
Las razas BON y SM presentaron heterocigosidades observadas (0.2388 y 0.3511 respectivamente) por debajo de las reportadas por Bedoya et ál. 2001 las cuales fueron de 0.67 y 0.73 respectivamente. Para todas las demás razas criollas se encontró que la heterocigosidad observada tiene valores similares a aquellas reportadas por Bedoya et ál. (2001) con valores entre 0.2388 para BON y 0.7666 para CEBU. También se observó que los valores encontrados en este trabajo comparados con los de Bedoya et ál.(2001)y Sastre (2003) muestran valores similares para la herocigosidad de la raza CEBU y CAS (valores entre 0.6691 y 0.7666 paraCEBU y 0.5 y 0.7273 para CAS).
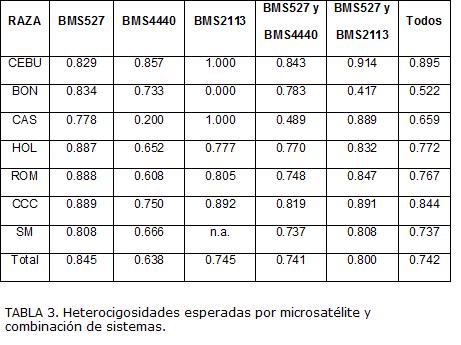
Con el propósito de observar posibles diferencias en los cálculos de heterocigosidad como resultado en la variación en el número de individuos y sistemas genotipificados, los análisis se hicieron por particiones como se muestra en la Tabla 3. En ésta se incluyen los valores de heterocigosidad para las diferentes combinaciones consideradas para cada una de las razas de estudio. Las pruebas no paramétricas no muestran diferencias significativas para las comparaciones realizadas (p=0.01).
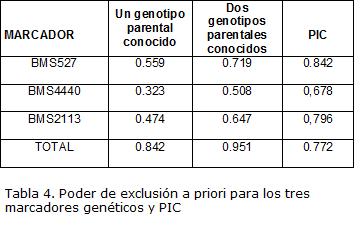
El poder de exclusión a priori calculado para cada uno de los microsatélites, mostró que el marcador con mayor poder de exclusión es el sistema BMS527, seguido de BMS2113. (Tabla 4). El poder de exclusión a priori para los tres marcadores en conjunto es de 0.9511. El contenido de información polimorfica es alto para el marcador BMS527 y el más bajo lo muestra el marcador BMS4440. Sin embargo, hay que tener en cuenta que el marcador BMS 527 se encuentra en desequilibrio de HW, por lo tanto estos valores hay que tomarlos con precaución.
Distancias Genéticas
La Fig 1 muestra tres dendogramas obtenidos con las distancias POSA (proporción de alelos compartidos), Nei Standard que asume el modelo mutacional de IAM y Delta µ que asume un modelo mutacional SMM. A pesar de supuestos biológicos distintos, la topología resultante es muy similar para las tres distancias analizadas. Las tres distancias utilizadas mostraron a Cebú como la raza más alejada, con un único cluster que incluye a las cinco razas criollas y HOL. Adicionalmente el cluster (HOL-CAS-ROM) se mantuvo con las tres distancias analizadas aunque con arreglos distintos.
Los coeficientes de mezcla que expresan el aporte genético de las razas CEBU y HOL a las razas de ganado criollo colombiano, analizado mediante el parámetro My, sugieren que la contribución de la raza Cebú a las razas colombianas está entre 8 y 22%, y la de Holstein entre 76 y 88% (Tabla 5), es decir los resultados sugieren que el aporte de la raza HOL en las razas colombianas es mayor al de CEBU.
DISCUSIÓN
Teniendo en cuenta individualmente los microsatélites utilizados, dos (BMS527 y BMS4440) permitieron identificar en 100 y 90.9% respectivamente de los alelos reportados, mientras que BMS2113 reportó el 81.8%. Por otra parte el número promedio de alelos (NPA) teniendo en cuenta los tres sistemas estudiados fue de 10.3, superior a los reportados en comparaciones de otras razas que utilizan mayor número de individuos y de sistemas. Por ejemplo, Machado et ál. (2003) reporta 7.11 alelos por locus utilizando tres razas brasileñas y Holstein analizando nueve sistemas genéticos. En razas francesas dicho valor varia entre 6 y 7.7 (Moazami-Goudarzi et ál, 1997), en italianas es de 6.5 y 8 (Ciampolini et ál, 1995), en británicas 3.4 y 3.8 (Machugh et ál, 1997) y en españolas 9.95 (Arranz et ál,1997). Con respecto a las razas criollas colombianas, el NPA encontrado es superior al encontrado por Bedoya et ál. (2001) quienes reportan un NPA de 8.8 para cinco sistemas en siete razas criollas. Es importante anotar que los NPA son obtenidos de sistemas distintos y en número de individuos y razas evaluadas variables. Bedoya et ál. 2001 utilizó 199 individuos, ocho razas y cinco sistemas, éste trabajo utiliza 105 individuos, siete razas y tres sistemas microsatelitales. Se observa que a diferencia del estudio de Bedoya et ál. 2001 éste trabajo reporta el mayor número promedio de alelos en la raza ROM (7.3 vs 3.75). El trabajo de Bedoya et ál. 2001 subestimó probablemente el verdadero NPA como resultado del bajo número de individuos muestreados (5) vs 20 de este trabajo. Por otra parte y a pesar del bajo número de individuos muestreados por Bedoya et ál. 2001 (4) para la raza CAS con respecto a los genotipicados en este trabajo (13), ambos reportan el menor NPA para las razas colombianas (3.0 y 4.0 respectivamente).
Los alelos únicos encontrados en este trabajo podrían ser útiles en un conjunto de marcadores específicos para la identificación de razas, en particular cuando se desea rastrear el origen de carne o en eventos de robo de ganado. Sin embargo, se requieren estudios de genotipos simulados para estimar la confiabilidad en la asignación de razas con alelos únicos en donde se deben probar distintas combinaciones de microsatélites. Por ejemplo, Machughet ál. (1998) reportagenotipos simulados para siete razas europeas con un máximo de 10 sistemas y un mínimo de dos. En general, a mayor número de microsatélites utilizados, la designación era más exitosa (entre 90 y 100%). Sorprendemente, Machughet ál (1998) logró la asignación correcta con un éxito del 89% utilizando solamente dos microsatélites y de 92% utilizando cuatro. En el primer caso uno de los sistemas utilizados corresponde a BMS2113 incluido en este trabajo en donde éste sistema demostró ser eficiente en las pruebas de exclusión para paternidad.
Cuando se analizaron razas individuales, se encontró que varios sistemas aparecían en desequilibrio de HW, particularmente para el primer sistema microsatelital (BMS527). Sin embargo, no parece ser el exceso de homocigotos el responsable del desequilibrio pues en general las heterocigosidades son altas junto con los valores de NPA. Es posible que fuerzas evolutivas como la deriva genética, la selección, la mutación y la migración pudieran estar rompiendo este equilibrio en las poblaciones de ganado criollo colombianas, en donde no se mantienen como poblaciones naturales sino en núcleos de conservación; dichas fuerzas evolutivas pueden estar jugando un papel importante en su acervo genético. Por una parte, el efecto de la deriva genética podría ser importante en estas poblaciones dado su tamaño relativamente limitado, y por otra es posible que dentro del ganado criollo existan alelos no neutros que representen una ventaja selectiva que contribuya a la divergencia de las poblaciones. Adicionalmente las mutaciones de baja frecuencia podrían contribuir a la diferenciación genética dentro de las poblaciones, medible solamente después de algunas generaciones. Finalmente, es importante aclarar, que el desequilibrio encontrado puede ser causado por un deficiente muestreo (Taberlet et ál, 1996).
No es de extrañar que haya desequilibrio para algunos sistemas en razas de bovinos criollos colombianos si se compara con la literatura existente. Por ejemplo Steiglederet ál. (2004) en una evaluación para diferentes razas de ganado criollo brasileño, utilizando 14 marcadores microsatelitales, mostraron que solo uno de los sistemas está en equilibrio de HW; sin embargo no se encontraron evidencias que sugieran que este desequilibrio sea debido a endogamia o a subdivisión de la población pues la heterocigosidad fue alta variando entre 0.51 y 0.89. En este caso, la atribución del desequilibrio se explicó probablemente por error de muestreo. En otro estudio Pimentel de Melo et ál (2003) caracterizaron genéticamente el ganado Aberdeen Angus del Brasil usando fragmentos de restricción y cuatro microsatélites. Se encontró que solo uno de los microsatélites estudiados (CSFM50) presentó desviación del equilibrio de HW, que los autores explican debido al uso intensivo de inseminación artificial y transferencia de embriones. Martín-Burriel et ál. (1999) analizaron la diversidad genética en seis razas nativas españolas utilizando 30 microsatélites, encontrando una de las razas estudiadas en desequilibrio de HW en cuatro de los 30 loci. Este resultado junto con la baja heterocigosidad podría deberse al efecto Wahlund, en donde la raza de toros de lidia en España se divide en pequeñas ganaderías, induciendo aislamiento reproductivo dentro de esta raza. Hansen et ál. (2002) al evaluar la diversidad genética con 15 microsatélites en razas de ganado de Canadá, y al compararlo con el ganado Pardo suizo, Holstein y Jersey de Canadá, encontraron la raza Holstein en desequilibrio en varios de los microsatélites utilizados por efectos probables de selección. Machughet ál. (1994) estudiaron la variabilidad genética en ganado Europeo, encontrando que tres de doce microsatélites presentaban desequilibrio de HW; uno de ellos localizado dentro del gen de la prolactina (PRL). En este caso, el desequilibrio podría ser explicado por efectos de selección para producción de leche dentro del ganado europeo.
Cuando se compara la información de marcadores en individuos distintos existe el problema de contar el número actual vs. el número observado de diferencias mutacionales pues dos individuos pueden compartir un alelo por ancestría común o simple convergencia. Adicionalmente, cuando dos individuos difieren en por ejemplo dos repeticiones en la longitud de un marcador microsatelital, la pregunta que surge es si se trata de una o dos mutaciones. Es decir, un conteo correcto es crítico, aunque la mayoría de las mutaciones de los microsatélites se asumea pasos únicos. Un primer modelo mutacional, modelo de alelos infinitos (Infinite allele model), asume que cada mutación que surge es única y sugiere que un número infinito de alelos es generado. Así, cada conteo es considerado como una nueva mutación. Por el contrario, el modelo de pasos (stepwise model) considera los cambios en longitud, permitiendo un paso hacia arriba o hacia abajo por cada mutación. En este trabajo, la evaluación de los dos modelos de evolución de microsatélites no evidenció que los datos se ajusten a uno más que a otro. Se presume una igual probabilidad para los eventos de deleción y adición, y existen evidencias basadas en la distribución de los alelos y análisis de pedigríes que muestran que la mayoría de las mutaciones son compatibles con el modelo stepwise.
Adicionalmente, muchos estudios han sugerido que las mutaciones tienen una tendencia hacia la expansión (Amos et ál, 1996), aunque la tendencia mutacional parece depender del tamaño del microsatélite. Dentro de un locus, los alelos más grandes tenderían a perder repeticiones y aquellos pequeños tenderían a ganarlas, lo que podría ser un mecanismo para impedir que estas regiones del genoma se expandan de manera infinita a lo largo de la evolución. Se requiere de estudios de pedigríes de bovinos para determinar las frecuencias mutacionales de distintos microsatélites, y determinar los mecanismos de generación de nuevos alelos para predecir cual modelo mutacional está operando en estas poblaciones.
La presencia de un alelo de 118 pares de bases en dos razas para el microsatélite BMS527, que está por fuera del rango esperado para éste (165-187), permite presumir que se pudo haber originado por uno o varios eventos de deleción en el interior del mismo, puesto que el alelo nuevo es mucho más pequeño. Se ha reportado en la literatura que cerca del 15 % de las mutaciones en humanos son de uno o dos pasos (Brinkmann et ál, 1998), sin embargo Huang et ál. (2002) en un estudio realizado en 53 pedigríes, encontró que cerca del 63% son mutaciones múltiples, pudiendo ser hasta de cinco o más pasoslo cual podría explicar el alelo de tamaño tan corto como el de 118 pares de bases encontrado en este trabajo.
Con el propósito de comparar las heterocigosidades esperadas incluyendo microsatélites individuales o combinaciones de los mismos, las pruebas de Mann-Witney-Wilcoxon revelaron que no hay diferencias significativas entre las heterocigosidades cuando se comparan todos los microsatélites y las combinaciones de los mismos (p <0.01). A pesar de las diferencias en el número de individuos analizados por microsatélite, los análisis demuestran que los sistemas permitieron identificar un alto porcentaje de los alelos reportados para cada sistema.
La diferencia en las heterocigosidades con respecto al trabajo de Bedoya et ál. (2001) puede ser debida al mayor número de individuos y de microsatélites, a la presencia de alelos nulos en los individuos analizados o a que los dos estudios utilizan sistemas microsatelitales diferentes.
En cuanto a las heterocigosidades observadas,solamente dos razas (BON y SM) mostraron valores relativamente distintos a los reportados por Bedoyaet ál,(2001).
Esto probablemente se debe a que para estas dos razas se tipificaron muy pocos individuos en alguno de los sistemas (BON n=1 para BMS4440, que resultó homocigoto). En el caso de las demás razas fue evidente que los valores reportados por Bedoyaet ál, (2001) o Sastre (2003) quienes utilizaron un mayor número de marcadores no difirieron de manera importante con respecto a los encontrados en este trabajo en el que se usaron tan solo tres sistemas. Esto sugiere que se pueden obtener datos informativos de una población aun en el caso de emplear pocos sistemas microsatelitales, probablemente debido al poder discriminatorio de los mismos.
De acuerdo con la historia, se sabe que las razas Españolas que entraron primero al continente Americano, debido a que estaban asentadas en el puerto de Cádiz, son las razasBerrenda, Retinta y Rubia Gallega (Beteta, 2000). Los primeros análisis bioquímicos muestran similitudes entre estas razas y las del ganado criollo colombiano (Hernández, 1976 citado en Beteta, 2000).Estudios iniciales tanto de similitud de color como de análisis de polimorfismo bioquímicos, muestran una clara ascendencia retinta para las razas colombianas ROM, CAS, CH y CCC. Puede evidenciarse una marcada cercanía entre las razas CAS y ROM, a través del análisis de dos de las tres distancias genéticas realizadas en este trabajo. Esta cercanía podría ser explicada por el ancestro único, común de la raza retinta española. Otras razas americanas como la criolla mexicana y la Long Horn de Estados Unidos, han mostrado tener ascendencia de las razas españolas Berrenda y Retinta (Beteta, 2000).
No hay un claro patrón de diferenciación entre razas lo cual es, probablemente, el resultado de la mezcla continuada llevada a cabo por décadas, entre razas criollas, y foráneas como la Cebú y la Holstein. Los coeficientes de mezcla encontrados en este trabajo sugieren que el aporte genético de la raza HOL fue mayor para todas las razas criollas que el de la raza CEBU. Similarmente, en un estudio realizado por Barrera et ál. (2006), no se encontraron haplotipos mitocondriales únicos para Bos indicus en la raza CEBU, los hallados compartían haplotipos de origen Bos taurus.
La raza que mostró mayor mezcla con CEBU fue la SM, la primera es resistente a la sequía y puede buscar alimento en grandes extensiones, lo cual pudo ser de interés para los ganaderos de la zona quienes cruzaron CEBU con SM, procurando mejorar la resistencia de la raza criolla a las sequías y la adaptación mediante la búsqueda de zonas de pastoreo en las grandes extensiones de los Llanos Orientales.
Se sabe que el desarrollo de la raza SM se centró en las sabanas secas de San Martín (Piedemonte Llanero del Departamento del Meta) y se les atribuye a los Jesuitas el inicio de la selección fenotípica de los ancestrales bovinos llaneros,a partir del siglo XVII. En 1950 para evitar la extinción de la raza por cruzamiento absorbente con toros CEBU, se estableció un núcleo en San Martín. Los coeficientes de mezcla encontrados en este trabajo, sugieren que el manejo del cruzamiento selectivo no ha sido suficiente para eliminar el componente CEBU de esta raza.
El porcentaje de mezcla de la raza CCC con CEBU, encontrado en este trabajo tiene un valor del 18%.Los primeros reportes de ganado CEBU en Colombia, aunque no son muy claros, muestran que el ganado Bos indicus fue traído inicialmente a Colombia a comienzo del siglo XX (Asocebú). Hacia el año 1913, dos toros, uno Guzerat y el otro Nerole, fueron importados de la India, para una ganadería de Zambrano, Bolívar y fueron mezclados con ganado de la costa, lo que podría explicar el porcentaje encontrado. Aunque en un momento de la historia se prohibió la importación de ganado proveniente de la India, debido a que se creía que era portador de enfermedades, ya habían transcurrido varias generaciones de cruces. Sin embargo, pocos años después se levantó la prohibición y continuaron entrando a Colombia ejemplares de Cebú, provenientes principalmente de los Estados Unidos, los cuales se dispersaron a través de la geografía colombiana y se mezclaron con las diferentes razas existentes aportando así a su patrimonio genético.Por otra parte, el bajo índice de mezcla encontrado entre la raza BON con CEBU, puede deberse a la poca adaptación de esta última a las condiciones geográficas y climáticas de la zona andina en donde se asienta el ganado BON, por lo que no se dieron las condiciones para que los ganaderos incentivaran la mezcla.
AGRADECIMIENTOS
Los autores expresan su agradecimiento a la Universidad Nacional de Colombia, a Corpoica por las instalaciones, a Colciencias por la financiación y a la Universidad de Texas por suministrar la secuencia de los cebadores y asesoria para la realización de este trabajo.
BIBLIOGRAFIA
AMOS W, RUBENSTAIN DC. Microsatellites are subject to directional evolution. Nature Genetics. 1996; 12: 13-14.
[ Links ]ARRANZ J, BAYON J, Y SAN PRIMITIVO F. Comparison ofprotein markers and microsatellites in differentiation of cattle populations.Animal Genetics. 1997;27:415-419.
[ Links ]BARRERA GP, MARTÍNEZ R, PÉREZ JE, POLANCO N, ARIZA, F. Evaluación de la variabilidad genética en ganado criollo colombiano mediante 12 marcadores microsatélites. Animal Genetics Resources Information. 2006;38: 35-45.
[ Links ]BEDOYA G, CARVAJAL L,BERMÚDEZ N, MORENO FL, MÁRQUEZ ME, et ál. Estructura molecular y poblacional del ganado criollo Colombiano (GCC). Rev.Co. Cienc.Pec. 2001; 14:2.
[ Links ]BETETA M. Llegada del vacuno español a Suramérica. Anales de la Real Academia de Ciencias Veterinarias. 2000, 7(7): 99-116.
[ Links ]BRINKMANN B, KLINTSCHARM, NEUHUBERF, HÜHNE J, BURKHARDR. Mutation rates in microsatellites: Influence of the structure and length of the tandem repeat. American J Human Genetics.1998; 62: 1408-1415.
[ Links ]CIAMPOLINI R, MOAZAMI G, VAIMAN D, DILLMAN C, MAZZATI E. Individual multilocus genotypes using microsatellite polymorphisms to permit the analysis of the genetic variability within and between Italian beef cattle. Journal of Animal Science. 1995;73: 3259-3568.
[ Links ]DANE. Encuesta Nacional Agropecuaria. Resultados (Internet). 2001 Disponible en: http://www.dane.gov.co/daneweb_V09/#twoj_fragment1-3.
[ Links ]DIERINGER D, SCHLÖTTERER C. Microsatellite analyzer (MSA): a plataform independent analysis tool for large microsatellite data sets. Molecular Ecology Notes .2003;3(1): 167-169.
[ Links ]DUPANLOUP I, BERTORELLE G. Inferring admixture proportions from molecular data: extension to any number of parental populations. Molecular Biology and Evolution. 2001; 18(4): 672-675.
[ Links ]EDIN H, MEUWISSEN TH. Marker-Based estimates of between and within population kinships for the conservation of genetic diversity. J Anim Breed Genet. 2001; 118(3) 141-159.
[ Links ]FAO. Food and Agricultural Organisation of the United Nations. Documento Líneas directrices- Caracterización, (MoDAD) (Internet). 2003. Disponible en: http://www.fao.org/dad-is/
[ Links ]HANSEN C, SHRESTHA JNB, PARKERRJ, CROWGH, MCALPINE PJ, DERRJN. Genetic diversity among Canadians, Brown Swiss, Holstein and Jersey cattle of Canada based on 15 bovine microsatellite markers. Genome. 2002; 45:897-904.
[ Links ]HERNÁNDEZ BG, PINZÓN ME, HUERTAS RH, GONZÁLEZ CR, MARTÍNEZ CG, GÓMEZ J. Y VALDERRAMA M. Razas bovinas criollas y colombianas. Banco Ganadero. 1996: 22p.
[ Links ]HUANG QY, XU FH, SHEN H, DENG HY, LIU YJ, et ál. Mutation patterns at dinuleotide microsatellite loci in humans. Am J Hum Genet. 2002;70:625-634.
[ Links ]KOLMOGOROV AN. Sulla determinazione empirial di una legge di distribuzione. Giornale dell Institute Italiano degli Altuari.1933;4:83-91
[ Links ]LEWIS PO, ZAYKIN D. Genetic Data Analysis: Computer program for the analysis of allelic data [programa de ordenador]. Version 1.0 (d16c).2001. Free program distributed by the authors over the Internet from http://hydrodictyon.eeb.uconn.edu/people/plewis/software.php
[ Links ]LIRON JO, PERO-GARCÍA P, GIOVAMBATTISTA G. Genetic Characterization of Argentine and Bolivian Creole Cattle Breeds Assessed through Microsatellites. Journal of Heredity. 2006; 97 (4): 331-339.
[ Links ]MACHADO MA, SCHUSTER I, MARTÍNEZ ML, CAMPOS AL. Genetic Diversity of four Cattle Breeds Using Microsatellite Markers. R. Bras. Zootec. 2003; 32: 93-98.
[ Links ]MACHUGH D, LOFTUS RT, BRADLEY D, SHARP PM, CUNNINGHAM P. Microsatellite DNA variation within and among European cattle breeds. Proc.R.Soc.Lond. 1994; 256: 25-31.
[ Links ]MACHUGH D, SHIVERMD, LOFTUSRT, CUNNINGHAM P, BRADLEYD. Microsatellite DNA variation and the Evolution, domestication and Phylogeography of the Taurine and Zebu Cattle (Bos Taurus and Bos indicus). Genetics. 1997; 146:1071-1086.
[ Links ]MACHUGH D, LOFTUS RT, CUNNINGHAM P, BRADLEY DG. Genetics structure of seven European cattle breeds assessed using 20 microsatellites markers. Animal Genetics. 1998; 29 (5): 333-340.
[ Links ]MANN HB Y WITNEYDR. On a test on whether one or two random variables is stochastically larger than the other. Annals of Mathematical statistics. 1947; 18: 50-60.
[ Links ]MARSHALL TC, SLATE J, KRUUK L, PEMBERTON JM. Statistical confidence for likelihood-based paternity inference in natural populations. Molecular Ecology. 1998; 7: 639-65.
[ Links ]MARTÍN-BURRIEL I, GARCÍA-MURO E, ZARAGOZA P. Genetic diversity analysis of six Spanish native cattle breeds using microsatellites. Animal Genetics. 1999; 30: 177-182.
[ Links ]MOAZAMI-GOUDARZIK, DLALOE, FURET JP, GROSCLAUDE F. Analysis of genetic relationships between 10 cattle breeds witp7 microsatellites. Animal Genetics. 1997; 338-345.
[ Links ]MORENO FL, DAVIES S, ESTRADA JL, MÁRQUEZ ME, CARVAJAL LG, et ál. Estudio Genético Molecular de Ganado Criollo Colombiano. Revista Colombiana de Ciencias Pecuarias. Colombia. 1999;12: 255-255.
[ Links ]MORENO F, BEDOYA G, DERR J N, CARVAJAL LG, BERMÚDEZ N, ZULUAGA FN, et al. Diversidad y relaciones filogenéticas del ganado criollo colombiano. Rev Corpoica. 2001;3(2):17-25.
[ Links ]PIMENTEL DE MELLO L, TAMBASCO-TALHARID, LEHMANN COUTINHO AL, DE ALMEIDA REGITANO LC. Genetic characterization of Aberdeen Angus cattle using molecular markers. Genetics and Molecular Biology.2003;26:2:33-137.
[ Links ]SAMBROOK J, FRITSCH EF, MANIATIS T. Molecular Cloning: a Laboratory Manual, 2nd edn. Cold Spring Harbor Laboratory Press, New York. 1989.
[ Links ]SASTRE HJ. Ganado criollo Casanareño, patrimonio genético bovino de la Orinoquía Colombiana. Descripción etnológica y estrategias para su rescate y conservación. (Tesis de doctorado). Facultad de Veterinaria. Universidad de Córdoba, España.2003.
[ Links ]SMIRNOV NV. Estimate of deviation between empirical distributions functions in two independent samples (In Russian). Bulletin Moscow University.1939; 2: 3-16.
[ Links ]STEIGLEDER CS, ALMEIDA EA, WEIMER TA. Genetics Diversity of Brazilian creole cattle based on fourteen microsatellite loci. Arch. Zootec. 2004; 53:3-11.
[ Links ]SWOFFORD DL. PAUP* Phylogenetic Analyses Using Parsimony,and other methods. Sinauer Associates, Sunderland, MA. 1999.
[ Links ]TABERLET P, GRIFFINS, GOOSEEN SB, QUESTIAU S, MANCEAU V, et ál. Reliable genotyping of samples with very low DNA quantities using PCR. Nucleic Acids Research. 1996; 26: 3189-3194.
[ Links ]













