Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.15 no.2 Bogotá Mayo/Aug. 2010
EXPRESIÓN DE LA PROTEÍNA Cry1Ac EN TEJIDOS DE LÍNEASTRANSGÉNICAS DE PAPA (Solanum tuberosum spp. ANDÍGENA)VAR. DIACOL CAPIRO.
Cry1Ac Protein Expression in Tissues of Potato (Solanumtuberosum spp. Andígena) Transgenic Lines Var. Diacol Capiro.
PABLO ANDRÉS VANEGAS ARAUJO1, M.Sc.; JENNIFER TERESA BLANCO MARTÍNEZ1, Bióloga; ALEJANDRO CHAPARRO-GIRALDO1, Ph. D. 1 Departamento de Biología e Instituto de Genética. Grupo de Ingeniería Genética de Plantas. Universidad Nacional de Colombia. Bogotá, Colombia. achaparrog@bt.unal.edu.co
Presentado 11 de septiembre de 2009, aceptado 26 de noviembre de 2009, correcciones 14 de diciembre de 2009.
RESUMEN
La papa (Solanum sp.) es el cuarto producto alimenticio más importante en el mundo. En Colombia anualmente se producen alrededor de 2,8 millones de toneladas, sirviendo como sustento económico a 90.000 familias. En el país, Tecia solanivora genera el mayor impacto económico en el cultivo con pérdidas de hasta el 100% en la producción de tubérculos. El fitomejoramiento vía introducción de genes Cry, que codifican para cristales proteicos insecticidas, constituye una alternativa para reducir el ataque de insectos en cultivos de interés comercial. En este trabajo se caracterizó la inserción, transcripción y expresión del gen Cry1Ac en diferentes tejidos y en tres etapas del desarrollo para dos líneas transgénicas de Solanumtuberosum spp. andígena variedad Diacol Capiro generadas previamente por transformación con Agrobacteriumtumefaciens. La caracterización se realizó a través de técnicas de PCR, RT-PCR y ELISA. Se corroboró la inserción y transcripción del gen utilizando primers que amplificaron una banda específica de 766pb para Cry1Ac. Los niveles de expresión de la proteína llegaron a ser mayores a 45µg/g y no mostraron diferencias significativas entre las líneas analizadas, ni entre las tres etapas del desarrollo. No se evidenciaron diferencias significativas entre las líneas transgénicas con respecto al control al hacer un análisis de algunas características fenotípicas relevantes. Los resultados encontrados sugieren la realización de seguimientos y ensayos de bioseguridad sobre este material, ya que los altos niveles de expresión en todos los tejidos analizados, pueden afectar a organismos no blanco.
Palabras clave: S. tuberosum, Diacol Capiro, Cry1Ac, caracterización molecular, cultivos transgénicos.
ABSTRACT
The potato plant is the fourth most important crop in the world. In Colombia around
2.8 million tons are produced annually economically supporting 90000 families. In the country, the major economic impact in the crop is caused by Tecia solanivora that originates loses up to 100% in the tuber production. The genetic plant breeding related to the introduction of Cry genes which codify insecticidal crystal proteins is an alternative for reducing the insect attack in commercial crops. In this work, the insertion, transcription and expression of Cry1Ac gen was characterized in different tissues and three development stages of two transgenic lines of Solanum tuberosum variety Diacol Capiro that were previously transformed by Agrobacterium tumefaciens method. The characterization was realized by PCR, RT-PCR and ELISA techniques. The gen insertion and transcription was confirmed using primers for Cry1Ac gen that amplified a specific band of 766 bp. The protein expression levels were higher than 45 µg/g and were not significantly different between the analyzed lines nor the three development stages. Furthermore, taking into account some relevant phenotypic features, no significant differences were found between transgenic lines and controls. The results suggest that monitoring and biosecurity assays are necessary with this vegetal material because their high level expression inside all the tissues analyzed that could affect non-targeted insects.
Key words: S. tuberosum, Diacol Capiro, Cry1Ac, molecular characterization, GM crops.
INTRODUCCIÓN
Los insectos son agentes bióticos limitantes de la productividad de cultivos de interés comercial. Para el manejo de estas plagas, se recurre al uso de productos fitosanitarios insecticidas, dentro de los cuales, los de mayor empleo en el cultivo de la papa son clorpirifos, metamidofos, carbofuran y carbosulfan (CEVIPAPA, 2008). Los dos primeros insecticidas son organofosforados y han generado problemas de salud. Según Hurtado y Gutiérrez, 2005, se ha reportado que al año hasta el 3% de los trabajadores agrícolas sufren intoxicación por estos compuestos. Por su parte, el uso de carbofuran y carbosulfan ha demostrado tener potenciales efectos mutagénicos y embrionarios en mamíferos (Giri, 2003).
Otra estrategia es el control biológico de insectos, que aunque genera menores impactos a nivel ambiental, no ha demostrado alto niveles de eficiencia. El uso de virus de la familia Baculoviridae, que es altamente específica para ciertas especies de insectos, se considera como una de las herramientas más promisorias para el control de plagas (CIP, 2000). Dentro de las prácticas culturales realizadas está la recolección manual de pupas, el control de malezas, la eliminación de residuos de la cosecha, las trampas para capturar adultos en los bordes de los lotes y la construcción de drenajes adecuados (López-Ávila, 2000). Dentro del contexto de manejo integrado de plagas se ha buscado obtener variedades vegetales que posean resistencia a los insectos plaga. Una tecnología que se viene explorando y que puede ofrecer soluciones parciales al problema es la utilización de plantas transformadas con genes que confieren resistencia a insectos (Chaparro et al., 2003). Se denominan plantas transgénicas a aquellas plantas que fueron modificadas por la introducción de uno o varios genes (con sus respectivas secuencias reguladoras) por técnicas moleculares, y que les confieren una(s) nueva(s) característica(s) (Byrne, 2004). Para la modificación de plantas que contengan características particulares se han buscado genes en diversas especies con características promisorias para los cultivos.
Dentro de las especies bacterianas con genes que pueden conferir resistencia a insectos se encuentra la bacteria Gram+ Bacillus thuringiensis (Bt)¸ la cual durante la fase de esporulación produce cristales paraesporales consistentes en una o más d-endotoxinas de 130 kDa denominadas proteínas Cry (Benítez, 2005). El uso del grupo de genes, que codifican para dichas proteínas, se remonta a 1981 cuando Schnepf y Whiteley clonaron y lograron expresión de uno de estos genes en Escherichia coli (Schnepf et al., 1998). Más adelante se logró la obtención de tomate, tabaco, maíz y plantas de algodón transformadas con estos genes. Por otra parte, los agricultores orgánicos usan B. thuringiensis en su presentación convencional como insecticida bacteriano (Betz, et al., 2000). En la tabla 1 se encuentran relacionados los eventos de transformación con los distintos genes cry en algunas especies vegetales, para contrarrestar el ataque de lepidópteros y coleópteros (Mentaberry, 2007).

La caracterización de los materiales transgénicos se realiza sistemáticamente mediante el estudio de la inserción y expresión del transgén en los materiales potencialmente transformados (Weng et al., 2004). Una de las técnicas que se utiliza con el propósito de evidenciar la inserción del transgén es la reacción en cadena de polimerasa (PCR), la amplificación de un fragmento de DNA a partir de primers específicos, evidencia la presencia del gen de interés (Weng et al., 2004; Valderrama et al., 2007; Smith et al. 2004). Estudios de expresión a nivel de mRNA, se pueden hacer utilizando la técnica de RT-PCR, en la cual se utiliza la retrotranscripción de RNA para producir cDNA y posteriormente ensayar la amplificación específica del cDNA de interés mediante PCR (Akhurst et al., 2003). Para estudiar la expresión a nivel de proteína, es posible usar pruebas comerciales específicas para la detección de proteínas como ELISA, con la cual se puede obtener la concentración estimada de proteína en distintos tejidos (Valderrama et al., 2007; Nayak, et al., 1997). El uso de cultivos transgénicos en la agricultura llegó en 2008 a 166 millones de hectáreas en 25 países, que representan el 11% de las 1500 millones de hectáreas cultivadas en el mundo. Colombia se encuentra en el puesto 16, siendo los cultivos de algodón y maíz los más importantes (James, 2008). En el mundo el principal cultivo transgénico es la soya con 65,8 millones de Ha, seguido del maíz con 37,3 millones de Ha, el algodón con una superficie de 15,5 millones de Ha, y la canola con 5,9 millones de Ha (James, 2008). Con esta tecnología se han reportado beneficios económicos cercanos a los 44.000 millones de dólares, y beneficios ambientales al reducir la utilización del principio activo de pesticidas en 359.000 toneladas métricas y disminuir la emisión de CO2 en 14,8 millones de kg (James, 2008).
El uso de cultivos transgénicos al igual que cualquier tecnología entraña riesgos potenciales, por lo cual se han creado estándares de bioseguridad que se basan en protocolos técnicos internacionales (Smith, 2004). Se han realizado diferentes estudios de bioseguridad en cultivos transformados con genes Cry para estimar el impacto de su consumo sobre el hombre, insectos no blanco y polinizadores asociados, en ellos no se han reportado efectos adversos o negativos (Fisher y Rosner, 1959; Sears, 2001; Betz, et al. 2000; Groot y Dicke, 2002; Hanley et al., 2003).
En este trabajo se realizó la caracterización, en tres estadios de desarrollo y tres tejidos, de dos líneas transgénicas de la variedad Diacol Capiro, desarrolladas por la Unidad de Biotecnología Vegetal (UBV) del convenio Universidad Nacional de Colombia Sede Medellín (UNCM) y la Corporación para Investigaciones Biológicas. La caracterización molecular fue realizada a nivel de DNA, RNA y Proteína. También se analizo si la transformación tuvo algún efecto en el fenotipo.
MATERIALES Y MÉTODOS
RECEPCIÓN Y MANTENIMIENTO DE CULTIVO VEGETAL
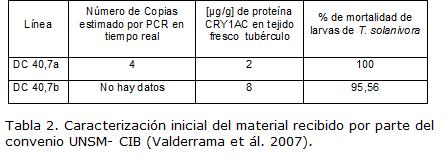
El material vegetal de partida fueron plántulas in vitro de líneas transgénicas de la variedad Diacol Capiro (DC) y Parda Pastusa (PP) de S. tuberosum, que expresan el gen Cry1Ac, provenientes de la UBV. El trabajo se realizo sobre las líneas transgénicas DC. Este material tuvo una caracterización inicial realizada por Valderrama et al., 2007 (Tabla 2). Se realizo la micropropagación mediante transferencia del meristemo apical y los dos primeros nudos a medio MS (Murashige y Skoog, 1962) fresco, suplementado con AG3 (0,2 mg/l), ácido D-pantoténico (1 mg/l) y Tiamina (0,4 mg/l). Los explantes se mantuvieron en un fitotrón -Versatil Environmental Test Chamber- (Sanyo®) con una temperatura de 17 ± 2 °C, humedad relativa (HR) de 60% ± 5% y un fotoperiodo 16 horas luz / 8 horas oscuridad.
CONFIRMACIÓN DE LA PRESENCIA DEL TRANSGÉN
La extracción del DNA de cada una de las líneas, se hizo siguiendo el protocolo descrito por Dellaporta, 1983, con modificaciones realizadas por el Grupo de Ingeniería Genética (IGP) de la Universidad Nacional de Colombia sede Bogotá (UNCB). A partir de este DNA se efectuaron los ensayos de PCR. Se obtuvo un promedio de 3,2 µg/µl de ADN con un grado aceptable de pureza para los ensayos de PCR, con una modificación realizada al protocolo, consistente en aumento de 30 minutos en el tiempo de precipitación con isopropanol.
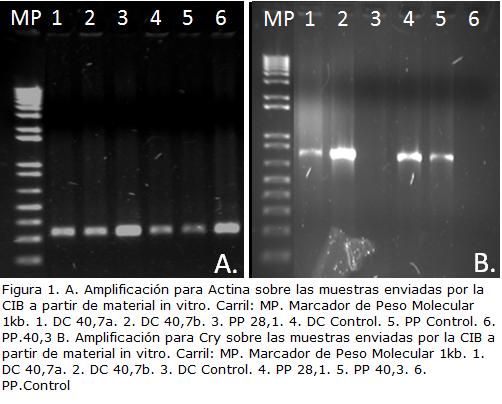
Las reacciones de amplificación se realizaron en el termociclador My Cycler® (BioRad), en un volumen final de 25 µl conteniendo 1 x de Buffer de reacción, 4 mM de MgCl2, 0,2 mM de dNTPs, 0,1 µM de cada uno de los primers, 1 U de Taq polimerasa (Invitrogen) y 5 µl de DNA. Para la amplificación de Cry1Ac se utilizaron las siguientes secuencias de primers: Forward 5’-ATCTTCACCTCAGCTGCTT-3’ y Reverse 5’-GGCACATTGTTG TTCTGTGG-3’ (Valderrama etal., 2007), a partir de los cuales se obtiene un amplicón de 766 pb. Como control interno de reacción se uso el gen actina cuyo set de primers fueron diseñados por el Grupo IGP: forward 5’-ATCGTCAGGGATGTGAAAGA-3’ y reverse 5’-ATACCGGGGAACATGGTAGT-3’, a partir de los cuales se obtiene un amplicón de 260 pb. La desnaturalización inicial se realizó a 95 ºC por 4 minutos, seguida por 35 ciclos de amplificación: desnaturalización (95 ºC por 30 s), anillamiento (55 ºC por 30 s) y extensión (72 ºC por 45 s). La elongación final se ejecutó por cinco minutos a 72 ºC. La detección de las bandas de amplificación se realizó por visualización en UV de un gel de Agarosa al 1% teñido con bromuro de etidio en concentración 0,5 µg/µl.
ENDURECIMIENTO DEL MATERIAL IN VITRO
Una vez se removió el agar de las raíces de las plántulas in vitro, se transfirieron a materas de 1 kg que contenían turba. Se colocaron tres plantas por matera y se cubrieron con un vaso plástico de 12 oz invertido. Se utilizaron 20 materas por línea transgénica y 20 por línea control. Se llevaron a un cuarto de crecimiento ubicado en el Instituto de Genética de la UNCB donde se mantuvieron con una temperatura de 17±3°C, HR de 60%, fotoperiodo 16/8 y riego cada dos días. Luego de diez días, se abrió un orificio en cada uno de los vasos plásticos, se repitió este proceso cinco días después y finalmente, a los 20 días de transferidas, se retiró el vaso de cada una de las materas. Se dejaron tres días adicionales para la aclimatación del material y luego se transfirieron las plantas a bolsas de vivero de 4 kg llenadas hasta la mitad con tierra de vivero con 5 g de fertilizante triple 15 y se llevaron a condiciones de confinamiento con variación en la temperatura con un mínimo de 4 °C en la noche y un máximo de 20 ºC en el día. Transcurridos 15 días se realizó el primer aporque y a los 30 días el segundo, junto con la segunda fertilización con 5 g triple 15 manteniéndose con riego constante hasta el momento en que se procedió a realizar la toma de los tejidos para las pruebas de laboratorio. El acceso al cuarto fue restringido y los materiales de desecho se esterilizaron con autoclave a 15 libras de presión y 120 ºC durante 15 minutos.
CARACTERIZACIÓN FENOTÍPICA
Se tomaron mediciones de variables cuantitativas (longitud total, peso fresco, número de hojas y número de ramas) a las plantas de cada línea transgénica y del control, con el fin de analizar si se presentaron cambios fenotípicos significativos. Estas mediciones fueron tomadas en los meses uno, tres y cinco de desarrollo de las plantas.
VERIFICACIÓN DE LA TRANSCRIPCIÓN DE CRY1AC
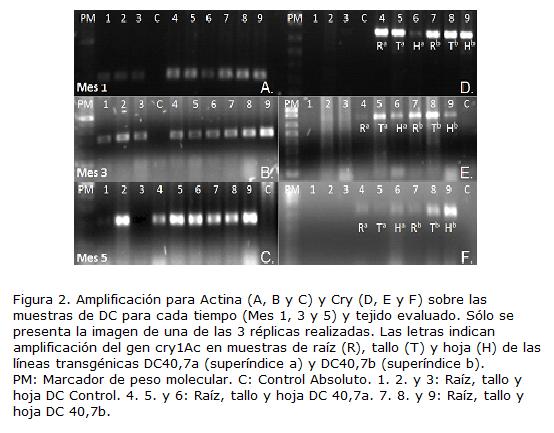
Se seleccionaron al azar tres materas para cada tratamiento y de cada matera se tomó al azar una de las plantas que fue lavada, secada y procesada para la extracción de RNA de los distintos tejidos siguiendo la metodología indicada por la casa comercial Invitrogen, 2008, para el uso del reactivo TRIzol®. Se realizó la síntesis de cDNA a partir de 3 µl de RNA y posteriormente se hizo la amplificación por PCR siguiendo el protocolo de RT-PCR recomendado por Larsen, 2005. El resultado fue analizado mediante electroforesis en gel de agarosa al 1%, 90 voltios por 40 minutos.
CARACTERIZACIÓN DE LA EXPRESIÓN CRY1AC
Sobre las líneas transgénicas que mostraron amplificación por PCR y trascripción del gen, se tomaron muestras en los meses uno, tres y cinco. Se seleccionaron al azar tres materas para cada tratamiento y de cada matera se tomó al azar una de las planta que fue lavada, secada y almacenada entre papel absorbente y bolsas Zip-Lock a -80 °C. Estas muestras fueron las utilizadas para los ensayos con la proteína transgénica. Sobre este material se realizó la prueba DAS-ELISA con el QualiPlate™ Kit for Cry1Ab/Cry1Ac (Envirologix™) siguiendo las recomendaciones del fabricante. Las lecturas de absor-bancia se hicieron en un espectrofotómetro SensidentScan (Merck) a 450 nm. Con el propósito de establecer los datos de concentración a partir de las absorbancias obtenidas, se realizaron lecturas de absorbancias de siete concentraciones definidas de la proteínaCry1Ac contenida en el Kit QualiPlate™ Kit for Cry1Ab/Cry1Ac, con estos datos se construyo la ecuación de la recta de la forma y=mx+b con la que se definieron los datos de concentraciones de los distintos tejidos para las absorbancias obtenidas en la lectura.
ANÁLISIS ESTADÍSTICO
El análisis estadístico de los resultados de las pruebas de ELISA se ejecutó mediante una prueba de ANOVA con el fin de realizar comparaciones entre los distintos factores evaluados teniendo en cuenta las siguientes tres unidades experimentales y niveles:
- a. Líneas: Niveles (Control; 40,7.a; 40,7b).
- b. Meses: Niveles (1,3,5).
- c. Tejidos: Niveles (Raíz, tallo, hoja).
Como variable respuesta se tuvo cada uno de los datos de concentración estimados a partir de los datos de absorbancia leídos por la prueba de DAS-ELISA. El modelo propuesto es un Diseño de bloques completamente aleatorizados (DBCA) y el modelo a seguir fue el siguiente:
Yijkl = µ+ τi+ γj+ (τγ)ij + βk(j) + εijkl donde τi es el efecto del tratamiento i-ésimo (i = 1, 2, 3); γj es el efecto del tejido j-ésimo (j = 1, 2, 3) y βk(j) es el efecto del mes k-ésimo (k = 1, 2, 3), m es la media general, εijkl corresponde al error dado por las repeticiones y las interacciones entre los tratamientos y los tejidos están dadas por la expresión (τγ)ij. Se realizó la validación de supuestos de normalidad, homocedasticidad e independencia para hacer la comprobación de las siguientes hipótesis: Ho: No existen diferencias entre las líneas (incluyendo el control); Ha: Existe al menos una diferencia entre líneas (podría ser dada por el control).
ANÁLISIS DE RIESGO BIOLÓGICO POTENCIAL
Con base en las concentraciones observadas de la proteína en los tejidos, se examinaron posibles escenarios de riesgo biológico con respecto a la entomofauna asociada al cultivo de la papa. En estos escenarios se analizó la posible incidencia que esta tecnología podía tener sobre insectos no blanco que están directa e indirectamente relacionados al cultivo, tanto para los benéficos como para las plagas.
RESULTADOS Y DISCUSIÓN
CONFIRMACIÓN DE LA PRESENCIA DEL TRANSGÉN
De la PCR para el gen actina endógeno (Fig. 1A) se puede observar que todas las muestras presentaron el amplicon específico de 267 pb, lo que indica que el protocolo utilizado es el apropiado. Este resultado se verifico para las líneas transgénicas DC, para las líneas transgénicas PP (carriles 1,2,3 y 6) y para las líneas control no transformadas de las mismas variedades (carriles 4 y 5). Los resultados de la PCR para el gen Cry1Ac (Fig. 1B) mostraron el amplicón esperado de 766pb en las líneas transgénicas DC (carriles 1 y 2) y en las líneas transgénicas PP (carriles 4y5). Este resultado no se observó en las líneas control no transformadas DC (Carril 3) y PP (Carril 6) (Fig. 1B). De esta manera se demostró la presencia física del transgen en el genoma de las líneas transformadas.
CARACTERIZACIÓN FENOTÍPICA
La caracterización fenotípica general se realizó con el fin de estudiar si la introducción del gen, al menos para los procesos involucrados en las características fenotípicas evaluadas, provocó algún cambio en la expresión de estas características. La pregunta es válida en tanto que el sistema de transformación mediado por Agrobacterium integra el transgén de forma aleatoria en cualquier cromosoma (Fernández, 2006). Se realizó la caracterización sobre el material de la variedad DC. Luego de comprobar el supuesto de normalidad en los datos para cada variable (KS: P>0,05), se realizó un ANOVA con dos factores (Tratamiento y Tiempo) sobre la hipótesis nula de no encontrar diferencias significativas entre los tratamientos, los tiempos, ni ninguna interacción entre dos factores para cada una de las variables analizadas (longitud medida en centímetros, peso medido en gramos, número de hojas y número de ramas) (Tabla 3).
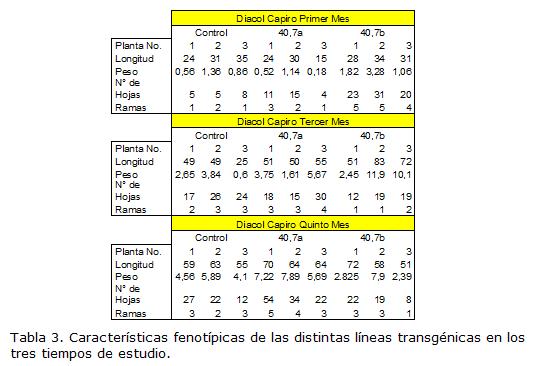
El análisis de los resultados indica que no existen diferencias significativas (P>0,05) entre las líneas transgénicas y el control para ninguna de las características fenotípicas analizadas. Con base en este resultado se postula que la inserción del gen Cry1Ac en el genoma de las líneas transgénicas examinadas no afectó las características fenotípicas analizadas. Esto se contrasta con el reporte de Senior, 1995, en Antirrhinus majus, donde observó anomalías en el desarrollo de las plantas transformadas presentándose enanismo, estimulación radical y en el número de flores. También con lo encontrado por Cattaneo et al., 2006, quienes reportaron mayor producción en el algodón genéticamente modificado.
Cuando se analizó el comportamiento de las variables con respecto al tiempo, se encontraron diferencias estadísticamente significativas en la longitud, el peso y el número de hojas de las plantas a través de los meses evaluados (P=0,000, P=0,002 y P=0,026, respectivamente). Estas diferencias fueron las esperadas en virtud a cambios estructurales y morfológicos que son el resultado del proceso de crecimiento vegetal, que a su vez, no se vio afectado por la inserción del gen Cry1Ac, dado que no se evidenció una interacción entre los factores tenidos en cuenta en el análisis. Para la variable número de ramas, no se encontraron diferencias significativas en el tiempo (P=0,328). Lo que se explica por que el número de ramas fue el mismo desde el inicio de las mediciones en el primer mes, hasta la última medición, en el quinto mes.
VERIFICACIÓN DE LA TRANSCRIPCIÓN DE CRY1AC
Se evidenció transcripción del gen Cry1Ac en las dos líneas transgénicas DC al observarse en los geles de agarosa los resultados del ensayo de RT-PCR, en los cuales hay evidencia del amplicón del gen de actina (266pb) para todas las muestras y únicamente hay evidencia de amplicones del gen Cry1Ac para las líneas transgénicas (Fig. 2). La evidencia de la transcripción del transgén en los distintos tejidos analizados, indica la inserción completa del casete de expresión en ambas líneas, dado que es necesaria la presencia tanto de la región promotora como de la región terminadora para que se efectúe correctamente la transcripción. Se asume que este patrón de expresión es debido al promotor constitutivo CaMV35S, que en las plantas transformadas determina la expresión en todos los tejidos la mayor parte del tiempo. Estos resultados concuerdan con el reporte de Greenplate, 1997, cuando caracterizó las concentraciones de proteína Cry1Ac en polen y en pétalos de algodón Bollgard™, y con el reporte de Yang y Christou, 1990, cuando observaron expresión GUS en raíces, tallos y hojas de plantas transgénicas de soya que tenían el mismo promotor.
CARACTERIZACIÓN DE LA EXPRESIÓN DE LA PROTEÍNA
En el ensayo de ELISA se siguió la recomendación del fabricante de la prueba y se observaron patrones de coloración que evidencian la actividad de la proteína de interés, que se corroboraron con los datos obtenidos por el lector de placas de ELISA para las absorbancias a 450 nm. A partir de estos datos se realizó la estimación de las concentraciones para cada tejido (datos no incluidos). Con las lecturas de absorbancia obtenidas se realizó una cuantificación de la proteína efectuando estimaciones directas por medio de una regresión lineal con las absorbancias obtenidas de unas diluciones de concentración (entre 0,3 µg- 3 µg) del control positivo contenido en el kit. Los rangos de concentraciones estimadas por tejido para las líneas transgénicas se encuentran relacionadas en la tabla 4. Se observa que las mayores mediciones fueron de 51,16 µg/g y 49,21 µg/g en hojas para las líneas 40,7a y 40,7b respectivamente, en contraste con los 8 µg/g encontrados en tubérculos por Valderrama et al., 2007. Tales diferencias son comparables con lo encontrado por Meiyalaghan et al., 2006, quienes cuantificaron proteína de líneas transgénicas de papa que contenían el transgén Cry1Ca5 y encontraron como valores máximos de 1,02 µg/g y 8,42 µg/g para tubérculo y hoja respectivamente. Esta diferencia de concentraciones entre los tejidos analizados en este estudio y el tubérculo puede ser el resultado de que en tubérculos, la extracción proteica y la reacción de la prueba de ELISA se vean afectadas por la alta cantidad de almidón que almacena éste órgano.
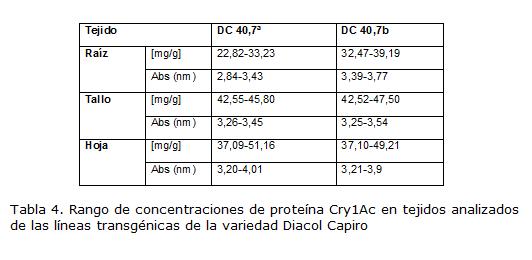
Las altas concentraciones encontradas para estas líneas, pueden ser el resultado del tipo de promotor utilizado en el casete de expresión (2x35S), esto si se compara con el los resultados encontrados en el estudio, anteriormente mencionado de papa que contenía el transgén Cry1Ca5 cuyo promotor es un 35S sencillo (Meiyalaghan, et al. 2006). Cañedo et al.,1999, cuando compararon en líneas transgénicas de papa Cry1Ab la expresión del promotor doble y el promotor sencillo 35S, encontraron concentraciones máximas de 10,87 µg/g y 6,26 µg/g para los tejidos de tubérculo, respectivamente.
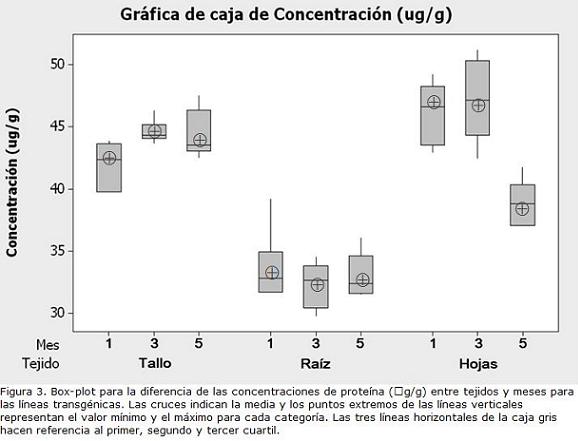
Los datos de concentración fueron analizados estadísticamente mediante ANOVA y se encontró un R2 del 97%, concluyéndose de este análisis que existen diferencias significativas dentro de los tratamientos (control y líneas), dentro de los tejidos y en los meses explicados en virtud a los tejidos analizados, además, existe una interacción estadísti-camente significativa entre tejido y líneas (P<0,05 para todos los casos). Se corroboró altas concentraciones en las líneas transgénicas con respecto al efecto basal encontrado en el control, pero entre ellas no se evidenciaron diferencias. Si se hace un análisis para las dos líneas transgénicas (Fig. 3) con respecto a los meses evaluados, no existe un patrón claro en la fluctuación de la proteína a través del tiempo. En cuanto a los tejidos, aparentemente las hojas presentan la mayor concentración de proteína Cry1Ac el primer y el tercer mes, pero desciende en el mes cinco. Sin embargo, en los otros tejidos, la concentración permanece sin variaciones significativas en el tiempo. Este resultado puede relacionarse con las etapas de desarrollo y partición de asimilados de la planta de papa descritas por Aldabe y Dogliotti, 1996, quienes reportaron que durante las primeras dos etapas de desarrollo de la planta (hasta el día 80 del cultivo) los fotoasimilados se direccionan principalmente hacia las hojas aumentando el metabolismo en este tejido, debido al aumento en la biomasa, en la tercera etapa hacia el día 110 la repartición de los fotoasimilados se direccionan hacia los tubérculos.
Para disminuir los potenciales riesgos de bioseguridad, el ideal sería obtener líneas transgénicas de papa que expresen la proteína de forma tejido específica. Así, la transformación de plantas para que expresen el transgen únicamente en tubérculos empleando promotores específicos, sería una metodología eficiente para evitar efectos negativos potenciales en especies no blanco que entren en contacto con tejidos florales (p. ej. Polinizadores) o foliares (p. ej. Fitófagos). Sin embargo, esto implica una inversión adicional de tiempo y dinero en la realización de estudios previos para identificar y analizar promotores específicos de tubérculo.
Por otro lado, además de los insectos no blanco se haría necesario realizar ensayos con insectos plaga que no fueran lepidópteros, ya que se han realizado estudios donde la tecnología Cry1Ac en algodón ha provocado mortalidad del díptero Lycoriella pleuroti de acuerdo con la concentración obtenida en el cultivo (Xu et al., 2006). De esta manera las concentraciones que presentan las líneas de papa examinadas podrían tener efecto sobre fitófagos plaga tales como Epitrix cucumeris, Lyriomyza quedrata, Copitarsia consueta, Frankliniella tuberosi,Thripspalmi, o la mosca blanca entre otros, o Phthorimaeaoperculella. En esta última especie, Cañedo etal., 1999, reportan mortalidad hasta del 100% en larvas después de 10 días de infestación, cuando fueron alimentadas en hojas de líneas transgénicas que contenían la proteína Cry1Ab a una concentración máxima de 10,87 µg/g.
ANÁLISIS DE RIESGO BIOLÓGICO POTENCIAL
Con respecto a la tecnología transgénica con genes Cry se ha escrito ampliamente, se ha reportado que el polen del maíz Bt no tiene efectos sobre Danaus plexippus (Sears et al., 2001), que el algodón Bt no tiene impacto sobre poblaciones de artrópodos no blanco (Head et al., 2005), que la Canola Bt no tiene efecto en el parasitoide del lepidóptero Plutella xylostella (Schuler, 2004) entre otros reportes. En estos estudios no se presenta la concentración expresada en los tejidos de la planta para la proteína. Para algodón Bollgard™ Adamczyk y Sumerford, 2001 reportan niveles de expresión 2ng/g, mientras que Biao et al., 2009, reportan datos de hasta 24,7 µg/g. Estos resultados junto con lo que se ha descrito anteriormente para el caso de la papa, muestran la alta variación en la expresión de la proteína transgénica, incluso dentro de la misma especie. Los niveles de expresión varían con el ambiente, Chen et al., 2005, reportaron que elevadas temperaturas degradan la proteína soluble presente en las hojas de algodón Bt, mientras que Coviella et al., 2002, reportaron que la producción de proteína CRY disminuye cuando se presentan concentraciones elevadas de CO2 y N. Los niveles de expresión son altamente sensibles y están regulados por diversos factores ambientales y factores inherentes a la variedad transformada, lo que demuestra que cada evento de transformación es caso específico. Para el proceso de liberación comercial de las líneas transgénicas examinadas, debe considerarse los niveles de proteína Cry1Ac reportados en este trabajo, como base de los estudios de bioseguridad pertinentes, en relación con los efectos potenciales sobre especies de insectos no blanco.
AGRADECIMIENTOS
A la Dirección de Investigación Sede Bogotá (DIB), Asohofrucol y CEVIPAPA, por el apoyo económico brindado a este proyecto. A Unidad de Biotecnología Vegetal del convenio UNSM-CIB por el material vegetal entregado para este estudio, especialmente al profesor Rafael Arango y al investigador Diego Villanueva. Al Laboratorio de Biología Molecular del Departamento de Biología y al Instituto de Genética de la UNCB, por las instalaciones para el desarrollo de esta investigación. A los miembros del grupo de Ingeniería Genética de Plantas de la UNCB, por sus aportes.
BIBLIOGRAFÍA
ADAMCZYK J, SUMERFORD D. Potential factors impacting season-long expression of CrylAc in 13 commercial varieties of Bollgard of cotton. J Insect Sci. 2001;1:1-6.
[ Links ]ALDABE L, DOGLIOTTI S. Bases fisiológicas del crecimiento y desarrollo del cultivo de papa (Solanum tuberosum L). Uruguay: Universidad de la República, Montevideo, Uruguay; 1996.
[ Links ]AKHURST R, JAMES W, BIRD L, BEARD C. Resistance to the Cry1Ac Endotoxin of Bacillus thuringiensis in the Cotton Bollworm, Helicoverpa armigera (Lepidoptera: Noctuidae). J Econ Entomol. 2003;96(4):1290-1299.
[ Links ]BENÍTEZ A. Avances Recientes en Biotecnología Vegetal e Ingeniería Genética de Plantas. Barcelona, España: Editorial Reverté; 2005.
[ Links ]BETZ F, HAMMOND B, FUCHS R. Safety and Advantages of Bacillus thuringiensis-Protected Plants to Control Insect Pests. Regul Toxicol Pharmacol. 2000;32:156-173.
[ Links ]BIAO L, CHANG S, XUE K, ZHOU K, LI X, etal. The oral toxicity of the transgenic Bt+ CpTI cotton pollen to honey bees (Apismellifera). Ecotoxicol Environ Saf. 2009; 72 (4): 1163- 1169.
[ Links ]BYRNE P, WARD S, HARRINGTON J. Transgenic crops: An introduction and resource Guide. History of plant breeding. Colorado State University; 2004. Disponible en http://cls.casa.colostate.edu/transgeniccrops/history.html .
[ Links ]CAÑEDO V, BENAVIDES J, GOLMIRZAIE A, CISNEROS F, GHISLAIN M, LAGNAOUI A. Assessing Bt-Transformed Potatoes for Potato Tuber Moth, Phthorimaea operculella (Zeller), Management. Impact on changing a world. Program report. International Potato Center. Lima, Perú; 1999.
[ Links ]CATTANEO M, YAFUSO C, SCHMIDT C, HUANG C, RAHMAN M, et al. Farm-scale evaluation of the impacts of transgenic cotton on biodiversity, pesticide use, and yield. Proc Natl Acad Sci USA. 2006;103(20):7571-7576.
[ Links ]CEVIPAPA. Consolidado de preguntas frecuentes; 2008. Disponible en: http://www.cevipapa.org.co/faq.php. [ Links ]
CHAPARRO A, SINISTERRA X, QUINTERO O, SANTOS J. Transformación de papa criolla variedad yema de huevo con un gen que codifica para un inhibidor de proteasas derivado del pomelo. Memorias Tecia solanivora. II Taller Nacional Cevipapa y Consejo Nacional de la papa. Colombia; 2003.
[ Links ]CHEN D, YE G, YANG C, CHEN Y, WU Y. The effect of high temperature on the insecticidal properties of Bt Cotton. Environ Exper Bot. 2005;53:333-342.
[ Links ]CIP (CENTRO INTERNACIONAL DE LA PAPA). Control biológico de La polilla de la papa con Baculovirus phtorimaea. Lima: Boletín de Capacitación CIP-2; 2000.
[ Links ]COVIELLA C, STIPANOVIC R, TRUMBLE J. Plant allocation to defensive compounds: interactions between elevated CO2 and nitrogen in transgenic cotton plants. J Exp Bot. 2002;53:323-33.
[ Links ]DELLAPORTA S, WOOD J, HICKS J. A plant DNA minipreparation: Version II. Plant Mol Biol Report. 1983;1:19-21.
[ Links ]FERNÁNDEZ F. Vegetales transgénicos: mitos y realidades desde una perspectiva técnica. Rev Fitotec Mex. 2006;29(2):95-102.
[ Links ]FISHER R, ROSNER L. Toxicology of the microbial insecticide, Thuricide. Agric Food Chem. 1959;7:686-688.
[ Links ]GIRI S, GIRI A, SHARMA G, PRASAD S. Induction of sister chromatid exchanges by cypermethrin and carbosulfan in bone marrow cells of mice in vivo. Mutagenesis. 2003;18:53-58.
[ Links ]GREENPLATE J. Response to reports of early damage in 1996 commercial Bt transgenic cotton (Bollgard™) plantings. Soc Invertebr Pathol Newslett. 1997;29:15-18.
[ Links ]GROOT A, DICKE M. Insect-resistant transgenic plants in a multi-trophic context. Plant J. 2002;31:387-406.
[ Links ]HANLEY A, HUANG Z, PETT W. Effects of dietary transgenic Bt corn pollen on larvae of Apis mellifera and Galleria mellonella. J Apic Res. 2003;42:77-81.
[ Links ]HEAD G, MOAR W, EUBANKS M, FREEMAN B, RUBERSON J, et al. Multiyear, large-scale comparison of arthropod populations on commercially managed Bt and non-Bt cotton fields. Environ Entomol. 2005;34:1257-1266.
[ Links ]HURTADO C, GUTIERREZ M. Enfoque del paciente con intoxicación aguda por plaguicidas organofosforados. Rev Fac Med. 2005;53(4):244-258.
[ Links ]INVITROGEN®. TRIzol® Reagent; 2008. Disponible en: http://www.invitrogen.com/contet/sfs/manuals/15596018%20pps%20Trizol%20Reagent%20061207.pdf. Consultada en septiembre de 2009.
[ Links ]JAMES C. Situación mundial de la comercialización de cultivos GM/transgénicos en 2008. Resumen ejecutivo. Brief 39. ISAAA International Service For The Acquisition Of Agri-Biotech Applications; 2008.
[ Links ]LARSEN K. Molecular cloning and characterization of a cDNA encoding endonuclease from Potato (Solanumtuberosum). J Plant Physiol. 2005;162(11):1263-1269.
[ Links ]LOPEZ AVILA A. Papas colombianas (Insectos plaga del cultivo de la papa en Colombia y su manejo). Revista al campo andino. 2000;3(1-2):152-159.
[ Links ]MEIYALAGHAN S, JACOBS J, BUTLER R, WRATTEN S, CONNER A. Transgenic Potato Lines Expressing cry1Ba1 or cry1Ca5 Genes are Resistant to Potato Tuber Moth. Potato research. 2006;49(3):203-216.
[ Links ]MENTABERRY A. Resistencia a insectos y nematodos. Curso de Agrobiotecno-logía. Buenos Aires: Depto. Fisiología, Biología Molecular y Celular. Universidad de Buenos Aires; Buenos Aires, Argentina; 2007.
[ Links ]MURASHIGE T, SKOOG F. A Revised Medium for Rapid Growth and Bio Assays with Tobacco Tissue Cultures. Physiol Plant. 1962;15:473-497.
[ Links ]NAYAK P, BASU D, DAS S, BASU A, GHOSH D, et al. Transgenic elite indica rice plants expressing CryIAc -endotoxin of Bacillus thuringiensis are resistant against yellow stem borer (Scirpophaga incertulas). Proc Natl Acad Sci. 1997;94:2111-2116.
[ Links ]SCHNEPF E, CRICKMORE N, VAN RIE J, LERECLUS D, BAUM J, et al. Bacillus thuringiensis and its pesticidal crystal proteins. Microbiol Mol Biol Ver. 1998; 62 (3): 775-806.
[ Links ]SCHULER T, DENHOLM I, CLARK S, STEWART C, POPPY G. Effects of Bt plants on the development and survival of the parasitoid Cotesia plutellae (Hymenoptera: Braconidae) in susceptible and Bt-resistant larvae of the diamondback moth, Plutella xylostella (Lepidoptera: Plutellidae). J Insect Physiol. 2004;50:435-443.
[ Links ]SEARS M, HELLMICH R, STANLEY-HORN D, OBERHAUSER K, PLEASANTS J, et al. Impact of Bt corn pollen on monarch butterfly populations: A risk assessment. Proc Natl Acad Sci USA. 2001;98(21):11937-11942.
[ Links ]SENIOR I, HOLFORD P, COOLEY R, NEWBURY H. Transformation of Antirrhinum majus using Agrobacterium rhizogenes. J Exp Bot. 1995;46:1233-1239.
[ Links ]SMITH J. Biotecnología. Zaragoza: Editorial Acribia S.A.; 2004.
[ Links ]SMITH D, MAXWELL P, DE BOER S. Method for the detection of syntheticcry3A in transgenic potato. J Agric Food Chem. 2004;52:809-815.
[ Links ]VALDERRAMA A, VELÁSQUEZ N, RODRIGUEZ E, ZAPATA A, ABBAS M, et al. Resistance to Tecia solanivora (Lepidoptera: Gelechiidae) in three transgenic andean varieties of potato expressing Bacillus thuringiensis Cry1Ac protein. J Econ Entomol. 2007; 100(1):172-179.
[ Links ]WENG H, PAN A, YANG L, ZHANG C, LIU Z, ZHANG D. Estimating number of transgene copies in transgenic rapeseed by real-time PCR assay with HMG I/Y as an endogenous reference gene. Plant Mol Biol Rep. 2004;22:289-300.
[ Links ]XU B, WANG Y. Effects of Cry1Ac and Secondary Metabolites in Bt Transgenic Cottonseed on Lycoriella pleuroti Yang et Zhang (Diptera: Sciaridae). Environ Entomol. 2006;35(3):807-810.
[ Links ]YANG N, CHRISTOU P. Cell type specific expression of a CaMV 35S-GUS gene in transgenic soybean plants. Dev Genet. 1990;11(4):289-293.
[ Links ]













