Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.16 no.1 Bogotá Jan./Apr. 2011
tRNASer (UCN) MITOCONDRIAL DE Lutzomyia hartmanni PREDICCIÓN DE LA ESTRUCTURA SECUNDARIA DEL tRNASer (UCN) MITOCONDRIAL DEL FLEBOTOMÍNEO Lutzomyia hartmanni (DIPTERA: PSYCHODIDAE)
Prediction of the Secondary Structure of the Mitochondrial tRNASer (UCN) of Lutzomyia hartmanni (Diptera: Psychodidae)
ALVEIRO PÉREZ-DORIA1, Biólogo; EDUAR E. BEJARANO1, M.Sc. 1 Grupo de Investigaciones Biomédicas, Universidad de Sucre. Carrera 14 # 16B-32. A.A. 406, Sincelejo, Sucre, Colombia. eduarelias@yahoo.com
Presentado 25 de junio de 2009, aceptado 1 de febrero de 2011, correcciones 14 de febrero de 2011.
RESUMEN
Lutzomyia (Helcocyrtomyia) hartmanni es un flebotomíneo implicado en la transmisión de Leishmania (Viannia) colombiensis, uno de los agentes etiológicos de leishmaniasis cutánea en Colombia. El objetivo de este trabajo fue explorar la utilidad potencial del RNA de transferencia mitocondrial para Serina (UCN) (tRNASer), en la discriminación taxonómica de L. hartmanni. El DNA mitocondrial se extrajo, amplificó y secuenció a partir de material entomológico recolectado en Envigado, Antioquia, Colombia. El gen tRNASer de L. hartmanni mostró una longitud de 68 pares de bases, con un contenido AT del 80,9%. Éste se diferencia de los demás tRNASer de Lutzomyia conocidos a la fecha tanto por sustituciones en la secuencia primaria de nucleótidos como por los cambios que éstas generan en la estructura secundaria. El número de apareamientos intracatenarios fue siete en el brazo aceptor del aminoácido, tres en el brazo dihidrouridina (DHU), cinco en el brazo del anticodón y cinco en el brazo ribotimidina-pseudouridina-citosina (T C). El tamaño de las lupas DHU, anticodón, variable y T C correspondió a cinco, siete, cuatro y ocho nucleótidos, respectivamente. La ausencia notoria de pares de bases no-Watson-Crick en los cuatro brazos del tRNASer de L. hartmanni, la distingue de otras especies de Lutzomyia.
Palabras clave: flebotomíneos, Lutzomyia hartmanni, ADN mitocondrial, leishmaniasis, Colombia.
ABSTRACT
Lutzomyia (Helcocyrtomyia) hartmanni is a sand fly that has been implicated in the transmission of Leishmania (Viannia) colombiensis, an etiologic agent of cutaneous leishmaniasis in Colombia. The objective of this work was to explore the potential usefulness of the mitochondrial serine transfer RNA (UCN) (tRNASer) in the taxonomic determination of L. hartmanni. Mitochondrial DNA was extracted, amplified and sequenced from entomological material collected in Envigado, Antioquia, Colombia. The tRNASer gene length was 68 nucleotide pairs, with an average adenine-thymine content of 80,9%. The studied tRNASer differs from other sand fly tRNASer known to date, on the basis of its primary and secondary structure. The observed number of intrachain base pairing was 7 in the acceptor arm, 3 in the dihydrouridine (DHU) arm, 5 in the anticodon arm, and 5 in the ribothymidine-pseudouridine-cytosine (T C) arm. The size of the DHU, anticodon, variable and T C loops was estimated to be 5, 7, 4, and 8 nucleotides, respectively. The notorious absence of non-Watson-Crick base pairs in the four arms of the tRNASer distinguishes that of L. hartmanni from others Lutzomyia spp.
Key words: sand flies, Lutzomyia hartmanni, mitochondrial DNA, leishmaniasis, Colombia.
INTRODUCCIÓN
Bajo el término de leishmaniasis se designa a una serie de enfermedades que se caracterizan por afectar, principalmente piel, mucosas o vísceras de algunos mamíferos, teniendo en común que se producen por protozoos del género Leishmania Ross, 1903, y se transmiten por insectos hematófagos de la subfamilia Phlebotominae Rondani, 1840 (Lainson y Shaw, 2005). Lutzomyia hartmanni (Fairchild y Hertig, 1957) es un díptero implicado en la transmisión de Le. (Viannia) colombiensis, parásito reconocido como agente etiológico de leishmaniasis cutánea (Kreutzer et al., 1991). Este insecto se encuentra presente en Costa Rica, Panamá, Colombia, Ecuador y Perú (Young y Duncan, 1994).
L. hartmanni es ubicada, taxonómicamente, en el subgénero Helcocyrtomyia Barretto, 1962, por presentar cinco espinas en el estilo, cuatro dientes horizontales en el cibario, ascoides simples en las antenas, tufo de setas en la base de la coxita, y espermatecas anuladas y delgadas, con los ductos espermáticos individuales más largos que el ducto común. De acuerdo con Galati y Cáceres, 1994, este subgénero está conformado por tres series de especies, incluidas las series sanguinaria Barretto, 1962, osornoi Galati y Cáceres, 1994, y peruensis Barreto, 1962. Aunque los miembros de este taxón pueden ser discriminados morfológicamente, algunas especies presentan isomorfismo en uno de los sexos (Ogusuku et al., 1997; Galati, 2003), lo que obstaculiza su determinación taxonómica. Los genes mitocondriales son considerados como una herramienta que complementa a la morfología en el estudio taxonómico de los flebotomíneos (Bejarano, 2001). No obstante, a la fecha sólo se han analizado segmentos parciales de un número limitado de genes mitocondriales en Lutzomyia França, 1924 (Ready et al., 1997; Suguri et al., 1997; Ishikawa et al., 1999; Uribe et al., 2001; Arrivillaga et al., 2002; Testa et al., 2002; Hodgkinson et al., 2003; Torgerson et al., 2003; Beati et al., 2004; Vivero et al., 2007; Pérez-Doria et al., 2008a; Pérez-Doria et al., 2008b; Bejarano et al., 2009) y falta aún explorar la utilidad taxonómica del resto del genoma mitocondrial, que incluye los genes que codifican para los RNA de transferencia (tRNA) necesarios para la síntesis de proteínas en la mitocondria.
Los genes mitocondriales que codifican para los tRNAs se caracterizan por exhibir una tasa mayor de mutación que los tRNAs del genoma nuclear, los cuales son altamente conservados en insectos (Simon et al., 1994). Sin embargo, los genes que codifican para tRNAs mitocondriales evolucionan más lentamente que los genes mitocondriales codificadores de proteínas, razón por la cual podrían ser útiles como marcadores moleculares a distintos niveles taxonómicos.
En insectos, los tRNAs se sintetizan inicialmente como moléculas monocatenarias de RNA, las cuales posteriormente experimentan apareamientos intracatenarios que dan origen a la estructura secundaria de doble cadena con forma de hoja de trébol (Alberts et al., 2002). Estos genes están sometidos a restricciones mutacionales que operan especialmente en sus dominios funcionales, por consiguiente, la utilidad de los tRNAs estaría no sólo en el polimorfismo de la secuencia primaria de nucleótidos, sino también en los cambios en la estructura secundaria de la molécula. Tales aspectos son abordados en el presente artículo, en el cual se describe por primera vez la estructura primaria y secundaria de uno de los tRNA mitocondriales para serina de L. hartmanni.
MATERIALES Y MÉTODOS
RECOLECCIÓN E IDENTIFICACIÓN DE FLEBOTOMÍNEOS
El material entomológico de L. hartmanni se recolectó con un aspirador bucal sobre cebo humano protegido en un bosque secundario de la vereda el Vallano (06° 08 N, 75° 34 W), municipio de Envigado, departamento de Antioquia. La zona de vida del sitio de recolección corresponde a bosque húmedo tropical, con una altura de 1909 msnm, una temperatura promedio anual de 16,8 °C y una humedad relativa del 85,8%. La determinación taxonómica de especie se llevó a cabo usando las claves de Young y Duncan, 1994 y Galati, 2003. El tórax, las patas, un ala y parte del abdomen de cada flebotomíneo, se utilizó para la extracción del material genético.
EXTRACCIÓN DEL DNA
La purificación del DNA se realizó de acuerdo al protocolo de Collins et al., 1987, con los ajustes descritos a continuación. Cada espécimen fue macerado en 60 µL del tampón de lisis (0,08 M NaCl, 0,16 M sacarosa, 0,06 M EDTA, 0,5% SDS, 0,1 M tris-HCL a pH de 7,5), e incubado a 65 °C durante 30 min. Posteriormente, se adicionaron 14 µL de acetato de potasio 8 M y se incubó sobre hielo por 30 min. Después se centrifugó a 12,000 x g durante 10 min y el sobrenadante se transfirió a un vial nuevo. El DNA se precipitó durante 18 h con 200 µL de etanol absoluto a -20°C y se centrifugó a 12,000 x g por 20 min. Seguidamente, se decantó el sobrenadante y se lavó el DNA dos veces con 200 µL de etanol al 70% y con 200 µL de etanol absoluto. Por último, se evaporó el etanol a temperatura ambiente y se resuspendió el DNA en 30 µL de agua estéril.
AMPLIFICACIÓN DEL GEN MITOCONDRIAL TRNASER(UCN)
El gen que codifica para el tRNA mitocondrial para serina (tRNASer) que reconoce al codón UCN, se amplificó con la técnica de PCR, usando el cebador delantero 5 -CA(T/C)ATTCAACC(A/T)GAATGATA-3 y el cebador reverso 5 -GGTA(C/T)(a/t) ttgcctcga(t/A)ttcg(t/a)TATGA-3 (Ready et al., 1997), los cuales son complementarios al extremo 3 del gen citocromo b y al extremo 3 del gen NAD1, respectivamente, que a su vez son codificados por la cadena pesada (L) y liviana (H) del genoma mitocondrial. La mezcla de PCR tuvo un volumen final de 50 µL, e incluyó buffer para PCR 1x (Promega Corporation, Madison, WI), 1,5 mM MgCl2, 0,2 mM de la mezcla de desoxirribonucleótidos (Promega), 0,3 mM de cada cebador, 1,5 U de Taq DNA polimerasa (Promega) y 6 mL de la solución de DNA (~10 ng/µL).
SECUENCIACIÓN DE NUCLEÓTIDOS Y ANÁLISIS MOLECULAR
La secuencia de nucleótidos del gen tRNASer se determinó para cada hebra de la doble hélice de DNA en un secuenciador automático de electrofóresis capilar ABI Prismâ 3730xl (Applied Biosystems), que emplea didesoxirribonucleótidos marcados con distintos fluorocromos (Big Dye Terminator Cycle Sequencing Ready Reaction Kit, Applied Biosystems, Foster City, CA).
El programa MEGA versión 3.1 (Kumar et al., 2004) se empleó para la revisión de los electroforegramas, el ensamblaje de la secuencia consenso de nucleótidos y la determinación de la composición nucleotídica del gen analizado. La estructura secundaria del tRNASer de L. hartmanni se infirió con el programa tRNAscan-SE 1.21 (Lowe y Eddy, 1997) y se editó de modo manual. Finalmente, la secuencia obtenida en este estudio se registró en la base de datos moleculares Genbank, con el número de acceso EF033635.
RESULTADOS Y DISCUSIÓN
El genoma mitocondrial de los insectos contiene 22 genes que codifican para igual número de tRNAs, uno por cada aminoácido, con la excepción de leucina y serina, los cuales poseen individualmente dos tipos de moléculas (Hanada et al., 2000). Los tRNASer que emplea la maquinaria traduccional de la mitocondria se ubican en el transcrito de la cadena H y L del DNA y reconocen los codones AGY y UCN, respectivamente, el último de los cuales se caracterizó en la presente investigación.
El gen tRNASer obtenido de L. hartmanni mostró una longitud de 68 pb, con una composición nucleotídica neta de 30 adeninas, 25 timinas, siete guaninas y seis citosinas, en la que se aprecia un alto contenido de adenina y timina propio de los genes mitocondriales (Beard et al., 1993). Este gen está flanqueado en su extremo 5 por el gen citocromo b y en el extremo 3 por el espacidor intergénico dos (IG2). Llama la atención que los dos primeros nucleótidos del extremo 5 del gen tRNASer, representados por adenina y timina, constituyeron a su vez las dos últimas bases del codón de parada (TAG) del gen citocromo b, mientras que los nucleótidos del extremo 3 fueron de codificación exclusiva. Esto implica la existencia de un traslape entre el gen citocromo b y tRNASer, que pone de manifiesto el perfil económico del genoma mitocondrial, y la inexistencia del espaciador intergénico uno presente en otras especies de Lutzomyia (Vivero et al., 2007; Pérez-Doria et al., 2008a; Pérez-Doria et al., 2008b).
La estructura secundaria inferida del tRNASer de L. hartmanni está conformada por el brazo aceptor del aminoácido, el brazo y la lupa dihidrouridina (DHU), el brazo y la lupa del anticodón, la lupa variable, y el brazo y la lupa ribotimidina-pseudouridinacitosina (T C; Fig. 1). En el brazo aceptor del aminoácido se observaron siete apareamientos, seis de los cuales correspondieron al dinucleótido A-U (adenina y uracilo) y uno a G-C (gua-nina y citosina). No obstante, durante su maduración in vivo, las dos últimas bases U-U del extremo 3 del tRNASer son reemplazadas por el trinucleótido CCA (citosina, citosina y adenina), conformando el sitio de unión del aminoácido respectivo.
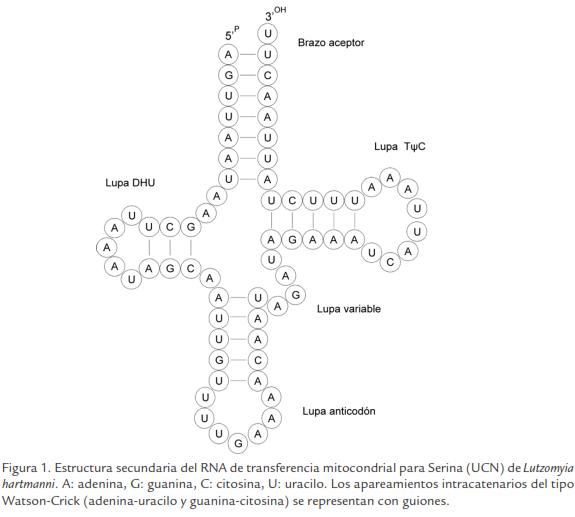
El número de apareamientos presentes en el brazo DHU fue 3, incluidos dos pares GC y un solo par A-U, mientras que tanto el brazo del anticodón como el brazo T C presentaron cinco pares de bases, cuatro de ellos constituidos por el dinucleótido A-U y uno por G-C. De otro lado, el tamaño de las lupas DHU, anticodón, variable y T C correspondió a 5, 7, 4 y 8 nucleótidos, respectivamente. Este flebotomíneo presentó además dos bases entre el brazo aceptor y el brazo DHU, y una base entre este último y el brazo del anticodón.
El tRNASer de L. hartmanni se distingue de los demás tRNASer de Lutzomyia conocidos a la fecha (Vivero et al., 2007; Pérez-Doria et al., 2008a; Pérez-Doria et al., 2008b) tanto por las sustituciones en la secuencia primaria de nucleótidos como por los cambios que éstas generan en la estructura secundaria. Primeramente, la secuencia CAUU (citosina, adenina, uracilo y uracilo) presente en la lupa T C es exclusiva de esta especie cuando se compara con L. cayennensis cayennensis (Floch y Abonnenc, 1941), L. columbiana (Ristorcelli y Van Ty, 1941), L. dubitans (Sherlock, 1962), L. evansi (Nunez-Tovar, 1924), L. gomezi (Nitzulescu, 1931), L. panamensis (Shannon, 1926), L. pia (Fairchild y Hertig, 1961), L. rangeliana (Ortiz, 1952), L. tihuiliensis Le Pont, Torrez-Espejo y Dujardin, 1997, y L. trinidadensis (Newstead, 1922; Vivero et al., 2007; Pérez-Doria et al., 2008a; Pérez-Doria et al., 2008b). Más aún, al alinearse con la región homóloga de estos taxones, la secuencia del gen tRNASer de L. hartmanni exhibe un evento indel (inserción-delección) entre el brazo y la lupa T C que no se observa en las demás especies (Fig. 2). Estos cambios indican la utilidad potencial de esta secuencia nucleotídica como marcador molecular.

Los pares de bases no-Watson-Crick determinan en gran parte la configuración terciaria de un RNA y contribuyen no sólo en las interacciones RNA-RNA sino también en los procesos de reconocimiento RNA-proteínas (Leontis et al., 2002; Chandrasekhar y Malathi, 2003). Sin embargo, al examinar la estructura de hoja de trébol del tRNASer de L. hartmanni se hace notoria la ausencia de pares de bases no-Watson-Crick en los cuatro brazos (Fig. 1), lo que la separa de las especies de Lutzomyia previamente mencionadas, las cuales exhiben un característico par de bases no-Watson-Crick U-U (uracilo y uracilo) en el extremo proximal del brazo del anticodón, con la excepción de L. rangeliana que presenta, al igual que L. hartmanni, el par de bases canónico U-A (uracilo y adenina) en dicha posición. No obstante, los tRNASer de las últimas dos especies se diferencian entre si por el tamaño de las lupas DHU y T C, que muestran, respectivamente, una extensión de cinco y ocho nucleótidos en L. hartmanni, y de seis y siete bases en L. rangeliana.
Es de resaltar que la secuencia de bases AGAU (adenina, guanina, adenina y uracilo) de la lupa variable del tRNASer de L. hartmanni es idéntica a la presente en las otras especies de Lutzomyia secuencidas (Vivero et al., 2007; Pérez-Doria et al., 2008a; Pérez-Doria et al., 2008b), con la excepción de L. rangeliana donde la lupa variable esta constituida por AUAU (adenina, uracilo, adenina y uracilo). En conclusión, la estructura primaria y secundaria del tRNASer de L. hartmanni difiere de la de otras especies de Lutzomyia, lo que indica su utilidad como marcador en taxonomía molecular, sin embargo, se requiere ampliar el estudio a otros taxones de la subfamilia Phlebotominae.
AGRADECIMIENTOS
Los autores agradecen la colaboración del Biólogo Boris Zuleta durante la recolección del material entomológico.
BIBLIOGRAFÍA
ALBERTS B, JOHNSON A, LEWIS J, RAFF M, ROBERTS K, WALTER P. Molecular biology of the cell. 4 ed. New York: Garland Science; 2002.
[ Links ]ARRIVILLAGA JC, NORRIS DE, FELICIANGELI MD, LANZARO GC. Phylogeography of the neotropical sand fly Lutzomyia longipalpis inferred from mitochondrial DNA sequences. Infect Genet Evol. 2002;2(2):83-95.
[ Links ]BEARD CB, HAMM DM, COLLINS FH. The mitochondrial genome of the mosquito Anopheles gambiae: DNA sequence, genome organization, and comparisons with mitochondrial sequences of other insects. Insect Mol Biol. 1993;2(2):103-124.
[ Links ]BEATI L, CÁCERES AG, LEE JA, MUNSTERMANN LE. Systematic relationships among Lutzomyia sand flies (Diptera: Psychodidae) of Peru and Colombia based on the analysis of 12S and 28S ribosomal DNA sequences. Int J Parasitol. 2004;34(2): 225-234.
[ Links ]BEJARANO EE, ROJAS W, URIBE S, VÉLEZ ID, PORTER CH. Genetic analysis of a recently detected urban population of Lutzomyia evansi (Diptera: Psychodidae) in Colombia. Rev Soc Entomol Argent. 2009;68(1-2):1-7.
[ Links ]BEJARANO EE. Nuevas herramientas para la clasificación taxonómica de los insectos vectores de leishmaniosis: utilidad de los genes mitocondriales. Biomedica 2001;21(2):182-191.
[ Links ]CHANDRASEKHAR K, MALATHI R. Non-Watson Crick base pairs might stabilize RNA structural motifs in ribozymes. A comparative study of group-I intron structures. J Biosci. 2003;28(5):547-555.
[ Links ]COLLINS FH, MENDEZ MA, RASMUSSEN MO, MEHAFFEY PC, BESANSKY NJ, FINNERTY V. A ribosomal RNA gene probe differentiates member species of the Anopheles gambiae complex. Am J Trop Med Hyg. 1987;37(1):37-41.
[ Links ]GALATI EAB, CÁCERES AG. Descrição de Lutzomyia pallidithorax, sp. n. e de Lutzomyia castanea, sp. n. do Peru e análise cladística das séries do subgênero Helcocyrtomyia Barretto (Diptera, Psychodidae). Rev Bras Entomol. 1994;38(2):471-488.
[ Links ]GALATI EAB. Morfologia, terminologia de adultos e identificação dos táxons da América. En: Rangel EF, Lainson R, editores. Flebotomíneos do Brasil. Rio do Janeiro: Editora Fiocruz; 2003. p. 53-175.
[ Links ]HANADA T, SUZUKI T, WATANABE K. Translation activity of mitochondrial tRNA with unusual secondary structure. Nucleic Acids Symp Ser. 2000;44:249-250.
[ Links ]HODGKINSON VH, BIRUNGI J, QUINTANA M, DEITZE R, MUNSTERMANN LE. Mitochondrial cytochrome b variation in populations of the visceral leishmaniasis vector Lutzomyia longipalpis across eastern Brazil. Am J Trop Med Hyg. 2003;69(4):386-392.
[ Links ]ISHIKAWA EA, READY PD, DE SOUZA AA, DAY JC, RANGEL EF, DAVIES CR, et al. A mitochondrial DNA phylogeny indicates close relationships between populations of Lutzomyia whitmani (Diptera: Psychodidae, Phlebotominae) from the rain-forest regions of Amazonia and northeast Brazil. Mem Inst Oswaldo Cruz. 1999;94(3):339-345.
[ Links ]KREUTZER RD, CORREDOR A, GRIMALDI G. Jr, GROGL M, ROWTON ED, YOUNG DG, et al. Characterization of Leishmania colombiensis sp. n (Kinetoplastida: Trypanosomatidae), a new parasite infecting humans, animals, and phlebotomine sand flies in Colombia and Panama. Am J Trop Med Hyg. 1991;44(6):662-675.
[ Links ]KUMAR S, TAMURA K, NEI M. MEGA 3: Integrated software for Molecular Evolutionary Genetics Analysis and sequence alignment. Brief Bioinform. 2004;5(2):150-163.
[ Links ]LAINSON R, SHAW JJ. New World leishmaniasis. En: Cox FEG, Wakelin D, Gillespie SH, Despommier D, editors. Parasitology, Topley - Wilson s Microbiology and Microbial Infections, 10th Edition. London: Hodder Arnold; 2005;(6):313-349.
[ Links ]LEONTIS NB, STOMBAUGH J, WESTHOF E. The non-Watson-Crick base pairs and their associated isostericity matrices. Nucleic Acids Res. 2002;30(16):3497-3531.
[ Links ]LOWE TM, EDDY SR. tRNAscan-SE: a program for improved detection of transfer RNA genes in genomic sequence. Nucleic Acids Res. 1997;25(5):955-964.
[ Links ]OGUSUKU E, CANALES J, PÉREZ E. Descripción de Lutzomyia gonzaloi n. sp., y de Lutzomyia monzonensis n. sp. (Diptera: Psychodidae: Phlebotorninae) y dos nuevos registros de Phlebotominae para el Perú. Rev Per Ent. 1997;40:71-78.
[ Links ]PÉREZ-DORIA A, BEJARANO EE, SIERRA D, VÉLEZ ID. Molecular evidence confirms the taxonomic separation of Lutzomyia tihuiliensis from Lutzomyia pia (Diptera: Psychodidae) and the usefulness of pleural pigmentation patterns in species identification. J Med Entomol. 2008a;45(4):653-659.
[ Links ]PÉREZ-DORIA A, BEJARANO EE, VÉLEZ ID. Descripción del ARN de transferencia mitocondrial para Serina (UCN) de Lutzomyia columbiana (Diptera, Psychodidae). Rev Bras Entomol. 2008b;52(4):591-594.
[ Links ]READY PD, DAY JC, DE SOUZA AA, RANGEL EF, DAVIES CR. Mitochondrial DNA characterization of populations of Lutzomyia whitmani (Diptera Psychodidae) incriminated in the peri-domestic and silvatic transmission of Leishmania species in Brazil. Bull Entom Res. 1997;87:187-195.
[ Links ]SIMON C, FRATI F, BECKENBACH A, CRESPI B, LIU H, FLOOK P. Evolution, weighting, and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers. Ann Entomol Soc Am. 1994;87(6):651-701.
[ Links ]SUGURI S, HARADA M, MARUNO M, HOSOKAWA A, SUD RA, GÓMEZ EAL, et al. A preliminary study on nucleotide sequence variations of Lutzomyia spp. in the cytochrome c oxidase subunit I gene. En: Hashiguchi Y, editor. Studies on New World leishmaniasis and its transmission, with particular reference to Ecuador. Kochi, Japan: Kyowa Printing Co. 1997;25-27.
[ Links ]TESTA JM, MONTOYA-LERMA J, CADENA H, OVIEDO M, READY PD. Molecular identification of vectors of Leishmania in Colombia: mitochondrial introgression in the Lutzomyia townsendi series. Acta Trop. 2002;84(3):205-218.
[ Links ]TORGERSON DG, LAMPO M, VELÁZQUEZ Y, WOO PTK. Genetic relationships among some species groups within the genus Lutzomyia (Diptera: Psychodidae). Am J Trop Med Hyg. 2003;69(5):484-493.
[ Links ]URIBE S, LEHMANN T, ROWTON ED, VÉLEZ ID, PORTER CH. Speciation and population structure in the morphospecies Lutzomyia longipalpis (Lutz - Neiva) as derived from the mitochondrial ND4 gene. Mol Phylogenet Evol. 2001;18(1):84-93.
[ Links ]VIVERO RJ, CONTRERAS-GUTIÉRREZ MA, BEJARANO EE. Análisis de la estructura primaria y secundaria del ARN de transferencia mitocondrial para Serina en siete especies de Lutzomyia. Biomedica. 2007;27(3):429-438.
[ Links ]YOUNG DG, DUNCAN MA. Guide to the identification and geographic distribution of Lutzomyia sand flies in Mexico, the West Indies, Central and South America (Diptera: Psychodidae). Mem Am Entomol Inst. 1994;54:1-881.
[ Links ]













