Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.16 no.3 Bogotá Sept./Dec. 2011
GENES Y EVOLUCIÓN EL DELGADO HILO QUE NOS CONECTA POR MILES DE MILLONES DE AÑOS
Genes and Evolution, the Thin Thread that Connects Us for Billions of Years
LUIS FERNANDO GARCÍA1, Ph. D. 1 Departamento de Biología. Facultad de Ciencias. Universidad Nacional de Colombia, Sede Bogotá. Bogotá-Colombia. lfgarciap@unal.edu.co
Presentado 24 de marzo de 2011, aceptado 28 de junio de 2011, correcciones 1 de julio de 2011.
RESUMEN
La escala temporal en nuestra cotidianidad refleja una pequeña fracción de la historia evolutiva de los organismos, incluidos los humanos. La edad del planeta se estima en 4.500 millones de años, donde ocurrieron profundos cambios en la posición de los continentes y en el clima del planeta. Sin embargo, los cambios más drásticos surgen solo en los últimos 500 millones de años. La genética ha sido una herramienta fundamental para inferir el cambio de los organismos a lo largo de millones de años, por ejemplo, mediante la estimación de tiempos de divergencia. Los seres vivos comparten el material genético como huellas moleculares para rastrear cambios en el pasado. Así, mediante el uso de secuencias de ADN y proteínas, los científicos estudian cómo los organismos han evolucionado, cuándo surgieron importantes novedades evolutivas, cómo interaccionan los genes, y determinan las relaciones evolutivas entre las formas del pasado y las contemporáneas. Las relaciones entre organismos son inferidas por simple similaridad (fenéticas), por compartir características únicas y mediante modelos matemáticos que describen como evolucionan las secuencias de ADN y las proteínas (filogenéticas). La genética también evidencia la dinámica de los genomas (duplicación, transposición, recombinación, inversiones), que resulta en ganancia y pérdida de genes, nuevas funciones, y transferencia horizontal de genes, mecanismos que incrementan la diversidad existente hoy. Aunque el ambiente es fundamental en la evolución de los organismos, es imprescindible reconocer que hay un hilo conductor muy fino y fuerte que nos conecta inevitablemente a esas primeras moléculas que se formaron en el pasado distante.
Palabras clave: evolución, genoma, genética, transferencia horizontal de genes, mutación.
ABSTRACT
The temporal scale of our daily life is a small fraction of the evolutionary history of organisms, including the humans. The planet age is estimated in 4.5 billion years, in which profound changes in the position of continents and the climate of the earth occurred. Nevertheless, most drastic changes arose just in the last 500 million years. The genetics has been a fundamental tool to infer the organismal change throughout billions, thousands and millions of years, for example through the estimation of divergence times. Living organisms share the genetic material as a molecular fingerprint to trace changes in the past. Thus, by using DNA and protein sequences, scientists may understand how the living organisms have evolved, when important evolutionary novelties arose, how genes interact, and determine the evolutionary relationships between past and contemporary forms. The relationships among organisms are inferred by simple similarity (phenetic relationships), by sharing unique features and throughout mathematical models that describe how DNA sequences or proteins evolve (phylogenetic relationships). Genetics also evidences the dynamics of genomes (duplication, transposition, recombination, inversions) that results in gene gain and loss, new functions, and horizontal gene transfer, mechanisms that increase today’s existing diversity. Although the environment is fundamental in the evolution of organisms, it is essential to recognize that there is a very thin and strong thread that connects us inevitably to those first molecules formed in the distant past.
Key words: Evolution, genome, genetics, horizontal gene transfer, mutation
INTRODUCCIÓN
Existen dos factores imprescindibles en el análisis, cuando se aborda el tema de genes y evolución: el primero involucra el tiempo que en la escala general se reduce en el mejor de los casos a milenios cuando por ejemplo se habla de la historia de la humanidad. En evolución se habla de escalas de tiempo mucho más largas que incluyen no solamente los milenios, sino los cientos de miles de años, los millones de años y aún los miles de millones de años. El segundo factor importante involucra la variación en dos sentidos: 1) El tiempo geológico (deriva continental y cambio climático), y 2) genético: en primer lugar el planeta tierra durante los últimos millones de años ha sufrido profundos cam-bios en la forma, tamaño y conectividad de nuestros continentes, por ejemplo aunque las posiciones de los polos y el ecuador no han cambiado, puesto que los continentes se han movido de manera muy dinámica, las regiones que son tropicales o polares hoy, no lo eran necesariamente en el pasado (Brown y Lomolino, 1998). Este dinamismo ha repercutido de manera importante en los patrones de distribución de las especies, lo que ha conllevado a distribuciones homogéneas, disyuntas o discontinuas (Wen, 1999; Sanmartín et al., 2001). Así, el movimiento de los continentes pudo separar las poblaciones de organismos que previamente estaban juntas; ello puede ser también atribuible a cambios en el nivel del mar, la retracción de hábitats por cambios climáticos e incluso puede tener una explicación reciente cuando ocurre por fragmentación de hábitat resultado de causas antropogénicas, como es el caso en muchas poblaciones de anfibios (Measey y Tolley, 2011).
El cambio climático es de alta importancia en evolución, pues se han modificado las condiciones de temperatura del planeta. Los ciclos de glaciación-interglaciación han ocurrido periódicamente durante los últimos millones de años (Berger y Lutre, 2002). Como respuesta, los organismos pueden optar por migrar hacia tierras más calidas modificando no solo su distribución, sino las interacciones interespecíficas. Cuando el clima cambia a condiciones más favorables, las especies pueden regresar a la distribución original de sus ancestros, modificando nuevamente sus patrones biogeográficos y su interacción con otras especies tales como comportamientos territoriales interespecíficos y competencia por los recursos, entre otros. Es posible también que ocurra incompatibilidad genómica cuando dos poblaciones de una misma especie divergen por aislamiento, formando dos nuevas especies, pero posteriormente se reencuentran y comparten un hábitat determinado (Lynch, 2002).
Otro elemento importante en la evolución corresponde al cambio interno del componente genético. Así como cambia el ambiente, el genoma también sufre modificaciones que incluyen procesos adaptativos, efectos selectivos y posiblemente patrones de especiación (de Aguiar et al., 2009). Aunque los genomas cambian de manera dinámica, mediante acumulación de mutaciones, duplicaciones génicas, inversiones, transposiciones, fusión y fisión de genes, una conclusión que se deriva de la secuenciación de genomas completos es que los ancestros de especies cercanas son más parecidos genéticamente que aquéllos más lejanos. Lo anterior resulta del origen monofilético de la vida (ancestría común) y de un proceso evolutivo mediado mayoritariamente por selección natural, como Darwin lo sugirió hace 150 años (Darwin, 1959). Así, el origen, continuidad y eventual extinción de especies animales, de plantas o microorganismos no solo depende de los genes. Es también el resultado de una profunda y compleja interacción entre organismos cuyos genes controlan maquinarias celulares y moleculares muy complejas para el correcto funcionamiento de un organismo y su influencia recíproca con el medio ambiente. Este artículo enfatiza en el tiempo (variación a escala geológica y cambio climático) y la variación (genética) como elementos fundamentales en el proceso evolutivo. Asimismo, destaca importantes cambios y tendencias a nivel genómico evidenciados gracias al desarrollo de nuevas tecnologías. El artículo corresponde a la conferencia presentada en la Cátedra José Celestino Mutis de la Universidad Nacional de Colombia y denominada -Todo lo que usted quiere saber de genética y nunca se atrevió a preguntar- que tuvo lugar en el año 2010 en el auditorio León de Greiff.
EVOLUCIÓN DESDE LA PRIMERA CÉLULA PROCARIOTA A LOS HUMANOS
Para dimensionar la escala evolutiva y la transformación orgánica ocurrida desde las primeras células hasta entidades más complejas y recientemente divergidas, es necesario entender la escala temporal. La edad de la tierra es aproximadamente 4.500 millones de años (MA) estimativo basado en datación por radiometría (Dalrymple, 2001); 1.000 MA después habría surgido la primera forma de vida (Schopfet al., 2002). Los científicos han dividido la escala evolutiva del tiempo en eras, periodos y épocas (Tabla. 1). En términos generales se puede definir un fósil como los restos o señales de la actividad de organismos pasados como esqueletos, conchas, caparazones e impresiones de plantas (Bentonet al., 2000). Los fósiles proveen el contexto temporal del devenir evolutivo de la biota (Benton et al., 2000), aunque dicho registro puede ser incompleto (Futuyma, 2009).
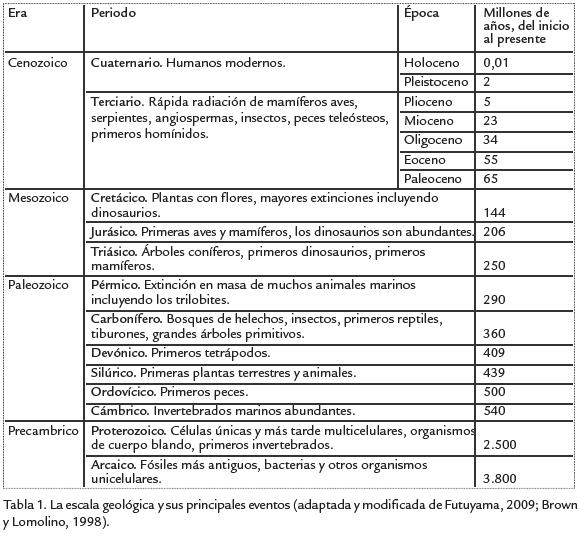
Las primeras células procariota (sin un núcleo celular diferenciado) surgen hace aproximadamente 3800 MA (Woese, 1998). Dentro de los grupos procariota se incluyen los dominios Archaea y Bacteria; el primero fue reconocido como domino taxonómico solo en la década de los 90 (Woese et al., 1990). Después de 1.000 millones de años surgieron las primeras células eucariota, que tiene su ADN en el interior del núcleo diferenciado (Zimmer, 2009). La formación de la membrana celular requirió la aparición de los fosfolípidos, que constituyen un filtro y un canal de comunicación que protege del medio ambiente circundante (Koch y Silver, 2005; Campbell, 2006). Al surgir las primeras células, evolucionan hacia bacterias con capacidad fotosintética (Olson, 2006), de la cual en buena parte depende el planeta entero.
De las células eucarióticas ancestrales surgen formas ameboides que eventualmente darán origen a plantas (Ruppert et al., 2004). Otras células empiezan a organizarse en colonias con células en su interior y células en su exterior, cada una con funciones específicas (Ruppert et al., 2004). Los organismos filtradores de alimento circundante en el medio acuático semejan pólipos, semejantes a los de los corales de hoy. Posteriormente evolucionan pequeños tentáculos que pueden llevar comida directamente a una boca primitiva. También ciertos organismos empiezan a desarrollar cutículas protectoras, con superficies espinosas, como armaduras (Ruppert et al., 2004).
Hace aproximadamente 550 MA, a partir de los filtradores, aparecen las branquias (agallas) caracterizadas por su mayor eficiencia pues extraen oxígeno disuelto en agua y excretan dióxido de carbono. Estos organismos incluyen formas que nadan libremente en el medio circundante ya sea como juveniles o adultos, mientras otros viven fijados al sustrato marino. Los ancestros de los vertebrados tienen estructuras similares a una columna vertebral, hace 500 MA surgen los primeros peces filtradores sin mandíbula (Forey, 1995; Mallatt y Sullivan, 1998). Estos pequeños ancestros gradualmente desarrollan ojos y mandíbulas lo cual representa una importante novedad evolutiva; tener mandíbula les permite comer y no solo filtrar, y tener branquias les da la posibilidad de respirar debajo del agua. Otra importante novedad evolutiva surge con la aparición de pulmones primitivos, lo que les permite independizarse del ambiente acuático. Los primeros anfibios evolucionaron hace aproximadamente 300-350 MA, tienen una cola tipo pez y sus extremidades les permiten explorar el medio terrestre (Carroll, 2009). La aparición de otra novedad evolutiva, consistente en la capa protectora para huevos, permitió la evolución de reptiles (Hirsch, 1994; Mikhailov, 1997). Una rama de reptiles dio origen a los dinosaurios emplumados cuyas plumas servían para vuelos cortos. Las aves constituyen un linaje descendiente de reptiles primitivos (Ostrom, 1973; Ostrom, 1976). Otros dinosaurios evolucionaron hacia tamaños colosales. Su extinción ocurrió hace unos 65 millones de años, al parecer por el impacto de un meteorito que ocasionó profundos cambios en la atmósfera del planeta (Frankel, 1999).
De los primeros ancestros reptilianos terrestres aparecen los primeros mamíferos, como los marsupiales. Sus crías se desarrollan en el interior de sus madres, aunque nacen inmaduras y totalmente dependientes del cuidado parental (Sears, 2009). Un organismo que semejaba a una musaraña fue el ancestro de todos los mamíferos placentados (Ahlberg y Milner, 1994), que a diferencia de los marsupiales nacen totalmente desarrollados (Vaughan, 1999). Una de esas líneas evolutivas (los monos) pudo escalar árboles, desarrolló mejor destreza en sus movimientos, una visión aguda, cerebros más grandes y una enorme curiosidad acerca del medio ambiente que le rodeaba. Los simios (monos sin cola) y los humanos tienen un ancestro común; es decir, que ambos linajes compartieron un mismo ancestro, del cual se diferenciaron (White et al., 2009; Green et al., 2010). Nuestros ancestros caminaron erguidamente, liberando sus manos para asirse y aferrar cosas, como herramientas e incluso para la defensa. Fueron organismos más altos, con volúmenes cerebrales más grandes, más inteligentes y eventualmente con aptitudes para comunicarse El grado de homología entre simios y humanos es muy alto tanto a nivel morfológico como genético, y es evidencia del hilo conductor que nos conecta. Si fuera posible comprimir los 4.500 MA de evolución en un año (12 meses, 8.760 horas, 525.600 minutos y 31’536.000 segundos), cada segundo sería equivalente a 142 años en tiempo real. Así, los 65 MA del pasado, cuando desaparecieron los dinosaurios, equivaldrían a tan solo 3,5 días del calendario evolutivo. En relación a nuestro ancestro, el género Homo que surgió hace aproximadamente 2,5 MA años, es decir, hace tan solo 3,2 horas y los primeros humanos modernos (Homo sapiens) que surgen hace aproximadamente 165.000 años, correspondería a una aparición de tan solo 3,2 horas. La última glaciación (10.000 años) equivaldría a tan solo unos 42 segundos.
DE RODINIA A LOS CONTINENTES ACTUALES
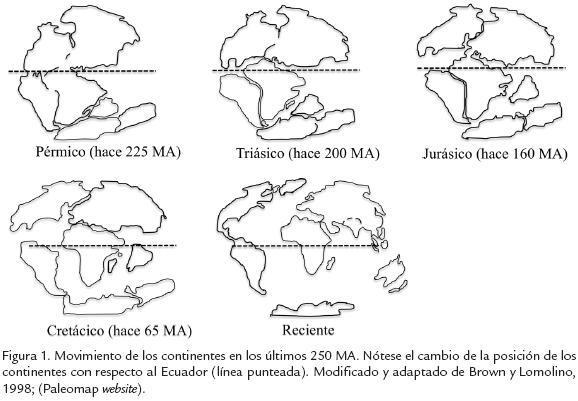
No solo el componente genético es importante en el desarrollo evolutivo, los cambios geográficos y climáticos también han tenido gran importancia en la distribución y divergencia organísmica. Se ha estimado que un primer supercontinente llamado Rodinia se formó aproximadamente hace 1.100 MA (Torsvik, 2003). El supercontinente Gondwana aparece en el Pérmico hace aproximadamente 250 MA (Fig. 1) que sufrió profundas modificaciones formando los continentes actuales y modificando a medida que se movía y fragmentaba la distribución de todos las formas de vida existentes. Por ejemplo, en el Triásico, los animales terrestres podrían migrar del polo sur al polo norte. El supercontinente Pangea empezó a fragmentarse en la mitad del Jurásico y al final del mismo el océano Atlántico separa África del este de la actual Norte América (Paleomapwebsite). India y Magadascar previamente conectadas se separan, e India migró hacia el norte para colisionar con Eurasia. Norte América estaba conectada con Europa y, Australia estaba aún unida a Antártica. Hace aproximadamente 50-55 MA India colisiona con Asia formando el plateau Tibetano y los Himalayas. Australia empieza a migrar rápidamente hacia el norte separándose de Antártica y los continentes empiezan a tener la posición que se conoce hoy (Paleomap website).
LA GENÉTICA Y LA EVOLUCIÓN
En 1865, Gregor Mendel, un monje austriáco diseñó de manera brillante unos experimentos de cruces de guisantes con distintas características, describiendo las proporciones en que resultaban las descendencias. Mendel pudo así predecir que la información o materia hereditaria que se transmitía entre generaciones estaba ubicada en unidades discretas y la manera como estas se separarían. Los experimentos de Mendel y las reglas de la herencia se mantienen hasta hoy día (Griffiths et al., 2000). Posteriormente en 1953, Watson y Crick, usando datos de difracción de rayos x, proponen la estructura del ADN y su organización en una doble hélice (Watson y Crick, 1953). Con la contribución de Watson y Crick, se da inicio al conocimiento sobre los elementos (nucleótidos) que forman el ADN, los enlaces y la complementariedad de las bases nitrogenadas, las ribosas y la geometría de la estructura de la molécula de la herencia. A partir del 2000 la era genómica cambió de manera importante cuando el borrador del genoma humano es publicado (Lander et al., 2001; Waterston et al., 2002). La era genómica también ha permitido poner a prueba las relaciones evolutivas entre entidades biológicas basadas en datos morfológicos.
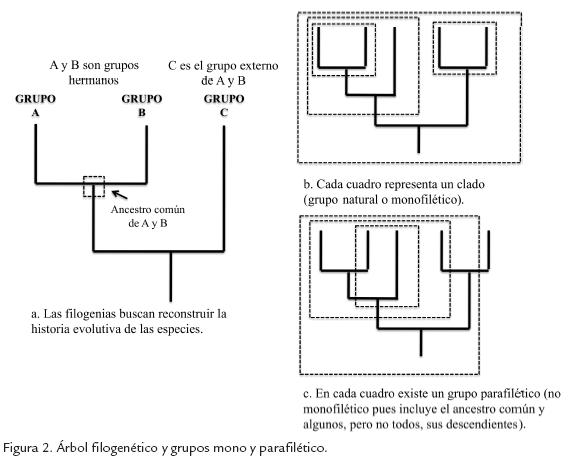
En biología, es posible agrupar organismos a través de distintas aproximaciones, una incluye similaridad (relaciones fenéticas) que no necesariamente implica una relación evolutiva pues muchos organismos pueden poseer características morfológicas comunes debido a que viven en ambientes similares, sin compartir un ancestro común (Futuyama, 2009). La Fig. 2 muestra una representación de una filogenia, se trata de buscar ancestros comunes en el grupo de interés, grupos hermanos y grupos naturales o monofiléticos (que incluyen el ancestro común y todos los descendientes; Fig. 2A y B). El descubrimiento de grupos no naturales o parafiléticos (incluyen el ancestro común y algunos, pero no todos los descendientes), permiten modificar constantemente la clasificación taxonómica de los organismos (Fig. 2C). Una hipótesis de filogenia no se limita a determinar las relaciones entre las entidades biológicas de interés, sino de usar esa información para hacer inferencias adicionales como la aparición de novedades evolutivas importantes. Por ejemplo, en la Fig. 3A se muestra la filogenia de vertebrados, que permite determinar el grado de relación entre los distintos grupos y proponer cuando aparecieron diferentes novedades de interés (pelo, membrana amniótica, entre otros).
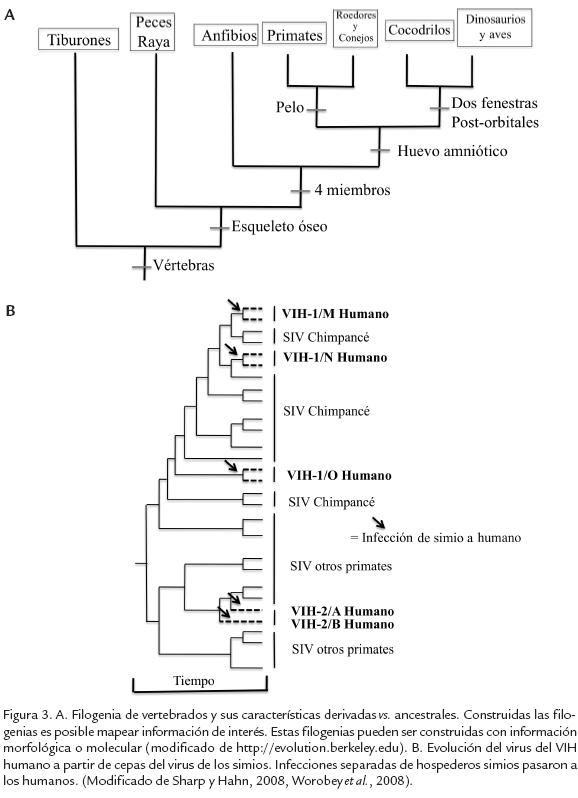
A una escala más reciente, este tipo de enfoque ha permitido entender el surgimiento y evolución de enfermedades que afectan poblaciones humanas. Por ejemplo, el VIH (virus de inmunodeficiencia humana) aparece recientemente en la escala evolutiva; fragmentos de VIH se han encontrado en cadáveres preservados desde 1960, sugiriendola presencia del virus en poblaciones humanas incluso desde principio del siglo XX (Worobey et al., 2008). Por reconstrucción filogenética usando secuencias de ADN, hoy se sabe que el virus está compuesto por cinco familias virales: los grupos M, N y O de VIH1, y los grupos A y B del VIH2 (Fig. 3B); todas descendientes del SIV (virus de inmunodeficiencia en simios, por sus siglas en inglés), cada una resultado de una invasión distinta en hospederos humanos y al parecer en dos eventos separados, el virus simio pasó a hospederos humanos.
LA EVOLUCIÓN DEL GENOMA
A lo largo de la historia evolutiva, el genoma de los organismos ha sufrido numerosos cambios. Estos incluyen mayor tamaño, incremento en el número de genes que pueden surgir por duplicación de formas existentes, nuevas funciones, al igual que intercambio de genes dentro y entre genomas. Como resultado, los genomas se hacen más complejos de modo tal que se realizan estudios comparados (genómica comparada), aunque aún falta avanzar en el entendimiento de la función génica. La paradoja del valor C, la duplicación y fusión de genes, el exon shuffling y la transferencia horizontal de genes son mecanismos interesantes que explican en parte, cómo ha surgido la amplia diversidad genómica existente hoy en día. Adicionalmente, el estudio de los genomas ha permitido establecer que distintas moléculas tienen tasas de mutación específicas. Tal constancia permite utilizarlas como relojes moleculares para estimar el grado de divergencia evolutiva, que junto con la evidencia fósil, amplían el conocimiento sobre el tiempo y modo de evolución.
La paradoja del valor C. El valor C se define como la cantidad de ADN por genoma haploide. Cuando se evalúa dicho valor para distintas especies, no existe relación entre el contenido de ADN y la complejidad organísmica (Tabla. 2) o el contenido de genes codificados por el organismo. Por ejemplo, varios protozoarios unicelulares poseen mucho más ADN que los mamíferos, que presumiblemente son más complejos (Li, 1997). La ausencia de una relación consistente entre el valor C y la complejidad de un organismo es llamado la paradoja del valor C. Aunque es cierto que en general organismos más complejos, tienen genomas más grandes, existe gran variación dentro de grupos (Patrushev y Minkevich, 2008). Por ejemplo, salamandras y peces pulmonados tienen genomas que sobrepasan los 120 pg de ADN por núcleo, mientras el genoma humano tiene 3,5 pg (1pg de ADN corresponde aproximadamente a 109 pares de bases). En plantas terrestres, predominan los genomas gigantes como el de Fritillaria assyriaca, una especie de la familia Liliaceae con 127 pg de ADN (Patrushev y Minkevich, 2008). Algunas de las hipótesis que explican el exceso de ADN con alguna posible función sugieren que las secuencias no codificantes de los genomas eucariota proporcionan genes con protección global y diferencial contra mutágenos químicos. De esa manera, formarían un nuevo sistema de protección de la información genética eucariota (Patrushev y Minkevich, 2008). Otra alternativa apunta a que podría tratarse de un mecanismo de regulación génica ya que si la mayoría de organismos requieren 30.000 a 40.000 genes para su funcionamiento, el exceso de ADN podría estar involucrado en regulación génica, aun por conocer. Por ejemplo, la cantidad de ADN no codificante en eucariotes puede variar de 3 Mb (1Mb=1’000.000 pares de bases) a 100.000 Mb, que abarca des-de un 30 hasta casi 100% del genoma (Cavalier-Smith, 1985).
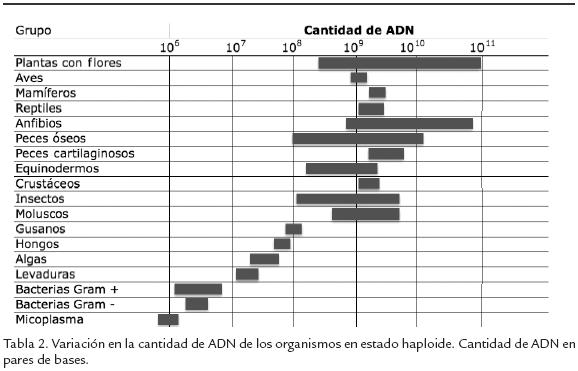
La información genómica reciente confirma que tampoco hay relación entre el número de genes y la complejidad de un organismo, por lo que ha surgido el concepto de paradoja del valor G (número de genes por enoma haploide), y el valor I (medida de información codificada por el genoma). Este último busca incorporar el número efectivo de genes en un organismo que tengan en cuenta fenómenos de corte y empalme alternativos, modificaciones postranscripcionales, proteínas multidominoio, redundancia de genes e interacción entre genes (Hahn y Wray, 2002). Otras teorías apuntan a una posible interacción coevolutiva entre volumen del núcleo y volumen celular. Es decir, la cantidad de ADN se ajustaría como mecanismo adaptativo a un cambio en el tamaño celular (Gregory, 2001). Es probable que en los próximos años, y como resultado de la gran cantidad de información de tipo genómico, surja evidencia a esta u otras teorías que expliquen el enigma de la enorme diversidad en el tamaño de los genomas de organismos eucariota.
Duplicación de genes. Este fenómeno (Fig. 4A) puede producirse en porciones de genes, genes completos, parte de un cromosoma, un cromosoma completo o todo el genoma (Li, 1997). Se considera el mecanismo más eficaz para generar nuevos genes y nuevas vías de control bioquímico que han permitido la divergencia adaptativa de organismos. Por ejemplo, se pueden producir muchas copias de un gen con igual función, como rRNAs o algunas proteínas (Li, 1997; Graur y Li, 2000). En plantas, la duplicación genómica es común, aumentando así el tamaño del genoma (Kellogg, 2003; Lockton y Gaut, 2005), en animales la duplicación es común y ocurrió incluso antes de la diferenciación de los cromosomas sexuales. En mamíferos, aves y reptiles, la duplicación genómica produce disrupción del mecanismo de determinación de sexo y es rápidamente eliminada (Li, 1997).
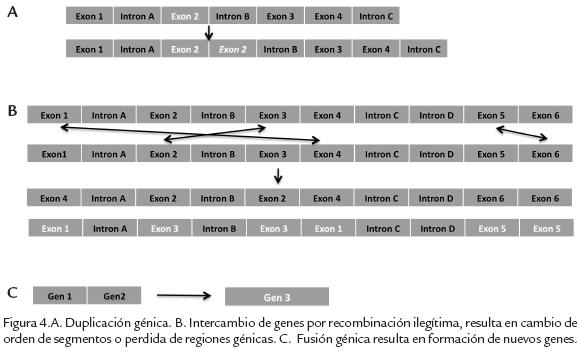
Cuando los genes se duplican, puede ocurrir que la copia sea un sustrato sobre el cual otros mecanismos evolutivos actúen para originar nuevas funciones. También puede ocurrir que la copia se silencie, es decir, pierda su funcionalidad mediante truncamiento de la transcripción o la traducción. Este último mecanismo explica en muchos casos el surgimiento de pseudogenes o copias no funcionales de un gen (D’Errico et al., 2004). La duplicación génica puede contribuir a la complejidad fisiológica o estructural en organismos (Li, 1997). Por ejemplo, hace 600-800 MA un primer evento de duplicación produce dos tipos de proteínas funcionales distintas, mioglobina y hemoglobina. La primera cumple la función de acumular oxígeno en músculo, la segunda, de trasportarlo en sangre. Un segundo evento de duplicación hace aproximadamente 450-500 MA origina las familias alfa y beta. Estas familias se ubican en cromosomas distintos (16 y 11 respectivamente) en humanos y se expresan diferencialmente en el desarrollo embrionario de mamíferos.
Exon Shuffling (Intercambio de exones). La recombinación sexual de genomas parentales es importante en la evolución eucariota, ocurre al azar pero en posiciones homólogas cuya ubicación depende en cierta medida de la longitud del ADN. En el genoma, los exones ocupan solo 1% del mismo (en humanos) y los intrones 24%; se considera que la mayoría de entrecruzamientos y recombinaciones ocurre entre exones. El proceso natural de crear nuevas combinaciones de exones por recombinación intrónica se conoce como intercambio de exones (exon shuffling; Kolkman y Stemmer, 2001). Tal intercambio de exones por recombinación ilegítima, tiene un efecto inmediato que altera el orden de los genes duplicados e incrementa la diversidad proteíca al generar multidominios (Fig. 4B; Vibranovski et al., 2005; Patty, 1999).
Fusión de genes. Nuevas funciones génicas pueden surgir cuando dos genes se fusionan en una única unidad transcripcional. Esta fusión puede ocurrir por recombinación de ADN (Long et al., 2003), como se ha evidenciado en primates (Marques et al., 2005; Akiva et al., 2006). Una vez generados los nuevos genes, se suceden cambios adaptativos rápidos a nivel de aminoácidos y fuertes restricciones funcionales; al parecer alejarse de las funciones ancestrales podría ser un paso crítico en la evolución de estos genes quiméricos surgidos por fusión génica (Jones y Begun, 2005).
La transferencia horizontal de genes (THG). La THG incluye un modo de transmisión genética horizontal (o lateral). Este fenómeno se diferencia de la vía vertical que se da en organismos con reproducción sexual. No existe necesariamente una relación evolutiva directa entre las entidades biológicas que interactúan bajo THG (Fig. 5A). El impacto de la transferencia horizontal de genes empieza a ser evidente cuando a partir de información genómica de muchos organismos procariota y de análisis filogenéticos, se ha encontrado que hay muchos organismos que han adquirido parte de sus genes por transferencia horizontal (Escobar-Páramo et al., 2003; Almeida et al., 2008).
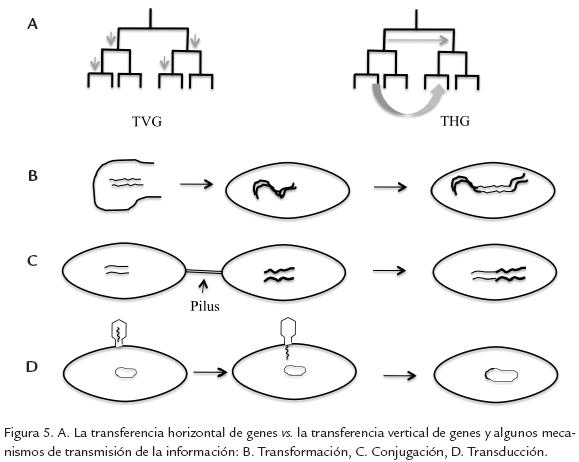
Existen tres mecanismos de THG. El primero es la transformación bacteriana, que involucra un fragmento de ADN desnudo de una célula donante. Este se libera y se introduce a la célula receptora a través de poros (Fig. 5B). Una de las hebras de ADN es digerida permitiendo la entrada de la otra hebra que se recombina con regiones homólogas del cromosoma bacteriano, permitiendo que la célula receptora adquiera la información de la célula donante. Un segundo mecanismo es por conjugación bacteriana e incluye contacto célula a célula (Fig. 5C). La célula donante forma unpilus de conjugación para hacer contacto con el receptor y por el cual ocurre la transmisión del material genético. Finalmente, la transducción ocurre cuando un bacteriófago infecta una bacteria e incorpora su ADN (Fig. 5D).
Las tasas de mutación y el reloj molecular. Las moléculas tienen distintas tasas de evolución y por lo tanto en un mismo periodo de tiempo, el número absoluto de cambios puede ser distinto para diferentes genes. Dicha velocidad de substitución corresponde a la tasa de mutación expresada como el número de cambios por periodo de tiempo. Así, para un rango de tiempo determinado, algunas moléculas con tasas de mutación rápida experimentarán numerosos cambios, otras con tasas intermedias experimentarán algunos cambios, e incluso algunas moléculas con bajas tasas de mutación no experimentarán cambio alguno. En general, se puede decir que moléculas que no tienen una restricción funcional cambian más frecuentemente que aquellas con una función importante. Por ejemplo, las histonas son moléculas que protegen el nucleosoma que recubre el ADN, su carga positiva recubre el ADN de naturaleza negativa, la restricción funcional no permite que haya cambios de aminoácidos que cambien la carga de la proteína. Por otra parte genes no codificadores, como el D-loop del ADN mitocondrial donde se inicia la replicación, tiene una de las tasas de cambio más altas (Lunt, 1998). Puesto que las moléculas cambian a una tasa constante a lo largo del tiempo, se dice que existe un reloj molecular (Zuckerkand y Pauling, 1965). Dicha tasa es utilizada para estimar tiempos de divergencia, usando normalmente un punto de calibración con evidencia fósil (Drummond et al., 2006). También es posible evaluar la constancia en la tasa de evolución molecular cuando no hay fósiles disponibles, mediante pruebas como la prueba relativa (Lemey y Posada, 2010) que evalúa si la tasa de cambio, después de un evento de divergencia, es la misma para las dos ramas resultantes.
Sin embargo, hoy en día se acepta que no existe un reloj universal. Debido a las restricciones funcionales y estructurales, evidenciadas con estudios de evolución molecular, al igual que efectos de selección, hoy en día se habla de relojes locales y no globales para describir las tasas de cambio de las moléculas (Drummond et al., 2006). Por ejemplo, el sistema de termorregulación, el tiempo de generación y la tasa metabólica de los organismos, tienen un efecto sobre la tasa de evolución molecular (Martin y Palumni, 1993). También hay evidencia sobre variaciones temporales en la tasa de evolución y que dichas tasas son específicas para cada gen (Bedford y Hartl, 2008). Muchos estudios evolutivos en la actualidad, buscan estimar tiempos de divergencia basados en la hipótesis del reloj molecular. Sin embargo, dada la enorme heterogeneidad en las tasas de evolución molecular, al estimar tiempos de divergencia se deben tener en cuenta relojes locales que aplican a grupos específicos y ser muy precavidos al supuesto de reloj universal.
CONCLUSIÓN
Los factores tiempo geológico y cambio o variación genética son fundamentales en evolución, aunque aparentemente imperceptibles juegan un papel definitivo en el proceso evolutivo. El genoma no es un simple compartimento de almacenamiento de información, sino que presenta un gran dinamismo por efectos estocásticos o por acción de la selección natural. Las modificaciones del genoma permiten rastrear la historia evolutiva de los organismos pues todos los organismos comparten ancestros comunes. En las representaciones gráficas de las filogenias existen ramas o linajes evolutivos que se mantienen, otros se extinguen, y otros surgen como resultado del proceso dedivergencia.
El avance de la genética ha permitido evidenciar los cambios genómicos que incluyen la alta diversificación proteica resultado de formación de nuevos genes. La diversidad genética de los organismos representada en diversidad de especies es resultado también del aislamiento geográfico, la tectónica de placas y el clima del planeta. Simulaciones de los movimientos tectónicos postulan que se está entrando en una fase de colisión continental que resultará en que Sur América se desprenderá de Norte y Centro América, y se unirá nuevamente a África. Por otra parte, el Atlántico se extenderá, África colisionará con Europa cerrando el Mediterráneo, y Australia colisionará con el sureste de Asia, Pangea Última se formará 250 MA en el futuro (Paleomap website). Aunque la distribución de las especies depende en gran medida del movimiento de los continentes donde habitan, estarán inevitablemente conectados por el proceso evolutivo pues hay un muy delgado hilo (la genética) que se extiende y nos conecta entre esas primeras moléculas que se formaron hace 4.500 MA. Dichas moléculas evolucionan a diferentes tasas de mutación, lo que permite estudiar procesos evolutivos a diferente escala, para así determinar tiempos de divergencia.
AGRADECIMIENTOS
BIBLIOGRAFÍA
AHLBERG PE, MILNER, AR. The Origin and Early Diversification of Tetrapods. Nature. 1994;368:507-514.
[ Links ]ALMEIDA FC, LESZCZYNIECK M, FISHER PB, DeSALLE R. Examining Ancient Inter-domain Horizontal Gene Transfer. Evol Bioinform Online. 2008;4:109-119.
[ Links ]AKIVA P, TOPORIK A, EDELHEIT S, PERETZ Y, DIBER A, SHEMESH, et al. Transcription-mediated Gene Fusion in the Human Genome. Genome Res. 2006;16:30-36.
[ Links ]BEDFORD T, HARTL DL. Overdispersion of the Molecular Clock: Temporal Variation of Gene Specific Substitution Rate in Drosophila. Mol Biol Evol. 2008;25:1631-1638.
[ Links ]BENTON MJ, WILLS MA, HITCHIN R. Quality of the Fossil Record Through Time. Nature. 2000;403:534-537.
[ Links ]BERGER A, LOUTRE MF. Climate: An Exceptionally Long Interglacial Ahead?. Science. 2002;297:1287-1278.
[ Links ]BROWN JH, LOMOLINO MV. Biogeography. 2 ed. Sunderland, MA: Sinauer Associates; 1998.
[ Links ]CAMPBELL NA, WILLIAMSON B, HEYDEN RJ. Biology: Exploring Life. New Jersey: Pearson Prentice Hall; 2006.
[ Links ]CARROLL R. The Rise of Amphibians: 365 Million Years of Evolution. Baltimore: The Johns Hopkins University Press; 2009.
[ Links ]CAVALIER-SMITH T. The evolution of genome size. New York: John Wiley; 1985.
[ Links ]DE AGUIAR MAM, BARANGER M, BAPTESTINI EM, KAUFMAN L, BAR-YAM Y. Global patterns of speciation and diversity. Nature 2009;460:384-387
[ Links ]D ERRICO I, GADALETA G, SACCONE C. Pseudogenes in Metazoa: Origin and Features. Brief Funct Genomic Proteomic. 2004;3(2):157-167.
[ Links ]DALRYMPLE GB. The Age of the Earth in the Twentieth Century: A Problem (Mostly) Solved. Special Publications. Geological Society of London. 2001;190:205-221.
[ Links ]DARWIN C. The Origin of Species by Means of Natural Selection, or the Preservation of Favored Races in the Struggle for Life. New York: Random House Value Publishing; 1959.
[ Links ]DRUMMOND AJ, HO SYW, PHILLIPS MJ, RAMBAUT A. Relaxed Phylogenetics and Dating with Confidence. PLoS Biol. 2006;4(5): e88.
[ Links ]FRANKEL C. The End of the Dinosaurs: Chicxulub Crater and Mass Extinctions. Cambridge: Cambridge University Press; 1999.
[ Links ]FOREY PL. Agnathans Recent and Fossil, and the Origin of Jawed Vertebrates. Rev fish biolfisher.1995;5:267-303.
[ Links ]FUTUYAMA D. Evolution. 2 ed. Massachussetts: Sinauer Associates; 2009.
[ Links ]ESCOBAR-PÁRAMO P, GIUDICELLI C, PARSOT C, DENAMUR E. The Evolutionary History of Shigella and Enteroinvasive Escherichia coli Revised. J Mol Evol. 2003;57:140-148.
[ Links ]GRAUR D, LI WH. Fundamentals of Molecular Evolution. 2 ed. Sunderland, MA: Sinauer Associates; 2000.
[ Links ]GREGORY, TR. Coincidence, Coevolution, or Causation? DNA Content, Cell Size, and the C value Enigma. Biol Rev. 2001;76:65-101.
[ Links ]GRIFFITHS AJF, MILLER, JH, SUZUKI DT, LEWONTIN RC, GELBART WM. An Introduction to Genetic Analysis. 7 ed. New York; WH Freeman; 2000.
[ Links ]GREEN RE, KRAUSE J, BRIGGS AW, MARICIC T, STENZEL U, KIRCHER M, et al. A Draft Sequence of the Neandertal Genome. Science. 2010;328:710-722.
[ Links ]HAHN MW, WRAY GA. The G-value Paradox. Evol Dev. 2002;4:73-75.
[ Links ]HIRSCH KF. The Fossil Record of Vertebrate Eggs. En: Donovan SK, editor. The Palaeobiology of Trace Fossils. Hoboken, NJ: John Wiley and Sons; 1994. p. 269-294.
[ Links ]JONES CD, BEGUN DJ. Parallel Evolution of Chimeric Fusion Genes. Proc Natl Acad Sci U S A. 2005;102(32):11373-11378.
[ Links ]KELLOGG EA. Genome Evolution: It is all Relative. Nature. 2003;422(6930):383-384.
[ Links ]KOCH AL, SILVER S. The First Cell. Adv Microb Physiol. 2005;50:227-259.
[ Links ]KOLKMAN JA, STEMMER W PC. Directed Evolution of Proteins by Exon Shuffling. Nat Biotechnol. 2001;19:423-428.
[ Links ]LANDER ES, LINTON LM, BIRREN B, NUSBAUM C, ZODY MC, BALDWIN J, et al., Initial Sequencing and Analysis of the Human Genome. Nature. 2001;409:860-921.
[ Links ]LEMEY P, POSADA D. Molecular Clock Analysis. En: Vandemme A, Salemi M, editors. The Phylogenetic Handbook. Cambridge: Cambridge University Press; 2010. p. 362-380.LI WH. Molecular Evolution. Sunderland, MA. Sinauer Associates; 1997.
[ Links ] [ Links ]LOCKTON S, GAUT BS. Plant Conserved Non-coding Sequences and Paralogue Evolution. Trends Genet. 2005;21(1):60-65.
[ Links ]LONG M, BETRAN E, THORNTON K, WANG W. The Origin of New Genes: Glimpses from the Young and Old. Nat Rev Genet. 2003;(4):865-875.
[ Links ]LUNT DH, WHIPPLE LE, HYMAN BC. Mitochondrial DNA Variable Number Tandem Repeats (VNTRs): Utility and Problems in Molecular Ecology. Mol Ecol. 1998;(7):1441-1455.
[ Links ]LYNCH, M. Gene Duplication and Evolution. Science. 2002;(297):945-947.
[ Links ]MALLAT J, SULLIVAN J. 28S and 18S Ribosomal DNA Sequences Support the Monophyly of Lampreys and Hagfishes. Mol Biol Evol. 1998;(15):1706-1718.
[ Links ]MARQUES AC, DUPANLOUP I, VINCKENBOSCH N, REYMOND A, KAESSMANN H. Emergence of Young Human Genes After a Burst of Retroposition in Primates. PloS Biol. 2005;3:1970-1979.
[ Links ]MARTIN AP, PALUMNI SR. Body Size, Metabolic Rate, Generation Time, and the Molecular Clock. Proc Natl Acad Sci U S A. 1993;90:4087-4091.
[ Links ]MEASEY GJ, TOLLEY KA. Investigating the Cause of the Disjunct Distribution of Amietophrynus pantherinus, the Endangered South African Western Leopard Toad. Conserv Genet. 2011;12(1):61-70.
[ Links ]MIKHAILOV KE. Fossil and Recent Eggshell in Amniotic Vertebrates: Fine Structure, Comparative Morphology and Classification. Speci Pap Paleontol. 1997;(56):1-80.
[ Links ]OLSON JM. Photosynthesis in the Archean Era. Photosynth Res. 2006;88(2):109-117.
[ Links ]OSTROM JH. The ancestry of Birds. Nature. 1973;242(5393):136.
[ Links ]OSTROM JH. Archaeopteryx and the Origin of Birds. Bioll Jl Linn Soc. 1976;8(2):91-182.
[ Links ]PALEOMAP website: http://www.scotese.com.
[ Links ]PATRUSHEV LI, MINKEVICH IG. The Problem of the Eukaryotic Genome Size. Biochemistry (Moscow). 2008;73(13):1519-1552.
[ Links ]PATTY L. Genome Evolution and the Evolution of Exon Shuffling, a Review. Gene. 1999;238 (1):103-14.
[ Links ]RUPPERT EE, FOX RS, BARNES RD. Invertebrate Zoology: A Functional Evolutionary Approach. 7 ed. Thomson, Brooks/Cole; 2004.
[ Links ]SANMARTÍN I, ENGOFF H. RONQUIST F. Patterns of Animal Dispersal, Vicariance and Diversification in the Holarctic. Biol J Linn Soc. 2001;73:345-390.
[ Links ]SCHOPF JW, KUDRYAVTSEV AB, AGRESTI DG, WDOWIAK TJ, CZAJA AD. Laser-Raman Imagery of Earth s Earliest Fossils. Nature. 2002;416(6876):73-76.
[ Links ]SEARS KE. Differences in the Timing of Prechondrogenic Limb Development in Mammals. The Marsupial-Placental Dichotomy Resolved. Evolution. 2009;63(8)2193-2220.
[ Links ]SHARP PM, HAHN B. Prehistory of HIV-1. Nature. 2008;455:605-606.
[ Links ]TORSVIK TH. The Rodinia Jigsaw Puzzle. Science. 2003;300(5624):1379-1381.
[ Links ]VAUGHAN T, RYAN J, CZAPLEWSKI N. Mammalogy. Philadelphia: Saunders College Publishing; 1999.
[ Links ]VIBRANOVSKI MD, SAKABE NJ, DE OLIVEIRA RS, De SOUZA SJ. Signs of Ancient and Modern Exon-Shuffling Are Correlated to the Distribution of Ancient and Modern Domains Along Proteins. J Mol Evol. 2005;61:341-350.
[ Links ]WATSON JD, CRICK FH. Molecular Structure of Nucleic Acids; A Structure for Deoxyribose Nucleic Acid. Nature. 1953;171(4356):737-738.
[ Links ]WATERSTON RH, LANDER ES, SULSTON JE. On the Sequencing of the Human Genome. Proc Natl Acad Sci U S A. 2002;99(6):3712-3716.
[ Links ]WEN J. Evolution of Eastern Asian and Eastern North American Disjunct Distributions of Flowering Plants. Annu Rev Ecol Syst. 1999;30:421-455.
[ Links ]WHITE TD, BERHANE A, BEYENE Y, HAILE-SELASSIE Y, LOVEJOY CO, SUWA G, WOLDEGABRIEL G. Ardipithecus ramidus and the Paleobiology of Early Hominids. Science. 2009;326(5949):75-86.
[ Links ]WOESE C. The Universal Ancestro. Proc Natl Acad Sci U S A.1998;95(12):6854-6859.
[ Links ]WOESE CR, KANDLER O, WHEELIS ML. Towards a Natural System of Organisms: Proposal for the Domains Archaea, Bacteria, and Eucarya. Proc. Natl. Acad. Sci. 1990;87(12):4576-4579.
[ Links ]WOROBEY M, GEMMEL M, TEUWEN DE, HASELKORN T, KUNTSMAN K, BUNCE M, et al. Direct Evidence of Extensive Diversity of HIV-1 in Kinshasa by 1960. Nature. 2008:455:661-665.
[ Links ]ZIMMER C. On the Origin of Eukaryotes. Science. 2009;325(5941):666-668.
[ Links ]ZUCKERKANDL E, PAULING L. Evolutionary Divergence and Convergence in Proteins. En: Bryson V, Vogel HJ, editors. Evolving Genes and Proteins. New York: Academic Press; 1965. p. 97-166.
[ Links ]













