Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.16 no.3 Bogotá Sept./Dec. 2011
GENÉTICA MOLECULAR Y BIOGERONTOLOGÍA EN LA ERA POSGENÓMICA: UN ENFOQUE EN LAS SIRTUINAS
Molecular Genetics of Aging in the Postgenomic Era: A Focus on Sirtuins
SHADAY MICHÁN1,*, Ph. D., JUAN E. CASTILLO1 1Instituto de Geriatría, Institutos Nacionales de Salud, Secretaría de Salud, México. Periférico Sur n.º 2767, Col. San Jerónimo Lídice, Del. Magdalena Contreras, México D.F., C.P. 10200. *Autor al que se debe dirigir la correspondencia: Shaday Michán, shaday.michan@salud.gob.mx. Teléfono (52) 55 5573 8686. Fax (52) 55 56229198
Presentado el 2 de agosto de 2011, aceptado el 5 de abril de 2011, corregido el 15 de mayo de 2011.
RESUMEN
La forma de estudiar la genética ha progresado notablemente en las últimas décadas. Sus orígenes se remontan al estudio de los caracteres hereditarios, seguido por el descubrimiento de los genes y los cromosomas hasta conocer la estructura del ADN. Este último evento impulsó el desarrollo de la tecnología del ADN recombinante y de la secuenciación masiva y automatizada, los cuales permitieron determinar posterior-mente la anatomía de los genomas. Todos estos descubrimientos han promovido la evolución de la biomedicina hacia las eras genómica y posgenómica en las que el uso de la genética reversa impera sobre la genética básica o directa. Además, surge la genética molecular, la genómica funcional y las diversas tecnologías -ómicas- que en conjunto pretenden comprender de manera integral la función de todos los componentes del genoma y sus productos. La biogerontología, disciplina que estudia los mecanismos biológicos del envejecimiento, es uno de los campos que se han desarrollado notoriamente en los últimos 15 años y refleja los avances científicos de la era posgenómica. Actualmente se han identificado varios gerontogenes y vías moleculares que modifican longevidad y regulan procesos y enfermedades relacionadas con envejecimiento. Dentro de estos genes se encuentran las sirtuinas, una familia de genes conservada evolutivamente que codifica para proteínas con actividad de desacetilasa dependiente de NAD+ y que tienen un papel importante en envejecimiento. En este trabajo revisamos diferentes aproximaciones de genética reversa que se han empleado para identificar algunas de las funciones de estos genes en mamíferos.
Palabras clave: envejecimiento, sirtuinas, modelos animales de enfermedad, ADN recombinante, genética, genómica, biología molecular, ingeniería genética, marcación de gen.
ABSTRACT
The way to study genetics has notably progressed in the last decades. Their origins date back to the study of hereditary features, followed by the discovery of genes and chromosomes up to the knowledge of DNA structure. This last event led to the development of recombinant DNA technology and the massive and automated sequencing, which allowed later to determine the anatomy of genomes. All of these discoveries have pushed the evolution of biomedicine towards the genomic and postgenomic eras, in which the use of reverse genetics prevails over the basic or direct one. Furthermore, it emerges the molecular genetics, the functional genomics and the diverse -omics- technologies that together pretend to understand, in an integrative way, the function of all of the genome components and its products. Biogerontology, discipline that studies the biological mechanisms of aging, is one of the fields that has developed notoriously in the last 15 years and reflects the scientific advances of the postgenomic era. Currently, there have been identified several gerontogenes and molecular pathways that modify and regulate age-related processes and diseases. Among these genes are the sirtuins, an evolutionarily preserved family of genes, which codify for proteins with NAD+ dependent deacetylase activity and that play an important role on aging. In this work, we review different reverse genetics approaches that have been used in order to identify some of the functions of these genes in mammals.
Key words: aging, sirtuins, disease animals models, recombinant DNA, genetics, genomics, molecular biology, genetic engineering, gene targeting.
INTRODUCCIÓN
DE LA GENÉTICA CLÁSICA A LA ERA GENÓMICA
Genes y ADN: los factores fundamentales de la herencia. El estudio de la herencia biológica ha sido un tema de interés a lo largo de la historia pero fue a mediados del siglo XIX con los experimentos realizados por Mendel cuando se lograron establecer las primeras respuestas a este enigma. Mendel observó que las características de los organismos eran heredables de padres a hijos y de una forma independiente, lo cual dio origen al concepto de unidades de la herencia, los genes (Mendel, 1865).
En la primera década del siglo XX, Thomas Morgan sugirió que los genes se localizaban físicamente en los cromosomas con base en observaciones de transmisión de caracteres ligados al sexo en la mosca de la fruta Drosophila melanogaster (Morgan, 1910; Bridges, 1914). En esa época, se desconocía cuál era la molécula responsable de la herencia y la forma de estudiar la genética era mediante el análisis de caracteres fenotípicos. Aunque Friedrich Miescher, 1871, aisló ácido desoxirribonucleico (ADN) pocos años después que su contemporáneo y compatriota, Mendel, presentó los resultados sobre sus experimentos de hibridación en plantas a la Sociedad de Historia Natural de Brünn, se desconocía cuál era la función de este, y no fue sino hasta 1944, cuando se identificó al ADN como la molécula portadora de la información genética (Avery et al., 1944). Este concepto fue confirmado ocho años más tarde por Hershey y Chase, 1952, en un experimento con el fago T2.
Posteriormente, las aportaciones de un conjunto de investigadores permitieron conocer en detalle la estructura del ADN (Franklin y Gosling, 1953; Levene, 1953; Watson y Crick, 1953; Wilkins et al., 1953), descubrimiento que marcó una transición importante en la forma de estudiar genética, desde el estudio clásico de la transmisión de los caracteres hereditarios hasta la era genómica.
La revolución genómica.
Descifrar forma en la que se encuentra codificada la información genética promovió el desarrollo de la tecnología de ADN recombinante, lo cual permitió aislar, manipular y editar material genético; obtener un gran número de copias de fragmentos específicos de este ácido nucleico para su estudio; clonar genes en vectores y estudiar su estructura, organización, función y expresión. Un cuarto de siglo posterior al descubrimiento de la estructura del ADN, se diseñaron los métodos para su secuenciación con el objetivo de conocer el orden de las bases nitrogenadas que constituyen los diversos genes de las diferentes especies, contribución importante que impulsó el desarrollo de la era genómica (Maxam y Gilbert, 1977; Sanger et al., 1977). Posterior-mente se describió la secuencia de todo el material genético o “genoma” contenido en los mismos, incluyendo el ADN del núcleo, mitocondria y los diferentes tipos de plástidos. En 1977 se publicó la secuencia completa del primer genoma, la del fago X174 de 5368 pb (Sanger et al., 1977) y pocos años después se dieron a conocer las 16.569 pb que constituyen el genoma de la mitocondria humana (Anderson et al., 1981).
Posteriormente se desarrolló la secuenciación automatizada, la cual permitió secuenciar ADN con mayor eficiencia, rapidez y a gran escala. Las bases de datos que alojaban secuencias de genes crecieron rápidamente, al tiempo que se iniciaron varios proyectos para secuenciar genomas de diversas especies, incluyendo el del ser humano, el cual empezó en 1990 y concluyó una década después (Roberts, 2001). A la fecha se conocen un total de 3.918 genomas completos de los cuales 40 pertenecen a eucariontes, 1.377 a procariontes, 2.460 a virus y 41 a viroides. Además, existen varios proyectos en curso que al concluirse sumarán un total de 6.710 genomas secuenciados de más de 60 especies. Los genomas difieren en tamaño y complejidad; por ejemplo, el más pequeño reportado a la fecha es el de la alfa-proteobacteria simbionte de cícadas Candidatus Hodgkinia cicadicola compuesto por 144 kbp (McCutcheon et al., 2009); los de los virus son de 2 a 500 kpb y los de las bacterias están compuestos por 500 a 10.000 kpb. Los eucariontes presentan genomas desde 672 kpb como el de Saccharomyces cerevisiae hasta 2,9 x 106 kpb como el del ser humano (Venter et al., 2001) o aún estos pueden ser más grandes como el de la planta Paris japónica con 149 x 106 kpb (Pellicer et al., 2010). Los genomas también varían en el número de copias que se presentan en los seres vivos; por ejemplo, los hongos en alguna etapa de su ciclo de vida cuentan únicamente con una copia del genoma, es decir son haploides, mientras los organismos poliploides pueden poseer un alto número de copias como el crisantemo que presenta hasta diez. Además, aunque los genomas de los seres vivos son únicamente de ADN, los virus, por su naturaleza, pueden tener genomas cuya información genética se encuentra codificada tanto en ADN como en ARN; en cadena doble o sencilla; y presentar una topología circular, propia de bacterias, o lineal, como eucariontes.
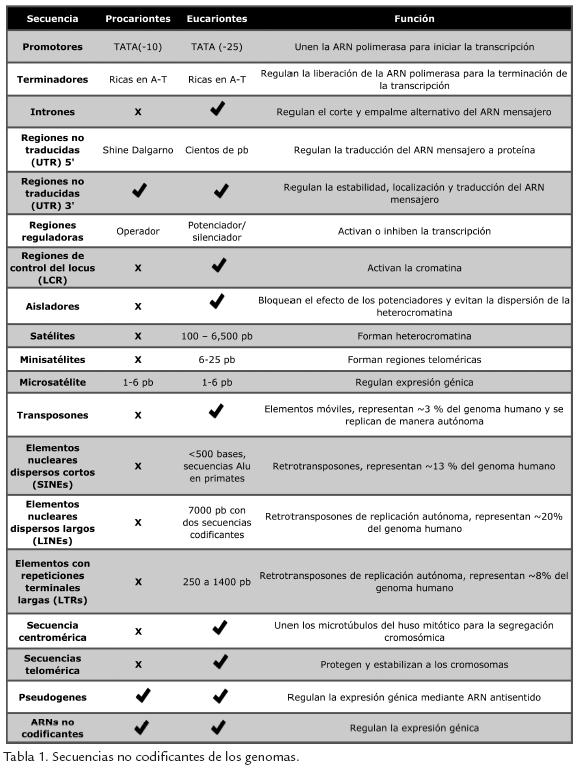
En la revolución genómica el objeto de estudio principal se enfocó a los genomas más que a los genes y este interés permitió esclarecer detalladamente la composición de la molécula esencial que forma a los seres vivos, dejando atrás aquello que hace un siglo y medio se planteaba como un enigma. El análisis de los genomas ha revelado que estos contienen menos genes de lo que se estimaba y que tienen una proporción significativa de ADN no codificante, el cual representa aproximadamente 10% del genoma de procariontes, de 10 a 40% en eucariontes unicelulares, de 70 a 90% en invertebrados, y más de 98% en mamíferos (Szymanski et al., 2005). Estas regiones están compuestas por diferentes secuencias que controlan la expresión génica de forma análoga en procariontes y eucariontes (Tabla. 1), como son promotores, terminadores, secuencias reguladoras y regiones no traducidas (UTR) 5’ y 3’. Además, en los eucariontes, donde las regiones no codificantes son más abundantes, también existen otros tipos de secuencias reguladoras de la expresión génica como los intrones, los aisladores, las regiones de control del locus; otras secuencias como ARNs no codificantes; pseudogenes o genes que han perdido la capacidad de expresión; y diversas secuencias repetitivas como microsatélites, minisatélites, satélites y secuencias dispersas cortas (SINES, por sus siglas en inglés) o largas (LINES, por sus siglas en inglés; Proudfoot, 1980). Todas estas últimas secuencias se denominaron y consideraron en un principio como “basura” pero en la actualidad son objeto de intensas investigaciones en diversos laboratorios del mundo, los cuales han demostrado que éstas desempeñan papeles muy importantes. Por ejemplo, los microsatélites participan en la función y estructura de los centrómeros, así como en la segregación de las cromátidas (Haaf, 1992); los telómeros protegen a los cromosomas de deterioro, mantienen la estabilidad genómica y regulan el envejecimiento, así como diversas enfermedades relacionadas con este proceso (Royle et al., 2009); y el ARN no codificante interfiere con expresión génica (Ørom et al., 2010).
Aunque la secuencia de los genomas ya es un conocimiento finito, aún sigue siendo inmensurable lo que desconocemos sobre la organización y función de todos los elementos contenidos en éstos, incluyendo los genes, diferentes secuencias codificantes y no codificantes; sus interacciones y su regulación transcripcional; el funcionamiento de los productos codificantes; los mecanismos traduccionales y postraduccionales que los regulan; y el diálogo que se establece entre éstos y sus reguladores para coordinar y mantener la homeostasis de las funciones celulares en los seres vivos.
LA GENÉTICA MOLECULAR Y GENÓMICA FUNCIONAL EN LA ERA POSGENÓMICA
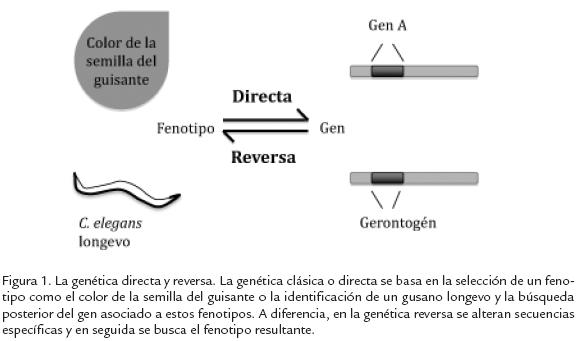
Genética directa y genética reversa. La capacidad de manipular ADN y conocer las secuencias de los genes en combinación con la necesidad de entender en detalle la estructura y función de los mismos, ha revolucionado la forma de aproximarnos a la genética y promovido el desarrollo de un nuevo campo de la biología, la genética molecular. El tipo de observaciones que Mendel y Morgan realizaron en sus experimentos son un claro ejemplo de la genética clásica o directa, en la que se observaba un fenotipo y éste se atribuía a un factor o gen responsable de dicho efecto. Aunque en ese entonces la naturaleza de este era desconocida, se tenía la certeza de que su segregación era independiente. Con el desarrollo posterior de las técnicas de ADN recombinante y la secuenciación fue posible identificar alteraciones específicas en las secuencias de ADN responsables de un fenotipo determinado. La genética directa ha evolucionado con el paso del tiempo y esto ha permitido descubrir la función de varios genes a partir de estudiar fenotipos singulares aislados naturalmente o inducidos con mutágenos (Stark y Gudkov, 1999). A principios de 1986, la clonación posicional o descubrimiento de genes basado en mapeo se convirtió en el método principal para elucidar las bases moleculares de varias enfermedades genéticas. La disponibilidad de la secuencia de los genomas y el desarrollo de métodos rápidos de secuenciación impulsó la evolución hacia la era posgenómica en la que nos encontramos actualmente y en la que impera el uso de aproximaciones en sentido contrario a la genética directa, es decir, se altera la secuencia específica que se desea estudiar y posteriormente se busca el fenotipo asociado con dicha modificación (Fig. 1). A esta forma de estudiar la función de secuencias de ADN se le conoce como genética reversa, en la cual se dirige una modificación determinada a una secuencia específica del genoma con el objetivo de reemplazarla o mutarla (knock-in), eliminarla (knock-out), o bien introducir una copia extra para incrementar su expresión (transgénico). También se puede utilizar la tecnología antisentido como el ARN de interferencia o los morfolinos (oligómeros formados por análogos de ácidos nucleicos) para disminuir o suprimir un producto génico y posteriormente analizar los cambios fenotípicos resultantes. La genética reversa actualmente se aplica en una gran variedad de organismos, desde bacterias hasta plantas y metazoarios como el gusano Caenorhabditis elegans, la mosca de la fruta Drosophila melanogaster y el pez cebra Danio rerio. En 1989, Capecchi, 1989, Evans y Smithies generaron el primer ratón knock-out por reemplazamiento génico, lo cual les mereció ser galardonados con el premio Nobel en Medicina y Fisiología 2007. Desde entonces, la genética reversa en roedores ha estado evolucionando y actualmente varios grupos participan en un proyecto internacional para obtener mutantes de cada uno de los genes contenidos en el genoma de ratón. Además, aunque los primeros knock-outs de rata se obtuvieron en 2003 por medio de mutagénesis inespecíficas (Zan et al., 2003), fue a finales de 2010 cuando se creó el primer knock-out en esta especie por medio de mutagénesis dirigida en células troncales. La pérdida de función se logró al reemplazar los exones 2-5 del gen p53 por medio de recombinación homóloga (Tong et al., 2010). Todos estos hallazgos en la era posgenómica permiten estudiar la función génica en modelos novedosos, abren la puerta hacia un sinfín de descubrimientos y prometen un avance importante en biomedicina y farmacología durante las próximas décadas.
La genómica funcional. En la era posgenómica también se desarrolla un nuevo campo de la biología molecular que es la genómica funcional, el cual tiene como objetivo entender las funciones del ADN a diferentes niveles incluyendo transcritos, secuencias no codificantes y productos de los genes, así como los mecanismos genéticos y moleculares que gobiernan las funciones de los seres vivos. La genómica funcional busca utilizar nuevas aproximaciones y tecnologías para contestar eficientemente la infinidad de preguntas que existen en biomedicina y las cuales están centradas en entender las relaciones entre las características del genoma de los organismos y su fenotipo. Los estudios de relación entre genotipo y fenotipo han permitido identificar varias alteraciones genéticas causantes de enfermedades. En 1981 se conocían menos de 25 genes responsables de alguna enfermedad y para el año 2000 se describieron 1.112 mutaciones relacionadas con más de 1.500 enfermedades. También, durante este mismo lapso de tiempo se lograron caracterizar molecularmente 1.430 desórdenes clínicos (Peltonen y McKusick, 2001).
En vez de aplicar aproximaciones para estudiar la función de un solo gen, en la actualidad la tendencia es realizar análisis globales para evaluar el comportamiento simultáneo de una gran cantidad de genes, proteínas, microARNs y demás componentes del genoma. Nuevas herramientas como los chips han sido la base para el desarrollo de diferentes tecnologías ómicas como la transcriptómica que se emplea para el estudio de los patrones de expresión génica; la proteómica para el análisis de las interacciones y de los niveles proteicos; y la metabolómica para la dinámica e integración de las vías metabólicas.
Esta área también incluye el estudio de mutaciones y variaciones en la secuencia del genoma conocidas como polimorfismos de un solo nucleótido (SNPs). A la fecha se han descrito 3 millones de sitios con SNPs en el genoma humano y el entender cómo estas variaciones genéticas regulan fenotipos celulares, tejidos y órganos es uno de los retos de la biomedicina en el siglo XXI.
AVANCES BIOGERONTOLÓGICOS EN LA ERA POSGENÓMICA
El descubrimiento de los gerontogenes. La biogerontología, disciplina que estudia los mecanismos biológicos de envejecimiento y las enfermedades relacionadas con este proceso, ha evolucionado notoriamente en las últimas décadas como resultado de los avances y la implementación de las diferentes aproximaciones científicas que han impulsado a las eras genómica y posgenómica. Por ejemplo, actualmente sabemos que el envejecimiento no es azaroso, sino que igual que muchos otros procesos biológicos es regulado dinámicamente por vías celulares clásicas, además, diversos factores como los dietéticos, genéticos y farmacológicos pueden modificar su curso (Kenyon, 2010; Michán, 2010). Puesto que la biogerontología es una disciplina joven, aún falta mucho por explorar y tiene el potencial de contribuir con el desarrollo de una vida humana saludable en edades avanzadas, uno de los retos biomédicos más urgentes en la actualidad en la que la esperanza de vida de la población mundial ha incrementado notoriamente y como consecuencia, la proporción de adultos mayores de 65 años y las enfermedades asociadas con el envejecimiento también van en aumento (Michán y Michán, 2010).
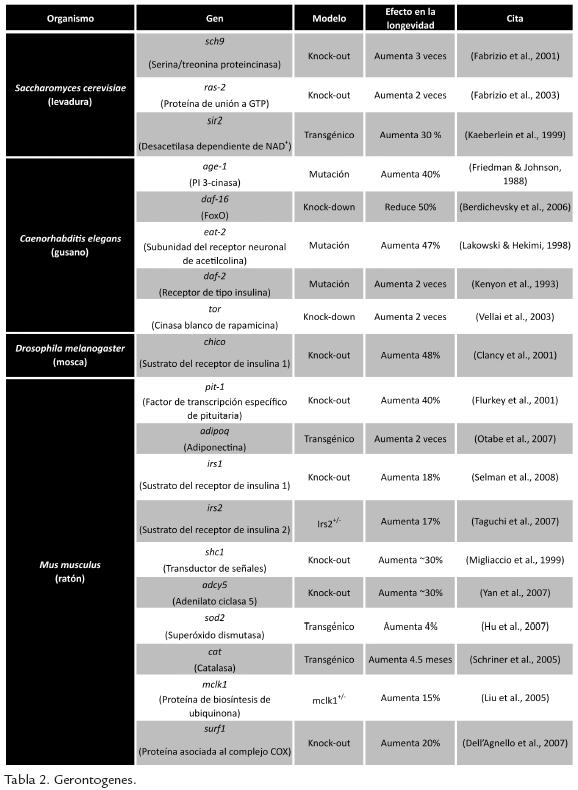
La genética molecular y la genómica funcional características de la era posgenómica, han permitido empezar a conocer algunos de los genes y mecanismos moleculares relacionados con el proceso de envejecimiento. Si bien, desde hace varias décadas se han reportado diversas intervenciones que regulan la esperanza de vida de los organismos como la descrita por McCay et al., 1935, quienes demostraron que la restricción calórica (RC) o disminución en el consumo de calorías en 30% duplicaba la esperanza de vida de ratas, no fue sino hasta 1983 cuando se realizaron los primeros estudios en el gusano C. elegans para la identificación de gerontogenes o genes que modifican la esperanza de vida de los organismos. En un tamizado genético Klass, 1983, identificó ochos mutantes de gusano con esperanza de vida más larga que la cepa silvestre. Años después, Friedman y Johnson, 1988, reportaron la primera mutación en el gen age-1 que codifica para la fosfoinosítido 3 (IP3) cinasa de C. elegans, responsable de incrementar hasta 60% su longevidad. A esto procedió el descubrimiento de daf-2, un gen que codifica para la única proteína homóloga al receptor de tipo insulina de mamíferos presente en éste nemátodo y en el que una mutación duplicó su esperanza de vida. A la fecha se han descrito gran variedad de gerontogenes en diferentes modelos biológicos dentro de los que se encuentran los que participan en las vías de señalización de detección de nutrientes como la de Tor y la de Ras, y genes que responden a estrés oxidativo como las enzimas mitocondriales superóxido dismutasa (Sod2) y catalasa (mCAT; Tabla. 2). Es importante notar que tanto la pérdida de la función como la sobreexpresión de gerontogenes pueden resultar en un aumento en la longevidad.
En la siguiente sección describimos diversos enfoques genéticos que han conducido al descubrimiento de las sirtuinas, una familia de gerontogenes conservados durante la evolución, que por regular la longevidad de organismos, han despertado el interés de biogerontólogos y promovido que numerosos laboratorios nos sumemos al esfuerzo para entender el papel que los productos de estos genes juegan en diferentes procesos celulares relacionados con envejecimiento.
Genética molecular para el estudio de las sirtuinas.
Con el uso de la genética directa y por medio de un tamizado para encontrar supresores del defecto de apareamiento en la levadura S. cerevisiae, hace más de treinta años se identificó un grupo de 4 genes, SIR (silent information regulator) 1-4, responsables del silenciamiento de los loci de tipo sexual (Rine y Herskowitz, 1987; Rine et al., 1979). Posteriormente se demostró que SIR1-4 también regulaba silenciamiento de los genes próximos a telómeros (Aparicioet al., 1991), inhibían transcripción y ayudaban a mantener la estabilidad genómica al formar parte del complejo que repara ADN por medio de la unión de extremos no homólogos (NHEJ, por sus siglas en inglés; Tsukamoto et al., 1997). La participación de los genes SIR en la regulación de silenciamiento génico, estabilidad genómica y reparación de ADN sugería que éstos podrían regular longevidad de la levadura, así que utilizando genética reversa Kaeberlein et al., 1999, obtuvieron mutantes de los diferentes genes SIR y evaluaron el envejecimiento del microorganismo. De los cuatro genes, solo la ausencia de SIR2 disminuyó 50% la esperanza de vida de la levadura, mientras que la adición de una copia extra del gen en la cepa silvestre aumentó su longevidad 30%. En S. cerevisiae, SIR2 también fue esencial para suprimir la recombinación en las repeticiones de rDNA (Gottlieb y Esposito, 1989), así como la escisión y formación de círculos extracromosomales de rDNA (ERCs, por sus siglas en inglés), las cuales promueven envejecimiento de la célula (Sinclair y Guarente, 1997). Además, SIR2, a diferencia de los otros genes SIR, también participa en procesos de desacetilación. Braunstein et al., 1993, demostraron que la sobreexpresión de esta causaba que las lisinas de histonas en las regiones silenciadas del genoma estuvieran hipoacetiladas y consecutivamente se comprobó que SIR2 poseía actividad de desacetilasa dependiente de NAD+ (Landry et al., 2000).
Considerando que la actividad de desacetilasa de SIR2 subyace a fenómenos de silenciamiento génico, los cuales se encuentran conservados desde bacterias hasta humano, era muy probable pensar que estos genes estuvieran presentes en otros organismos. En 1995, esta hipótesis fue corroborada por Brachmann et al., 1995, quienes demostraron la presencia de homólogos de SIR2 en diferentes especies desde bacterias hasta vertebrados. Los miembros de la familia de genes/proteínas homólogas de SIR2 recibieron el nombre de sirtuinas y estudios filogenéticos dentro de los cuales el más reciente incluye 77 especies y 240 sirtuinas con representantes de todos los fila, clasifican a estas con base en sus secuencias de aminoácidos en cinco grupos principales: I, II, III, IV y U y dan a conocer las siguientes peculiaridades de esta familia de proteínas. Las sirtuinas se presentan en la mayoría de las especies desde bacterias hasta humanos, con excepción de dos algas rojas y algunas archaea. Además, existe un grupo de estas no relacionado con las encontradas en vertebrados pero que están presentes en bacterias y archaea (U), un subgrupo de sirtuinas que figura únicamente en los hongos (Ic) y otro encontrado únicamente en animales, SIRT3 (Greiss y Gartner, 2009).
Estudios adicionales han demostrado que funcionalmente las sirtuinas regulan envejecimiento y diversas patologías asociadas con este proceso (Michán y Sinclair, 2007). Mediante el uso de genética reversa se ha logrado adicionar o eliminar genes de sirtuinas en metazoarios para estudiar la variedad de funciones de esta familia. Por ejemplo, en C. elegans una duplicación del ortólogo de SIR2, sir2.1, aumentó 50% la esperanza de vida al regular la vía de señalización de insulina río arriba al factor de transcripción daf-16 (Tissenbaum y Guarente, 2001; Berdichevsky et al., 2006). Además, la sobreexpresión de dSir2 en D. melanogaster con niveles de ARN mensajero cuatro veces mayores al endógeno, incrementó longevidad 29% en hembras y 18% en machos (Rogina y Helfand, 2004). En mamíferos se han identificado siete sirtuinas, SIRT1-7, con diferentes localizaciones subcelulares y de las cuales ya se conocen algunas funciones.
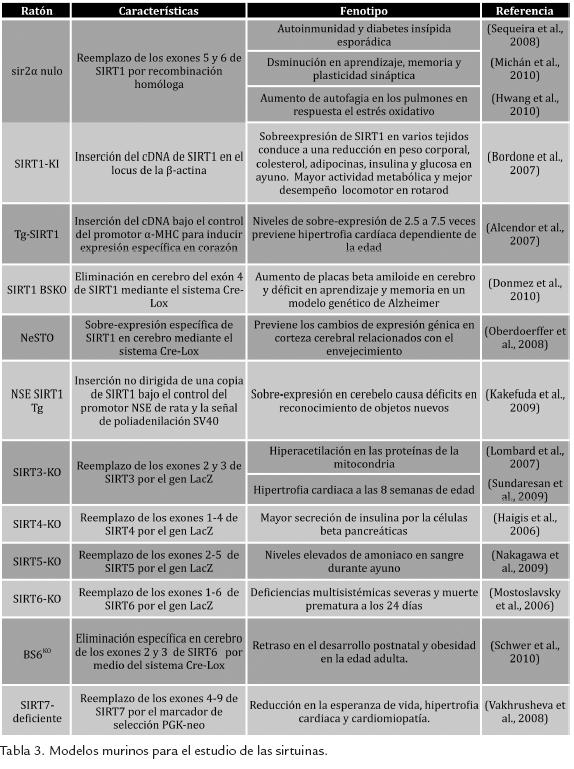
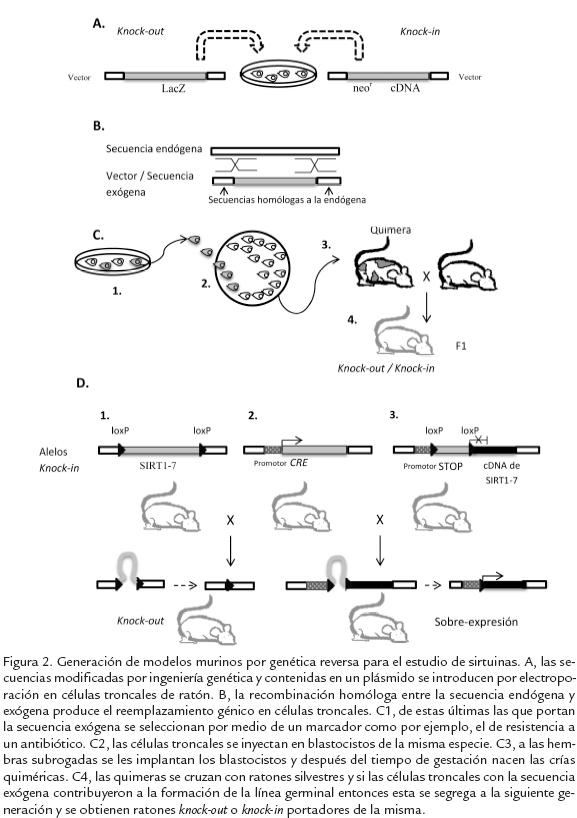
Varias estrategias de genética reversa se han empleado para manipular ADN y estudiar el funcionamiento de las sirtuinas en modelos de ratón (Tabla. 3). Las secuencias específicas posibles de ser analizadas se pueden eliminar o mutar por medio de reemplazamiento génico en células troncales de la misma especie, el cual se logra como resultado de recombinación homóloga entre secuencias endógenas y exógenas (Fig. 2B). Estas últimas se manipulan previamente por ingeniería genética e inmovilizan en un vector o plásmido de ADN para ser introducidas por medio de choque eléctrico a dichas células (Fig. 2A). De éstas, las que experimentan recombinación homóloga son seleccionadas por medio de un marcador contenido en la construcción exógena (Fig. 2C 1) e inyectadas en blastocistos (Fig. 2C 2), los cuales posteriormente se implantan en hembras subrogadas de la misma especie (Fig. 2C 3). Como resultado se obtiene un ratón quimérico en el que si las células madre inyectadas contribuyeron al desarrollo de la línea germinal, entonces el ADN exógeno se segregará a la siguiente generación y cada una de las células del ratón F1 contendrá en su genoma una copia de este (Fig. 2C 4). Así, se pueden obtener ratones de línea germinal tanto knock-out como knock-in que sobreexpresen una determinada sirtuina para analizar su función (Fig. 2C).
Otra aproximación de genética reversa para generar modelos murinos es por medio del uso del sistema de recombinación Cre-loxP, el cual permite regular de manera espacial y temporal la recombinación de fragmentos de ADN flanqueados por la secuencia loxP en presencia de la recombinasa Cre. Para esto se requiere una cepa portadora del alelo condicional que se genera por medio de ingeniería genética utilizando un plásmido al que se le introduce el material genético de interés flanqueado por secuencias loxP. Posteriormente las células troncales se transforman con dicho vector y siguiendo los mismos pasos descritos anteriormente para la generación de knock-out/knock-in de línea germinal, se obtiene una cepa de ratón que contiene el alelo de interés flanqueado por secuencias loxP (Fig. 3A; Fig. 3B).
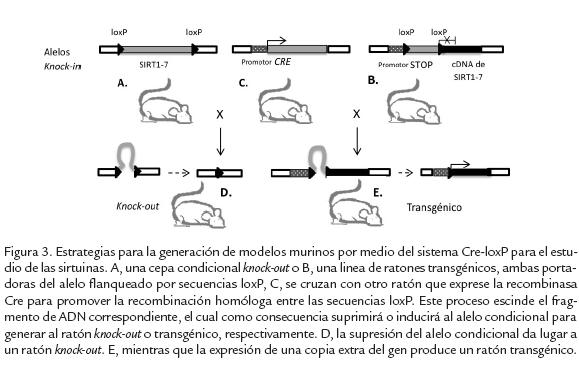
Por otra parte se requiere una cepa que exprese la recombinasa Cre, la cual promueva recombinación homóloga entre secuencias loxP y escinda el fragmento de ADN correspondiente para inducir o suprimir al alelo condicional (Fig. 3C). En la actualidad existe una colección de ratones transgénicos que expresan recombinasa Cre bajo la dirección de una gran variedad de promotores, los cuales son distribuidos a nivel comercial por los laboratorios Jackson (http://www.jax.org/) o Taconic (http://www.taconic.com/wmspage. cfm?parm1=16). De tal forma que dependiendo del tipo de estudio que se va a realizar y del tejido que se requiere analizar, se utiliza el ratón apropiado para cruzarlo con la cepa que contiene el alelo condicional. Por ejemplo, si el objetivo es analizar la función de la sirtuina en el organismo completo, entonces se puede elegir un ratón que exprese Cre de manera ubicua utilizando transgénicos con diferentes promotores como el de β-actina humana; el del virus de tumor mamario de ratón (MMTV); el de adenovirus EIIa; el de ubiquitina C humana (UBC); el CAG compuesto por el promotor y potenciador de β-actina de pollo acoplado al potenciador de CMV; el del prión de ratón (Prnp); o el del citomegalovirus humano (CMV) que se induce en todos los tejidos incluyendo la línea germinal. En caso de que el estudio se requiera llevar a cabo de forma específica en algún tipo de tejido o grupo de células, entonces se deberá elegir un ratón que exprese Cre bajo el control de promotores que únicamente sean activos en el tejido de interés. Esta es una de las formas en las que se pueden generar alelos condicionales para inducir la expresión de un transgén de sirtuina o la eliminación de sus exones para generar un knock-out tejido específico. Por ejemplo, existen ratones que expresan CRE en hígado (Alb-Cre), músculo estriado (ACTA1-Cre), células beta pancreáticas (Ins-Cre) o glándulas mamarias (Wap-Cre), entre muchos otros y algunos de los cuales se han utilizado para descubrir diferentes funciones de las sirtuinas que discutiremos en detalle más adelante.
También, es importante considerar que la expresión de la recombinasa Cre, dependiendo del promotor, puede inducirse durante las etapas embrionarias tempranas y acarrear problemas de desarrollo, interfiriendo con algunos estudios de envejecimiento. Para estos casos se pueden utilizar sistemas de expresión inducibles mediante diversos compuestos que brindan la posibilidad de elegir el tiempo en el que se desea iniciar la expresión, y dependiendo del promotor estos pueden ser ubicuos o tejido-específicos. Por ejemplo, los sistemas que permiten esta regulación son el promotor Mx1 que se induce por interferón, el receptor de estrógeno (ERT) inducido por tamoxifeno, el receptor de progesterona inducido por RU 486 y el sistema TetO inducido por tetraciclina.
El uso de todas estas diferentes aproximaciones de genética reversa han ayudado a dilucidar varias de las funciones de las sirtuinas de mamíferos. Por ejemplo, utilizando ratones knock-out de línea germinal a los que se les eliminó por reemplazamiento génico los exones 5 y 6 del gen que codifica para SIRT1, demostramos que esta sirtuina que tiene la mayor similitud a Sir2 de levadura y que se localiza en el núcleo de neuronas del hipocampo, es esencial para la adquisición y consolidación de la memoria hipocampodependiente a corto y largo plazo, así como para el mantenimiento de la plasticidad sináptica. Sin embargo, esta no modifica las propiedades sinápticas basales, la arquitectura de las espinas dendríticas, ni los niveles de proteínas sinápticas. Además, nuestros resultados mostraron que alteraciones en los árboles dendríticos neuronales, en la vía de señalización de la cinasa ERK1/2 y en la expresión génica son algunos de los mecanismos por los cuales SIRT1 puede regular aprendizaje, memoria y plasticidad sináptica (Michán et al., 2010).
Estudios in vitro con cultivos celulares e in vivo con modelos murinos han demostrado que SIRT1 también desempeña un papel importante en la protección contra diferentes patologías relacionadas con envejecimiento. Por ejemplo, Donmez et al., 2010, sobreexpresaron SIRT1 en células de neuroblastoma al transfectar estas con un plásmido que contenía una copia editada de este gen conocida como cDNA, la cual poseía únicamente los exones y estaba bajo la dirección de un promotor de mamíferos. Por otra parte, para la supresión génica utilizaron tecnología antisentido y a las células les introdujeron una horquilla pequeña de ARN (shRNA) para interferir con el proceso de traducción del mensajero de SIRT1. En ambos casos las células fueron expuestas a los péptidos que forman las placas β-amiloides, consideradas unas de las causas de la enfermedad de Alzheimer. La sobreexpresión de SIRT1 disminuyó la presencia de dichas placas, en contraste, las células tratadas con el shRNA mostraron un depósito mayor de las mismas. Para estudiar el efecto in vivo de la pérdida de la función de SIRT1 en la enfermedad de Alzheimer, generaron un modelo de ratón con tres alelos, el que porta una mutación para esa enfermedad, el alelo condicional knock-out de SIRT1 y el transgén Nestin-Cre con expresión específica de Cre en cerebro. De acuerdo con lo observado in vitro, los resultados con estos ratones demostraron que la carencia de SIRT1 aumentaba la presencia de placas β-amiloide en el cerebro. En contraste, los ratones transgénicos de línea germinal que sobreexpresaban SIRT1 como resultado de recombinación homóloga e inserción de una copia de cDNA de este gen en seguida del promotor de β-actina endógeno, presentaron menores depósitos de las mismas placas. Investigaciones adicionales indican que SIRT1 desacetila el receptor de ácido retinoico, el cual activa el gen de α-secretasa. Este a su vez promueve escisión del precursor de proteína amiloidea, evita formación de placas β-amiloide y protege contra la neurodegeneración producida por la enfermedad de Alzheimer.
Por otra parte, utilizando una cepa Villin-Cre para sobreexpresar de forma específica SIRT1 en el epitelio del intestino del modelo genético murino de cáncer de colon APCmin, demostramos que esta sirtuina disminuye la incidencia de adenomas en ani-males propensos a desarrollar cáncer debido a que portan una mutación análoga en el gen del supresor tumoral APCmin, causante de cáncer de colon familiar y de 80% de los casos de cáncer esporádico en humano (Firestein et al., 2008).
Otros experimentos mostraron que ratones transgénicos que sobreexpresaban niveles moderados de SIRT1 (de 2,5 a 7,5 veces arriba del nivel basal) bajo el control del promotor específico de corazón α-MHC presentaban cambios atenuados asociados a la edad en este órgano, como son una disminución en la incidencia de cardiomiopatías, en la presencia de alteraciones histológicas y en los niveles de proteínas que forman parte de la familia INK4/ARF, los cuales son inductores de senescencia celular y marcadores de envejecimiento. Además, el aumento en SIRT1 promovió la resistencia del corazón a estrés oxidativo (Alcendor et al., 2007).
Nuestros estudios también demuestran que SIRT1 regula la estabilidad genómica y protege contra linfoma en el modelo de pérdida de heterocigosidad p53+/-. Mediante cruzas en una línea de ratón se combinó el alelo mutante de p53, el transgén Mx-Cre para expresar Cre en las células linfoides de ratones adultos y cDNA de expresión condicional de SIRT1 adyacente a un casete de paro de transcripción flanqueado por secuencias loxP. Los animales inyectados con el inductor de interferón poly(I)poly(C) que expresaron Cre, recombinaron las secuencias loxP, perdieron el casete de paro adyacente al alelo condicional y por lo tanto sus células linfoides produjeron niveles elevados de SIRT1, tuvieron una supervivencia 46% mayor después de ser expuestos a radiación gamma en comparación con ratones control p53+/-que no expresaban la sirtuina (Oberdoerffer et al., 2008).
Otras funciones atribuidas a SIRT1 son la formación de heterocromatina facultativa (Vaquero et al., 2004), la regulación de la diferenciación celular (Zhao et al., 2005), la supresión de apoptosis en respuesta a estrés (Vaziri et al., 2001), la resistencia celular durante deficiencia de oxígeno (Chen et al., 2006) y la reparación de ADN inducida por luz UV (Wei et al., 2008).
Una de las funciones de sirtuina nuclear SIRT6 fue descrita por Mostoslavsky et al., 2006, en un modelo knock-out de línea germinal que se generó por medio de reemplazo de los exones 1-6 en un proceso de recombinación homóloga. La ausencia de dichas secuencias génicas, además de un marcado retraso en el crecimiento corporal, también promovió bajos niveles de grasa subcutánea, colitis, linfopenia, osteopenia, entre otras deficiencias metabólicas, efectos que condujeron a muerte prematura de los ratones al día 24 de vida. Sin embargo, los ratones que carecían de SIRT6 específicamente en cerebro, alcanzaron su tamaño corporal normal a la sexta semana pero presentaron retraso en el desarrollo postnatal, obesidad durante la etapa adulta y reducción en los niveles hipotalámicos de proopiomelanocortina (POMC), del homólogo 1 de la proteína single-minded (Sim1) y del factor neurotrófico derivado de cerebro (BDNF), todos estos factores relacionados con obesidad en humanos.
SIRT7 es una de las tres sirtuinas que también se localiza en núcleo, específicamente dentro del nucléolo. Para el estudio de su función Vakhrusheva et al., 2008, generaron un modelo knock-out mediante reemplazo génico, el cual presentó hipertrofia cardiaca, cardiomiopatía inflamatoria y disminución en longevidad. Experimentos in vitro demostraron que p53 es sustrato de SIRT7 y que el mecanismo molecular que subyace al fenotipo cardiaco observado en este ratón está relacionado con la hiperacetilación y activación de dicho factor transcripcional, y en consecuencia con un aumento en la apoptosis de células de corazón debido a la ausencia de esta sirtuina.
SIRT2 se localiza en citoplasma y regula ciclo celular (North y Verdin, 2007), tumorogénesis (Hiratsuka et al., 2003) y adipogénesis (Jing et al., 2007). Esta sirtuina interacciona con el factor de transcripción HOXA10 que regula embriogénesis y desarrollo (Bae et al., 2004). Además, inhibidores de SIRT2 en diversos modelos han demostrado que esta juega un papel importante en neurodegeneración (Garske et al., 2007). Lombard et al., 2007, desarrollaron ratones knock-out para las tres sirtuinas mitocondriales, SIRT3, SIRT4 y SIRT5. Aun cuando estos animales parecen normales y únicamente la deficiencia de SIRT3 causó hiperacetilación de proteínas mitocondriales, se han logrado identificar diversas funciones de estas sirtuinas. Nuestros datos demuestran que SIRT3 se regula dinámicamente por la dieta y el ejercicio en músculo esquelético (Palacios et al., 2009). Experimentos in vitro e in vivo indican que SIRT3 reduce la producción de especies reactivas de oxígeno, protege de muerte celular inducida por daño oxidativo, y a través de este mismo mecanismo media los efectos antienvejecimiento de la restricción calórica que previenen la pérdida auditiva dependiente de la edad (Shi et al., 2005; Someya et al., 2010). Además, se ha demostrado que SIRT3 regula la oxidación de ácidos grasos (Hirschey et al., 2010), la hipertrofia cardiaca (Sundaresan et al., 2009), y juega un papel importante como supresor tumoral (Kim et al., 2010). Los ratones deficientes de SIRT4 presentan niveles de secreción de insulina mayores en respuesta a glucosa y aminoácidos en comparación con animales que presentan alelos silvestres (Haigis et al., 2006). El ratón mutante de SIRT5 presenta desregulación en la actividad de carbomoil fosfato sintetasa 1 (CPS1), alteraciones en el ciclo de la urea y altos niveles de amoniaco en sangre durante el ayuno (Nakagawa et al., 2009). En resumen, las sirtuinas desempeñan funciones diversas a nivel celular, muchas de ellas relacionadas con los procesos de envejecimiento en mamíferos.
CONCLUSIONES
La era posgenómica que estamos viviendo actualmente es el resultado de una serie de avances científicos y tecnológicos que han revolucionado la biomedicina y la forma de aproximarnos a la genética. Ya conocemos en detalle la anatomía de los genomas y el reto al que nos enfrentamos en la actualidad es entender la función de cada uno de los componentes de éste, incluyendo a los genes, a otras secuencias codificantes y a las diferentes regiones de ADN no codificante que contienen en abundancia los genomas de los mamíferos. Desconocemos aún la función de todos los genes, la causa de una gran variedad de enfermedades y apenas estamos empezando a entender algunos de los mecanismos biológicos que participan en envejecimiento. La biogerontología promete un importante desarrollo en las próximas décadas, en donde nuevos gerontogenes, múltiples blancos e interacciones moleculares faltan por descubrir. De las sirtuinas, si bien para algunas se han reportado varias funciones como es el caso de SIRT1, de otras, como por ejemplo de las mitocondriales SIRT3, SIRT4, SIRT5, se desconocen muchas de sus funciones a pesar que la falta de SIRT3 incrementa acetilación global de proteínas mitocondriales. Diferentes aproximaciones genéticas siguen desarrollándose y en varios modelos biológicos, los cuales abrirán las puertas hacia nuevos descubrimientos que sin duda alguna continuarán revolucionando la biomedicina y biogerontología.
AGRADECIMIENTOS
Los autores agradecen a la Dra. Martha Zentella de Piña, al Dr. Enrique Piña Garza, al Dr. Edgar Zenteno Galindo y al Departamento de Bioquímica de la Facultad de Medicina de la Universidad Nacional Autónoma de México por el apoyo que les brindaron durante la realización de este trabajo.
BIBLIOGRAFÍA
ALCENDOR RR, GAO S, ZHAI P, ZABLOCKI D, HOLLE E, YU X, et al. Sirt1 Regulates Aging and Resistance to Oxidative Stress in the Heart. Circ Res. 2007;100(10):1512-1521.
[ Links ]ANDERSON S, BANKIER AT, BARRELL BG, BRUIJN MHL DE, COULSON AR, DROUIN J, et al. Sequence and Organization of the Human Mitochondrial Genome. Nature. 1981;290(5806):457-465.
[ Links ]APARICIO O, BILLINGTON B, GOTTSCHLING D. Modifiers of Position Effect are Shared Between Telomeric and Silent Mating-type Loci in S. cerevisiae. Cell. 1991;66(6):1279-1287.
[ Links ]AVERY OT, MACLEOD CM, MCCARTY M. Studies on the Chemical Nature of the Substance Inducing Transformation of Pneumococcal Types. J Exp Med. 1944;79(2):137-158.
[ Links ]BAE NS, SWANSON MJ, VASSILEV A, HOWARD BH. Human Histone Deacetylase SIRT2 Interacts with the Homeobox Transcription Factor HOXA10. J Biochem. 2004;135(6):695-700.
[ Links ]BERDICHEVSKY A, VISWANATHAN M, HORVITZ H, GUARENTE L. C. Elegans SIR-2.1 Interacts with 14-3-3 Proteins to Activate DAF-16 and Extend Life Span. Cell. 2006;125(6):1165-1177.
[ Links ]BRACHMANN C, SHERMAN J, DEVINE S, CAMERON E, PILLUS L, BOEKE J. The SIR2 Gene Family, Conserved from Bacteria to Humans, Functions in Silencing, Cell Cycle Progression, and Chromosome Stability. Genes Dev. 1995;9(23):2888-2902.
[ Links ]BRAUNSTEIN M, ROSE A, HOLMES S, ALLIS C, BROACH J. Transcriptional Silencing in Yeast is Associated with Reduced Nucleosome Acetylation. Genes Dev. 1993;7(4):592-604.
[ Links ]BRIDGES C. Direct Proof Through Non-disjunction that the Sex-linked Genes of Drosophila are Borne by the X-chromosome. Science. 1914;40(1020):107-109.
[ Links ]CAPECCHI MR. The new mouse genetics: altering the genome by gene targeting. Trends Genet. 1989;5(3):70-76.
[ Links ]CHEN B, NELSON MM, SADOVSKY Y. N-myc down-regulated Gene 1 Modulates the Response of Term Human Trophoblasts to Hypoxic Injury. J Biol Chem. 2006;281(5):2764-2772.
[ Links ]DONMEZ G, WANG D, COHEN DE, GUARENTE L. SIRT1 Suppresses Beta-amyloid Production by Activating the Alpha-secretase Gene ADAM10. Cell. 2010;142(2):320-332.
[ Links ]FIRESTEIN R, BLANDER G, MICHÁN S, OBERDOERFFER P, OGINO S, CAMPBELL J, et al. The SIRT1 Deacetylase Suppresses Intestinal Tumorigenesis and Colon Cancer Growth. Plos one. 2008;3(4):e2020+.
[ Links ]FRANKLIN RE, GOSLING RG. Molecular Configuration in Sodium Thymonucleate. Nature. 1953;171(4356):740-741.
[ Links ]FRIEDMAN D, JOHNSON T. A Mutation in the Age-1 Gene in Caenorhabditis elegans Lengthens Life and Reduces Hermaphrodite Fertility. Genetics. 1988;118(1):75-86.
[ Links ]GARSKE AL, SMITH BC, DENU JM. Linking SIRT2 to Parkinson s Disease. ACS Chem Biol. 2007;2(8):529-532.
[ Links ]GOTTLIEB S, ESPOSITO R. A new role for a yeast transcriptional silencer gene, SIR2, in regulation of recombination in ribosomal DNA. Cell. 1989;56(5):771-776.
[ Links ]GREISS S, GARTNER A. Sirtuin/Sir2 Phylogeny, Evolutionary Considerations and Structural Conservation. Mol Cells. 2009;28(5):407-415.
[ Links ]HAAF T. Integration of Human -Satellite DNA Into Simian Chromosomes: Centromere Protein Binding and Disruption of Normal Chromosome Segregation. Cell. 1992;70(4):681-696.
[ Links ]HAIGIS MC, MOSTOSLAVSKY R, HAIGIS KM, FAHIE K, CHRISTODOULOU DC, MURPHY AJ, et al. SIRT4 Inhibits Glutamate Dehydrogenase and Opposes the Effects of Calorie Restriction in Pancreatic Cells. Cell. 2006;126(5):941-954.
[ Links ]HERSHEY AD, CHASE M. Independent Functions of Viral Protein and Nucleic Acid in Growth of Bacteriophage. J Gen Physiol. 1952;36(1):39-56.
[ Links ]HIRATSUKA M, INOUE T, TODA T, KIMURA N, SHIRAYOSHI Y, KAMITANI H, et al. Proteomics-based Identification of Differentially Expressed Genes in Human Gliomas: Down-regulation of SIRT2 Gene. Biochem Biophys Res Commun. 2003;309(3):558-566.
[ Links ]HIRSCHEY MD, SHIMAZU T, GOETZMAN E, JING E, SCHWER B, LOMBARD DB, et al. SIRT3 Regulates Mitochondrial Fatty-acid Oxidation by reversible Enzyme Deacetylation. Nature. 2010;464(7285):121-125.
[ Links ]JING E, GESTA S, KAHN RR. SIRT2 Regulates Adipocyte Differentiation Through FoxO1 Acetylation/Deacetylation. Cell Metab. 2007;6(2):105-114.
[ Links ]KAEBERLEIN M, MCVEY M, GUARENTE L. The SIR2/3/4 Complex and SIR2 Alone Promote Longevity in Saccharomyces cerevisiae by Two Different Mechanisms. Genes Dev. 1999;13(19):2570-2580.
[ Links ]KENYON CJ. The genetics of Ageing. Nature. 2010;464(7288):504-512.
[ Links ]KIM HSS, PATEL K, MULDOON-JACOBS K, BISHT KS, AYKIN-BURNS N, PENNINGTON JD, et al. SIRT3 is a Mitochondria-localized Tumor Suppressor Required for Maintenance of Mitochondrial Integrity and Metabolism During Stress. Cancer cell. 2010;17(1):41-52.
[ Links ]KLASS M. A Method for the Isolation of Longevity Mutants in the Nematode Caenorhabditis elegans and Initial Results. Mech Ageing Dev. 1983;22(3-4):279-286.
[ Links ]LANDRY J, SUTTON A, TAFROV S, HELLER R, STEBBINS J, PILLUS L, et al. The Silencing Protein SIR2 and its Homologs are NAD-dependent Protein Deacetylases. P Natl Acad Sci U S A. 2000;97(11):5807-5811.
[ Links ]LEVENE H. Genetic Equilibrium when More than One Ecological Niche is Available. Am Nat. 1953;87(836).
[ Links ]LOMBARD DB, ALT FW, CHENG H-L, BUNKENBORG J, STREEPER RS, MOSTOSLAVSKY R, et al. Mammalian Sir2 Homolog SIRT3 Regulates Global Mitochondrial Lysine Acetylation. Mol Cell Biol. 2007;27(24):8807-8814.
[ Links ]MAXAM AM, GILBERT W. A New Method for Sequencing DNA. Proc Natl Acad Sci U S A. 1977;74(2):560-564.
[ Links ]MCCAY CM, CROWELL MF, MAYNARD LA. The Effect of Retarded Growth Upon the Length of Life Span and Upon the Ultimate Body Size. Nutrition. 1935;10(1):63-79.
[ Links ]MCCUTCHEON JP, MCDONALD BR, MORAN NA. Origin of an Alternative Genetic Code in the Extremely Small and GC-Rich Genome of a Bacterial Symbiont. Plos Genet. 2009;5(7):e1000565+.
[ Links ]MENDEL G. Versuche über Plflanzenhybriden. Verhandlungen des naturforschenden Vereines in Brünn, Bd. IV für das Jahr 1865, Abhandlungen, 3-47.
[ Links ]MICHÁN L, MICHÁN S. El desarrollo de la biogerontología y geriatría de inicios del siglo XX a la actualidad. In: Gutiérrez Robledo LM, Gutiérrez Ávila JH, editor(s). México: Institutos Nacionales de Salud; 2010. p. 137-145.
[ Links ]MICHÁN S, LI Y, CHOU MM-HM, PARRELLA E, GE H, LONG JM, et al. SIRT1 is Essential for Normal Cognitive Function and Synaptic Plasticity. J Neurosci. 2010;30(29):9695-9707.
[ Links ]MICHÁN S, SINCLAIR D. Sirtuins in Mammals: Insights Into their Biological Function. Biochem J. 2007;404(1):1-13.
[ Links ]MICHÁN S. Biogerontología y mecanismos biológicos del envejecimiento. México: Instituto de Geriatria, Secretaria de Salud; 2010. p. 57-66.
[ Links ]MIESCHER F. Üeber die chemische Zusammensetzung der Eiterzellen. Med Chem Unters. 1871;4:486-501.
[ Links ]MORGAN T. Sex Limited Inheritance in Drosophila. Science. 1910;32(812):120-122.
[ Links ]MOSTOSLAVSKY R, CHUA KF, LOMBARD DB, PANG WW, FISCHER MR, GELLON L, et al. Genomic Instability and Aging-like Phenotype in the Absence of Mammalian SIRT6. Cell. 2006;124(2):315-329.
[ Links ]NAKAGAWA T, LOMB DJ, HAIGIS MC, GUARENTE L. SIRT5 Deacetylates Carbamoyl Phosphate Synthetase 1 and Regulates the Urea Cycle. Cell. 2009;137(3):560-570.
[ Links ]NORTH BJ, VERDIN E. Interphase Nucleo-cytoplasmic Shuttling and Localization of SIRT2 During Mitosis. Plos one. 2007;2(8):
[ Links ]OBERDOERFFER P, MICHÁN S, MCVAY M, MOSTOSLAVSKY R, VANN J, PARK S-K, et al. SIRT1 Redistribution on Chromatin Promotes Genomic Stability but Alters Gene Expression during Aging. Cell. 2008;135(5):907-918.
[ Links ]ØROM UA, DERRIEN T, BERINGER M, GUMIREDDY K, GARDINI A, BUSSOTTI G, et al. Long Noncoding RNAs with Enhancer-like Function in Human Cells. Cell. 2010;143(1):46-58.
[ Links ]PALACIOS OM, CARMONA JJ, MICHÁN S, CHEN KYY, MANABE Y, WARD JLL, et al. Diet and Exercise Signals Regulate SIRT3 and Activate AMPK and PGC-1alpha in Skeletal Muscle. Aging; 2009;1(9):771-783.
[ Links ]PELLICER J, FAY MF, LEITCH IJ. The Largest Eukaryotic Genome of Them All? Bot J Linn Soc. 2010;164(1):10-15.
[ Links ]PELTONEN L, MCKUSICK VA. Dissecting Human Disease in the Postgenomic Era. Science. 2001;291(5507):1224-1229.
[ Links ]PROUDFOOT N. PSEUDOGENES. Nature. 1980;286(5776):840-841.
[ Links ]RINE J, HERSKOWITZ I. Four Genes Responsible for a Position Effect on Expression from HML and HMR in Saccharomyces cerevisiae. Genetics. 1987;116(1):9-22.
[ Links ]RINE J, STRATHERN J, HICKS J, HERSKOWITZ I. A Suppressor of Mating-type Locus Mutations in Saccharomyces cerevisiae: Evidence for and Identification of Cryptic Mating-type Loci. Genetics. 1979;93(4):877-901.
[ Links ]ROBERTS L. The Human Genome. Controversial from the Start. Science. 2001;291(5507):1182-1188.
[ Links ]ROGINA B, HELFAND SL. Sir2 Mediates Longevity in the Fly Through a Pathway Related to Calorie Restriction. Proc Natl Acad Sci U S A. 2004;101(45):15998-16003.
[ Links ]ROYLE NJ, MÉNDEZ-BERMÚDEZ A, GRAVANI A, NOVO C, FOXON J, WILLIAMS J, et al. The Role of Recombination in Telomere Length Maintenance. Biochem Soc Trans. 2009;37(3):589-5
[ Links ]SANGER F, AIR GM, BARRELL BG, BROWN NL, COULSON AR, FIDDES JC, et al. Nucleotide sequence of bacteriophage X174 DNA. Nature. 1977;265(5596):687-695.
[ Links ]SANGER F, NICKLEN S, COULSON A. DNA Sequencing with Chain-terminating Inhibitors. Proc Natl Acad Sci U S A. 1977;74(12):5463-5467.
[ Links ]SHI T, WANG F, STIEREN E, TONG Q. SIRT3, a Mitochondrial Sirtuin Deacetylase, Regulates Mitochondrial Function and Thermogenesis in Brown Adipocytes. J Biol Chem. 2005;280(14):13560-13567.
[ Links ]SINCLAIR DA, GUARENTE L. Extrachromosomal rDNA Circles A Cause of Aging in Yeast. Cell. 1997;91(7):1033-1042.
[ Links ]SOMEYA S, YU W, HALLOWS WC, XU J, VANN JM, LEEUWENBURGH C, et al. Sirt3 Mediates Reduction of Oxidative Damage and Prevention of Age-Related Hearing Loss under Caloric Restriction. Cell. 2010;143(5):802-812.
[ Links ]STARK GR, GUDKOV AV. Forward Genetics in Mammalian Cells: Functional Approaches to Gene Discovery. Hum Mol Genet. 1999;8(10):1925-1938.
[ Links ]SUNDARESAN NR, GUPTA M, KIM G, RAJAMOHAN SB, ISBATAN A, GUPTA MP. Sirt3 Blocks the Cardiac Hypertrophic Response by Augmenting Foxo3a-dependent Antioxidant Defense Mechanisms in Mice. J Clin Invest. 2009;119(9):2758-2771.
[ Links ]SZYMANSKI M, BARCISZEWSKA M, ERDMANN V, BARCISZEWSKI J. A New Frontier for Molecular Medicine: Noncoding RNAs. Biochim Biophys Acta - Reviews on Cancer. 2005;1756(1):65-75.
[ Links ]TISSENBAUM HA, GUARENTE L. Increased Dosage of a Sir-2 Gene Extends Lifespan in Caenorhabditis elegans. Nature. 2001;410(6825):227-230.
[ Links ]TONG C, LI P, WU NL, YAN Y, YING Q-L. Production of p53 Gene Knockout Rats by Homologous Recombination in Embryonic Stem Cells. Nature. 2010;467(7312):211-213.
[ Links ]TSUKAMOTO Y, KATO J-ICHI, IKEDA H. Silencing Factors Participate in DNA Repair and Recombination in Saccharomyces cerevisiae. Nature. 1997;388(6645):900-903.
[ Links ]VAKHRUSHEVA O, SMOLKA C, GAJAWADA P, KOSTIN S, BOETTGER T, KUBIN T, et al. Sirt7 Increases Stress Resistance of Cardiomyocytes and Prevents Apoptosis and Inflammatory Cardiomyopathy in Mice. Circ Res. 2008;102(6):703-710.
[ Links ]VAQUERO A, SCHER M, LEE D, ERDJUMENT-BROMAGE H, TEMPST P, REINBERG D. Human SirT1 Interacts with Histone H1 and Promotes Formation of Facultative Heterochromatin. Mol Cell. 2004;16(1):93-105.
[ Links ]VAZIRI H, DESSAIN S, NG EATON E, IMAI S, FRYE R, PANDITA T,et al. hSIR2(SIRT1) Functions as an NAD-dependent p53 Deacetylase. Cell. 2001;107(2):149-159.
[ Links ]VENTER JC, ADAMS MD, MYERS EW, LI PW, MURAL RJ, SUTTON GG, et al. The Sequence of the Human Genome. Science. 2001;291(5507):1304-1351.
[ Links ]WATSON JD, CRICK FH. Molecular Structure of Nucleic Acids; a Structure for Deoxyribose Nucleic Acid. Nature. 1953;171(4356):737-738.
[ Links ]WEI M, FABRIZIO P, HU J, GE H, CHENG C, LI L, et al. Life Span Extension by Calorie Restriction Depends on Rim15 and Transcription Factors Downstream of Ras/ PKA, Tor, and Sch9. Plos Genet. 2008;4(1):
[ Links ]WILKINS MH, STOKES AR, WILSON HR. Molecular Structure of Deoxypentose Nucleic Acids. Nature. 1953;171(4356):738-740.
[ Links ]ZAN Y, HAAG JD, CHEN K-S, SHEPEL LA, WIGINGTON D, WANG Y-R, et al. Production of Knockout Rats Using ENU Mutagenesis and a Yeast-based Screening Assay. Nat Biotechnol. 2003;21(6):645-651.
[ Links ]ZHAO X, STERNSDORF T, BOLGER TA, EVANS RM, YAO T-PP. Regulation of MEF2 by Histone Deacetylase 4-and SIRT1 Deacetylase-mediated Lysine Modifications. Mol Cell Biol. 2005;25(19):8456-8464.
[ Links ]













