Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.16 no.3 Bogotá Sept./Dec. 2011
CÓDIGOS DE BARRAS DE LA VIDA: INTRODUCCIÓN Y PERSPECTIVA
DNA Barcode of Life: An Introduction and Perspective
ANDREA PAZ1, Bióloga; MAILYN GONZALEZ1, Ph. D.; ANDREW J. CRAWFORD1,2 , Ph. D. 1 Departamento de Ciencias Biológicas, Universidad de los Andes, A.A. 4976, Bogotá, Colombia.2 Instituto Smithsonian de Investigaciones Tropicales, Apartado 0843-03092, Balboa, Ancón, República de Panamá. Autor para comunicación: Andrea Paz, Instituto de Genética de Poblaciones y Filogeografía, M1-306, Departamento de Ciencias Biológicas, Universidad de los Andes, Carrera 1E n.º. 18A-10, A.A. 4976, Bogotá, Colombia. Teléfono: +57 1 339-4949 ext. 2794, Fax: +57 1 332-4069. a.paz263@uniandes.edu.co
Presentado 24 de febrero de 2011, aceptado 2 de mayo de 2011, correcciones 1 de julio de 2011.
RESUMEN
El término -Código de Barras de ADN- se basa en el uso de una región de ADN estandarizada, la cual sirve como una etiqueta para la identificación rápida y de especies. El propósito de un sistema de identificación más eficaz es facilitar la conservación, conocimiento y uso sustentable de la biodiversidad. Después de ocho años de discusión y producción en la literatura científica, el tema sigue generando controversia, debido en parte a la falta de homogeneidad en la definición y el alcance del método entre los autores. En este artículo enfatizamos la definición y metodología de los códigos de barra de ADN, así como su uso para contestar preguntas nuevas en los campos de la ecología, la evolución y la conservación.
Palabras clave: bases de datos, bioinformática, códigos de barras del ADN taxonómico, sistemática, genética, taxonomía.
ABSTRACT
DNA barcode of life is a global initiative to populate a database of short, standardized DNA fragments from most eukaryotic species to facilitate the identification of whole or fragmented biological samples. Increasing the ability of non-experts to indentify unknown samples should improve our collective understanding and management of the world s biodiversity. In the eight years following its proposal, DNA barcoding remains controversial in the scientific literature due, in part, to a lack of consensus on how DNA barcoding is defined and what activities are included under this rubric. In the present paper we review the definition, methods and goals of DNA barcoding, highlighting how the massive data sets generated by various DNA barcode of life campaigns may be applied to fundamental and novel questions in ecology, evolution and conservation.
Key words: Bioinformatics, Databases, DNA barcode, Genetics, Molecular diagnosis, Systematics, Taxonomy.
INTRODUCCIÓN
La biodiversidad comprende la variedad de ecosistemas, especies y genes (SCBD, 2011) y, debido al marcado efecto antropogénico sobre todos estos niveles de variación biológica, es una prioridad global acelerar su descripción y conservación. Se ha estimado que la tasa de extinción actual de especies es entre cien y mil veces superior a las tasas observadas durante los cincos episodios de extinción masiva que precedieron la aparición del ser humano (Pimm et al., 1995). Ante la acelerada pérdida de biodiversidad en todo el mundo debido al inadecuado uso de la tierra, la deforestación, el cambio climático, las especies invasoras y los patógenos emergentes, entre otras causas (Hoffmann et al., 2010), las medidas de gestión de los recursos naturales se han enfocado en acelerar el inventario de la diversidad biológica, de comprender su funcionamiento e integrar su uso en el desarrollo sostenible de la sociedad humana (SCBD, 2011). En este contexto, el año 2010 fue nominado como -Año Internacional de la Biodiversidad- por el Convenio de Diversidad Biológica (CDB), durante la cual se propuso hacer un llamado de atención a la comunidad con el fin de mejorar nuestro conocimiento, uso y valoración de la biodiversidad que nos rodea (SCBD, 2011).
El conocimiento taxonómico actual reconoce alrededor de 1,8 millones de especies entre los 10-15 millones de especies estimadas (May y Harvey, 2009). Esta cifra no es exacta ya que continuamente se están descubriendo y describiendo nuevas especies (Bisby et al., 2011). La falta de información sobre el número -exacto- de especies en el mundo se debe en parte a la falta de exploración en ciertas áreas del planeta, a la escasez de estudios taxonómicos en algunos grupos biológicos, y a la dificultad que representa la identificación morfológica de algunos organismos (i.e., la similitud morfológica que constituye la base de la taxonomía tradicional; Packer et al., 2009). En particular, la descripción taxonómica es incompleta en ciertos grupos como insectos y plantas (Blaxter, 2003). De por sí, la identificación de especímenes a partir de su morfología puede ser muy difícil para los investigadores, debido a problemas intrínsecos en ciertos grupos con plasticidad fenotípica, diferencias ontogénicas y dimorfismo sexual, entre otros (Hebert et al., 2003c; Packer et al., 2009). Existen, además, situaciones donde la identificación basada en caracteres morfológicos no puede ser aplicada. Entre los ejemplos más comunes podemos mencionar el caso de ciertos estadios larvales, fragmentos de tejido como restos de hojas, semillas, sangre, plumas, carne o pieles, los cuales no presentan suficientes caracteres diagnósticos para una acertada identificación (Eaton et al., 2010). Sumado a estas limitaciones, la identificación de especímenes por parte de expertos es de difícil acceso y puede requerir de mucho tiempo ya que existen pocos taxónomos especializados para cada grupo de organismos. No obstante, el tiempo necesario para identificar con certeza un espécimen es de gran importancia en situaciones como control de tráfico ilegal de especies en fronteras o la identificación de especies invasoras y plagas que pueden generar graves alteraciones ecológicas y económicas. En Colombia, el tráfico ilegal de especies es un problema persistente y es necesario que los especímenes sean identificados con rapidez y exactitud por parte de los agentes que realizan los controles.
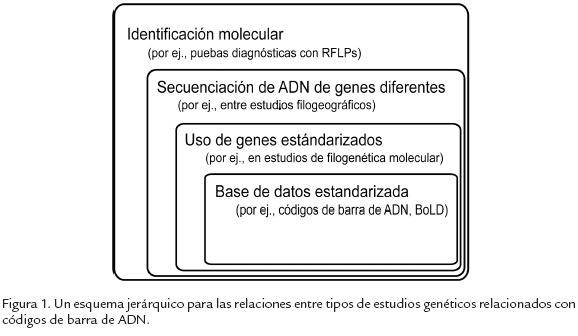
Desde hace más de tres décadas, la biología molecular se ha convertido en una herramienta valiosa en la identificación de individuos y especies y ha sido ampliamente aplicada para el estudio de las relaciones filogenéticas de organismos, lo cual ha tenido implicaciones dramáticas para la taxonomía (Fig. 1; Baker et al., 1996; Teletchea et al., 2008). Una ventaja de la información genética es que puede ser obtenida a partir de un pequeño fragmento del organismo. Dentro de las metodologías que en biología molecular permiten aproximarse a la identificación de individuos y/o de especies, los códigos de barras de ADN son un método de identificación y reconocimiento de especies que utiliza una base de datos de secuencias de ADN estandarizadas (Hebert et al., 2003c; Hebert et al., 2004b)
El uso de códigos de barras de ADN ha sido presentado como herramienta de identificación taxonómica de especímenes y como mecanismo para descubrir especies crípticas. Los códigos de barras de especímenes desconocidos que no concuerden con ninguna especie presente en la base de datos de referencia pueden sugerir que la base de datos está incompleta o que se trata de una posible nueva especie. Por otro lado los códigos de barras pueden revelar divergencia genética muy alta dentro de una misma especie putativa sugiriendo la existencia de especies crípticas.
La propuesta de utilizar códigos de barras de ADN como herramienta de identificación generó, desde sus inicios, gran controversia en la comunidad científica. El problema radicó en parte en la confusión que existe entre esta herramienta de identificación molecular y una propuesta distinta, la taxonomía molecular o -taxonomía por ADN- (Tautz et al., 2003; Blaxter, 2004). En la taxonomía por ADN, la clasificación de los organismos ya no dependería de características morfológicas (caracteres visibles) sino únicamente de las secuencias de ADN. Por ejemplo, en microbiología, la taxonomía por ADN es una metodología aceptada (Fraser et al., 2009), pero en la sistemática de eucariotas, este concepto ha sido poco bienvenido (Seberg et al., 2003). Ambas aproximaciones metodológicas, códigos de barra de ADN y taxonomía por ADN, proponen colectar secuencias de ADN que reflejen la variación intraespecífica de un gran número de especies en el mundo.
Bajo nuestro concepto (y tal vez el concepto más aceptado actualmente), el desarrollo de códigos de barra de ADN no excluye a la taxonomía que se basa en caracteres morfológicos diagnósticos, por el contrario, se presenta como una herramienta complementaria basada en técnicas de biología molecular para la correcta identificación de las especies (Padial y De la Riva, 2007).
CÓDIGOS DE BARRAS DE ADN
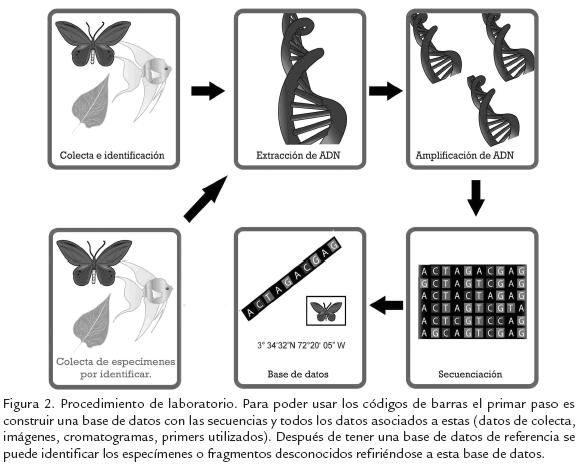
Los códigos de barras de ADN constituyen una herramienta diseñada para identificar de forma rápida y precisa las especies a partir de una corta y estándar secuencia de ADN (Fig. 2; Floyd et al., 2002; Hebert et al., 2003c). Para que una región del genoma pueda ser utilizada como código de barras de ADN es necesario que esta permita distinguir entre variación interespecífica (i.e., entre especies cercanamente relacionadas) y variación intraespecífica (i.e., dentro de la misma especie). De lo anterior, se genera el concepto de -barcoding gap-, que es la diferencia que existe entre la variación interespecífica e intraespecífica. Cuanto mayor sea la diferencia entre estas la discriminación específica será más acertada (Meyer y Paulay, 2005).
¿Qué región escoger como código de barras? La metodología de código de barras de ADN se propuso inicialmente para identificar animales a partir de un fragmento del gen mitocondrial citocromo c oxidasa subunidad 1 (COI), de aproximadamente 648 pares de bases (pb). La región COI presenta una alta tasa de sustitución, lo que se manifiesta en alta variación de la secuencia entre especies del mismo género (Hebert et al., 2003a; Hebert et al., 2003b; Luo et al., 2011). En los últimos ocho años, numerosos estudios han mostrado que para varios grupos de aves, peces, mamíferos, lepidópteros y otros insectos este fragmento COI presenta una variación interespecífica suficientemente amplia, permitiendo buena correspondencia entre la identificación molecular y la identificación basada en caracteres morfológicos de las especies (Hebert et al., 2003c; Hebert et al., 2004a; Ward et al., 2005; Borisenko et al., 2008). Sin embargo, algunos autores han sugerido que el solapamiento entre variación intraespecífica e interespecífica aumenta cuando se realizan muestreos con un mayor número de taxa emparentados, disminuyendo la fiabilidad del código de barras de ADN (Moritz y Cicero, 2004; Meyer y Paulay, 2005). En ciertos grupos, como anfibios, cuando varios individuos de una especie provienen de regiones geográficas distantes, su variación intraespecífica puede superar la variación interespecífica observada entre especies del mismo género, haciendo difícil la delimitación de especies con solo la secuencia de COI (Vences et al., 2005). No obstante, en otros grupos como lepidópteros y aves se ha demostrado que al integrar individuos y taxa de amplias regiones geográficas no se reduce el éxito de identificación basado en COI (Kerr et al., 2009; Lukhtanov et al., 2009). Si bien el código de barras basado en la secuencia del gen COI es apropiado para la mayoría de grupos de animales, existen dificultades en usar este gen para identificar especies de algunos grupos. COI parece evolucionar lentamente en cnidarios y esponjas, y como consecuencia la distancia genética entre especies cercanas es demasiado pequeña para permitir la identificación confiable a nivel de especie (Shearer et al., 2002; Hebert et al., 2003d; Wörheide, 2006; Huang et al., 2008). Por esta razón, en esponjas se ha sugerido secuenciar un fragmento más largo de COI e incluir el gen nuclear, Internal Transcribed Spacer (ITS). Para cnidarios, el gen mitocondrial 16S ha sido recomendado como marcador complementario al gen COI (Moura et al., 2008).
En el caso de plantas y de hongos COI no es utilizable como código de barras de ADN lo que ha llevado a buscar secuencias alternativas. En plantas la tasa de sustitución en la región COI es muy baja a diferencia de animales, lo cual, se traduce en la ausencia de variación a nivel de la secuencia entre especies del mismo género (Palmer y Herbon, 1988; Cowan et al., 2006). La búsqueda de un código de barras para plantas se enfocó en el genoma nuclear y plastídico y se concluyó que un método multilocus sería necesario en este grupo de organismos (Kress et al., 2005; Cowan et al., 2006). En el 2009, después de un largo debate, el CBOL (Consortium for the Barcode of Life, Plant Working Group et al., 2009), aprobó a las regiones plastídicas matK y rbcL como regiones barcode para las plantas. Ambas regiones son < 800 pb y han sido ampliamente utilizadas en estudios de filogenia por lo cual tienen una importante representación en las bases de referecia. Sin embargo, algunos estudios han mostrado que estos marcadores, aun combinados, no presentan una variación suficiente para diferenciar especies cercanas en algunas familias de Angiospermas por lo cual se ha propuesto la utilización de otros marcadores como la región transgénica psbA-trnH, el intron trnL y la región nuclear ITS para estos casos (Kress et al., 2005; Taberlet et al., 2007; Gonzalez et al., 2009; Yao et al., 2010). En el caso de plantas esta metodología tiene especial importancia por la dificultad en la identificación taxonómica de algunas especies de plantas medicinales debido a su similitud morfológica que impide su completa caracterización genética y química y así dificulta el control de calidad de los fitomedicamentos y el aprovechamiento racional y sustentable de los recursos genéticos. En el caso de los hongos, se ha sugerido el uso del ITS y la Large subunit de ADNr (LSU) como código de barras (Seifert, 2009; Eberhardt, 2010; Jargeat et al., 2010). Aún no hay consenso sobre el marcador estándar para códigos de barras de ADN en hongos (Begerow et al., 2010) pero se encuentra en preparación un propuesta al CBOL sugiriendo el uso del ITS o de ITS y LSU (Eberhardt, 2010).
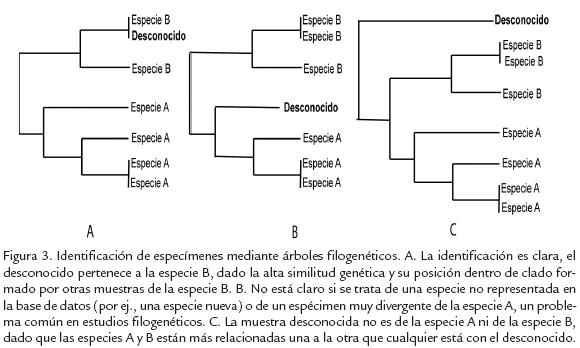
Procedimiento de identificación. Asumiendo que existe una base de datos de referencia de los códigos de barras ADN suficientemente completa entonces debemos definir cómo se identifica un espécimen a partir de su secuencia código de barras ADN. Existen varias propuestas metodológicas (Goldstein y DeSalle, 2011) para analizar los datos provenientes de un proyecto de códigos de barras de ADN, sin embargo, aquí describimos únicamente las tres más frecuentes. Una primera aproximación es la identificación basada en distancia o divergencia genética, la cual está determinada por el porcentaje de sitios nucleotídicos que varían entre una secuencia desconocida y las secuencias que se encuentran en la base de datos de referencia, corregida con el modelo de sustitución nucleotídica Kimura 2 parámetros (Kimura, 1980). Diversos trabajos han encontrado valores umbrales de las distancias genéticas en diferentes taxa, para sugerir que la secuencia nueva proviene de una especie distinta (Hebert et al., 2004a). Por ejemplo, en el caso de aves y lepidópteros se propuso que una divergencia entre secuencias de COI mayor a 2% indica la posible presencia de especies distintas (Hebert et al., 2004a; Janzen et al., 2005). La segunda aproximación posible es la identificación por medio de árboles filogenéticos (Fig. 3); en este caso la identificación es dada por la posición de la secuencia desconocida en una filogenia, basado en su cercanía evolutiva con las otras secuencias de ese grupo monofilético (Elias et al., 2007). La última aproximación frecuentemente usada consiste en identificar uno o más caracteres moleculares cuyo estado sea de sinapomorfía (carácter nuevo compartido por dos o más individuos), permitiendo un diagnóstico de especies por estados derivados de nucleótidos (Sass et al., 2007; Rach et al., 2008). Una ventaja de este método es que estos caracteres usados en la identificación podrían formar también una parte de la descripción primaria de especies nuevas (Goldstein y DeSalle, 2011).
Podemos esperar ver en el futuro más aplicaciones de modelos explícitamente evolutivos a los datos de códigos de barras para la identificación de muestras y para un tema relacionado: la delimitación de especies. Un método nuevo propone identificar grupos o clusters de códigos de barras de ADN por medio de análisis coalescentes, con el propósito de detectar cambios significativos en las tasas de coalescencia a través de una genealogía de códigos de barras (Pons et al., 2006). Aunque este método también es controversial (Lohse, 2009; Papadopoulou et al., 2009), vale la pena investigar y desarrollar más métodos estadísticos y evolutivos con aplicaciones al análisis de códigos de barras (Nielsen y Matz, 2006).
APLICACIONES DE LOS CÓDIGOS DE BARRAS DE ADN MÁS ALLÁ DE LA IDENTIFICACIÓN DE ESPECIES
Múltiples disciplinas como genética de poblaciones (Craft et al., 2010), evolución y ecología (Valentini et al., 2008) se benefician actualmente de la disponibilidad de los datos moleculares. Por ejemplo, Kerr et al., 2009 basados en los códigos de barras de COI compararon el tiempo de divergencia entre las especies de aves del Neotrópico y Norte América. Los códigos de barras permitieron concluir en este caso particular que la biodiversidad del Neotrópico no parece ser explicada por radiaciones recientes. En ecología, una de las aplicaciones más interesantes es la posibilidad de analizar la dieta de diferentes animales a partir de su contenido estomacal o de sus heces (Valentini et al., 2008). De esta manera, se identificaron la planta huésped de la cual se alimentaban 76 especies de escarabajos y Valentini et al., 2008 describieron el régimen alimenticio de la marmota (Marmota caudata) y el oso (Ursus arctos) presentes en el parque Dusai en Pakistán. La posibilidad de identificar qué comen los animales es crucial para entender el funcionamiento de los ecosistemas y el papel que juegan las especies en las redes tróficas (Soininen et al., 2009).
La aplicación de códigos de barras ha abierto nuevas posibilidades de estudios principalmente en sistemas altamente biodiversos como son las comunidades tropicales (Kress y Erickson, 2008). Por un lado, la integración de esta metodología a los inventarios de diversidad y en complemento con la información morfológica y ecológica ha permitido por ejemplo, el descubrimiento de nuevas especies de lepidópteros en Costa Rica (Janzen et al., 2009). Por otro lado, la posibilidad de inventariar grandes áreas de bosque tropical permite abordar preguntas acerca de la ecología de comunidades como por ejemplo evaluar la estructura filogenética de las especies en coexistencia. En este sentido, Gonzalez et al., 2009 y Kress et al., 2005 mostraron como a partir de secuencias código de barras era posible establecer la filogenia de una comunidad local o regional y estudiar en qué medida los procesos de filtro ambiental y competencia moldean las comunidades vegetales de un bosque en la Guyana Francesa y en Panamá.
En el bosque tropical en particular, la colecta de datos de códigos de barras a nivel de comunidades permitirá homogenizar las identificaciones y cuantificar con mayor precisión los cambios en la estructura y diversidad de las mismas. Por ejemplo, los códigos de barras pueden ser útiles para comprender el cambio en la composición de las comunidades debido a fenómenos como el cambio climático (Kress y Erickson, 2008; Dick y Kress, 2009). Los códigos de barras han permitido también cuantificar el efecto de una epidemia que ha causado una mortalidad masiva en anfibios, a pesar de que la taxonomía de los miembros de la comunidad estaba incompleta (Crawford et al., 2010). La disponibilidad de datos masivos de ADN que permitan identificar especímenes y grupos que necesitan estudios taxonómicos más detallados puede además ayudar a generar un mejor manejo de la biodiversidad y de los planes de conservación (Neigel et al., 2007; Francis et al., 2010; Vernooy et al., 2010).
Desde un punto de vista económico, una de las aplicaciones más importantes de los códigos de barras de ADN se presenta en el manejo de pestes, especies invasoras y en el control del tráfico ilegal de especies. Por ejemplo durante un inventario en aguas del Pacifico de Canadá fue detectada la presencia del alga Gracilaria vermiculophylla, una especie invasora cuya plasticidad fenotípica dificulta su reconocimiento a partir de caracteres morfológicos (Saunders, 2009). Otro ejemplo de la utilidad de los códigos de barras es el estudio de Crawford et al., 2011 donde usaron códigos de barras para identificar una nueva rana invasora en la Ciudad de Panamá. Recientemente, el estudio de Eaton et al., 2010 mostró que con secuencias de COI era posible identificar vertebrados objeto de tráfico ilegal en África y América del Sur a partir de material manufacturado (p.e. pieles, bolsos, correas). De esta forma, el código de barras de ADN se convierte en una herramienta con aplicaciones legales y utilidad directa para los agentes que regulan el comercio de especies o de partes de ellas.
LIMITACIONES DE LOS CÓDIGOS DE BARRAS DE ADN
Una de las mayores críticas realizadas al código de barras de ADN es que se basa en la información proveniente de un solo marcador o grupo de ligamiento genético (mitocondrial en animales y plastídico en plantas). El uso de uno o varios genes provenientes de un genoma haploide puede afectar la identificación correcta de una especie. Estos problemas se pueden dar en casos como sorteo incompleto de linajes por radiaciones recientes (Elias et al., 2007), la incorporación de genes de una población genéticamente distinta o introgresión (Liu et al., 2010), o presencia de pseudogenes (Rubinoff, 2006). En el caso de introgresión se ha visto que el ADN mitocondrial (ADNmt) de una especie puede reemplazar completamente el de otra especie como es el caso de Drosophila yakuba y D. santomea (Llopart et al., 2005). Se ha reportado también la posibilidad de coamplificar la región deseada junto con duplicaciones génicas (Campbell y Barker, 1999), pseudogenes generados por integración de este marcador molecular en el genoma nuclear (Zhang y Hewitt, 1996; Bensasson et al., 2001; Calvignac et al., 2011) o con fragmentos heteroplásticos (presencia de copias mutadas en un mismo individuo; Frey y Frey, 2004). En artrópodos en particular, una infección por la bacteria endosimbiótica, Wolbachia, puede causar una barrida selectiva que puede eliminar la variabilidad del ADNmt causando una incongruencia marcada en la historia genealógica del genoma nuclear y el ADNmt (Raychoudhury et al., 2010). El investigador usando datos de códigos de barras de ADN tiene que tomar en cuenta todas estas limitantes y revisar estas posibles fuentes de error antes de llegar a sus conclusiones.
Sin importar el marcador utilizado, un gran problema para la aplicación de los códigos de barras de ADN es disponer de una base de datos de referencia completa. Por ejemplo, no se puede identificar un criminal solo por sus huellas digitales ni por su ADN si esta persona no se encuentra en una base de datos de referencia. De la misma manera, a pesar de la posibilidad de obtener ADN de unas botas de piel de cocodrilo, no se podría identificar de qué especie de cocodrilo proviene si no existe una base pública de referencia de datos genéticos de las diferentes especies de cocodrilo. A pesar de los resultados satisfactorios en muchos taxa para dilucidar la diagnosis de especies usando código de barras de ADN, lamentablemente, hasta el momento no existe una o un conjunto de regiones que permitan la identificación de todos los grupos conocidos de animales o plantas (Hebert et al., 2004a; Kerr et al., 2007; Roy et al., 2010), y puede ser que todavía estemos subestimando la cantidad de especies que existen (Hickerson et al., 2006).
CONSTRUCCIÓN DE UNA BASE DE DATOS DE REFERENCIA GLOBAL
La identificación de especies a partir de códigos de barras requiere de una gran base de datos de referencia que permita asignar secuencias de procedencia desconocida a secuencias de organismos previamente descritos con una alta probabilidad de acierto (Hajibabaei et al., 2005; Ratnasingham y Hebert, 2007). La base de datos proporciona el enlace clave entre una secuencia de ADN de origen no-conocido y el mundo de especímenes, literatura taxonómica y los datos asociados de colecta y de laboratorio. Es por esto que los primeros esfuerzos para establecer los códigos de barras de la vida deben estar encaminados a recopilar estas secuencias de códigos de barras para especies ya conocidas.
Para llenar una biblioteca de referencia en códigos de barra de ADN, se necesitan no solo fragmentos de ADN sino que cada secuencia provenga de un espécimen -voucher- depositado en una colección de historia natural que permita una mejor identificación de los organismos por taxónomos expertos (Ratnasingham y Hebert, 2007; Goldstein y DeSalle, 2011). La biblioteca de referencia tiene que estar disponible para otros investigadores y el público en general. Una base de datos extensa que contenga una gran cantidad de información y que sea de fácil acceso implica además la estandarización de los datos. Esta no se refiere solo a la secuencia código de barras sino a la forma como se registran los datos de colecta y laboratorio para poder ser compartidos (Hajibabaei et al., 2005; Ratnasingham y Hebert, 2007). Con el propósito de ordenar, almacenar y compartir estos datos fue creada la base de datos de Barcode of Life Data Systems o BoLD (Hajibabaei et al., 2005; Ratnasingham y Hebert, 2007). BoLD es una base de datos pública que recopila los siete elementos esenciales de los códigos de barras de ADN que son: la información taxonómica, los datos del voucher, la información de colecta incluyendo datos geográficos, el identificador del espécimen, la secuencia de COI de mínimo 500 pb, los primers y los cromatogramas (datos crudos de ADN que provienen del proceso de secuenciación automática por el método de Sanger; Ratnasingham y Hebert, 2007). Además, permite incluir fotografías de los especímenes más secuencias de otros marcadores con sus respectivos primers y cromatogramas. Esta base de datos permite no solo ensamblar los datos requeridos para la generación de códigos de barras de ADN sino que además incluye diversas herramientas para el manejo y análisis de los mismos (Ratnasingham y Hebert, 2007).
Alrededor del mundo se han generado múltiples iniciativas para obtener grandes cantidades de códigos de barras y construir así una base de datos de referencia cada vez más amplia. Una de las iniciativas más grande es denominada International Barcode of Life o iBOL la cual pretende recopilar 5 millones de códigos de barra de 500.000 especies en cinco años. El iBOL fue lanzado oficialmente en 2010 y comprende 25 países cuya participación varía según su posición como nodos principales, regionales o nacionales. De los países de América Latina, México, Brasil y Argentina actúan como nodos regionales, y como nodos nacionales están participando Panamá, Costa Rica y Perú. Por su parte Ecuador también está recopilando grandes cantidades de códigos de barras. En Colombia, varias universidades e institutos han venido compilando de manera independiente cantidades modestas de códigos de barras de ADN en organismos como mosquitos, anfibios, lepidópteros, plantas y hongos. Existe actualmente una voluntad a nivel nacional de unir esfuerzos y consolidarnos como un nodo nacional de iBOL, de manera que se puedan generar de forma más eficiente y concertada los códigos de barras de la biodiversidad del país. No obstante, en Colombia, la iniciativa de códigos de barras de ADN se ve limitada principalmente por el acceso a los permisos para recolectar especímenes y para acceder a recursos genéticos, así como por la necesidad de obtener financiación que apoye este tipo de iniciativas. A nivel nacional existe también el debate acerca de la importancia de publicar los datos a nivel nacional, de manera paralela a su publicación en bases de referencia internacionales. Países como México pasaron por discusiones similares y optaron por crear un espejo de BoLD para mantener una copia de los datos en su país sin crear una base de datos aislada ni redundante.
No todos los estudios genéticos de biodiversidad pueden ser considerados códigos de barras de ADN (Fig. 1). La característica más importante de los códigos de barras de ADN es que son estándares y los datos son de acceso libre. Aunque el éxito de los códigos de barras de ADN no ha sido igual en todos los grupos taxonómicos, en general las campañas actuales han sido exitosas en la generación de grandes cantidades de datos y en la caracterización de la biodiversidad. Además, de la identificación de especímenes, la generación de cantidades masivas de datos genéticos estandarizados, tiene el potencial de revolucionar los estudios ecológicos y evolutivos de fauna y flora, sobre todo en comunidades con una alta riqueza. Con o sin códigos de barras, el nuevo siglo está convirtiéndose en el siglo de la genética, la genómica y la metagenómica. Esperamos que en Colombia las entidades gubernamentales, académicas y privadas involucradas en el manejo y estudio de la altísima diversidad del país tomen decisiones adecuadas para que Colombia sea un líder en la aplicación de nuevas técnicas y aproximaciones para el estudio de la biodiversidad.
AGRADECIMIENTOS
Gracias al Departamento de Biología, Facultad de Ciencias de la Universidad Nacional de Colombia, Sede Bogotá por la invitación a participar en la Cátedra de Sede José Celestino Mutis: -Todo lo que usted quiere saber de genética y nunca se atrevió a preguntar.- Gracias a S. V. Flechas y dos revisores anónimos por sus valiosos comentarios sobre el manuscrito.
BIBLIOGRAFÍA
BAKER CS, CIPRIANO F, PALUMBI S R. Molecular Genetic Identification Of Whale and Dolphin Products from Commercial Markets in Korea and Japan. Mol Ecol. 1996;5(5):671-685.
[ Links ]BEGEROW D, NILSSON H, UNTERSEHER M, MAIER W. Current State and Perspectives of Fungal DNA Barcoding and Rapid Identification Procedures. Appl Microbiol Biotechnol. 2010;87(1):99-108.
[ Links ]BENSASSON D, ZHANG D-X. HARTL DL, HEWITT G M. Mitochondrial Pseudogenes: Evolution s Misplaced Witnesses. Trends Ecol Evol. 2001;16(6):314-321.
[ Links ]BISBY FA, ROSKOV YR, ORRELL TM, NICOLSON D, PAGLINAWAN LE, BAILLY, N. et al. Species 2000 & ITIS Catalogue of Life: 2011 Annual Checklist, Digital Resource at www.catalogueoflife.org/annual-checklist/2011/. 2011.
[ Links ]BLAXTER M. Molecular Systematics: Counting Angels with DNA. Nature. 2003;421(6919):122-124.
[ Links ]BLAXTER ML. The Promise of a DNA Taxonomy. Philos Trans R Soc Lond B Biol Sci 2004;359(1444):669-679.
[ Links ]BORISENKO AV, LIM BK, IVANOVANV, HANNER RH, HEBERT PDN. DNA Barcoding in Surveys of Small Mammal Communities: A Field Study in Suriname. Mol Ecol Resour. 2008;8(3):471-479.
[ Links ]CALVIGNAC S, KONECNY L, MALARD F, DOUADY CJ. Preventing the Pollution of Mitochondrial Datasets with Nuclear Mitochondrial Paralogs (numts). Mitochondrion. 2011;11(2):246-254.
[ Links ]CAMPBELL NJ, BARKER SC. The Novel Mitochondrial Gene Arrangement of the Cattle Tick, Boophilus microplus: Fivefold Tandem Repetition of a Coding Region. Mol Biol Evol. 1999;16(6):732-740.
[ Links ]COWAN R S, CHASE MW, KRESS WJ, SAVOLAINEN V. 300,000 Species to Identify: Problems, Progress, and Prospects in DNA Barcoding of Land Plants. Taxon. 2006;55:611-616.
[ Links ]CRAFT KJ, PAULS SU, DARROW K, MILLER SE, HEBERT PDN, HELGEN LE et al. Population Genetics of Ecological Communities with DNA Barcodes: An Example from New Guinea Lepidoptera. Proc Natl Acad Sci USA. 2010:107(11):5041-5046.
[ Links ]CRAWFORD AJ, ALONSO R, JARAMILLO CA, SUCRE S, IBÁÑEZ R. DNA Barcoding Identifies a Third Invasive Species of Eleutherodactylus (Anura: Eleutherodactylidae) in Panama City, Panama. Zootaxa 2011; En prensa.
[ Links ]CRAWFORD AJ, LIPS KR, BERMINGHAM E. Epidemic Disease Decimates Amphibian Abundance, Species Diversity, and evolutionary History in the Highlands of Central Panama. Proc Natl Acad Sci USA. 2010;107(31):13777-13782.
[ Links ]DICK CW, KRESS WJ. Dissecting Tropical Plant Diversity with Forest Plots and a Molecular Toolkit. BioScience. 2009;59(9):745-755.
[ Links ]EATON M, MEYERS G, KOLOKOTRONIS S-O, LESLIE M, MARTIN A, AMATO G. Barcoding Bushmeat: Molecular Identification of Central African and South American Harvested Vertebrates. Conserv Genet. 2010;11(4):1389-1404.
[ Links ]EBERHARDT UA Constructive Step Towards Selecting a DNA Barcode for Fungi. New Phytol. 2010;187(2):265-268.
[ Links ]ELIAS M, HILL RI, WILLMOTT KR, DASMAHAPATRA KK., BROWER AVZ, MALLET J. et al. Limited Performance of DNA Barcoding in a Diverse Community of Tropical Butterflies. Proc R Soc B. 2007;274(1627):2881-2889.
[ Links ]FLOYD R, ABEBE E, PAPERT A, BLAXTER M. Molecular Barcodes for Soil Nematode Identification. Mol Ecol. 2002;11(4):839-850
[ Links ]FRANCIS CM, BORISENKO AV, IVANOVA NV, EGER JL, LIM BK, GUILLÉN-SERVENT A. et al. The Role of DNA Barcodes in Understanding and Conservation of Mammal Diversity in Southeast Asia. PLoS One. 2010;5(9):e12575
[ Links ]FRASER C, ALM EJ, POLZ MF, SPRATT BG, HANAGE WP. The Bacterial Species Challenge: Making Sense of Genetic and Ecological Diversity. Science. 2009;323(5915):741-746.
[ Links ]FREY JE, FREY B. Origin of Intra-individual Variation in MR-amplified Mitochondrial Cytochrome Oxidase I of Thrips tabaci (Thysanoptera: Thripidae): Mitochondrial Heteroplasmy or Nuclear Integration? Hereditas. 2004;140(2):92-98.
[ Links ]GOLDSTEIN PZ, DESALLE R. Integrating DNA Barcode Data and Taxonomic Practice: Determination, Discovery, and Description. Bioessays. 2011;33(2):135-147.
[ Links ]GONZALEZ MA, BARALOTO C, ENGEL J, MORI SA, PÉTRONELLI P, RIÉRA B. et al. Identification of Amazonian Trees with DNA Barcodes. PLoS One. 2009;4(10):e7483.
[ Links ]HAJIBABAEI M, DEWAARD JR, IVANOVA NV, RATNASINGHAM S, DOOH R. T, KIRK SL. et al. Critical Factors for Assembling a High Volume of DNA Barcodes. Philos T RoySoc B. 2005;360(1462):1959-1967.
[ Links ]HEBERT P, CYWINSKA A, BALL S, DEWAARD J. Barcode of Life: Identifying Species with DNA Barcoding Biological identifications through DNA barcodes. Proc R Soc Lond B Biol Sci. 2003a;270:313-321.
[ Links ]HEBERT P, RATNASINGHAM S, DEWAARD J. Barcoding Animal Life: Cytochrome c Oxidase Subunit 1 Divergences Among Closely Related Species. Proc R Soc Lond B Biol Sci. 2003b;270:S96S99.
[ Links ]HEBERT PDN, CYWINSKA A, BALL SL, DEWAARD JR. Biological Identifications through DNA Barcodes. Proc R Soc Lond B Biol Sci. 2003c;270(1512):313-321.
[ Links ]HEBERT PDN, RATNASINGHAM S, DE WAARD JR. Barcoding Animal Life: Cytochrome c Oxidase Subunit 1 Divergences Among Closely Related Species. Proc R Soc B. 2003d;270(Sl 1):S96-S99.
[ Links ]HEBERT PDN, STOECKLE MY, ZEMLAK TS, FRANCIS C. M. Identification of Birds through DNA Barcodes. PLoS Biol. 2004a;2(10):e312.
[ Links ]HEBERT PDN, STOECKLE MY, ZEMLAK TS, FRANCIS C. M. Identification of Birds through DNA Barcodes. PLoS Biol. 2004b;2(10):e312.
[ Links ]HICKERSON M, MEYER C, MORITZ C. DNA Barcoding Will Often Fail to Discover New Animal Species Over Broad Parameter Space. Syst Biol. 2006;55:729-739.
[ Links ]HOFFMANN M, HILTON-TAYLOR C, ANGULO A, BOHM M, BROOKS TM, BUTCHART SHM. et al. The Impact of Conservation on the Status of the World s Vertebrates. Science. 2010;330(6010):1503-1509.
[ Links ]HUANG D, MEIER R, TODD P, CHOU LM. Slow Mitochondrial COI Sequence Evolution at the Base of the Metazoan Tree and Its Implications for DNA Barcoding. J Mol Evol. 2008;66(2):167-174.
[ Links ]JANZEN DH, HAJIBABAEI M, BURNS JM, HALLWACHS W, REMIGIO E, HEBERT PDN. Wedding Biodiversity Inventory of a Large and Complex Lepidoptera fauna with DNA Barcoding. Philos Trans R Soc Lond B Biol Sci. 2005;360(1462):1835-1845.
[ Links ]JANZEN DH, HALLWACHS W, BLANDIN P, BURNS JM, CADIOU J-M, CHACON I. et al. Integration of DNA Barcoding Into an Ongoing Inventory of Complex Tropical Biodiversity. Mol Ecol Resour. 2009;9(s1):1-26.
[ Links ]JARGEATP, MARTOS F, CARRICONDE F, GRYTA H, MOREAU P-A, GARDES M. Phylogenetic Species Delimitation in Ectomycorrhizal Fungi and Implications for Barcoding: The Case of the Tricholoma scalpturatum complex (Basidiomycota). Mol Ecol. 2010;19(23):5216-5230.
[ Links ]KERR KCR, LIJTMAER DA, BARREIRA AS, HEBERT PDN, TUBARO PL. Probing Evolutionary Patterns In Neotropical Birds Through DNA Barcodes. PloS One. 2009;4(2):e4379.
[ Links ]KERR KCR, STOECKLE MY, DOVE CJ, WEIGT LA, FRANCIS CM, HEBERT PDN. Comprehensive DNA Barcode Coverage of North American Birds. Mol Ecol Notes. 2007;7(4):535-543.
[ Links ]KIMURA M. A Simple Method for Estimating Evolutionary Rate of Base Substitutions Through Comparative Studies of Nucleotide Sequences. J Mol Evol. 1980;16:111-120
[ Links ]KRESS WJ, ERICKSON DL. DNA Barcoding-a Windfall for Tropical Biology? Biotropica. 2008;40(4):405-408.
[ Links ]KRESS WJ, WURDACK KJ, ZIMMER EA, WEIGT LA, JANZEN DH. Use of DNA Barcodes to Identify Flowering Plants. Proc Natl Acad Sci USA. 2005;102(23):8369-8374.
[ Links ]LIU K, WANG F, CHEN W, TU L, MIN,M-S, BI K. et al. Rampant Historical Mitochondrial Genome Introgression Between Two Species of Green Pond Frogs, Pelophylax nigromaculatus and P. plancyi. BMC Evolutionary Biology. 2010;10(1):201.
[ Links ]LLOPART A, LACHAISE D, COYNE JA. Multilocus Analysis of Introgression Between Two Sympatric Sister Species of Drosophila: D. yakuba and D. santomea. Genetics. 2005;171:197-210.
[ Links ]LOHSE K. Can mtDNA Barcodes Be Used to Delimit Species? A Response to Pons et al. (2006). Syst Biol. 2009;58(4):439-442.
[ Links ]LUKHTANOV VA, SOURAKOV A, ZAKHAROV EV, HEBERT PDN. DNA Barcoding Central Asian Butterflies: Increasing Geographical Dimension does not Significantly Reduce the Success of Species Identification. Mol Ecol Resour. 2009;9(5):1302-1310.
[ Links ]LUO A, ZHANG A, HO S, XU W, ZHANG Y, SHI W. et al. Potential Efficacy of Mitochondrial Genes for Animal DNA Barcoding: A Case Study Using Eutherian Mammals. BMC Genomics. 2011;12(1):84.
[ Links ]MAY RM, HARVEY PH. Species Uncertainties. Science. 2009;323(5915):687-687.
[ Links ]MEYER CP, PAULAYG. DNA Barcoding: Error Rates Based on Comprehensive Sampling. PLoS Biol. 2005;3(12):e422.
[ Links ]MORITZ C, CICEROC. DNA Barcoding: Promise and Pitfalls. PLoS Biol. 2004;2(10):e354.
[ Links ]MOURA CJ, HARRIS DJ, CUNHA MR, ROGERS AD. DNA Barcoding Reveals Cryptic Diversity in Marine Hydroids (Cnidaria, Hydrozoa) from Coastal and Deep-sea Environments. Zool Scr. 2008;37(1):93-108.
[ Links ]NEIGEL J, DOMINGO A, STAKE J. DNA Barcoding as a Tool for Coral Reef Conservation. Coral Reefs. 2007;26(3):487-499.
[ Links ]NIELSEN R, MATZ M. Statistical Approaches for DNA Barcoding. Syst Biol. 2006;55(1):162-169.
[ Links ]PACKER L, GIBBS J, SHEFFIELD C, HANNER R. DNA Barcoding and the Mediocrity of Morphology. Mol Ecol Resour. 2009;9:42-50.
[ Links ]PADIAL JM, DE LA RIVA I. Integrative Taxonomists Should Use and Produce DNA Barcodes. Syst Biol. 2007;1586:67-68.
[ Links ]PALMER J, HERBON L. Plant Mitochondrial DNA Evolved Rapidly in Structure, but Slowly in Sequence. J Mol Evol. 1988;28(1):87-97.
[ Links ]PAPADOPOULOU A, MONAGHAN MT, BARRACLOUGH TG, VOGLER AP. Sampling Error does not Invalidate the Yule-coalescent Model for Species Delimitation. A Response to Lohse (2009). Syst Biol. 2009;58(4):442-444.
[ Links ]PIMM SL, RUSSELL GJ, GITTLEMAN JL, BROOKS TM. The Future of Biodiversity. Science. 1995;269(5222):347-350.
[ Links ]PONS J, BARRACLOUGH TG, GOMEZ-ZURITA J, CARDOSO A, DURAN DP, HAZELL S. et al. Sequence-based Species Delimitation for the DNA Taxonomy of Undescribed Insects. Syst Biol. 2006;55(4):595-609.
[ Links ]RACH J, DESALLE R, SARKAR IN, SCHIERWATER B, HADRYS H. Character-based DNA Barcoding Allows Discrimination of Genera, Species and Populations in Odonata. Proc R Soc B. 2008;275(1632):237-247.
[ Links ]RATNASINGHAM S, HEBERT PDN. BoLD: The Barcode of Life Data System (http://www.barcodinglife.org). Mol Ecol Notes. 2007;7(3):355-364.
[ Links ]RAYCHOUDHURY R, GRILLENBERGER BK, GADAU J, BIJLSMA R, VAN DE ZANDE L, WERREN JH, et al. Phylogeography of Nasonia vitripennis (Hymenoptera) Indicates a Mitochondrial-Wolbachia Sweep in North America. Heredity. 2010;104(3):318-326.
[ Links ]ROY S, TYAGI A, SHUKLA V, KUMAR A, SINGH UM, CHAUDHARY LB, et al. Universal Plant DNA Barcode Loci May Not Work in Complex Groups: A Case Study with Indian Berberis Species. PloS One. 2010;5(10):e13674.
[ Links ]RUBINOFF D. Utility of Mitochondrial DNA Barcodes in Species Conservation. Conserv Biol. 2006;20(4):1026-1033.
[ Links ]SASS C, LITTLE DP, STEVENSON DW, SPECHT CD. DNA Barcoding in the Cycadales: Testing the Potential of Proposed Barcoding Markers for Species Identification of Cycads. PloS One. 2007;2(11):e1154.
[ Links ]SAUNDERS GW. Routine DNA Barcoding of Canadian Gracilariales (Rhodophyta) Reveals the invasive Species Gracilaria vermiculophylla in British Columbia. Mol Ecol Resour. 2009;9:140-150.
[ Links ]SCBD. Convention on Biological Diversity. [en línea] 2011 [citado 13 Feb 2011]; Disponible en: URL: http://www.cbd.int/
[ Links ]SEBERG O, HUMPHRIES CJ, KNAPP S, STEVENSON DW, PETERSEN G, SCHARFF N, et al. Shortcuts in Systematics? A Commentary on DNA-based Taxonomy. Trends Ecol Evol. 2003;18(2):63-65.
[ Links ]SEIFERT KA. Progress Towards DNA Barcoding of Fungi. Mol Ecol Resour. 2009;9:83-89.
[ Links ]SHEARER TL, VAN OPPEN MJH, ROMANO SL, WÖRHEIDE G. Slow Mitochondrial DNA sequence Evolution in the Anthozoa (Cnidaria). Mol Ecol. 2002;11(12):2475-2487.
[ Links ]SOININEN E, VALENTINI A, COISSAC E, MIQUEL C, GIELLY L, BROCHMANN C, et al. Analysing Diet of Small Herbivores: The Efficiency of DNA Barcoding Coupled with High-throughput Pyrosequencing for Deciphering the Composition of Complex Plant Mixtures. Front Zool. 2009;6(1):16.
[ Links ]TABERLET P, COISSAC E, POMPANON FO, GIELLY L, MIQUEL C, VALENTINI A, et al. Power and Limitations of the Chloroplast trnL (UAA) Intron for Plant DNA Barcoding. Nucleic Acids Res. 2007;35(3):e14-e14.
[ Links ]TAUTZ D, ARCTANDER P, MINELLI A, THOMAS RH, VOGLER AP. A Plea for DNA Taxonomy. Trends Ecol Evol. 2003;18(2):70-74.
[ Links ]TELETCHEA F, BERNILLON J, DUFFRAISSE M, LAUDET V, HÄNNI C. Molecular Identification of Vertebrate Species by Oligonucleotide Microarray in Food and Forensic Samples. J Appl Ecol. 2008;45(3):967-975.
[ Links ]VALENTINI A, POMPANON F, TABERLET P. DNA Barcoding for Ecologists. Trends Ecol Evol. 2008;24(2):110-117.
[ Links ]VENCES M, THOMAS M, VAN DER MEIJDEN A, CHIARI Y, VIEITES DR. Comparative Performance of the 16S rRNA Gene in DNA Barcoding of Amphibians. Front Zool. 2005;2(1):5.
[ Links ]VERNOOY R, HARIBABU E, MULLER MR, VOGEL JH, HEBERT PDN, SCHINDEL DE, et al. Barcoding Life to Conserve Biological Diversity: Beyond the Taxonomic Imperative. PLoS Biol. 2010;8(7):e1000417.
[ Links ]WARD RD, ZEMLAK TS, INNES BH, LAST PR, HEBERT PDN. DNA Barcoding Australia s Fish Species. Philos Trans R Soc Lond B Biol Sci. 2005;360:1847-1857.
[ Links ]WÖRHEIDE G. Low Variation in Partial Cytochrome Oxidase Subunit I (COI) Mitochondrial Sequences in the Coralline Demosponge Astrosclera willeyana Across the Indo-Pacific. Mar Biol. 2006;148(5):907-912.
[ Links ]YAO H, SONG J, LIU C, LUO K, HAN J, LI Y, et al. Use of ITS2 Region as the Universal DNA Barcode for Plants and Animals. PloS One. 2010;5(10):e13102.
[ Links ]ZHANG D-X, HEWITT GM. Nuclear Integrations: Challenges for Mitochondrial DNA markers. Trends Ecol Evol. 1996;11(6):247-251.
[ Links ]













