Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.17 no.1 Bogotá Jan./Apr. 2012
FILOGEOGRAFÍA COMPARADA: CONCEPTOS, MÉTODOS Y PATRONES GENERALES EN AVES NEOTROPICALES
Comparative Phylogeography: Concepts, Methods and General Patterns in Neotropical Birds
ENRIQUE ARBELÁEZ-CORTÉS1, M.Sc.
1 Museo de Zoología, Departamento de Biología Evolutiva, Facultad de Ciencias y Posgrado en Ciencias Biológicas, Universidad Nacional Autónoma de México. Ciudad Universitaria 04510, México. enriquearbelaez@gmail.com
Presentado 17 de agosto de 2011, aceptado 7 de febrero de 2012, correcciones 8 de febrero de 2012.
RESUMEN
Entender los patrones y procesos de diversificación de linajes intraespecíficos en el tiempo y en el espacio es el objetivo de la filogeografía. La comparación de estos patrones filogeográficos entre especies co-distribuidas muestra indicios de la historia de una comunidad. Aquí, reviso los conceptos y la metodología de la filogeografía comparada, un campo muy activo pero con métodos de análisis heterogéneos. Para dar un marco de referencia de la filogeografía en el Neotrópico, comento los patrones filogeográficos generales de las aves de esta región. La revisión de más de 100 estudios realizados en los últimos 25 años indica que los patrones filogeográficos de cada especie a pesar de coincidir en ciertos puntos con el de otras especies co-distribuidas tienen aspectos idiosincrásicos que indican que su historia es única.
Palabras clave: aves, comunidades, filogeografia, neotrópico.
ABSTRACT
Understanding the patterns and processes involved in intraspecific lineages diversification in time and space is the aim of phylogeography. The comparison of those phylogeographic patterns among co-distributed species shows insights of a community history. Here I review the concepts and methodologies of comparative phylogeography, an active research field that has heterogeneous analytical methods. In order to present a framework for phylogeography in the Neotropics, I comment the general phylogeographic patterns of the birds from this region. This review is based on more than 100 studies conducted during the last 25 years and indicate that despite different co-distributed species seem to share some points in their phylogeographic pattern they have idiosyncratic aspects, indicating an unique history for each one.
Key words: Birds, communities, Neotropics, phylogeography.
INTRODUCCIÓN
Un resultado reiterado al estudiar la variación de caracteres en las poblaciones de una especie es que, tal variación, no es geográficamente aleatoria; sino que tiene estructura, permitiendo ver la evolución en su nivel espacial más básico (Gould y Johnston, 1972). En los estudios de filogeografía (Avise et al., 1987) se utilizan secuencias de ADN de varios individuos de una especie para reconstruir las relaciones históricas de sus poblaciones y luego interpretarlas geográficamente, enfatizando en la influencia de eventos geológicos o paleoecológicos que asocien la historia de esa especie con la de su ambiente (Riddle, 1996; Avise, 1998; Arbogast y Kenagy, 2001). La filogeografía, aunque con sesgo hacia los animales, ha crecido rápidamente y se ha puesto al frente de estudios microevolutivos (Avise, 2000; Soltis et al., 2006; Beheregaray, 2008; Hickerson et al., 2010) usando nuevos métodos y enfoques (Garrick et al., 2010; Hickerson et al., 2010; Chan et al., 2011).
La filogeografía comparada estudia los patrones filogeográficos de varias especies codistribuidas de un modo cualitativo o usando métodos explícitos, muy heterogéneos, que vale la pena revisar (e.g. Bermingham y Avise, 1986; Sullivan et al., 2000; Zink, 2002; Cartens et al., 2005; Lapointe y Rissler, 2005; Carnaval et al., 2009). El mismo término también se ha usado para denominar otro tipo de estudios que comparan los patrones filogeográficos de especies muy relacionadas pero alopátricas (e.g. Pastorini et al., 2003; Albach et al., 2006; Chapple et al., 2008) que no consideraré aquí. La filogeografía comparada ha permitido visualizar patrones y proponer procesos relacionados con el origen de la diversidad en zonas de alta riqueza de especies como el Neotrópico (Solomon et al., 2008; Carnaval et al., 2009) y en grupos taxonómicos como las aves que tiene linajes con historias naturales dispares (Burney y Brumfield, 2009; Weir, 2009). A pesar de que las aves han sido el grupo de vertebrados menos estudiado desde una perspectiva filogeográfica (Beheregaray, 2008) los patrones que han mostrado ejemplifican la ocurrencia de diversos procesos evolutivos en su historia reciente, algo muy relevante para entender la diversidad presente en zonas como el Neotrópico (e.g., Cadena, 2007; Milá et al., 2009; González et al., 2011). En esta revisión mis objetivos son: 1) exponer los conceptos fundamentales de la filogeografía comparada y su marco metodológico y 2) revisar el estado del conocimiento de la filogeografía de las aves Neotropicales, comentando algunos de sus patrones más generales.
MATERIALES Y MÉTODOS
Para esta revisión realicé una búsqueda del término comparative phylogeography en el contenido de varias revistas científicas tales como: Molecular Phylogenetics and Evolution, Molecular Ecology, Evolution, Biological Journal of the Linnean Society y Journal of Biogeography. Examiné los resultados de estas búsquedas, seleccionando los artículos que trataran directamente de estudios de filogeografía comparada e hice énfasis en trabajos con vertebrados terrestres. Registros adicionales fueron obtenidos de la literatura citada en estos artículos así como de búsquedas puntuales en otras revistas. Adicionalmente, con el fin de mostrar un panorama de la filogeografía de aves neotropicales, compilé todos los artículos de filogeografía que encontré para este grupo de vertebrados llevados a cabo en dicha zona, incluyendo algunos trabajos de filogeografía comparada. El Neotrópico lo defino aquí como la mitad norte de Sur América, Centro América y la mitad sur de México y la búsqueda de trabajos la limito a especies continentales cuya distribución esté total o principalmente dentro de esta zona. Como resultado obtuve 94 artículos sobre filogeografía comparada y 30 trabajos de filogeografía en aves Neotropicales publicados durante 25 años (1986 mayo de 2011).
FILOGEOGRAFÍA COMPARADA: FUNDAMENTOS CONCEPTUALES
De acuerdo con los artículos revisados, la filogeografía comparada busca congruencias en los patrones filogeográficos y en los procesos demográficos de especies que comparten, total o parcialmente, sus distribuciones geográficas, y contrasta tales patrones con información geográfica, paleoecológica o biogeográfica (Bermingham y Avise, 1986; Zink, 1991; Joseph et al., 1995; Avise, 2000; Arbogast y Kenagy, 2001; Zink, 2002; Lapointe y Rissler, 2005; Carstens y Richards, 2007; Garrick et al., 2008; Castoe et al., 2009). Esta búsqueda de congruencia se fundamenta en la idea de que las especies que solapan sus distribuciones tienen una historia compartida (Bermingham y Avise, 1986; Zink, 2002; Lapointe y Rissler, 2005; Morrone, 2009). Implícita en esta interpretación está la asunción de que las especies co-distribuidas actualmente debieron estarlo también en el pasado (Carstens y Richards, 2007), por lo cual se esperaría que los mismos eventos ecológicos o geológicos hayan afectado en sincronía a esas especies, haciendo que las historias de sus poblaciones sean semejantes. Bajo este escenario, al reconstruir la historia de las especies de una región, usando métodos filogeográficos, se debería recuperar: 1) congruencia topológica, 2) congruencia temporal y 3) congruencia demográfica. A continuación comento cada uno de estos tipos de congruencia.
La congruencia topológica implica que las especies de un área geográfica respondieron concertadamente ante los mismos eventos históricos por lo que deben tener patrones filogeográficos semejantes (Sullivan et al., 2000). La congruencia topológica se consideró como la hipótesis fundamental a poner a prueba en filogeografía comparada, siendo la incongruencia explicada por diferencias ecológicas entre especies (Joseph et al., 1995; Turner et al., 1996; Zink, 1996; Rocha et al., 2002; Crawford et al., 2007; Qu et al., 2010). Sin embargo, varios trabajos muestran que la incongruencia topológica es frecuente (e.g. Turner et al., 1996; Kelemen y Moritz, 1999; Rocha et al., 2002; Lourie et al., 2005; Roberts, 2006; Bell et al., 2007; Kirchman y Franklin, 2007; Guzik et al., 2009; Moussalli et al., 2009; Qu et al., 2010) y la evidencia paleontológica indica que las comunidades pueden variar de un modo no concertado (Bennett, 1990; Barnosky y Shabel, 2005). La interpretación de la congruencia topológica en presencia de barreras geográficas conspicuas es sencilla mientras que la incongruencia plantea el reto de identificar la razón de las diferencias entre especies. Actualmente se sabe que la incongruencia topológica puede originarse por diferencias entre especies en cuanto a: selección natural, tasas de mutación molecular, tamaño efectivo poblacional, capacidad de dispersión, diferencias sutiles entre distribuciones, factores demográficos, extinciones locales (Avise, 1998; Rocha et al., 2002; Cartens et al., 2005; Steele y Storfer, 2007; Garrick et al., 2008) o por la aleatoriedad implícita en el proceso de coalescencia de los genes (Hugall et al., 2002; Garrick et al., 2008). También, se ha propuesto la idea (interesante pero poco explorada) de que la incongruencia topológica indica que no hubo simpatría histórica entre especies (Zink, 1996; Zink, 2002). Actualmente, la dicotomía congruencia-incongruencia topológica se considera simplista, y las semejanzas en los patrones filogeográficos de especies simpátricas son vistos como puntos a lo largo de un gradiente complejo cuyos extremos son: congruencia total e incongruencia absoluta (Sullivan et al., 2000; Zink, 2002; Carstens y Richards, 2007; Steele y Storfer, 2007; Garrick et al., 2008), siendo el grado de congruencia un indicativo de la estabilidad histórica de la comunidad estudiada (Zink, 2002).
La congruencia temporal en la división de linajes intraespecíficos entre especies simpátricas también la estudia la filogeografía comparada y su identificación se ha potenciado con el refinamiento de métodos de dataje usando información molecular (Bermingham y Avise, 1986; Avise, 1992; Cartens et al., 2005; Barber y Klicka, 2010). Su fundamento es que si dos especies comparten su patrón filogeográfico es porque un mismo evento histórico las afectó al mismo tiempo. Sin embargo, patrones filogeográficos similares pueden resultar de diferentes eventos asincrónicos (Avise, 1992; Barber et al., 2006; Soltis et al., 2006; Barber y Klicka, 2010); lo cual es conocido como pseudocongruencia (Soltis et al., 2006). La pseudocongruencia indica que las especies pueden responder independientemente a eventos similares repetidos en un mismo lugar. Finalmente, la congruencia demográfica puede buscarse analizando indicios de cambios poblacionales históricos debidos, por ejemplo, a que varias especies aumentaron su tamaño poblacional al expandirse por una nueva área tras un cambio ambiental (Hewitt, 2000, 2001, 2004; Lessa et al., 2003, 2010). Esta congruencia puede encontrarse, incluso, entre especies que no presenten estructura filogeográfica (Zink, 2002).
FILOGEOGRAFÍA COMPARADA: MÉTODOS
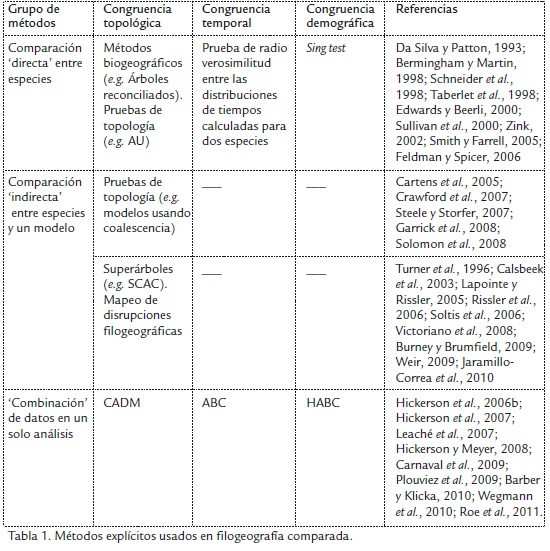
Tradicionalmente la identificación de congruencia en filogeografía comparada se ha hecho mediante inspección 'visual' o cualitativa de los resultados de análisis filogeográficos independientes para cada especie (Zink, 2002; Cartens et al., 2005; Carstens y Richards, 2007). A pesar de ser subjetivo (Sullivan et al., 2000; Soltis et al., 2006), este método se encuentra tácito en casi todos los estudios de filogeografía comparada (e.g. Bermingham y Avise, 1986; Kelemen y Moritz, 1999; Zink et al., 2001; Dawson et al., 2002; Hugall et al., 2002; Rocha et al., 2002; Dooh et al., 2006; Barber et al., 2006; Roberts, 2006; Taylor y Hellberg, 2006; Bell et al., 2007; Kirchman y Franklin, 2007; Crandall et al., 2008; Castoe et al., 2009; Guzik et al., 2009; Qu et al., 2010; Beatty y Provan, 2011; Oshida et al., 2011; Saeki et al., 2011) y solo algunos autores indican su uso (Schneider et al., 1998; Schäuble y Moritz, 2001; Sullivan et al., 2000). Tal vez las comparaciones 'a ojo' siguen vigentes dada la dificultad (o ¿imposibilidad?) de formalizar procesos históricos como la evolución. No obstante, existen métodos explícitos que dan rigor a la filogeografía comparada (Tabla 1.). Para explicar sus generalidades agrupé estos métodos considerando el modo como usan la información filogeográfica y siguiendo el orden de ideas de Garrick et al. (2008). A continuación comento su uso para identificar cada tipo de congruencia en filogeografía comparada.
La congruencia topológica puede examinarse directamente usando cladogramas de áreas (Da Silva y Patton, 1993; Bermingham y Martin, 1998; Taberlet et al., 1998; Schneider et al., 1998; Sullivan et al., 2000; Zink, 2002; Feldman y Spicer, 2006), que se generan sustituyendo las ramas terminales del cladograma de cada especie por la localidad/región del individuo al que corresponde. Estos cladogramas se comparan, o combinan, usando métodos biogeográficos tales como: parsimonia de Brooks (Taberlet et al., 1998), análisis de componentes (Bermingham y Martin, 1998), árboles reconciliados (Bermingham y Martin, 1998), análisis de descomposición (Schneider et al., 1998) o tree mapping (Feldman y Spicer, 2006; Sullivan et al., 2000) para generar un cladograma general de áreas; que es un consenso que muestra congruencias y diferencias entre especies. Aunque explícitos, estos métodos pierden utilidad cuando hay demasiada incongruencia topológica (Sullivan et al., 2000). Otra forma de 'medir' la congruencia topológica es ajustando el patrón filogeográfico de una especie en el de otra (Bermingham y Martin, 1998; Sullivan et al., 2000; Steele y Storfer, 2007) con métodos estadísticos como: Kishino-Hasegawa-Templeton test (Templeton, 1983; Kishino y Hasegawa, 1989), bootstrap paramétrico (Efron y Tibshirani, 1993), ShimodairaHasegawa test (Shimodaira, 2002) o approximately unbiased test (Shimodaira y Hasegawa, 2001). Estos métodos miden diferencias de valores de 'optimalidad' entre un árbol óptimo (generado para una especie) y un árbol ajustado a la hipótesis que se está examinando (el de otra especie) usando una distribución nula para derivar una probabilidad estadística (Sullivan et al., 2000). Así se obtiene un estimador para la congruencia entre topologías junto con la probabilidad de que, este, sea mayor al esperado de comparar árboles al azar. Una aproximación similar usa el patrón de estructura genética de una especie o una estructura hipotética (ya no la topología) para evaluar los datos de otra especie, usando un análisis molecular de varianza (AMOVA), y determinar si ese agrupamiento explica bien los resultados (Schäuble y Moritz, 2001; Lourie et al., 2005). Sin embargo, estos métodos solo permiten comparaciones pareadas entre especies y a pesar de haber sido usados en comunidades de vertebrados de diferentes regiones (Bermingham y Martin, 1998; Schneider et al., 1998; Sullivan et al., 2000; Feldman y Spicer, 2006) no han indicado congruencia topológica total.
Una manera 'indirecta' de buscar congruencia topológica es comparando los patrones filogeográficos de cada especie contra una topología hipotética (modelo); que es la esperada si las especies de un área hubieran sido afectadas por eventos históricos conocidos (Cartens et al., 2005; Crawford et al., 2007; Steele y Storfer, 2007; Garrick et al., 2008; Solomon et al., 2008). La congruencia entre los datos de cada especie y el modelo indica cuales especies se ajustan a la historia conocida del área y por lo tanto comparten sus patrones filogeográficos. Varios modelos pueden ser propuestos para representar diferentes historias (Cartens et al., 2005). El ajuste de los datos al modelo se hace usando las pruebas comentadas en el párrafo anterior y otras basadas en inferencia Bayesiana (Steele y Storfer, 2007) o en coalescencia (Knowles y Maddison, 2002). Un ejemplo de esta aproximación es el de Carstens y colaboradores (Cartens et al., 2005) quienes encontraron que diferentes especies en un bosque templado de Estados Unidos podían asignarse a uno de tres modelos alternativos propuestos a partir de información ad hoc de la historia de esa comunidad. A pesar de su utilidad estos modelos podrían sesgarse por las expectativas del investigador. Para disminuir tal sesgo se puede usar una aproximación que combina modelos de nicho ecológico (ENM) proyectados geográficamente en diferentes tiempos (condiciones paleoclimáticas) que al ser visualizados mediante sistemas de información geográfica permiten proponer modelos menos subjetivos (Chan et al., 2011).
Para evitar sesgos por parte del investigador al buscar congruencia topológica también se pueden 'aglomerar', en un solo análisis, todos los patrones filogeográficos mediante un superárbol regional que sintetiza la señal filogeográfica de cada especie (Lapointe y Rissler, 2005). Si no hay congruencia topológica entre especies el superárbol no tendrá ningún patrón. Existen varios métodos para hacer superárboles (Bininda-Emonds, 2004). En filogeografía comparada se han usado tres: MRP (matrix representation with parsimony; Lapointe y Rissler, 2005; Victoriano et al., 2008), SAC (supertree area cladogram) y SCAC (sequence concatenation area cladogram; Weir, 2009). En general se hace lo siguiente: Se diseña una matriz codificando la presencia y ausencia de los linajes de cada especie sobre una subdivisión del área de estudio (e.g. subregiones o una gradilla) y se analiza con parsimonia para encontrar el superárbol. También puede hacerse un superárbol (poco ortodoxo) acoplando secuencias de ADN de diferentes especies de una misma subregión y haciendo un análisis filogenético (SCAC; Weir, 2009) donde los terminales son las subregiones o los cuadros de la gradilla. Una ventaja de los superárboles es que no requieren que todas las especies sean totalmente simpátricas (Lapointe y Rissler, 2005), pero solo muestran patrones generales. Estos métodos se han implementado en regionalizaciones que buscan identificar áreas de endemismo (Weir, 2009) y zonas de 'diversificación activa' (Lapointe y Rissler, 2005) con fines de conservación.
Identificar la existencia de barreras geográficas compartidas entre especies es otra manera de buscar congruencia topológica. Esto se hace identificando divisiones filogeográficas, en el espacio geográfico, para cada especie (e.g. SAMOVA o Barrier; Dupanloup et al., 2002; Manni et al., 2004) cuya superposición en un mapa muestra las zonas con divisio- nes compartidas (Jaramillo-Correa et al., 2010). También se puede comparar el grado de diferenciación genética entre poblaciones, de varias especies, a través de barreras geográficas predefinidas (Turner et al., 1996; Burney y Brumfield, 2009; Weir, 2009) o no (Calsbeek et al., 2003; Soltis et al., 2006), para observar patrones compartidos. Un método reciente llamado congruence among distance matrices (CADM; Campbell et al., 2011; Roe et al., 2011) permite identificar congruencia global entre varias especies y, a posteriori , congruencia entre pares de especies usando matrices de distancia genética y de distancia geográfica al modo de un test de Mantel extendido. Otro método basado en el uso de sistemas de información geográfica permite dar mayor detalle a la representación espacial de la diferenciación genética entre localidades/poblaciones usando la superposición de 'paisajes genéticos', de especies co-distribuidas, creados a partir de un algoritmo de interpolación espacial y una matriz de distancias genéticas (Vandergast et al., 2010).
Vale anotar que estos métodos se basan en distancias genéticas y no en patrones filogeográficos, por lo que carecen de la señal histórica que caracteriza a la filogeografía (Avise, 1992; Templeton, 1998; Avise, 2000; Templeton et al., 2000). Este aspecto histórico puede integrarse poniendo en un mapa la ubicación de las divergencias topológicas más evidentes de cada especie (Rissler et al., 2006).
La congruencia temporal ha sido evaluada estimando tiempos de divergencia para los linajes de cada especie y examinando, luego, si los datajes de diferentes especies están dentro de un periodo semejante (e.g. inicios del Pleistoceno; Schäuble y Moritz, 2001; Wares y Cunningham, 2001; Calsbeek et al., 2003; Mateos, 2005; Castoe et al., 2009). Como esta aproximación no tiene rigor estadístico Edwards y Beerli, 2000, propusieron usar la distribución de valores alrededor del valor máximo de verosimilitud que se obtiene usando métodos bayesianos de dataje molecular y así detectar la congruencia temporal entre especies. La comparación de estas distribuciones entre especies se hace con una prueba de radio-verosimilitud que indica si el tiempo de divergencia de las poblaciones de dos especies es significativamente diferente o no. Sin embargo, existen parámetros que varían entre las especies comparadas como son: el tamaño poblacional ancestral, la migración post-divergencia, la heterogeneidad en tasas evolutivas y la subdivisión en la población ancestral; que dan incertidumbre al dataje de divergencia reciente (Edwards y Beerli, 2000). Por esto Hickerson y colaboradores (Hickerson et al., 2006a; Hickerson et al., 2006b; Hickerson et al., 2007) presentan un método que considera estos parámetros al poner a prueba la hipótesis de divergencia temporal simultánea entre dos poblaciones de varias especies a través de una misma barrera geográfica. El método emplea aproximate bayesian computation (ABC) y simulaciones basadas en coalescencia. Este método básicamente lo que hace es calcular hiperparámetros que describen procesos entre las especies y subparámetros que permiten que haya variación demográfica en cada especie (Hickerson et al., 2006a; Hickerson et al., 2006b; Hickerson et al., 2007; Csilléry et al., 2010). Luego simula una serie de datos bajo un modelo (e.g. divergencia sincrónica) usando coalescencia y permitiendo variaciones en el tamaño poblacional. Con los datos simulados calcula pseudoparámetros que reiteradamente se comparan con los parámetros de los datos reales y se aceptan o rechazar según un criterio de cercanía. Con la muestra de pseudoparámetros se construye la distribución a posteriori del hiperparámetro deseado que en este caso es el número de eventos de vicarianza. Lo que destaca al ABC, sobre otros métodos, es que permite el análisis de múltiples grupos de datos filogeográficos simultáneamente, considerando diferencias demográficas entre taxones (Carnaval et al., 2009). Su implementación ha mostrado que entre especies afectadas por una misma barrera geográfica no ha ocurrido un único evento simultáneo de vicarianza (Hickerson et al., 2006b; Leaché et al., 2007; Plouviez et al., 2009; Barber y Klicka, 2010; Chan et al., 2011).
Por último, en cuanto a la congruencia demográfica, los análisis demográficos por lo general indican crecimiento (+) o disminución (-) en el tamaño poblacional de una especie (Kuhner, 2008), de modo que cualquier sesgo significativo hacia una de las dos posibilidades indicaría congruencia demográfica entre especies. La identificación de un sesgo crecimiento/disminución puede hacerse usando el sign test, una prueba sencilla comparable a las tablas de contingencia (Smith y Farrell, 2005). Otro modo de poner a prueba hipótesis explicitas que involucran aspectos demográficos entre especies es usar hierarchical approximate bayesian computation (HABC) un método similar al ABC, que puede integrar múltiples parámetros (e.g. migración) para crear modelos evolutivos más flexibles (Hickerson y Meyer, 2008; Carnaval et al., 2009; Chan et al., 2011). Aunque la congruencia demográfica es la menos estudiada en filogeografía comparada, su detección ha indicado procesos que han afectado comunidades enteras (Wares y Cunningham, 2001; Lessa et al., 2003; Smith y Farrell, 2005; Crandall et al., 2008; Garrick et al., 2008; Lessa et al., 2010). La identificación de esta congruencia en combinación con otros métodos fue parte de un elegante estudio con fines de conservación en el bosque Atlántico brasilero (Carnaval et al., 2009).
GENERALIDADES DEL DISEÑO DE LOS ESTUDIOS DE FILOGEOGRAFÍA COMPARADA
Vale la pena decir que uno o varios de estos métodos los encontré implementados en solo el 20% de los trabajos de filogeografía comparada que revisé (n = 94). A pesar de que la filogeografía comparada aun tiene un sesgo hacia lo descriptivo los problemas que ha abordado son variados. Los estudios se han realizado con: entre una especie (contraintuitivo, pero es el caso de una especie de salmón con poblaciones simpátricas alocrónicas; Churikov y Gharrett, 2002) y 55 especies (Calsbeek et al., 2003) en el caso de revisiones de varios trabajos para una región, siendo incluidas en la mayoría de estudios entre dos y cuatro especies. Llama la atención que estos trabajos han incluido desde especies muy relacionadas filogenéticamente (Turner et al., 1996; Dawson et al., 2002; Feldman y Spicer, 2006) hasta taxones muy diferentes (Taberlet et al., 1998; Calsbeek et al., 2003; Cartens et al., 2005). En cada extremo de esta disparidad taxonómica los autores argumentan diferente, diciendo que en los estudios de filogeografía comparada hay que limitar taxonómica y ecológicamente las especies estudiadas para poder encontrar patrones compartidos (Feldman y Spicer, 2006) o que mientras más disparidad exista las congruencias encontradas tendrán mayor significado (Taberlet et al., 1998; Cartens et al., 2005). Geográficamente, los estudios van desde locales (Garrick et al., 2008; Guzik et al., 2009) hasta continentales (Zink, 1996; Joseph y Wilke, 2007; Weir, 2009). También hay trabajos estudiando interacciones ecológicas marcadas que a pesar de ser ejemplos de coevolución no han mostrado la congruencia filogeográfica esperada entre esas especies (e.g. Parker et al., 2004; Richards et al., 2007; Roe et al., 2011). A pesar de que las secuencias de ADN han sido la fuente principal de datos, se han incluido también otros marcadores moleculares como AFLPs y microsatélites como parte de la filogeografía comparada (e.g. Calsbeek et al., 2003; Guzik et al., 2009; Maliouchenko et al., 2007), haciendo posible una aproximación integrativa a varios 'niveles temporales' (Garrick et al., 2008; Garrick et al., 2010; Wegmann et al., 2010). Adicionalmente, éstos estudios se han complementado usando reconstrucciones paleoecológicas, principalmente modelos de nicho ecológico proyectados en condiciones ambientales del pasado (Joseph et al., 1995; Hugall et al., 2002; Bell et al., 2007; Carstens y Richards, 2007; Solomon et al., 2008; Carnaval et al., 2009; Moussalli et al., 2009; Beatty y Provan, 2011), aunque es necesario indicar que existen limitaciones de esta última aproximación (Nogués-Bravo, 2009)
FILOGEOGRAFÍA DE AVES NEOTROPICALES: UN EJEMPLO DE PATRONES GENERALES EN LA HISTORIA DE UNA BIOTA DIVERSA
A pesar de que encontré 30 estudios filogeográficos que incluyen 29 especies pertene- cientes a diez familias y cinco órdenes de aves (Brawn et al., 1996; Marks et al., 2002; González et al., 2003; García-Moreno et al., 2004; Lovette, 2004; Solórzano et al., 2004; Cheviron et al., 2005; Aleixo, 2006; Cabanne et al., 2007; Cadena et al., 2007; Chaves et al., 2007; Nyari, 2007; Bonaccorso et al., 2008; Cabanne et al., 2008; Cortés-Rodríguez et al., 2008a,b; Miller et al., 2008; Navarro-Sigüenza et al., 2008; Puebla-Olivares et al., 2008; Weir et al., 2008; Caparroz et al., 2009a; Caparroz et al., 2009b; Milá et al., 2009; Sanín et al., 2009; Vázquez-Miranda et al., 2009; Arbeláez-Cortés et al., 2010; Pérez-Emán et al., 2010; Cadena et al., 2011; D'Horta et al., 2011; González et al., 2011), en la mayoría de casos la distribución de estas especies no coincide, impidiendo hacer un análisis filogeográfico comparativo de todas ellas. No obstante existen cinco estudios comparativos (Bates et al., 2003; Bates et al., 2004; Burney y Brumfield, 2009; Weir, 2009; Barber y Klicka, 2010) que incluyen especies adicionales pero aunque su muestreo no es intensivo (ver más adelante) si abordan de manera explícita la filogeografía comparada de las aves. Por consiguiente, me limitaré a comentar cualitativamente estos trabajos y a presentar algunos de los patrones filogeográficos generales que parecen emerger para la avifauna Neotropical.
En estos estudios el número de individuos incluidos varía entre 21 y 160. Todos los estudios han usado secuencias de ADNmt, y únicamente dos incluyeron también secuencias nucleares (Cabanne et al., 2008; D'Horta et al., 2011) y tres usaron otros marcadores moleculares además de secuencias de ADN (Caparroz et al., 2009a; Milá et al., 2009; González et al., 2011). La escala geográfica va de regional (González et al., 2003; Chaves et al., 2007; Milá et al., 2009) a continental (Marks et al., 2002; Cheviron et al., 2005; Cadena et al., 2007; Nyari, 2007; Weir, 2009). Aquí consideraré las especies como de montaña (>1500 m) o de tierras bajas (<1500 m). Estos ecosistemas, aunque comparten grupos taxonómicos, representan dos tipos de avifaunas con aspectos ecológicos y evolutivos diferentes (Weir, 2006; Cadena et al., 2007).
Algunos estudios de filogeografía de aves Neotropicales incluyen dataje molecular (García-Moreno et al., 2004; Lovette, 2004; Cheviron et al., 2005; Chaves et al., 2007; Miller et al., 2008; Weir et al., 2008; Caparroz et al., 2009a; Caparroz et al., 2009b; D'Horta et al., 2011; González et al., 2011) que indican que los linajes intraespecíficos de estas especies se originaron hace entre cinco millones de años y 40.000 años, siendo la mayoría originados durante los últimos dos millones de años. Teniendo en cuenta las diferencias entre los métodos usados, los datos parecen señalar que los linajes filo-geográficos de aves Neotropicales se originaron entre el Plioceno y el Pleistoceno. Precisamente el proceso histórico más comúnmente considerado para explicar los patrones filogeográficos han sido las fluctuaciones climáticas del Cuaternario (García-Moreno et al., 2004; Aleixo, 2006; Chaves et al., 2007; Cabanne et al., 2008; Miller et al., 2008; Caparroz et al., 2009b; Arbeláez-Cortés et al., 2010; Pérez-Emán et al., 2010; D'Horta et al., 2011; González et al., 2011) que han hecho que los hábitats, montanos y de tierras bajas, se fragmenten y promuevan la diferenciación genética entre poblaciones alopátricas. Para especies de tierras bajas la formación de grandes ríos en Sur América (Marks et al., 2002; Bates et al., 2003; Bates et al., 2004; Cheviron et al., 2005; Nyari, 2007; Cabanne et al., 2008) y de cadenas montañosas (Lovette, 2004; Cheviron et al., 2005; Chaves et al., 2007; Milá et al., 2009) así como incursiones marinas (Brawn et al., 1996; González et al., 2003; Lovette, 2004) también se han considerado como modeladores de su estructura filogeográfica. No obstante, estos procesos no actuaron independientemente pero su efecto es difícil de separar (Cheviron et al., 2005; Cabanne et al., 2007; Cabanne et al., 2008). Adicionalmente, estudios que incluyen datos fenotípicos y ambientales así como información de otros marcadores moleculares han permitido identificar otros procesos, como selección natural o diferencias en flujo génico entre machos y hembras (Chaves et al., 2007; Caparroz et al., 2009a; Milá et al., 2009; González et al., 2011) como parte de la historia de estas aves. Finalmente, hay evidencia de crecimiento demográfico reciente de varias especies de aves (Cheviron et al., 2005; Aleixo, 2006; Cabanne et al., 2008; CortesRodríguez et al., 2008; Caparroz et al., 2009b; Pérez-Emán et al., 2010) que es interpretado como una respuesta a cambios ambientales 'favorables'. También se han señalado los Andes (Cheviron et al., 2005; Miller et al., 2008; Milá et al., 2009), y las tierras bajas del Istmo de Tehuantepec (Cadena et al., 2007; Cortés-Rodríguez et al., 2008; Pérez-Emán et al., 2010) como barreras para especies de tierras bajas y montanas, respectivamente. No obstante el efecto de esas barreras no ha sido igual para todas las especies (Marks et al., 2002; Miller et al., 2008; Arbeláez-Cortés et al., 2010).
Algunas de las especies estudiadas tienen distribuciones geográficas compartidas que permiten comparar sus patrones filogeográficos con detalle. Por ejemplo, hay tres especies de montaña, con datos filogeográficos para México y Centro América. Estas especies son: Arremon brunneinucha (Cadena et al., 2007; Navarro-Sigüenza et al., 2008), Chlorospingus ophthalmicus (García-Moreno et al., 2004; Bonaccorso et al., 2008; Weir et al., 2008) y Lepidocolaptes affinis (Arbeláez-Cortés et al., 2010); y a pesar de ser simpátricas su congruencia topológica es poca. Solo ciertos detalles, para México, como el aislamiento del macizo de los Tuxtlas y del norte de la sierra Madre del Sur coinciden, pero la diferenciación de la sierra Madre Oriental observada en A. brunneinucha y C. ophthalmicus no es evidente en L. affinis. Para Centro América el istmo de Tehuantepec es importante en la diferenciación de A. brunneinucha y C. ophthalmicus, pero para L. affinis la depresión de Nicaragua es la barrera más clara. Estos resultados son esperados por diferentes razones, según discutí al principio. No obstante en este caso la falta de congruencia topológica podría deberse a un factor que no se ha considerado previamente, y es que las unidades taxonómicas no son comparables, por ejemplo como resultado de estos análisis A. brunneinucha y C. ophthalmicus parecen representan más de una especie (e.g., García-Moreno et al., 2004; Cadena et al., 2007).
Finalmente, existen al menos cinco estudios de filogeografía comparada de aves Neotropicales que examinan los niveles de diferenciación genética entre poblaciones (Bates et al., 2004; Bates et al., 2003; Burney y Brumfield, 2009; Weir, 2009, Barber y Klicka, 2010); aunque en tres de ellos no se ponen a prueba hipótesis de congruencia entre especies. Estos estudios encontraron que, para las especies de montaña, las tierras bajas (e.g. valle del río Marañón en Perú e istmo de Tehuantepec en México; Weir, 2009; Barber y Klicka, 2010) afectan su patrón filogeográfico pero que no necesariamente la vicarianza a través de estas barreras fue sincrónica. Para aves de tierras bajas se encontró que las diferencias genéticas a través de cadenas montañosas y de ríos se relacionan bastante con la ecología de las especies, siendo las aves de dosel las que presentan menor diferenciación (Burney y Brumfield, 2009). Para especies de zonas menos boscosas (e.g. el cerrado entre Brasil y Bolivia) se encontró poca diferenciación genética, a pesar de una gran separación geográfica, que es atribuida a alto flujo génico o a una expansión poblacional reciente (Bates et al., 2003). Aunque patrones filogeográficos compartidos como estos empiezan a emerger para las aves Neotropicales; la representación en estos trabajos de grupos diversos y representativos del Neotrópico, como por ejemplo las tangaras (familia Thraupidae), es prácticamente nula. Por lo tanto los patrones filogeográficos que están emergiendo podrían no ser generales.
CONCLUSIÓN
La filogeografía comparada se ha establecido como uno de los campos más activos de investigación en evolución reciente de las biotas. A pesar de que sus conclusiones se han basado principalmente en comparaciones cualitativas existe ahora un grupo de métodos que permiten poner a prueba, explícitamente, hipótesis sobre la historia compartida de especies simpátricas. Los estudios filogeográficos de la avifauna Neotropical empiezan a mostrar patrones compartidos entre especies. Sin embargo el número de estos trabajos es muy bajo en comparación con la diversidad de esta región. La combinación de diferentes métodos e información, dentro del marco de la filogeografía comparada, resultará en un entendimiento detallado de la historia de las comunidades permitiendo conocer aspectos relacionados con su estabilidad y con su respuesta a cambios ambientales o procesos históricos que han dejado indicios en el patrón filogeográfico de sus especies.
AGRADECIMIENTOS
Agradezco al CONACyT-México por una beca de estudios de posgrado (número de becario: 210543). Agradezco, también, a Susana Magallón-Puebla por plantearme y discutir conmigo algunas de las ideas presentes en este manuscrito.
BIBLIOGRAFÍA
ALBACH DC, SCHÖNSWETTER P, TRIBSCH A. Comparative phylogeography of the Veronica alpina complex in Europe and North America. Mol Ecol. 2006;15(11):3269-3286. [ Links ]
ALEIXO A. Historical diversification of floodplain forest specialist species in the Amazon: A case study with two species of the avian genus Xiphorhynchus (Aves: Dendrocolaptidae). Biol J Linn Soc Lond. 2006;89(2):383-395. [ Links ]
ARBELÁEZ-CORTÉS E, NYÁRI ÁS, NAVARRO-SIGÜENZA AG. The differential effect of lowlands on the phylogeographic pattern of a Mesoamerican montane species (Lepidocolaptes affinis, Aves: Furnariidae). Mol Phylogenet Evol.2010;57(2):658-668. [ Links ]
ARBOGAST BS, KENAGY GJ. Comparative phylogeography as an integrative approach to historical biogeography. J Biogeogr. 2001;28(7):819-825. [ Links ]
AVISE JC. Molecular population structure and biogeographic history of a regional fauna: A case history with lessons for conservation and biology. Oikos. 1992;63(1):62-76. [ Links ]
AVISE JC. The history and purview of phylogeography: A personal reflection. Mol Ecol. 1998;7(4):371-379. [ Links ]
AVISE JC. Phylogeography: The history and formation of species. Cambridge, Harvard University Press; 2000. [ Links ]
AVISE JC, ARNOLD J, BALL RM, BERMINGHAM E, LAMB T, et al. Intraspecific phylogeography: The mitochondrial DNA bridge between population genetics and systematics. Annu Rev Ecol Syst. 1987;18:489-522. [ Links ]
BARBER BR, KLICKA J. Two pulses of diversification across the Isthmus of Tehuantepec in a montane Mexican bird fauna. Proc R Soc Lond B Biol Sci. 2010;277(1694):2675-2681. [ Links ]
BARBER PH, ERDMANN MV, PALUMBI SR. Comparative phylogeography of three codistributed stomatopods: Origins and timing of regional lineage diversification in the coral triangle. Evolution. 2006;60(9):1825-1839. [ Links ]
BARNOSKY AD, SHABEL AB. Comparison of mammalian species richness and community structure in historic and mid-Pleistocene times in the Colorado Rocky Mountains. Proc Calif Acad Sci. 2005;56(Supp1):50-61. [ Links ]
BATES JM, HAFFER J, GRISMER E. Avian mitochondrial DNA sequence divergence across a headwater stream of the rio Tapajós, a major Amazonian river. J Ornithol. 2004;145(3):199-205. [ Links ]
BATES JM, TELLO JG, SILVA JMCD. Initial assessment of genetic diversity in ten bird species of South American Cerrado. Stud Neotrop Fauna Environ. 2003;38(2):87-94. [ Links ]
BEATTY G, PROVAN J. Comparative phylogeography of two related plant species with overlapping ranges in Europe, and the potential effects of climate change on their intraspecific genetic diversity. BMC Evol Biol. 2011;11(1):29. [ Links ]
BEHEREGARAY LB. Twenty years of phylogeography. The state of the field and the challenges for the southern hemisphere. Mol Ecol. 2008;17(17):3754-3774. [ Links ]
BELL KL, MORITZ C, MOUSSALLI A, YEATES DK. Comparative phylogeography and speciation of dung beetles from the Australian wet tropics rainforest. Mol Ecol. 2007;16(23):4984-4998. [ Links ]
BENNETT KD. Milankovitch cycles and their effects on species in ecological and evolutionary time. Paleobiology. 1990;16(1):11-21. [ Links ]
BERMINGHAM E, AVISE JC. Molecular zoogeography of freshwater fishes in the southeastern United States. Genetics. 1986;113(4):939-965. [ Links ]
BERMINGHAM E, MARTIN AP. Comparative mtDNA phylogeography of Neotropical freshwater fishes: Testing shared history to infer the evolutionary landscape of lower Central America. Mol Ecol. 1998;7(4):499-517. [ Links ]
BININDA-EMONDS ORP. The evolution of supertrees. Trends Ecol Evol. 2004;19:315-322. [ Links ]
BONACCORSO E, NAVARRO SIGÜENZA AG, SANCHEZ-GONZALEZ LA, PETERSON AT, GARCIA-MORENO J. Genetic differentiation of the Chlorospingus ophthalmicus complex in Mexico and Central America. J Avian Biol.2008;39:311-321. [ Links ]
BRAWN JD, COLLINS TM, MEDINA M, BERMINGHAM E. Associations between physical isolation and geographical variation within three species of Neotropical birds. Mol Ecol. 1996;5(1):33-46. [ Links ]
BURNEY CW, BRUMFIELD RT. Ecology predicts levels of genetic differentiation in Neotropical birds. Am Nat. 2009;174(3):358-368. [ Links ]
CABANNE GS, D'HORTA FM, SARI EHR, SANTOS FR, MIYAKI CY. Nuclear and mitochondrial phylogeography of the Atlantic forest endemic Xiphorhynchus fuscus (Aves: Dendrocolaptidae): Biogeography and systematics implications. Mol Phylogenet Evol.2008;49(3):760-773. [ Links ]
CABANNE GS, SANTOS FR, MIYAKI CY. Phylogeography of Xiphorhynchus fuscus (Passeriformes, Dendrocolaptidae): Vicariance and recent demographic expansion in southern Atlantic forest. Biol J Linn Soc Lond. 2007;91(1):73-84. [ Links ]
CADENA CD. Testing the role of interspecific competition in the evolutionary origin of elevational zonation: An example with Buarremon brush-finches (Aves, Emberizidae) in the Neotropical mountains. Evolution. 2007;61(5):1120-1136. [ Links ]
CADENA CD, GUTIÉRREZ-PINTO N, DÁVILA N, TERRY CHESSER R. No population genetic structure in a widespread aquatic songbird from the Neotropics. Mol Phylogenet Evol.2011;58(3):540-545. [ Links ]
CADENA CD, KLICKA J, RICKLEFS RE. Evolutionary differentiation in the Neotropical montane region: Molecular phylogenetics and phylogeography of Buarremon brush-finches (Aves, Emberizidae). Mol Phylogenet Evol. 2007;44(3):993-1016. [ Links ]
CALSBEEK R, THOMPSON JN, RICHARDSON JE. Patterns of molecular evolution and diversification in a biodiversity hotspot: The California floristic province. Mol Ecol. 2003;12(4):1021-1029. [ Links ]
CAMPBELL V, LEGENDRE P, LAPOINTE F-J. The performance of the congruence among distance matrices (CADM) test in phylogenetic analysis. BMC Evol Biol. 2011;11(1):64. [ Links ]
CAPARROZ R, MIYAKI CY, BAKER AJ. Contrasting phylogeographic patterns in mitochondrial DNA and microsatellites: Evidence of female philopatry and male-biased gene flow among regional populations of the blue-and-yellow macaw (Psittaciformes: Ara ararauna) in brazil. Auk 2009a;126(2):359-370. [ Links ]
CAPARROZ R, SEIXAS GHF, BERKUNSKY I, COLLEVATTI RG. The role of demography and climatic events in shaping the phylogeography of Amazona aestiva (Psittaciformes, Aves) and definition of management units for conservation. Divers Distrib. 2009b;15(3):459-468. [ Links ]
CARNAVAL AC, HICKERSON MJ, HADDAD CLFB, RODRIGUES MT, MORITZ C. Stability predicts genetic diversity in the Brazilian Atlantic forest hotspot. Science. 2009;323(5915):785-789. [ Links ]
CARSTENS BC, RICHARDS CL. Integrating coalescent and ecological niche modeling in comparative phylogeography. Evolution. 2007;61(6):1439-1454. [ Links ]
CARTENS BC, BRUNSFELD SJ, DEMBOSKI JR, GOOD JM, SULLIVAN J. Investigating the evolutionary history of the Pacific Northwest mesic forest ecosystem: Hypothesis testing within a comparative phylogeographic framework. Evolution 2005;59(8):1639-1652. [ Links ]
CASTOE TA, DAZA JM, SMITH EN, SASA MM, KUCH U, et al. Comparative phylogeography of pitvipers suggests a consensus of ancient Middle American highland biogeography. J Biogeogr. 2009;36(1):88-103. [ Links ]
CHAN LM, BROWN JL, YODER AD. Integrating statistical genetic and geospatial methods brings new power to phylogeography. Mol Phylogenet Evol.2011;59(2):523-537. [ Links ]
CHAPPLE DG, DAUGHERTY CH, RITCHIE PA. Comparative phylogeography reveals pre-decline population structure of New Zealand Cyclodina (Reptilia: Scincidae) species. Biol J Linn Soc Lond. 2008;95(2):388-408. [ Links ]
CHAVES JA, POLLINGER JP, SMITH TB, LEBUHN G. The role of geography and ecology in shaping the phylogeography of the speckled hummingbird (Adelomyia melanogenys) in Ecuador. Mol Phylogenet Evol.2007;43(3):795-807. [ Links ]
CHEVIRON ZA, HACKETT SJ, CAPPARELLA AP. Complex evolutionary history of a Neotropical lowland forest bird (Lepidothrix coronata) and its implications for historical hypotheses of the origin of Neotropical avian diversity. Mol Phylogenet Evol. 2005;36(2):338-357. [ Links ]
CHURIKOV D, GHARRETT AJ. Comparative phylogeography of the two pink salmon broodlines: An analysis based on a mitochondrial DNA genealogy. Mol Ecol. 2002;11(6):1077-1101. [ Links ]
CORTES-RODRÍGUEZ N, HERNÁNDEZ-BAÑOS BE, NAVARRO-SIGÜENZA AG, OMLAND KE. Geographic variation and genetic structure in the streak-backed oriole: Low mitochondrial DNA differentiation reveals recent divergence. Condor. 2008a;110(4):729-739. [ Links ]
CORTÉS-RODRÍGUEZ N, HERNÁNDEZ-BAÑOS BE, NAVARRO-SIGÜENZA AG, PETERSON AT, GARCÍA-MORENO J. Phylogeography and population genetics of the amethyst-throated hummingbird (Lampornis amethystinus). Mol Phylogenet Evol. 2008b;48(1):1-11. [ Links ]
CRANDALL ED, FREY MA, GROSBERG RK, BARBER PH. Contrasting demographic history and phylogeographical patterns in two indo-pacific gastropods. Mol Ecol. 2008;17(2):611-626. [ Links ]
CRAWFORD AJ, BERMINGHAM E, POLANÍA C. The role of tropical dry forest as a long-term barrier to dispersal: A comparative phylogeographical analysis of dry forest tolerant and intolerant frogs. Mol Ecol. 2007;16(22):4789-4807. [ Links ]
CSILLÉRY K, BLUM MGB, GAGGIOTTI OE, FRANÇOIS O. Approximate Bayesian computation (ABC) in practice. Trends Ecol Evol. 2010;25(7):410-418. [ Links ]
D'HORTA FM, CABANNE GS, MEYER D, MIYAKI CY. The genetic effects of late quaternary climatic changes over a tropical latitudinal gradient: Diversification of an Atlantic forest passerine. Mol Ecol. 2011;20(9):1923-1935. [ Links ]
DA SILVA MNF, PATTON JL. Amazonian phylogeography: mtDNA sequence variation in arboreal echimyid rodents (Caviomorpha). Mol Phylogenet Evol. 1993;2(3):243-255. [ Links ]
DAWSON MN, LOUIE KD, BARLOW M, JACOBS DK, SWIFT CC. Comparative phylogeography of sympatric sister species, Clevelandia ios and Eucyclogobius newberryi (Teleostei, Gobiidae), across the California transition zone. Mol Ecol. 2002;11(6):1065-1075. [ Links ]
DOOH RT, ADAMOWICZ SJ, HEBERT PDN. Comparative phylogeography of two North American 'glacial relict' crustaceans. Mol Ecol. 2006;15(14):4459-4475. [ Links ]
DUPANLOUP I, SCHNEIDER S, EXCOFFIER L. A simulated annealing approach to define the genetic structure of populations. Mol Ecol. 2002;11(12):2571-2581. [ Links ]
EDWARDS SV, BEERLI P. Perspective: Gene divergence, population divergence, and the variance in coalescence time in phylogeographic studies. Evolution. 2000;54(6):1839-1854. [ Links ]
EFRON B, TIBSHIRANI RJ. An introduction to the bootstrap. New York, Chapman & Hall; 1993. [ Links ]
FELDMAN CR, SPICER GS. Comparative phylogeography of woodland reptiles in California: Repeated patterns of cladogenesis and population expansion. Mol Ecol. 2006;15(8):2201-2222. [ Links ]
GARCÍA-MORENO J, NAVARRO-SIGÜENZA AG, PETERSON AT, SANCHEZGONZALEZ LA. Genetic variation coincides with geographic structure in the common bush-tanager (Chlorospingus ophthalmicus) complex from Mexico. Mol Phylogenet Evol. 2004;33(1):186-196. [ Links ]
GARRICK RC, CACCONE A, SUNNUCKS P. Inference of population history by coupling exploratory and model-driven phylogeographic analyses. Int J Mol Sci. 2010;11(4):1190-1227. [ Links ]
GARRICK RC, ROWELL DM, SIMMONS CS, HILLIS DM, SUNNUCKS P, et al. Fine-scale phylogeographic congruence despite demographic incongruence in two lowmobility saproxylic springtails. Evolution. 2008;62(5):1103-1118. [ Links ]
GONZÁLEZ C, ORNELAS J, GUTIERREZ-RODRIGUEZ C. Selection and geographic isolation influence hummingbird speciation: Genetic, acoustic and morphological divergence in the wedge-tailed sabrewing (Campylopterus curvipennis). BMC Evol Biol. 2011;11(1):38. [ Links ]
GONZÁLEZ MA, EBERHARD JR, LOVETTE IJ, OLSON SL, BERMINGHAM E. Mitochondrial DNA phylogeography of the bay wren (Troglodytidae: Thryothorus nigricapillus) complex. Condor. 2003;105(2):228-238. [ Links ]
GOULD SJ, JOHNSTON RF. Geographic variation. Annu Rev Ecol Syst. 1972;3:457-498. [ Links ]
GUZIK MT, COOPER SJB, HUMPHREYS WF, AUSTIN AD. Fine-scale comparative phylogeography of a sympatric sister species triplet of subterranean diving beetles from a single calcrete aquifer in Western Australia. Mol Ecol. 2009;18(17):3683-3698. [ Links ]
HEWITT GM. The genetic legacy of the Quaternary ice ages. Nature. 2000;405:907-913. [ Links ]
HEWITT GM. Speciation, hybrid zones and phylogeography - or seeing genes in space and time. Mol Ecol. 2001;10(3):537-549. [ Links ]
HEWITT GM. Genetic consequences of climatic oscillations in the quaternary. Philos T R Soc B. 2004;359(1442):183-195. [ Links ]
HICKERSON MJ, CARSTENS BC, CAVENDER-BARES J, CRANDALL KA, GRAHAM CH, et al. Phylogeography's past, present, and future: 10 years after Avise, 2000. Mol Phylogenet Evol.2010;54(1):291-301. [ Links ]
HICKERSON MJ, DOLMAN G, MORITZ C. Comparative phylogeographic summary statistics for testing simultaneous vicariance. Mol Ecol. 2006a;15(1):209-223. [ Links ]
HICKERSON MJ, STAHL EA, LESSIOS HA. Test for simultaneous divergence using approximate Bayesian computation. Evolution 2006b;60(12):2435-2453. [ Links ]
HICKERSON MJ, STAHL E, TAKEBAYASHI N. MSBAYES: Pipeline for testing comparative phylogeographic histories using hierarchical approximate Bayesian computation. BMC Bioinformatics. 2007;8:268. [ Links ]
HICKERSON MJ, MEYER CP. Testing comparative phylogeographic models of marine vicariance and dispersal using a hierarchical Bayesian approach. BMC Evol Biol. 2008;8:322. [ Links ]
HUGALL A, MORITZ C, MOUSSALLI A, STANISIC J. Reconciling paleodistribution models and comparative phylogeography in the wet tropics rainforest land snail Gnarosophia bellendenkerensis (Brazier 1875). Proc Nat Acad Sci U S A. 2002;99(9):6112-6117. [ Links ]
JARAMILLO-CORREA JP, GRIVET D, TERRAB A, KURT Y, DE-LUCAS AI, et al. The strait of Gibraltar as a major biogeographic barrier in mediterranean conifers: A comparative phylogeographic survey. Mol Ecol. 2010;19(24):5452-5468. [ Links ]
JOSEPH L, MORITZ C, HUGALL A. Molecular support for vicariance as a source of diversity in rainforest. Philos T Roy Soc B. 1995;260(1358):177-182. [ Links ]
JOSEPH L, WILKE T. Lack of phylogeographic structure in three widespread Australian birds reinforces emerging challenges in Australian historical biogeography. J Biogeogr. 2007;34(4):612-624. [ Links ]
KELEMEN L, MORITZ C. Comparative phylogeography of a sibling pair of rainforest Drosophila species (Drosophila serrata and D. birchii). Evolution. 1999;53(4):1306-1311. [ Links ]
KIRCHMAN JJ, FRANKLIN JD. Comparative phylogeography and genetic structure of Vanuatu birds: Control region variation in a rail, a dove, and a passerine. Mol Phylogenet Evol. 2007;43(1):14-23. [ Links ]
KISHINO H, HASEGAWA M. Evaluation of the maximum likelihood estimate of the evolutionary tree topologies from DNA sequence data, and the branching order in Hominoidea. J Mol Evol. 1989;29(2):170-179. [ Links ]
KNOWLES LL, MADDISON WP. Statistical phylogeography. Mol Ecol. 2002;11(12):2623-2635. [ Links ]
KUHNER MK. Coalescent genealogy samplers: Windows into population history. Trends Ecol Evol. 2008;24(2):86-93. [ Links ]
LAPOINTE F-J, RISSLER LJ. Congruence, consensus, and the comparative phylogeography of codistributed species in California. Am Nat. 2005;166(2):290-299. [ Links ]
LEACHÉ AD, CREWS SC, HICKERSON MJ. Two waves of diversification in mammals and reptiles of Baja California revealed by hierarchical Bayesian analysis. Biol Letters. 2007;3(6):646-650. [ Links ]
LESSA EP, COOK JA, PATTON JL. Genetic footprints of demographic expansion in North America, but not Amazonia, during the late Quaternary. Proc Nat Acad Sci USA. 2003;100(18):10331-10334. [ Links ]
LESSA EP, D'ELÍA G, PARDIÑAS UFJ. Genetic footprints of late Quaternary climate change in the diversity of Patagonian-Fueguian rodents. Mol Ecol. 2010;19(15):3031-3037. [ Links ]
LOURIE SA, GREEN DM, VINCENT ACJ. Dispersal, habitat differences, and comparative phylogeography of Southeast Asian seahorses (Syngnathidae: Hippocampus). Mol Ecol. 2005;14(4):1073-1094. [ Links ]
LOVETTE IJ. Molecular phylogeny and plumage signal evolution in a trans Andean and circum Amazonian avian species complex. Mol Phylogenet Evol. 2004;32(2):512-523. [ Links ]
MALIOUCHENKO O, PALMÉ AE, BUONAMICI A, VENDRAMIN GG, LASCOUX M. Comparative phylogeography and population structure of European Betula species, with particular focus on B. pendula and B. pubescens. J Biogeogr. 2007;34(9):1601-1610. [ Links ]
MANNI F, GUERARD E, HEYER E. Geographic patterns of genetic, morphologic, linguistic) variation: How barriers can be detected by using Monmonier's algorithm. Hum Biol. 2004;76(2):173. [ Links ]
MARKS BD, HACKETT SJ, CAPPARELLA AP. Historical relationships among Neotropical lowland forest areas of endemism as determined by mitochondrial DNA sequence variation within the wedge-billed woodcreeper (aves: Dendrocolaptidae: Glyphorynchus spirurus). Mol Phylogenet Evol.2002;24(1):153-167. [ Links ]
MATEOS M. Comparative phylogeography of livebearing fishes in the genera Poeciliopsis and Poecilia (Poeciliidae: Cyprinodontiformes) in Central Mexico. J Biogeogr. 2005;32(5):775-780. [ Links ]
MILÁ B, WAYNE RK, FITZE P, SMITH TB. Divergence with gene flow and finescale phylogeographical structure in the wedge-billed woodcreeper, Glyphorynchus spirurus, a Neotropical rainforest bird. Mol Ecol. 2009;18(14):2979-2995. [ Links ]
MILLER MJ, BERMINGHAM E, KLICKA J, ESCALANTE P, AMARAL FSRD, WEIR JT, et al. Out of Amazonia again and again: Episodic crossing of the Andes promotes diversification in a lowland forest flycatcher. Proc R Soc Lond B Biol Sci. 2008;275(1639):1133-1142. [ Links ]
MORRONE JJ. Evolutionary biogeography: An integrative with case studies. New York, Columbia University Press, 2009. [ Links ]
MOUSSALLI A, MORITZ C, WILLIAMS SE, CARNAVAL AC. Variable responses of skinks to a common history of rainforest fluctuation: Concordance between phylogeography and palaeo-distribution models. Mol Ecol. 2009;18(3):483-499. [ Links ]
NAVARRO-SIGÜENZA AG, PETERSON AT, NYARI A, GARCÍA-DERAS GM, GARCÍA-MORENO J. Phylogeography of the Buarremon brush-finch complex (Aves, Emberizidae) in Mesoamerica. Mol Phylogenet Evol. 2008;478(1):21-35. [ Links ]
NOGUÉS-BRAVO D. Predicting the past distribution of species climatic niches. Global Ecol. Biogeogr. 2009;18(5):521-531.. [ Links ]
NYARI A. Phylogeographic patterns, molecular and vocal differentiation, and species limits in Schiffornis turdina (Aves). Mol Phylogenet Evol.2007;44(1):154-164. [ Links ]
OSHIDA T, LIN L-K, CHANG S-W, CHEN Y-J, LIN J-K. Phylogeography of two sympatric giant flying squirrel subspecies, Petaurista alborufus lena and P. philippensis grandis (Rodentia: Sciuridae), in Taiwan. Biol J Linn Soc Lond. 2011;102(2):404-419. [ Links ]
PARKER MA, DOYLE JL, DOYLE JJ. Comparative phylogeography of Amphicarpaea legumes and their root-nodule symbionts in Japan and North America. J Biogeogr. 2004;31(3):425-434. [ Links ]
PASTORINI J, THALMANN U, MARTIN RD. A molecular approach to comparative phylogeography of extant Malagasy lemurs. Proc Nat Acad Sci U S A. 2003;100(10):5879-5884. [ Links ]
PÉREZ-EMÁN J, MUMME RONALD L, JABÅONSKI PG. Phylogeography and adaptive plumage evolution in Central American subspecies of the slate-throated redstart (Myioborus miniatus); Ornithol Monogr. 2010; 67:90-102. [ Links ]
PLOUVIEZ S, SHANK TM, FAURE B, DAGUIN-THIEBAUT C, VIARD F, et al. Comparative phylogeography among hydrothermal vent species along the east Pacific rise reveals vicariant processes and population expansion in the south. Mol Ecol. 2009;18(18):3903-3917. [ Links ]
PUEBLA-OLIVARES F, BONACCORSO E, DE LOS MONTEROS AE, OMLAND KE, LLORENTE-BOUSQUETS JE, et al. Speciation in the emerald toucanet (Aulacorhynchus prasinus) complex. Auk 2008;125(1):39-50. [ Links ]
QU Y, LEI F, ZHANG R, LU X. Comparative phylogeography of five avian species: Implications for Pleistocene evolutionary history in the Qinghai-Tibetan plateau. Mol Ecol. 2010;19(2):338-351. [ Links ]
RICHARDS VP, THOMAS JD, STANHOPE MJ, SHIVJI MS. Genetic connectivity in the Florida reef system: Comparative phylogeography of commensal invertebrates with contrasting reproductive strategies. Mol Ecol. 2007;16(1):139-157. [ Links ]
RIDDLE BR. The molecular phylogeographic bridge between deep and shallow history in continental biotas. Trends Ecol Evol. 1996;11(5):207-211. [ Links ]
RISSLER LJ, HIJMANS RJ, GRAHAM CH, MORITZ C, WAKE DB. Phylogeographic lineages and species comparisons in conservation analyses: A case study of California herpetofauna. Am Nat. 2006;167(5):653-664. [ Links ]
ROBERTS TE. History, ocean channels, and distance determine phylogeographic patterns in three widespread Philippine fruit bats (pteropodidae). Mol Ecol. 2006;15(8):2183-2199. [ Links ]
ROCHA LA, BASS AL, ROBERTSON DR, BOWEN BW. Adult habitat preferences, larval dispersal, and the comparative phylogeography of three Atlantic surgeonfishes (Teleostei: Acanthuridae). Mol Ecol. 2002;11(2):243-252. [ Links ]
ROE AD, RICE AV, COLTMAN DW, COOKE JEK, SPERLING FAH. Comparative phylogeography, genetic differentiation and contrasting reproductive modes in three fungal symbionts of a multipartite bark beetle symbiosis. Mol Ecol. 2011;20(3):584-600. [ Links ]
SAEKI I, DICK CW, BARNES BV, MURAKAMI N. Comparative phylogeography of red maple (Acer rubrum l.) and silver maple (Acer saccharinum l.): Impacts of habitat specialization, hybridization and glacial history. J Biogeogr. 2011;38(5):992-1005. [ Links ]
SANÍN C, CADENA CD, MALEY JM, LIJTMAER DA, TUBARO PL, CHESSER RT. Paraphyly of Cinclodes fuscus (Aves: Passeriformes: Furnariidae): Implications for taxonomy and biogeography. Mol Phylogenet Evol.2009;53(2):547-555. [ Links ]
SCHÄUBLE CS, MORITZ C. Comparative phylogeography of two open forest frogs from Eastern Australia. Biol J Linn Soc Lond. 2001;74(2):157-170. [ Links ]
SCHNEIDER CJ, CUNNINGHAM M, MORITZ C. Comparative phylogeography and the history of endemic vertebrates in the wet tropics rainforests of Australia. Mol Ecol. 1998;7(4):487-498. [ Links ]
SHIMODAIRA H. An approximately unbiased test of phylogenetic tree selection. Syst Biol. 2002;51(3):492-508. [ Links ]
SHIMODAIRA H, HASEGAWA M. Consel: For assessing the confidence of phylogenetic tree selection. Bioinformatics. 2001;17(12):1246-1247. [ Links ]
SMITH CI, FARRELL BD. Range expansions in the flightless longhorn cactus beetles, Moneilema gigas and Moneilema armatum, in response to Pleistocene climate changes. Mol Ecol. 2005;14(4):1025-1044. [ Links ]
SOLOMON SE, BACCI M, JR., MARTINS J, JR., VINHA GG, MUELLER UG. Paleodistributions and comparative molecular phylogeography of leafcutter ants (Atta spp.) provide new insight into the origins of Amazonian diversity. Plos One. 2008;3(7):e2738. [ Links ]
SOLÓRZANO S, BAKER AJ, OYAMA K. Conservation priorities for resplendent quetzals based on analysis of mitochondrial DNA control region sequences. Condor. 2004;106(3):449-456. [ Links ]
SOLTIS DE, MORRIS AB, MCLACHLAN JS, MANOS PS, SOLTIS PS. Comparative phylogeography of unglaciated Eastern North America. Mol Ecol. 2006;15(14):4261-4293. [ Links ]
STEELE CA, STORFER A. Phylogeographic incongruence of codistributed amphibian species based on small differences in geographic distribution. Mol Phylogenet Evol. 2007;43(2):468-479. [ Links ]
SULLIVAN J, ARELLANO E, ROGERS DS. Comparative phylogeography of Mesoamerican highland rodents: Concerted versus independent response to past climatic fluctuations. Am Nat. 2000;155(6):755-768. [ Links ]
TABERLET P, FUMAGALLI L, WUST-SAUCY A-G, COSSON J-F. Comparative phylogeography and postglacial colonization routes in Europe. Mol Ecol. 1998;7(4):453-464. [ Links ]
TAYLOR MS, HELLBERG ME. Comparative phylogeography in a genus of coral reef fishes: Biogeographic and genetic concordance in the Caribbean. Mol Ecol. 2006;15(3):695-707. [ Links ]
TEMPLETON AR. Nested clade analyses of phylogeographic data: Testing hypotheses about gene flow and population history. Mol Ecol. 1998;7(4):381-397. [ Links ]
TEMPLETON AR. Phylogenetic inference from restriction endonuclease cleavage site maps with particular reference to the evolution of humans and the apes. Evolution. 1983;37(2):221-244. [ Links ]
TEMPLETON AR, MASKAS SD, CRUZAN MB. Gene trees: A powerful tool for exploring the evolutionary biology of species and speciation. Plant Spec Biol. 2000;15(3):211-222. [ Links ]
TURNER TF, TREXLER JC, KUHN DN, ROBISON HW. Life-history variation and comparative phylogeography of darters (Pisces: Percidae) from the North American central highlands. Evolution.1996;50(5):2023-2036. [ Links ]
VANDERGAST AG, PERRY WM, LUGO RV, HATHAWAY SA. Genetic landscapes GIS Toolbox: tools to map patterns of genetic divergence and diversity. Mol Ecol Res. 2010;1(1):158-161. [ Links ]
VÁZQUEZ-MIRANDA H, NAVARRO-SIGÜENZA AG, OMLAND KE. Phylogeography of the rufous-naped wren (campylorhynchus rufinucha): Speciation and hybridization in Mesoamerica. Auk. 2009;126(4):765-778. [ Links ]
VICTORIANO PF, ORTIZ JC, BENAVIDES E, ADAMS BJ, SITES JJW. Comparative phylogeography of codistributed species of Chilean Liolaemus (Squamata: Tropiduridae) from the central-southern Andean range. Mol Ecol. 2008;17(10):2397-2416. [ Links ]
WARES JP, CUNNINGHAM CW. Phylogeography and historical ecology of the north Atlantic intertidal. Evolution. 2001;55(12):2455-2469. [ Links ]
WEGMANN D, LEUENBERGER C, NEUENSCHWANDER S, EXCOFFIER L. ABCtoolbox: A versatile toolkit for approximate Bayesian computations. BMC Bioinformatics. 2010;11(1):116. [ Links ]
WEIR JT. Divergent timing and patterns of species accumulation in lowland and highland Neotropical birds. Evolution. 2006;60(4):842-855. [ Links ]
WEIR JT. Implications of genetic differentiation in Neotropical montane forest birds. Ann Mo Bot Gard. 2009;96(3):410-433. [ Links ]
WEIR JT, BERMINGHAM E, MILLER MJ, KLICKA J, GONZÁLEZ MA. Phylogeography of a morphologically diverse Neotropical montane species, the common bush-tanager (Chlorospingus ophthalmicus). Mol Phylogenet Evol. 2008;47(2):650-664. [ Links ]
ZINK RM. The geography of mitochondrial DNA variation in two sympatricsparrows. Evolution. 1991;45(2):329-339. [ Links ]
ZINK RM. Comparative phylogeography in North American birds. Evolution. 1996;50(1):308-317. [ Links ]
ZINK RM. Methods in comparative phylogeography, and their application to studying evolution in the North American aridlands. Integr Comp Biol. 2002;42(5):953-959. [ Links ]
ZINK RM, KESSEN AE, LINE TV, BLACKWELL-RAGO RC. Comparative phylogeography of some aridland bird species. Condor 2001;103(1):1-10. [ Links ]