Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.17 no.2 Bogotá May/Aug. 2012
PATRONES DE REPLICACIÓN Y BANDEO NO DIFERENCIAL EN ARDILLAS COLOMBIANAS DEL GÉNERO Sciurus (RODENTIA, SCIURIDAE)
Replication Patterns and no Differential Banding in Colombian Squirrels, Sciurus (Rodentia, Sciuridae)
CAROLINA ARANGO1, M.Sc.; MARTA LUCÍA BUENO1,2, M.Sc.
1Laboratorio de Citogenética. Instituto de Genética. Universidad Nacional de Colombia. Bogotá. carangor@unal.edu.co
2 Departamento de Biología. Facultad de Ciencias. Universidad Nacional de Colombia. Bogotá.
Presentado el 12 de novimebre de 2010, aceptado el 11 de noviembre de 2011, correcciones el 17 de julio de 2012.
RESUMEN
Las ardillas colombianas muestran una gran variabilidad citogenética lo que ha despertado el interés en ahondar aspectos evolutivos en el grupo. Para esto, varias herramientas de bandeo cromosómico convencional como bandas diferenciales Q y R y bandas no diferenciales C y NOR fueron analizadas permitiendo, por primera vez en las ardillas colombianas, detectar señales de cromosómicas inusuales de replicación e inactivación de heterocromatina y cromosomas marcadores específicos. Se explica la utilidad de cada uno de estos complementos en el análisis citogenético de especies y las observaciones para los citotipos de las ardillas colombianas.
Palabras clave: citogenética, Sciurus, ardilla, cromosoma, heterocromatina.
ABSTRACT
Colombian Squirrels cytogenetics showed a great variability which has renewed the interest in evolutionary aspects within the group. Many chromosome banding tools must be analyzed carefully in addition to the classical G-banding G technique. These techniques include other differential bands like Q and R banding and nondifferential banding (C and NOR). In this article the use of each of these supplements in the cytogenetic analysis of species and cytotypes observations for the Colombian squirrels is explained.
Key words: Cytogenetics, Sciurus, squirrel, chromosome, heterocromatin.
INTRODUCCIÓN
El orden Rodentia, perteneciente a los mamíferos, se caracteriza por su gran diversidad e importancia en la investigación clínica, en especial los roedores múridos. Aunque son numerosos los estudios genéticos en este grupo, los estudios evolutivos en estas especies no son concluyentes debido al gran número de rearreglos cromosómicos y polimorfismos encontrados, generando relaciones filogenéticas inciertas entre los diferentes grupos. Los roedores no múridos (Sciuridae) muestran un genoma más conservado (Graphodatsky et al., 2008), por lo cual representan un modelo ideal para el análisis genético y en específico para el estudio genómico comparativo cuyo fin es la búsqueda del genoma ancestral de los roedores (Stanyon et al., 2003; Li et al., 2006a; Li et al., 2006b, Beklemisheva, VR et al., 2011). Estas investigaciones encuentran su apogeo en especies europeas (Beklemisheva et al., 2011) y norteamericanas (Stanyon et al., 2003) que mediante estudios de citogenética molecular o ZooFish han logrado aproximaciones relevantes al estudio del genoma ancestral de los roedores. Sin embargo, son escasos los reportes de familias de roedores no muridos en latinoamérica, limitándose a estudios citogenéticos clásicos (Banda C, Banda NOR) en especies brasileñas como Sciurus alphonsei y Sciurus spadiceus (Lima y Langguth, 2002; Fagundes et al., 2003). Aunque las especies del género Sciurus hasta el momento reportadas presentan un cariotipo de 2n = 40, en Colombia se encuentra un cariotipo de 2n = 42 (Nadler y Hoffmann, 1970; Arango et al., 2007) generando una curiosidad creciente sobre las características citogenéticas de la especie colombiana. Para la correcta evaluación cromosómica de una especie, no solamente es necesario establecer el número cromosómico (2n) y Número Fundamental (NF); también es indispensable la caracterización del cariotipo mediante bandas diferenciales que permitan la clasificación inequívoca del cariotipo de una especie y genere las bases para la argumentación de análisis comparativos a nivel citogenético. Existen tres tipos de bandas cromosómicas diferenciables: Bandas Q, Bandas G y Bandas R. Dentro de las técnicas de bandeo G y Q, las bandas oscuras o fluorescentes son indicativas de un alto contenido en Adenina-Timina, mientras que las claras contienen Citocina-Guanina (Schreck et al., 1973; Drets, 2002). La importancia de esta observación radica en la densidad génica de cada banda. A mediados de la década de los 60 se estableció el término de isocoros para definir regiones de ADN según su densidad génica. Se nombraron entonces los isocoros de baja densidad (L1 y L2) como aquellas regiones de ADN con 1 gen cada 150 kb y los isocoros de alta densidad (H1, H2 y H3) como secciones de ADN con 1 gen cada 9 kb (Costantini et al., 2006). Cuando se establece una comparación de las bandas cromosómicas con respecto a la ubicación de los isocoros se observa que los isocoros de baja densidad muestran correspondencia con la ubicación de bandas oscuras o fluorescentes provenientes del bandeo G y/o Q respectivamente, en cualquier par cromosómico. De igual manera los isocoros de alta densidad se ubicaron en las bandas claras u opacas. Estas observaciones confirmaron que las regiones oscuras o fluorescentes a nivel cromosómico, ricas en Adenina-Timina, son entonces regiones de baja densidad génica. Por otro lado, la técnica de bandeo R, que recibe su nombre al generar una coloración reversa a la observada en la banda G, exhibe en los cromosomas bandas claras que determinan regiones ricas en Adenina-Timina y oscuras ricas en Citocina-Guanina. De este modo, la técnica de bandeo R permite visualizar las regiones ricas en actividad génica en cada uno de los cromosomas de la especie analizada, mediante la visualización de región como bandas oscuras a lo largo del cromosoma. Una de las características más relevantes en esta técnica de bandeo radica en la facilidad de observación del cromosoma X inactivo en las hembras de los mamíferos al presentarse este de coloración clara en una mayor proporción con respecto a su homólogo. La inactivación de este cromosoma se origina por procesos complejos a nivel molecular que tienen como objetivo generar un RNA de función silenciadora denominado RNAXist encargado de cubrir el cromosoma X inactivo génicamente (Plath, 2002). Citogenéticamente este hecho se observa como un cromosoma X sin coloración positiva u oscura en bandeamiento R (Schmidt et al., 1982), lo que confirma su baja actividad génica.
Otras técnicas adicionales de bandeo permiten la detección de características puntuales a nivel cromosómico recibiendo el nombre de Bandeo No Diferencial, porque no hace distinción entre los cromosomas de una célula y por lo tanto no permite la organización de un cariotipo. Este tipo de bandeo permite una sencilla visualización de segmentos de heterocromatina la cual tiene una característica básica sugerida y es el silenciamiento de los genes que allí se encuentren. Aunque la funcionabilidad de la heterocromatina aún se encuentra en discusión, Jackson, 1971, durante su disertación en "The Karyotype in Systematics", enfatiza numerosos ejemplos biológicos en donde se sugiere a la heterocromatización como un mecanismo de defensa para el silenciamiento de genes mutados que en caso de permanecer activos acarrearían serios problemas en el desarrollo biológico del individuo y en su fertilidad. Las técnicas clásicas de bandeo cromosómico descritas para la visualización de heterocromatina son las Bandas C y para la visualización de RNA ribosomal, las Bandas NOR (Verma y Babu, 1995; Drets, 2002). Estas señales pueden ser distintivas en cada especie, encontrándose aumentadas en una o disminuídas en número y/o tamaño en otras, e incluso con cambio en posición cromosómica, lo que permite la identificación de cromosomas marcadores (portadores de la señal), útiles para la diferenciación entre especies.
Para las ardillas colombianas los patrones de bandeo G han sido descritos con anterioridad (Arango et al., 2007). Sin embargo, las observaciones acerca del comportamiento de otros tipos de bandeamiento cromosómico sobre las ardillas colombianas es incipiente, encontrándose solo una referencia (Arango, et al., 2007).
MATERIALES Y MÉTODOS
Toma de muestra y procesamiento para obtención de cromosomas metafásicos a partir de sangre periférica. Se obtuvo la colaboración de la Unidad de rehabilitación y rescate de animales silvestres de la Universidad Nacional de Colombia (URRAS) y del Centro de recepción de fauna de la Secretaría de Ambiente (CRRF), en Engativá, Bogotá. Para cada unidad se realizó un documento explicativo del trabajo a realizar y se llevó a cabo una reunión formal y presencial para exponer la problemática de la investigación. Una vez aceptados los términos entre las entidades se comenzó con el muestreo con fines puramente académicos.
Identificación, registro fotográfico de ejemplares de ardillas colombianas alojadas y establecimiento de morfotipos. El registro fotográfico se realizó junto con la toma de datos provenientes de la historia clínica de cada ejemplar al momento de tomar la muestra, registrando número de identificación, posible procedencia, género, edad, tamaño corporal y patrón de coloración del pelo en la zona ventral, dorsal y cola. Toma de una muestra de sangre periférica de ejemplares de ardillas colombianas para análisis citogenético. El médico veterinario de cada centro de rescate animal (CRRF - URRAS) realizó una sedación con ketamina a cada ejemplar para minimizar el estrés producido por el procedimiento y luego realizó una venopunción por ejemplar para extraer 1 ml de sangre periférica heparinizada. La muestra de cada ejemplar recibió un código numérico único para ser procesada dentro de las instalaciones del Laboratorio de Citogenética, Instituto de Genética, Universidad Nacional de Colombia, con el fin de obtener cromosomas metafásicos. Adicionalmente, se registró el número de la historia clínica, tatuaje, y/o microship subcutáneo, con el fin de remitir los informes pertinentes a los centros que participaron en el estudio.
Procesamiento de la muestra sanguínea heparinizada para la obtención de cromosomas metafásicos. Se realizaron dos medios de cultivo para el crecimiento de linfocitos. Cada medio de cultivo se procesó en un frasco estéril marcado con el código de la muestra y constó de 4 mL de medio RPMI 1640, 1 mL de suero fetal bovino, 100 ul penicilina, 100 µL del agente mitogénico (favina - lectina extraída de Vicia faba) y 0,5 mL de la muestra del ejemplar. No se utilizó como agente mitogénico fitohemaglutinina M al no proporcionar un índice mitótico óptimo para el análisis de metafases. Ambos cultivos fueron incubados a 37 °C por 72 h. Uno de los cultivos recibió un pulso terminal con 40 µL de bromodeoxiuridina de 5 mg/mL (Sigma®) por 7 horas para obtener bandeo diferencial R. Completo el tiempo de incubación de ambos cultivos se procedió a la extracción de cromosomas metafásicos, añadiendo 100 µL de colchicina (25 g/mL) a cada cultivo por un tiempo de 25 minutos, 9 ml de solución hipotónica KCL 0,075 M por un tiempo de 20 minutos y fijando el contenido celular con fijador Carnoy (metanol: ácido acético Merck® en proporción 3 a 1). La muestra procesada del cultivo con pulso terminal de bromodeoxiuridina se extendió en láminas portaobjetos limpias para posteriores técnicas de bandeo cromosómico con banda R mientras que la muestra procesada del cultivo sin pulso terminal se extendió para las técnicas de bandeo G, Q, C, NOR. Se extendió una lámina por técnica de bandeo.
TÉCNICAS DE BANDEO CROMOSÓMICO
Bandeo R. Para esta técnica se siguió el protocolo propuesto por Ieshima et al., 1984, con modificaciones. Las láminas extendidas fueron expuestas a una solución de flourocromo Hoescht 33258 (0,2 mg/mL) por 25 minutos. Las láminas lavadas y secadas se cubrieron con una solución de 2XSSC y se incubaron en una cámara cerrada con lámpara de mercurio por 8 minutos. Se procedió a la tinción con colorante Giemsa a 4 % en agua destilada y metanol, por 7 minutos. Finalmente, las láminas se lavaron con agua y se secaron al aire para su observación al microscopio óptico.
Bandeo Q. Para esta técnica se siguió el protocolo propuesto por Caspersson et al., 1970, con modificaciones. Las láminas extendidas fueron expuestas a una solución de Quinacrina al 0,05 % por 15 minutos. Las láminas lavadas y secadas al aire se cubrieron con buffer fosfato pH 6,8 para ser observadas en el microscopio de fluorescencia. Bandeo C. Las láminas trabajadas en esta técnica fueron previamente analizadas en Bandeo Q para la correcta identificación de cromosomas. Se siguió el protocolo propuesto por Sumner, 1972, con modificaciones. Las láminas extendidas fueron expuestas a una solución de HCl 0,4 N por 50 minutos. Las láminas lavadas y secadas se expusieron luego a una solución de BaOH a 3 % por 24 minutos, para luego ser deshidratadas en alcohol a 70 % y a 96 % por 5 minutos. Cada lámina fue incubada en 2 x SSC a 59 °C por 16 horas, para luego realizar una nueva deshidratación en alcohol a 70 % y a 96 % por 5 minutos y colorear con colorante Giemsa a 5 % en buffer fosfato pH 6,8 por 15 minutos. Finalmente, las láminas se lavaron y secaron al aire para su observación en el microscopio óptico.
Bandeo NOR. Las láminas trabajadas en esta técnica fueron previamente analizadas en Bandeo Q para la correcta identificación de cromosomas. Se siguió el protocolo propuesto por Goodpasture y Bloom, 1975, con modificaciones. Las láminas extendidas fueron cubiertas con una solución de AgNO3 a 50 % p/v en agua destilada. Cada preparación se incubó en cámara húmeda a 37 °C durante 48 horas. Las láminas fueron teñidas con colorante Giemsa a 2 % en agua destilada por 15 segundos para su observación en el microscopio óptico.
REGISTRO FOTOGRÁFICO DE METAFASES Y ORGANIZACIÓN DE CARIOTIPO
Se tomaron microfotografías digitales a las metafases analizadas en cada técnica citogenética, mediante el uso de un microscopio óptico y el software para obtención y análisis de imágenes Lucia Karyotyping®. Cada imagen fue organizada para la obtención del cariotipo del ejemplar, tomando como base el cariotipo publicado para Sciurus granatensis (Arango et al., 2007)
ANÁLISIS DE RESULTADOS
Los cariotipos de cada ejemplar en cada una de las técnicas de bandeo cromosómico fueron comparados para establecer diferencias cromosómicas, en cuanto rearreglos o polimorfismos. Se establecieron citotipos y se analizaron las variaciones cromosómicas entre citotipos y morfotipos.
RESULTADOS
Morfológicamente y a través de la observación de la coloración del manto, se lograron establecer dos morfotipos principales (morfotipo A y B; Fig. 1) diferenciables por la presencia o ausencia de una banda gruesa de coloración negra en el dorso del animal, adicional a la gran variedad de tonalidades presentes en los ejemplares de ardillas colombianas y que fueron descritas con anterioridad por Emmons, 1990.
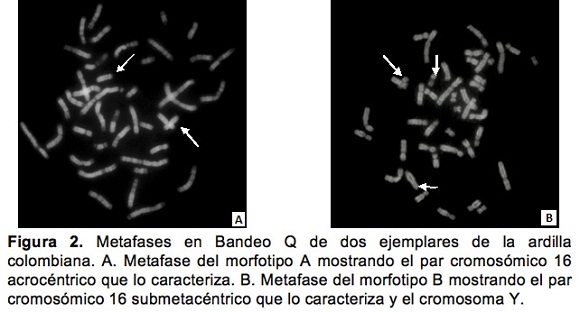
Luego del bandeamiento cromosómico utilizando técnicas diferenciales Q y R, se estableció para todos los ejemplares un número cromosómico de 2n=42 con grupos cromosómicos según las características descritas por Arango et al., 2007. Para cada uno de los dos morfotipos (A y B) se detectó una morfología acrocéntrica o metacéntrica del cromosoma 16 registradas mediante microfotografía digital con ayuda del software Lucia Karyotyping® (Fig. 2; Fig. 3). Lo anterior permitió observar que únicamente los ejemplares del morfotipo B, presentaban un par cromosómico 16 de morfología submetacéntrica, mientras que en los individuos del morfotipo A, la morfología del cromosoma 16 era acrocéntrica, confirmando así la presencia de dos citotipos en Colombia (Arango et al., 2007). Otra característica citogenética detectada en los ejemplares del morfotipo A fue una variación en la morfología y el patrón de bandeo de un par cromosómico que luego de la organización del cariotipo fue localizado en la posición 17. Esta observación sugiere la presencia de varios citotipos en el grupo.
Para cada uno de los morfotipos se establecieron las características del patrón de bandeo C (Fig. 4; Fig. 5; Fig. 6) y NOR, con el fin de localizar heterocromatina centromérica y regiones organizadoras nucleolares (Fig. 7). Adicionalmente se encontraron diferencias en el patrón de bandeo C de los citotipos del morfotipo A, según morfología del cromosoma 17 (Fig. 5).
DISCUSIÓN
Las técnicas de bandeo diferenciales tienen como fin el reconocimiento y la organización del cariotipo de una especie. En algunos mamíferos, específicamente en los humanos y en algunos primates, el cromosoma Y tiene en su porción terminal una región bastante rica en contenido de Adenina y Timina, evidenciándose como una porción de fluorescencia intensa mediante la técnica de Bandeo Q (Schmid et al., 1992). En el caso de las ardillas colombianas, el tamaño del cromosoma Y supondría una gran repetición de Adenina y Timina que, debido a la afinidad con la quinacrina, proporcionaría una luminosidad fuerte en la metafase mediante bandeo Q. Sin embargo, esta suposición no se detectó en ninguno de los ejemplares macho estudiados. De hecho ningún otro cromosoma presentó fluorescencia aumentada (Fig. 2).
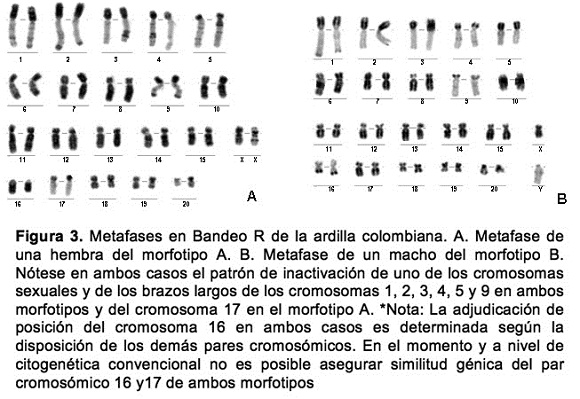
Por otro lado, la banda R permitió detectar no solo la inactivación de uno de los cromosomas X en las hembras (Fig. 3A), sino también un patrón de inactivación en el cromosoma Y (Fig. 3B), patrones de inactivación no esperados en los brazos largos de los cromosomas 1, 2, 3, 4, 5 y 9 (Fig. 3B) en ambos morfotipos y patrón de inactivación en brazos largos de los cromosomas 17 de algunos ejemplares del morfotipo A (Fig. 3A). Los patrones de inactivación en uno de los cromosomas sexuales, son patrones esperados que demuestran los mecanismos de compensación génica, por medio de silenciamiento (Plath, 2002). Sin embargo, no es común encontrar una gran variedad de autosomas con estos patrones de inactivación en mamíferos. Generalmente se tiende a relacionar una banda C positiva con un patrón de inactivación claro en bandas R, debido a la baja expresión génica en esas zonas. Sin embargo, las regiones de inactivación en autosomas observadas en estas ardillas no corresponden a bandas C positivas (Fig. 4; Fig. 5; Fig. 6), por lo que una explicación a este hecho puede deberse no solo a la cantidad de heterocromatina presente en estas zonas sino también a la baja densidad y expresión de los genes que allí se encuentran. Llama la atención que en todos los citotipos del morfotipo A en donde el par cromosómico 17 presenta un alto heteromorfismo, este se encuentre siempre inactivo en banda R. En este caso se podría sugerir que el genoma utiliza mecanismos de silenciamiento a este nivel para proteger a la especie de estos cambios cromosómicos que pueden llegar a traducirse en cambios génicos posiblemente deletéreos (Jackson, 1971). Siguiendo este planteamiento, al momento de observar la gran variabilidad citogenética intraespecífica en ardillas colombianas, es de suponer que estas deban poseer un mecanismo de protección a cambios negativos para la perpetuidad de su especie, por lo que los mecanismos de silenciamiento podrían ser la clave.
En los cromosomas existen regiones que de hecho, no tienen o poseen una muy baja expresión génica. Estas regiones reciben el nombre de heterocromatina y es definida como el material cromosómico de mayor condensación, rico en Adenina y Timina en bastantes repeticiones, que se observa al microscopio por medio de la técnica de Banda C (Drets, 2002). La distribución de la heterocromatina en el genoma puede variar entre las especies (Jackson, 1971). De hecho, las diferencias en la cantidad, posición y propiedades de la heterocromatina entre especies relacionadas son muy comunes y pueden ser utilizadas como marcadores específicos para la identificación. La observación de la pérdida o ganancia de heterocromatina en los cromosomas puede sugerir la acción de un mecanismo cromosómico involucrado durante la diferenciación de una especie base a otra relacionada. Un ejemplo es la pérdida de heterocromatina causada por una fusión centromérica. En este caso la formación de un cromosoma metacéntrico o submetacéntrico por medio de una fusión centromérica origina la pérdida de cierta cantidad de heterocromatina de alguno o de ambos cromosomas acrocéntricos involucrados en el rearreglo. En las ardillas colombianas se pudieron observar características relevantes a nivel de bandeo C. En primer lugar, el cromosoma Y no presentó señal positiva evidente para esta técnica de bandeo (Fig. 4), hecho que generalmente se observa en los cromosomas Y de los mamíferos debido a su alto contenido de Adenina y Timina. Todos los cromosomas presentaron señales positivas en las constricciones primarias, confirmando la presencia de centrómero. Únicamente el cromosoma 16 del morfotipo A no evidenció de forma clara una tinción C positiva a nivel de centrómero (Fig. 4), lo que sugiere que el origen de este cromosoma pudo darse por posible fisión centromérica previa en un cromosoma submetacéntrio o metacéntrico del cariotipo ancestral, ocasionando una pérdida parcial a nivel de centrómero (Jackson, 1971), confirmando así la sugerencia de Arango y Bueno, en prensa, en donde se supone que uno de los primeros rearreglos cromosómicos estructurales que permitieron la especiación de la ardilla colombiana fue la fisión céntrica de un cromosoma metacéntrico de una especie relacionada. Una observación interesante en este tipo de técnica, concierne a la asociación entre el tipo de bandeo C presente en el cromosoma 17 y su morfología cromosómica. Según documentos anteriores (Arango y Bueno, en prensa), las ardillas colombianas presentan varios citotipos diferenciados por la morfología y patrón de bandas de los cromosomas 16 y 17. En el caso específico del cromosoma 17, la banda C muestra distintos patrones de bandeo dependiendo de la variación morfológica que este tenga. Por tal motivo, en algunos citotipos específicamente con cromosomas 17 tipo a o d, se pueden encontrar cromosomas 17 con banda C positiva únicamente en la constricción primaria (centrómero; Fig. 5A), mientras que en otros se pueden detectar señales positivas tanto para centrómero como para regiones intersticiales de brazo largo (Fig. 5B). Sin embargo, sin importar la variación citogenética del cromosoma 17, se encontraron en todos los ejemplares señales de bandeo C positivas relevantes, como por ejemplo, una señal de heterocromatina centromérica aumentada en el brazo largo de los cromosomas 3 e inversión pericentromérica en los cromosomas 14 (Fig. 6). Solamente para los ejemplares del morfotipo A se encontraron señales intersticiales en el brazo largo de los cromosomas 5 (Fig. 6). Estos hallazgos tienen interés en aspectos evolutivos porque pueden estar indicando procesos cromosómicos involucrados en fenómenos de especiación. Por ejemplo, la señal intersticial en el brazo largo del par 5 de la ardilla colombiana, supondría un rearreglo cromosómico tipo fusión en eventos previos, posiblemente luego de la primera fisión céntrica comentada ya, en relación con el origen posible del cromosoma 16 acrocéntrico del morfotipo A de la ardilla colombiana. De igual manera, las inversiones pericentroméricas juegan un papel relevante para la generación de nuevas morfologías cromosómicas en el cariotipo de la especie que incluso pueden afectar el número fundamental de la misma (Jackson, 1971; Sumner, 2003). En este caso, una inversión pericentromérica cambia el tamaño tanto del brazo largo como del corto del cromosoma, pudiendo generar un cromosoma submetacéntrico a partir de un metacéntrico y viceversa. En el caso de las ardillas colombianas, el cromosoma 14 presenta una inversión pericentromérica originando un cromosoma metacéntrico con brazo corto posiblemente de mayor tamaño que el de la especie parental. Debido a que todos los citotipos evaluados tienen esta misma característica, es probable que estos rearreglos que involucran la cantidad y disposición de la heterocromatina centromérica sean predecesores de los rearreglos tipo fisión o fusión cromosómica ya explicados.
Las regiones organizadoras del nucleólo (NOR) forman una constricción secundaria dentro del cromosoma que puede ser identificada mediante la utilización de nitrato de plata (Drets, 2002). Son zonas altamente activas transcripcionalmente y su funcionabilidad radica en la capacidad de producir ARN ribosomales necesarios para la síntesis de proteínas en la célula durante un ciclo celular. Los genes de ARN ribosomales se encuentran dentro de estas regiones organizadoras y pueden encontrarse en un solo sitio en un par de cromosomas homólogos dentro de la especie. Por ejemplo, los NOR de los ratones han sido ubicados hasta en seis pares de cromosomas, mientras que en los humanos se han encontrado NOR en 5 pares cromosómicos. La localización del NOR dentro de un cromosoma puede ser generalmente cerca de las partes terminales aunque también se han encontrado reportes de observaciones NOR en zonas intersticiales (Sumner, 2003). Dentro de las ardillas colombianas se encontraron 4 pares de cromosomas con tinciones NOR positivas: cromosomas 1, 3 y 4 con sitios NOR en la región subterminal del brazo largo y cromosoma 20 con tinción NOR positiva en la región terminal del brazo largo (Fig. 7). Estas tinciones se mantuvieron en todos los ejemplares analizados, permitiendo establecer estas regiones como marcadoras para la ardilla colombiana en general. No se encontraron diferencias de posición de NOR dentro del grupo analizado. Sin embargo, existen cuestionamientos interesantes sobre si las bandas NOR realmente concretan la visualización de la totalidad de las regiones organizadoras nucleolares, porque en algunos casos, por efecto de la dominancia o recesividad los sitios NOR inactivos pueden no expresar las proteínas argentofilicas responsables de la presencia de la tinción característica (Dobigny et al., 2002). Por lo tanto sería prudente confirmar la localización de los sitios NOR encontrados en las ardillas colombianas y determinar si es posible la detección de otros sitios NOR no activos, mediante técnicas de hibridización fluorescente utilizando sondas específicas para marcadores de regiones organizadoras nucleolares (Phillips y Reed, 1996).
Con lo anterior es claro que a nivel citogenético son muchas las herramientas que pueden ser utilizadas para la explicación de las diferencias genéticas entre especies relacionadas y que el correcto uso y la combinación de las mismas pueden generar datos o respuestas relevantes a inquietudes no solo sistemáticas sino también evolutivas.
AGRADECIMIENTOS
Los investigadores desean extender sus agradecimientos a las entidades financiadoras División de Investigación Bogotá (DIB) de la Universidad Nacional de Colombia por la adecuación de infraestructura en laboratorio, a la Fundación para la Promoción de la Investigación y la Tecnología del Banco de la República por el suministro de materiales, a los centros de rescate de fauna silvestre URRAS (Unidad de rehabilitación y rescate de Animales Silvestres de la Universidad Nacional de Colombia) y CRRF (Centro de Recepción de Fauna de la Secretaría de Ambiente), por permitir el acceso a los ejemplares de estudio y al Instituto de Genética de la Universidad Nacional de Colombia por proporcionar los equipos necesarios para la investigación.
BIBLIOGRAFÍA
ARANGO C, BUENO ML, CHACÓN MN. Variación cariológica en diferentes fenotipos de Sciurus granatensis (Rodentia, Sciuridae). Acta biol.Colomb. 2007;12:3-12. [ Links ]
BEKLEMISHEVA VR, ROMANENKO SA, BILTUEVA LS, TRIFONOV VA, VOROBIEVA NV, SERDUKOVA NA, et al. Reconstruction of karyotype evolution in core Glires. I. The genome homology revealed by comparative chromosome painting. Chromosome Res. 2011;19:549-565. [ Links ]
CASPERSSON T, ZECH L, JOHANSSON C. Differential binding of alkylating fluorochromes in human chromosomes. Exp Cell Res. 1970;60:315-319 [ Links ]
CONSTATINI M, CLAY O, AULETTA F, BERNARDI G. An isochore map of human chromosomes. Genome Res. 2006;16:536-541. [ Links ]
DOBIGNY G, OZOUF COSTAZ C, BONILLO C, VOLOBOUEV V. Ag-NORs are not always true NORs:new evidence in mammals. Cytogenet Genome Res. 2002;98:75-77. [ Links ]
DRETS ME. Una saga citogenética: El descubrimiento de los métodos de bandeo. Rev Med Uruguay. 2002;18:107-121. [ Links ]
EMMONS LH. Neotropical Rainforest Mammals: a field Guide. The University of. Chicago Press; 1990. [ Links ]
FAGUNDES V, CHRISTOFF AU, AMARO-GHILARD RC, SCHEIBLER DR, YONENAGAYASSUDA Y. Multiple interstitial ribosomal sites (NORs) in the Brazilian squirrel Sciurus aestuans ingrami (Rodentia, Sciuridae) with 2n = 40. An overview of Sciurus cytogenetics. Genet Mol Biol. 2003;26(3):253-257. [ Links ]
GRAPHODATSKY AS, YANG F, DOBIGNY G, ROMANENKO SA, BILTUEVA LS, PERELMAN PL, et al. Tracking genome organization in rodents by Zoo-FISH. Chromosome Res. 2008;16(2):261-274. [ Links ]
GOODPASTURE C, BLOOM SE. Visualization of Nucleolar Organizer Regions in Mammalian Chromosomes Using Silver Staining. Chromosomes. 1975;53:37-50. [ Links ]
IESHIMA A, YORITA T, TAKESHITA K. A simple R-banding technique by BrdU- Hoechst treatment and Giemsa staining following heating and ultraviolet exposure. J Hum Genet. 1984;29(2):133-138. [ Links ]
JACKSON RC. The karyotype in systematics. Ann Rev Ecol Syst. 1971;2:327-368. [ Links ]
LI T, WANG J, SU W, YANG F. Chromosomal mechanisms underlying the karyotype evolution of the oriental voles (Muridae, Eothenomys). Cytogenet Genome Res. 2006a;114:50-55. [ Links ]
LI T, WANG J, SU W, NIE W, YANG F. Karyotypes Evolution of the Family Sciuridae. Inferences from the Genome Organizations of Ground Squirrels. Cytogent Genome Res. 2006b;112:270-276. [ Links ]
LIMA JF, LANGGUTH A. Karyotypes of Brazilian Squirrels: Sciurus spadiceus and Sciurus alphonsei. Folia Zool. 2002;5(3):201-204. [ Links ]
NADLER CF, HOFFMANN RS. Chromosomes of Some Asian and South American Squirrels (Rodentia, Sciuridae). Experientia. 1970; 26:1383-1386. [ Links ]
PHILLIPS RB, REED KM. Application of fluorescence in situ hybridization (FISH) techniques to fish genetics: a review. Aquaculture. 1996;140:197-216. [ Links ]
PLATH K, MLYNARCZYK EVANS S, NUSINOW DA, PANNING B. Xist RNA and the mechanism of X chromosome inactivation. 2002. Annu Rev Genet. 2002;36:233-78. [ Links ]
SCHMIDT M, STOLZMANN WM, BARANOVSKAYA LI. Replication Variants of the Human Inactive X Chromosome. I. Variability within Lymphocytes of Single Individuals. Chromosoma. 1982;85:405-412. [ Links ]
SCHMID M, STEINLEIN C, FEICHTINGER W, SCHMIDT M, VISBAL A, FERNANDEZ A. An intriguing Y chromosome in Heteromys anomalus (Rodentia, Heteromyidae). Hereditas. 1992;117(3):209-214. [ Links ]
SCHRECK RR, WARBURTON D, MILLER OJ, BEISER SM, ERLANGER BF. Chromosome Structure as Revealed by a Combined Chemical and Immunochemical Procedure. Proc Nat Acad Sci U S A. 1973;70(3):804-807. [ Links ]
STANYON R, STONE G, GARCIA M, FROENICKEA L. Reciprocal Chromosome Painting Shows that Squirrels, Unlike Murid Rodents, Have a Highly Conserved Genome Organization. Genomics. 2003; 82:245-249. [ Links ]
SUMNER AT. A simple technique for demonstrating centromeric heterochromatin. Exp Cell Res.1972;75:304-306. [ Links ]
SUMNER AT. Chromosomes: Organization and Function. United Kingdom: First Edition Blackwell Publishing; 2003. [ Links ]
VERMA RS, BABU A. Human Chromosomes. Principles and techniques. Second Edition. USA: McGraw-Hill Inc.; 1995. [ Links ]