Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.19 no.2 Bogotá May/Aug. 2014
https://doi.org/10.15446/abc.v19n2.41010
http://dx.doi.org/10.15446/abc.v19n2.41010
ANALIZANDO DATOS DE RNA-Seq EN PROCARIOTAS: UNA REVISIÓN PARA NO EXPERTOS
RNA-Seq Data Analysis in Prokaryotes: A Review for Non-experts
ANDRÉS EDUARDO RODRÍGUEZ CUBILLOS1, Biólogo; LAURA PERLAZA JIMÉNEZ1, M. Sc.; ADRIANA JIMENA BERNAL GIRALDO1, Ph. D.
1 Grupo de Micología y Fitopatología, Departamento de Ciencias Biológicas, Universidad de los Andes. Carrera 1 # 18A - 12. Laboratorio J-204. Bogotá, Colombia.
Autor de correspondencia: Adriana Bernal, abernal@uniandes.edu.co
Presentado el 28 de noviembre de 2013, aceptado el 31 de enero de 2014, fecha de reenvío el 12 de febrero de 2014.
Citation / Citar este artículo como: RODRÍGUEZ CUBILLOS AE, PERLAZA JIMÉNEZ L, BERNAL GIRALDO AJ. Analizando datos de RNA-Seq en procariotas: una revisión para no expertos. Acta biol. Colomb. 2014;19(2):131-142.
RESUMEN
La secuenciación de transcritos con RNA-Seq es hoy en día una de las técnicas más populares en los estudios transcriptómicos. Relativamente reciente, esta técnica ha permitido la secuenciación de transcritos de RNA en una escala y profundidad no alcanzada por otras técnicas anteriores. Sin embargo, el alcance de las conclusiones que se pueden sacar depende estrictamente de un proceso adecuado, desde el diseño experimental hasta el análisis bioinformático de los datos. Dadas las diferencias en el proceso transcripcional de las células eucariotas y procariotas, el análisis de RNA-Seq deberá tener ciertas consideraciones dependiendo del tipo de organismo estudiado. En esta revisión se exponen los principales factores a tener en cuenta para lograr un análisis de RNA-Seq consistente, replicable y concluyente, enfocándose específicamente en organismos procariotas.
Palabras clave: análisis bioinformático, diseño experimental, procariotas, RNA-Seq.
ABSTRACT
RNA-Seq is nowadays the method of choice for the sequencing of transcripts and transcriptomes in the field of molecular biology and gene expression assays. Until recently, this technique has allowed for the sequencing of RNA transcripts in an unprecedented scale and depth never reached in previous years; nevertheless, the reach and validity of the conclusions generated will depend strictly on an adequate experimental design and a robust analysis of the data. Given the inherent differences between prokaryotes and eukaryotes, the RNA-Seq analysis should take into account the type of organism studied. In this review we present the main factors to take into consideration when designing a consistent analysis for this type of data in prokaryotes, from the experimental design to the in silico analysis of the generated data.
Keywords: analysis, experimental design, prokaryotes, RNA-Seq.
INTRODUCCIÓN
El estudio del transcriptóma de un organismo en una condición dada puede realizarse con diferentes técnicas; estas pueden ser basadas en hibridación o secuenciación. Las técnicas basadas en hibridación, como los microarreglos, requieren el conocimiento previo de los transcritos de interés para generar las sondas. Además de esto, introducen ruido por posibles hibridaciones cruzadas, inherente al proceso de diseño de sondas, y limita el rango de detección a genes previamente descritos (Wang et al., 2009; McClure et al., 2013). Por su lado, las técnicas basadas en secuenciación tienen mayor alcance; técnicas como SAGE (Serial Analysis of Gene Expression), CAGE (Cap Analysis of Gene Expression) y MPSS (Massively Parallel Signature Sequencing) se basan en la secuenciación de cDNA, o librerías de ESTs con Sanger. Sin embargo, debido a que estas técnicas son de bajo rendimiento, se requiere dirigir la secuenciación de forma específica para producir resultados precisos, elevando los costos significativamente (Wang et al., 2009; Ozsolak y Milos, 2011; McClure et al., 2013).
La aparición de las plataformas de secuenciación masiva paralelizadas, o técnicas de secuenciación de nueva generación (NGS, Next-generation sequencing), han generado la producción de datos a gran escala. Debido a esto, los costos de secuenciación se han reducido drásticamente de entre dos a tres órdenes de magnitud en los últimos diez años (Shendure y Ji, 2008; Tucker et al., 2009). La reducción en los costos de NGS ha aumentado las posibilidades de estudiar cambios en el campo transcriptómico de forma más flexible y global que lo permitido por técnicas anteriores. El nuevo enfoque de interés en el tema de la transcriptómica es la secuenciación masiva y profunda de RNAs, cuya técnica se denomina RNASeq. El principal objetivo de RNA-Seq es catalogar todos y cada uno de los transcritos (RNA) expresados por una célula en una condición especifica; una técnica altamente cuantitativa y de alto rendimiento que ha encontrado diversas aplicaciones en la actualidad (Hoen et al., 2008; Wang et al., 2009; Chen et al., 2011; Ozsolak y Milos, 2011; McClure et al., 2013; Van Verk et al., 2013). Estas técnicas basadas en secuenciación masiva eliminan el ruido introducido por la correcta hibridación de sondas dependiente del diseño empleado, ofrecen un mayor rango dinámico de detección al evitar la saturación por señales de fluorescencia y permiten obtener un panorama más global de los genes activados y/o reprimidos sin restringirse a genes previamente caracterizados (Hoen et al., 2008; Bradford et al., 2010; Nookaew et al., 2012; Soneson y Delorenzi, 2013; Van Verk et al., 2013).
Aunque el alcance del RNA-Seq es mucho mayor a técnicas anteriormente usadas su poder informativo se ve limitado por cuatro factores importantes: 1) el diseño del experimento del cual se obtienen los datos a secuenciar, 2) la información requerida sobre los datos secuenciados, 3) la calidad de la secuenciación y 4) el análisis bioinformático de los datos (Agarwal et al., 2010; Bradford et al., 2010; Oshlack et al., 2010; Soneson y Delorenzi, 2013). Dada la cantidad de información recopilada en una corrida de RNA-Seq, este último punto puede llegar a ser abrumador si se desconoce una metodología adecuada. En esta revisión se sigue un flujo de trabajo para el análisis de organismos procariotas debido a que se excluyen los pasos necesarios para analizar datos de RNA-seq que contienen información sobre RNA producto de splicing alternativo, exclusivo de células eucariotas. También se entiende que el principal objetivo del análisis de RNA-Seq es detectar genes diferencialmente expresados (DEGs); sin embargo se incluyen recomendaciones para cumplir otros objetivos, como la detección de RNAs no codificantes y anotación de genes novedosos.
Diseño experimental
El diseño experimental para el análisis de RNA-Seq es un paso de gran relevancia, porque luego de secuenciadas las muestras cualquier error en el diseño experimental solo podrá corregirse reemplazando las muestras con una nueva secuenciación, lo cual implica altos costos y extiende el tiempo de la investigación. El diseño experimental de un análisis transcriptómico no está muy alejado del diseño de otros experimentos en investigación comparativa; sin embargo cobra gran importancia debido al origen de la variación encontrada en los resultados. En los datos de RNA-Seq se encuentran dos orígenes de variación: el biológico, y el técnico. Si el diseño experimental no se realiza adecuadamente se podrían confundir las variaciones técnicas con las biológicas y llegar a conclusiones erradas que ningún análisis estadístico elaborado podría evitar (Auer y Doerge, 2010). Una manera frecuente de controlar la variación técnica es utilizar Barcoding. Los Barcodes son pequeños fragmentos de secuencias específicas agregados a la secuencia original del transcrito que sirven como etiqueta para cada muestra de RNA, permitiendo diferenciar cada tratamiento del experimento; estas muestras luego son procesadas en la misma reacción de secuenciación para reducir al mínimo la variabilidad técnica, maximizar los recursos utilizados y, a la vez, permitir el diseño experimental en bloques (Auer y Doerge, 2010; Strickler et al., 2012).
Capturar la variabilidad biológica entre tratamientos se logra realizando el número de réplicas adecuadas. Sin embargo, cuando no se conoce la variabilidad del organismo, definir qué numero de réplicas biológicas es el adecuado resulta difícil; el método estadístico empleado también definirá un mínimo de réplicas a utilizar. Lo que se recomienda, en general, es tener tres réplicas biológicas por tratamiento, es decir tres tratamientos idénticos por separado (Anders y Huber, 2010; Soneson y Delorenzi, 2013; Tarazona et al., 2013). Esto permite evaluar la variabilidad biológica dentro de cada tratamiento y entre tratamientos. No obstante, aunque los trabajos de RNA-Seq sin réplicas biológicas son comunes en la literatura, a medida que crecen los estudios con esta técnica la estadística aplicada es mas estricta y los resultados sin réplicas biológicas pueden resultar obsoletos, sobretodo por la incapacidad de poder extrapolar los resultados a una población biológica (Anders y Huber, 2010; Auer y Doerge, 2010; Soneson y Delorenzi, 2013; Tarazona et al., 2013).
Además de tomar en cuenta la variabilidad técnica y biológica, es importante entender que la precisión en la estimación de transcritos diferencialmnete expresados está relacionada, también, con la profundidad de secuenciación; es decir, a mayor número de datos menos varianza existirá entre ellos. En este caso particular, los transcritos más afectados por una baja profundidad de secuenciación serán aquellos con bajos niveles de expresión y longitudes reducidas. Actualmente se ha propuesto que para un genoma de mamífero se requieren 700 millones de lecturas para obtener una cuantificación confiable de > 95 % de los transcritos expresados (Bradford et al., 2010; Tarazona et al., 2011; Cai et al., 2012). Según el consorcio de investigación ENCODE, lanzado por el Instituto Nacional de Investigación del Genoma Humano (NHGRI), la profundidad de secuenciación adecuada varía dependiendo del objetivo del proyecto. Para experimentos cuyo propósito es evaluar la similitud entre dos perfiles transcriptómicos, se recomienda tener treinta millones de lecturas pareadas de una longitud mayor a treinta nucleótidos. Si el experimento busca descubrir nuevos transcritos, o cuantificarlos de forma robusta, un mínimo de 100-200 millones de lecturas mayores a setenta pares de bases es recomendado.
Información requerida: tipos de librerías
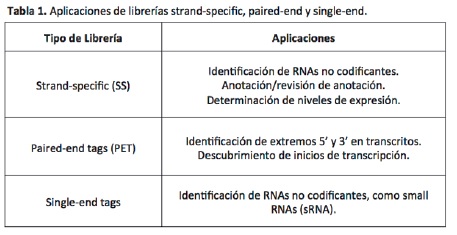
Los datos obtenidos en el experimento serán representados por secuencias, denominadas lecturas, y la agrupación de estas generan librerías de secuenciación. Además de contener lecturas, las librerías incluyen información particular de estas. Por esta razón, resulta crucial definir el tipo de librería deseada previo a la secuenciación. Dependiendo del tipo de librería se puede obtener información adicional a la expresión diferencial de genes, como la anotación de genes novedosos o la identificación de RNAs no codificantes. Adicionalmente, la escogencia adecuada de la librería tendrá un impacto sobre la precisión de los resultados finales. Para generar análisis de transcriptomas completos en una condición dada es recomendable la construcción de librerías paired-end tags, conocidas por sus siglas en inglés como librerías tipo PET (Tabla 1). Estas librerías secuencian dos regiones por cada fragmento de RNA, un fragmento a partir de cada extremo, produciendo lecturas pareadas (Ruan y Ruan, 2012). Esto es importante a la hora de realizar el ensamblaje de transcritos; es decir, la unión de lecturas para formar transcritos hipotéticos. Dicho proceso se logra, normalmente, mediante la identificación de lecturas contiguas alineadas a un genoma de referencia. Al contar con dos lecturas por fragmento, este tipo de librería disminuye las posibilidades de encontrar alineamientos en un genoma de referencia por azar, aumentando la precisión a la hora de identificar transcritos hipotéticos. Por otro lado, al obtener dos lecturas por fragmento se aumenta el área de cobertura en el genoma de referencia estudiado; por esta razón, se suele considerar que la cobertura de este tipo de librería es superior (Fullwood et al., 2009). Por su lado, las librerías single-end se recomiendan para el análisis y predicción de RNAs no codificantes (Tabla 1), los cuales son moléculas de menor longitud. En estos casos, las librerías tipo paired-end resultan en la sobreestimación de transcritos y complejizan el ensamblaje de los mismos debido al riesgo inherente de secuenciar por duplicado cada transcrito.
También existe la posibilidad de generar librerías hebra-específica (strand-specific) con información sobre la dirección de transcripción. Este tipo de información resulta muy útil a la hora de descubrir RNAs no codificantes, los cuales pueden ser transcritos en sentido contrario de su blanco, anotar de forma correcta genes novedosos (previamente no predichos por herramientas bioinformáticas) y, asimismo, obtener niveles de expresión más precisos debido a la capacidad de discernir mejor a qué transcrito pertenece una secuencia en regiones con genes superpuestos (Kapranov et al., 2007; He et al., 2008; Parkhomchuk et al., 2009). Aunque existen varios métodos para la construcción de una librería hebra-específica, se ha reportado que el método dUTP, donde toda la secuenciación se hace a partir de la primera hebra de cDNA, genera los resultados más confiables (Levin et al., 2010).
El tamaño de las librerías también resulta crucial porque determina la profundidad de secuenciación del ensayo. En casos donde las muestras de RNA no puedan ser obtenidas de forma pura (aislamiento a partir de tejidos hospederos), la cantidad limitante de RNA puede ser un obstáculo para aclanzar una profundidad de secuenciación efectiva. En estos casos, se suelen amplificar las librerías mediante PCR antes de la secuenciación. No obstante, este paso previo suele introducir ruido adicional debido a la posible generación de dímeros de primer's y/o la amplificación de productos inespecíficos (Tang et al., 2011). Como alternativa, se puede emplear una ampificación lineal, utilizada en la metodología CEL-seq (Hashimshony et al., 2012). Dicha aproximación emplea una transcripción in vitro (IVT) a partir de cDNAs de doble cadena con un promotor muy específico de la RNA polimerasa T7. Esta reacción isotérmica evita los productos inespecíficos y dímeros de primer's de la amplificación exponencial pero requiere de una mayor cantidad de muestra inicial; por esta razón, previo a la IVT, las muestras se juntan con códigos de barras (Barcodes) específicos para sumar las cantidades de RNA y poder, a su vez, discernir las muestras originales. Aún así, esta metodología muestra un fuerte sesgo hacia la detección de transcritos con altos niveles de expresión y no parece ofrecer una buena cobertura del transcriptoma, por lo que la mejor manera de obtener suficiente RNA a partir de muestras limitantes continúa siendo debatible (Bhargava et al., 2014).
Secuenciación
Teniendo en cuenta la información anterior, las muestras deben ser procesadas y enviadas a secuenciar, especificando qué tipo de librería se requiere. La secuenciación RNA-Seq de las muestras puede realizarse mediante cuatro tipos diferentes de plataforma: Illumina, SOLiD, 454 y Ion Torrent (Ozsolak y Milos, 2011; McClure et al., 2013). Sin embargo, para términos prácticos de este documento se asume que los datos fueron obtenidos a través de la plataforma de secuenciación Illumina, considerada como una de las tecnologías más dominantes en el mercado (Metzker, 2010). Para garantizar una buena calidad de secuenciación, es de vital importancia obtener buenas muestras del RNA a secuenciar. Para esto se debe lograr extraer RNA de buena calidad en las condiciones requeridas. La calidad del RNA suele medirse mediante dos parámetros: pureza e integridad. La pureza se puede analizar en un espectrofotómetro, como el NanoDrop; se espera que las medidas de absorbancia estén dentro del rango 1,8 y 2 para asegurar unas muestras sin contaminación de proteínas o compuestos fenólicos, normalmente introducidos durante el proceso de extracción. El bioanalizador realiza una electroforesis capilar que permite determinar la integridad del RNA mediante dos tipos de lecturas: el número de integridad del RNA (RIN) y las proporciones de RNA ribosomal (rRNA) presentes (23S/16S). La primera se calcula a través de un software que analiza todo el patrón electroforético de corrida y el segundo método calcula las proporciones de las intensidades de banda correspondientes al rRNA (las más visibles en una electroforesis de RNA total).
RINs de buena calidad se sitúan por encima de seis, en un rango de uno a diez, y buenas proporciones de rRNA son iguales o superiores a uno. Luego de asegurarse que la extracción se realice de la manera adecuada, las muestras se envían a secuenciar. Desde este punto la calidad de la secuenciación queda en manos de la compañía secuenciadora.
Análisis bioinformático
Después del proceso de secuenciación se obtienen los datos en forma de lecturas contenidos en archivos informáticos. Estos archivos pueden llegar a ser muy pesados; entre más grande sea el genoma del organismo estudiado mayor será el tamaño de los archivos, por lo cual se recomienda tener un clúster o un servidor para poder analizarlos de forma eficiente. Se recomienda tener, preferiblemente, un servidor con 16Gb de RAM o más; si no es posible, al menos 4Gb de RAM y una capacidad de almacenamiento superior al tamaño de las librerías debido a que los archivos resultantes del análisis bioinformático también suelen ser pesados (Van Verk et al., 2013). A lo largo de esta revisión se explicarán los pasos para analizar los datos utilizando un sistema operativo basado en Unix (sea GNU/Linux o Mac OS X). Los datos suelen estar en formato .fastq. Este formato contiene las secuencias de las lecturas y sus correspondientes valores de calidad de secuenciación. Los pasos básicos en el análisis bioinformático son: (1) la preparación de las lecturas, (2) el mapeo y ensamblaje, y (3) la identificación de genes diferencialmente expresados. Adicionalmente, se pueden identificar RNAs no codificantes aunque esta revisión se enfocará en la identificación de transcritos codificantes.
Preparando las lecturas para el análisis
Antes de centrarnos en identificar los transcritos diferencialmente expresados, se debe realizar una limpieza y ensamblaje de dichos transcritos. Para ello es necesario primero responder dos preguntas: 1)Cuál es el estado de la secuenciación de nuestras lecturas? 2) ¿Qué información dentro de las librerías es pertinente incluir en el análisis?
Para responder la primera pregunta se pueden visualizar las lecturas utilizando la herramienta bioinformática fastqC, disponible en http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (Van Verk et al., 2013). Esta herramienta (Fig. 1) permite observar la calidad de las lecturas, la distribución por nucleótido, el contenido GC, las secuencias sobrerrepresentadas, la frecuencia de k-meros y el nivel de duplicación, entre otros. Con esta información se identifica el estado de las lecturas y se toman medidas para optimizar las mismas; a esto se le denomina limpieza de las lecturas.
Existen varios criterios para identificar qué limpieza se debe realizar a las lecturas. Por ejemplo, la calidad de las lecturas debe estar por encima de 28 (quality score) y se deben eliminar las porciones de secuencias que incrementen la desviación estándar de la muestra. Se deben tener en cuenta la distribución y frecuencia de los nucleótidos; una inconstancia en estas características usualmente implica la presencia de fragmentos de los adaptadores ligados durante el proceso de secuenciación. Muchas veces dentro del servicio de secuenciación, las compañías incluyen el servicio de limpieza de adaptadores. Sin embargo, siempre es útil realizar este análisis debido a que no siempre la limpieza es realizada con los mismos estándares. Se puede decir que estos son los principales criterios a observar, sin embargo si está interesado en conocer otros criterios sugerimos visitar la página de los desarrolladores: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/.
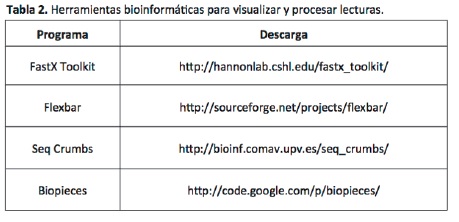
Para realizar dicha limpieza de lecturas se puede emplear el programa FastX Toolkit disponible gratis en la web del Laboratorio Hannon (http://hannonlab.cshl.edu/fastxtoolkit/). Para filtrar las secuencias por una calidad mínima de secuenciación se debe emplear el paquete Quality Filter de FastX Toolkit. Para eliminar regiones extremas con un contenido de bases anormal se puede utilizar Trimmer (paquete de FastX Toolkit). Después de filtrar por calidad y remover regiones extremas con proporciones anormales de bases es recomendable mirar los archivos nuevamente mediante fastqC para verificar se haya mejorado la calidad y se hayan eliminado las regiones correspondientes a los adaptadores. Además de FastX Toolkit, existen otras herramientas bioinformáticas disponibles para eliminar adaptadores, filtrar secuencias por calidad y recortar una longitud de secuencia especificada por el usuario (Tabla 2).
Para responder qué información dentro de las librerías es pertinente incluir en el análisis se debe tener en cuenta que los kit de limpieza de RNA ribosomal (rRNA) utilizados durante la extracción no son 100 % eficientes (Giannoukos et al., 2012). Uno de los grandes retos del analisis de RNA-Seq, sobretodo en procariotas, consiste en la eliminación adecuada de este tipo de RNA que no suministra información relevante sobre genes de interés diferencialmente expresados y suele ser muy abundante. Por ende, es recomendable realizar una filtración del rRNA in silico (métodos bioinformáticos). Para eliminar las secuencias correspondientes al rRNA es recomendable utilizar un alineador eficiente como Bowtie2 (Langmead y Salzberg, 2012). Bowtie2 ahorra memoria porque comprime los datos utilizando una transformación de Burrows- Wheeler, brindando una mayor eficiencia al lado de otros alineadores como SOAP y Maq; Bowtie es 351 veces más rápido que SOAP y 107 veces más rápido que Maq. La clave de la transformación Burrows-Wheeler radica en su flexibilidad; además de reducir el tamaño, en este caso, de un genoma, evita la pérdida de información asociada normalmente con la compresión de archivos (Langmead et al., 2009). Para eliminar el rRNA se deben indexar las secuencias de rRNA correspondientes al organismo de estudio con el fin de suministrárselo a Bowtie2 como un genoma de referencia. El siguiente paso será mapear las lecturas contra este "genoma de referencia" y eliminar todo aquello que alinee para conservar únicamente las lecturas que no corresponden a rRNA. Si se tienen lecturas pareadas (PETs) es importante realizar la limpieza por cada pareja de archivos en conjunto para evitar eliminar lecturas de una sola pareja únicamente (ver anexos para recomendaciones técnicas). Para obtener todos los detalles sobre las opciones y comandos de Bowtie2 se recomienda leer el manual del programa disponible en http://bowtie-bio.sourceforge.net/bowtie2/manual.shtml
Mapeo y ensamblaje de transcritos
Para analizar las lecturas de forma apropiada y poder descubrir genes diferencialmente expresados en un tratamiento específico se deben seguir tres pasos (Fig. 2): 1) mapeo de lecturas a un genoma de referencia, 2) ensamble y estimación de las abundancias de los transcritos y 3) identificación de transcritos diferencialmente expresados. Para realizar el mapeo de las lecturas a un genoma de referencia se puede utilizar TopHat; esta herramienta utiliza Bowtie2 para alinear lecturas a un genoma de referencia y ofrece las ventajas adicionales de permitir ensayos strand-specific (hebra-específico) e identificar genes novedosos por splicing alternativo, útil en organismos eucariotas (Trapnell et al. 2009). Para el mapeo es importante tener en cuenta que aproximadamente el 90 % del genoma procariota es codificante (Kuo y Ochman, 2009), por lo que el transcriptóma debería tener una alta cobertura del genoma.
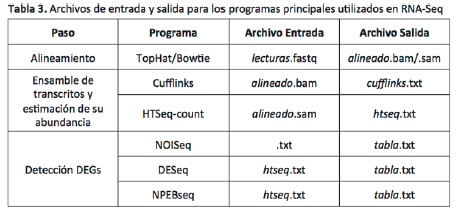
Para ensamblar transcritos a partir de lecturas y estimar sus abundancias se recomienda el uso de Cufflinks y Cuffmerge (Trapnell et al., 2010) o, en caso de querer utilizar DESeq, HTSeq-count (Tabla 3); este último puede ser descargado desde la página de los desarrolladores http://www.huber.embl.de/users/anders/HTSeq/doc/count.html. Es importante resaltar que para utilizar HTSeq-count se debe tener un archivo SAM de entrada y TopHat normalmente arroja resultados en formato binario BAM para ahorrar espacio (ver anexo para mayor información sobre compatibilidad de archivos y sugerencias técnicas). Otros programas como BWA, CLC o IDB-UD son alternativas adicionales para alinear lecturas y ensamblar transcritos (Li y Durbin, 2009; Peng et al., 2012).
Adicionalmente al genoma de referencia, es importante saber si se cuenta con un genoma anotado en formato .gtf o .gff. En caso de tener este tipo de archivo, es aconsejable suministrarlo en el proceso de alineamiento y ensamble para guiar dichos procesos y obtener los nombres anotados de los transcritos identificados (de lo contrario Cufflinks establece un código como nombre a cada transcrito especificando el rango de coordenadas que cubre dentro del genoma). Además de obtener genes previamente anotados, también se pueden identificar transcritos novedosos siempre y cuando se le permita a Cufflinks ensamblar transcritos diferentes a los ya anotados (ver anexo para recomendaciones técnicas).
Identificación de Genes Diferencialmente
Expresados (DEGs) Existen diversidad de programas capaces de identificar genes diferencialmente expresados (DEGs, por sus siglas en inglés). La selección del programa dependerá, en gran medida, del número de réplicas biológicas disponibles en el ensayo. En esta revisión nos enfocaremos principalmente en las ventajas y desventajas de los programas más comúnmente utilizados y entraremos a detallar mejor las aproximaciones paramétricas y no paramétricas empleadas por DESeq (Anders y Huber, 2010), NOISeq (Tarazona et al., 2011; Tarazona et al., 2012) y NPEBseq (Bi y Davuluri, 2013).
Antes de poder identificar DEGs se deben "normalizar" los datos de las abundancias de los transcritos hipotéticos. El término "normalización" suele emplearse de forma errada en los análisis de RNA-Seq para referirse al método por el cual se busca minimizar el ruido técnico introducido en los datos durante el proceso de secuenciación con el fin de volverlos comparables entre sí; no busca transformar los datos para que sigan una distribución normal. Existen numerosos métodos de normalización dentro de los cuales los más ampliamente utilizados son la normalización por tamaño de librería y por longitud del fragmento o transcrito hipotético. Normalizar por el tamaño de librería implica llevar a una misma escala todas las librerías correspondientes a cada tratamiento para evitar falsos positivos (una librería con mayor profundidad de secuenciación tiene más probabilidad de tener genes diferencialmente sobreexpresados respecto a otra librería sin ser consecuencia del tratamiento). De la misma forma, un transcrito largo tendrá mayor probabilidad de ser secuenciado y de tener un número mayor de lecturas alineadas que uno corto, implicando una mayor probabilidad de ser detectado como un DEG (Oshlack y Wakefield, 2009; Dillies et al., 2013). El método de normalización que emplea ambas correcciones mencionadas se conoce como FPKM por sus siglas en inglés (Fragments Per Kilobase Of Transcript Per Million Mapped Reads), parecido al RPKM (Reads Per Kilobase Of Transcript Per Million Mapped Reads). La única diferencia entre ambas es que una utiliza fragmentos y la otra lecturas; una librería tipo PET tiene dos lecturas por fragmento, razón por la cuál se emplea la terminología FPKM al trabajar con este tipo de librerías (Mortazavi et al., 2008).
También resulta útil tomar en cuenta las variaciones biológicas naturales entre tratamientos no atribuibles a los tratamientos en sí. Por esta razón, Robinson et al. (2010) crearon una corrección para eliminar el ruido introducido por la variación biológica entre muestras. Este tipo de normalización busca llevar a una misma escala los valores de expresión promedio entre las diferentes muestras bajo el supuesto que "la mayoría de genes no se encuentran diferencialmente expresados". Este tipo de normalización se conoce como TMM, por sus siglas en inglés Trimmed mean of M values, y podría ser útil si se encuentran demasiados genes diferencialmente expresados al final de un análisis. Vale la pena resaltar, sin embargo, las asunciones en las que se basa esta normalización: solo una minoría de genes se encuentran diferencialmente expresados y el número de genes sobreregulados será similar al número de genes reprimidos (Robinson y Oshlack, 2010; Dillies et al., 2013; Soneson y Delorenzi, 2013).
Al emplear Cufflinks para ensamblar transcritos a partir de lecturas alineadas se obtendrán abundancias normalizadas por FPKMs para cada transcrito hipotético. HTSeq-count, por otro lado, arroja abundancias crudas sin ningún tipo de normalización por lo que no se recomienda utilizar estas abundancias para detectar DEGs sin antes haberlas normalizado (los programas para la detección de genes diferencialmente expresados suelen tener pasos previos de normalización).
Debido al gran número de genes presentes en cada ensayo de RNA-Seq se requiere una corrección para las múltiples comparaciones (una por cada gen entre dos tratamientos) con el fin de evitar falsos positivos; a medida que aumentan las comparaciones aumenta la probabilidad de encontrar diferencias por azar. Inicialmente, el concepto de tasa de falsos descubrimientos (FDR por sus siglas en inglés), desarrollado por (Benjamini y Hochberg, 1995), fue ampliamente utilizado para controlar este tipo de error. No obstante, la mayoría de los cálculos estadísticos iniciales para determinar DEGs se basaban en un comportamiento normal de los datos, idea que normalmente no se cumple. Además, es importante resaltar que la matriz de datos obtenida después de una corrida de RNA-Seq se compone de números con valores positivos (no son números reales), por lo que una aproximación basada en un comportamiento gaussiano no sería apropiada (Li et al., 2012). Paralelamente, la estimación correcta de una FDR requiere de valores p precisos y estos valores de significancia se basan en una distribución teórica de los datos. Por ende, si dicha distribución teórica no se cumple, será difícil rechazar falsos positivos de forma acertada. Debido a esto, se desarrollaron métodos que, en vez de asumir una distribución normal, asumen una distribución de Poisson (Marioni et al., 2008) o una binomial negativa (Anders y Huber, 2010) para controlar mejor la sobredispersión observada entre réplicas técnicas y biológicas, respectivamente. Aún así, estas presunciones empleadas por programas paramétricos como EdgeR (Robinson et al., 2010) y DESeq (Anders y Huber, 2010) siguen asumiendo una distribución teórica de los datos y basan sus cálculos en una estimación de la relación existente entre media y varianza; parámetros difíciles de estimar por separado con pocas réplicas biológicas. Por ende, estas técnicas pueden ser sensibles a la variabilidad presente entre réplicas biológicas si no se cuenta con el número adecuado (Bullard et al., 2010).
Aunque EdgeR y DESeq funcionan bajo los mismos supuestos, DESeq emplea una regresión local de las dispersiones presentes en los datos que le brinda mayor flexibilidad ante varianzas inestables; esto ha sido demostrado experimentalmente por (Robles et al., 2012). Sin embargo, si un gen tiene una dispersión muy elevada en el tratamiento problema y una dispersión baja en el tratamiento de referencia, la regresión trazada de dispersión será más elevada en el tratamiento problema con respecto al tratamiento de referencia. De la misma forma, el promedio de expresión de un gen en un tratamiento será influenciado en gran medida por un valor extremo en alguna réplica y un gen podría ser elegido como un DEG a pesar de no tener un nivel de expresión similar entre las réplicas biológicas utilizadas. Por ende, la confiabilidad de DESeq y otros programas paramétricos se reduce con pocas réplicas biológicas debido a la imprecisión que existe a la hora de estimar los parámetros de media y varianza; utilizar estos programas es recomendable únicamente cuando se tienen tres o, preferiblemente, más de tres réplicas biológicas por tratamiento (Soneson y Delorenzi, 2013).
Para evitar supuestos en la distribución de los datos y disminuir la tasa de falsos positivos se pueden transformar los datos o emplear programas no paramétricos que no asumen una distribución establecida para los datos. Anders et al. (2010) desarrollaron un método donde los datos son transformados para estabilizar las varianzas y generar homocedasticidad. También existe una transformación utilizada originalmente para datos de microarreglos que ha sido empleada en algunos experimentos de RNA-Seq (Smyth, 2004; Soneson y Delorenzi, 2013). No obstante, los programas no paramétricos han tenido gran acogida en los últimos años y existen dos muy conocidos que han demostrado tener una baja tasa de falsos positivos: SAMseq (Li y Tibshirani, 2011) y NOISeq (Tarazona et al., 2011; Tarazona et al., 2012).
Recientemente, NOISeq ha tenido varias mejoras que incluyen la versión NOISeqBIO diseñada para trabajar con réplicas biológicas. NOISeq genera una distribución de ruido basada en los factores de cambio existentes entre réplicas de un mismo tratamiento. Posteriormente, los factores de cambio calculados entre tratamientos son contrastados contra la distribución de ruido para discernir entre cambios atribuibles a los tratamientos o al ruido existente entre réplicas (Tarazona et al., 2011; Tarazona et al., 2012).
NOISeqBIO genera una distribución nula mediante aleatorización de los datos de expresión. Esta distribución corresponde a la hipótesis nula de genes invariantes entre tratamientos (al aleatorizar los datos se rompe cualquier tipo de relación). Posteriormente, los factores de cambio calculados entre tratamientos son contrastados contra los calculados entre réplicas (ruido) y contra los calculados para la distribución nula con el fin de determinar si los cambios de expresión son significativos o no.
Sin embargo, el número de combinaciones posibles (permutaciones) para la construcción de la distribución nula dependerá del número de datos por gen (réplicas biológicas). Por ende, la distribución nula puede quedar "deficiente" con pocas réplicas biológicas. En caso de tener pocas réplicas biológicas (menos de cuatro), NOISeqBIO agrupa los genes con expresión similar (teniendo en cuenta todas las réplicas biológicas disponibles) y genera clústers de genes (k) donde cada (k) se toma como una condición y se aleatoriza con base en el número de genes contenidos para generar una distribución nula más robusta que se utiliza para contrastar los factores de cambio calculados entre tratamientos sin aleatorizar (Tarazona et al., 2011; Tarazona et al., 2012). Esta aproximación es más robusta que la estimación de la media y la varianza empleada por programas paramétricos si se tienen menos de cuatro o tres réplicas biológicas (Tarazona et al., 2011; Soneson y Delorenzi, 2013). Sin embargo, en caso de no tener réplicas, NOISeq también tiene una versión capaz de simular un número de réplicas biológicas establecida por el usuario (NOISeqSim), aunque las conclusiones de dicho análisis no podrán ser extrapoladas a nivel poblacional (Tarazona et al., 2013).
SAMseq también ha demostrado tener una tasa muy baja de falsos positivos. No obstante, estudios previos han recalcado que su capacidad para detectar DEGs se ve limitada al uso de al menos cuatro réplicas biológicas por tratamiento (Soneson y Delorenzi, 2013). La estadística no paramétrica también ha sido combinada con la estadística bayesiana en el pasado, aunque solo recientemente se ha implementado para la identificación de genes diferencialmente expresados en microarreglos (Smyth, 2004). No obstante, estos métodos no pueden ser aplicados directamente a datos de RNA-Seq debido a las diferencias en la naturaleza de los datos (Auer et al., 2012). El programa más reciente en emplear este tipo de estadística es NPEBseq desarrollado por Yingtao Bi et al., (2013). La estadística bayesiana infiere un parámetro (en este caso, la media de expresión génica) con base en una distribución a priori y la probabilidad que los datos se den bajo dicha hipótesis/modelo (verosimilitud).
La estadística bayesiana paramétrica asume distribuciones a priori paramétricas a diferencia de la estadística bayesiana no paramétrica. Por otro lado, la estadística bayesiana empírica no asume una distribución a priori; la construye con base en la media y la varianza observada de los datos para calcular una media estimada que se aproxime más a la media poblacional (desconocida). NPEBseq emplea estadística bayesiana empírica no paramétrica para definir genes diferencialmente expresados y se puede descargar a través de la página web del Instituto Wistar (http://bioinformatics.wistar.upenn.edu/NPEBseq). Este programa prueba la hipótesis que la diferencia en los niveles de expresión entre las condiciones A y B están por encima de un umbral de significancia biológico definido por el usuario (Bi y Davuluri, 2013).
Este umbral se suministra para disminuir la distancia que separa un resultado estadísticamente significativo de uno biológicamente significativo; es más probable secuenciar y detectar genes con niveles de expresión elevados, dejando por fuera genes con bajos niveles de expresión que también pueden estar diferencialmente expresados entre tratamientos (McIntyre et al., 2011). Programas paramétricos basados en la distribución normal dan predilección a genes con altos niveles de expresión independiente de las diferencias significativas entre tratamientos (Wu et al., 2010). No obstante, no queda claro cómo establecer este umbral de significancia biológica en NPEBseq para un set de datos específicos. La ventaja que ofrece NPEBseq es que no tiene supuestos paramétricos y es robusto incluso cuando se tiene una réplica biológica; ventajas que también ofrece NOISeqSim (Tarazona et al., 2011). Una ventaja adicional de NPEBseq, sin embargo, es que permite detectar el uso diferencial de exones, o isoformas, para cada gen (útil en organismos eucariotas) (Bi y Davuluri, 2013). Vale la pena resaltar, sin embargo, que el tiempo computacional requerido para correr NPEBseq es mucho mayor respecto a otros programas (entre estos DESeq y NOISeq). Asimismo, NPEBseq y DESeq están diseñados para trabajar con abundancias de lecturas crudas porque la robustez de su estadística depende, en gran medida, de sus métodos de normalización; NOISeq, por otro lado, ofrece diferentes métodos de normalización y no exige datos crudos para empezar (Tabla 3).
CONCLUSIONES
La secuenciación de RNAs (RNA-Seq) brinda un panorama global del transcriptoma de un organismo bajo una condición específica, generando datos transcriptómicos cuantificables a escalas antes no logradas. Sin embargo, la rapidez con la que se generan los datos hoy en día no va al mismo ritmo que los métodos disponibles para su análisis. Esta desigualdad en el ritmo de evolución entre las técnicas y sus métodos de análisis obligan al investigador a buscar siempre una imagen general y amplia de todas las posibilidades dentro de su investigación. Las nuevas herramientas para el análisis, como RNA-seq, surgen con sus propias limitaciones y ventajas. Por esta razón, resulta necesario conocer todos los factores que aseguran el éxito de la técnica, ya que de esto depende el buen uso de la misma. Para el investigador, una visión general como esta le permite sumergirse de una manera guiada dentro de las posibilidades metodológicas para tomar decisiones más acertadas.
En el análisis de datos de RNA-Seq en procariotas existen diversos métodos para llegar a los resultados finales y la escogencia de cada paso influenciará en gran medida el enfoque y la validez de los resultados obtenidos; desde el tipo de librería y el tratamiento de los datos crudos hasta los programas empleados para la identificación de genes diferencialmente expresados. En esta revisión se recopiló información sobre diferentes herramientas disponibles para realizar análisis de datos de RNA-Seq teniendo como base un genoma de referencia, brindando información suficiente para lograr un correcto diseño y análisis experimental de los datos así como alternativas y comparaciones entre aproximaciones estadísticas actualmente utilizadas para la identificación de DEGs.
Aunque este tipo de aplicación es reciente en el campo de la transcriptómica, ofrece grandes ventajas respecto a los microarreglos (evita el ruido introducido por el diseño de sondas, ofrece un panorama global con un mayor rango dinámico de detección y no se restringe a lo conocido). Debido a su eficiencia, resolución y ventajas económicas conforme pasan los años, RNA-Seq se ha convertido en la herramienta más poderosa y versátil de la actualidad para ensayos de expresión génica y estimación de abundancia de transcritos.
AGRADECIMIENTOS
A la Facultad de Ciencias de la Universidad de los Andes y al Laboratorio de Micología y Fitopatología Uniandes (LAMFU).
ANEXOS
Anexo 1. Sugerencias Técnicas Limpieza de lecturas tipo PET. Si se tienen PETs, es recomendable limpiar el rRNA de cada pareja de forma conjunta para evitar eliminar reads de una pareja únicamente. De esta forma también se aprovecha la mayor precisión que ofrecen dos secuencias por lectura y se evitan falsos positivos. Para eliminar todas las lecturas pareadas que alineen contra rRNAs indexados utilizando Bowtie2 se debe especificar el archivo de la primera pareja con -1 <ruta del archivo>, el de la segunda pareja con -2 <ruta del archivo> y guardando todas las lecturas pareadas que no alineen en un solo archivo con --un-conc <ruta destino>. El comando final sería:
bowtie2 -t -x <ruta rRNA indexado> -1 <ruta reads pareja1> -2 <ruta reads pareja2> --un-conc <ruta archivo salida>
Formatos de salida en los alineamientos. Al alinear lecturas contra un genoma de referencia se genera un archivo en formato SAM (Sequence Alignment Map). Este archivo suele ser muy pesado y se puede comprimir mediante código binario en un formato más liviano conocido como BAM (para hacer este tipo de conversión se puede emplear SAMtools disponible en http://samtools.sourceforge.net). Algunos progra-mas encargados de ensamblar transcritos a partir de lecturas alineadas y estimar sus abundancias utilizan archivos BAM de entrada (Cufflinks). Otros, sin embargo, únicamente aceptan archivos SAM (HTSeq-count). De la misma forma, los alineadores suelen tener un formato predeterminado de salida (TopHat produce archivos BAM). En caso de utilizar TopHat y HTSeq-count, por ejemplo, habrá un problema de compatibilidad y SAMtools no permite convertir un archivo BAM en SAM. Por consiguiente, resulta útil saber qué tipo de archivo queremos para poder especificarlo en el alineador que estemos utilizando.
Para evitar que TopHat convierta los resultados SAM en BAM (comportamiento predeterminado) se debe utilizar el comando --no-convert-bam (http://tophat.cbcb.umd.edu) a través de la terminal. También es recomendable especificar el tipo de librería utilizada en el momento de alinear y ensamblar las lecturas; en el caso de TopHat y Cufflinks -- library-type=fr-firststrand corresponde al método dUTP (Trapnell et al., 2012).
Identificación de transcritos novedosos con Cufflinks.
En algunas ocasiones, además de contar con el genoma del organismo estudiado, también se tendrá un archivo de anotación (formato .gtf o .gff). Este archivo suele tener información sobre los genes codificados y resulta muy útil para la identificación de genes diferencialmente expresados porque se puede suministrar como una guía para facilitar el ensamble de transcritos hipotéticos. Para utilizar un archivo de anotación (recomendado si se tiene) en Cufflinks se debe emplear -g o -G. Si únicamente se está interesado en la identificación de transcritos anotados, se debe suministrar el genoma de referencia con -G a través de la terminal; -g permite la identificación adicional de transcritos no anotados, aunque existe el riesgo de asignar transcritos previamente anotados como novedosos (dichos transcritos no serán encontrados en todos los tratamientos). En caso de tener problemas o dudas respecto al formato de anotación (.gff3), existe una aplicación en línea para validar este tipo de archivos (http://modencode.oicr.on.ca/cgi-bin/validategff3online). También se recomienda leer el manual de Cufflinks disponible en la página de los desarrolladores (http://cufflinks.cbcb.umd.edu).
BIBLIOGRAFÍA
Agarwal A, Koppstein D, Rozowsky J, Sboner A, Habegger L, Hillier LW, et al. Comparison and calibration of transcriptome data from RNA-Seq and tiling arrays. BMC Genomics. 2010;11:383. DOI:10.1186/1471-2164-11-383. [ Links ]
Anders S, Huber W. Differential expression analysis for sequence count data. Genome Biol. 2010;11(10):R106. DOI:10.1186/gb-2010-11-10-r106. [ Links ]
Auer PL, Doerge RW. Statistical design and analysis of RNA sequencing data. Genetics. 2010;185(2):405-416. DOI: 10.1534/genetics.110.114983. [ Links ]
Auer PL, Srivastava S, Doerge RW. Differential expression-- the next generation and beyond. Brief Funct Genomics. 2012;11(1):57-62. DOI:10.1093/bfgp/elr041. [ Links ]
Benjamini Y, Hochberg Y. Controlling the false discovery rate: a practical and powerful approach to multiple testing. J R Stat Soc Ser B. 1995;57(1):289-300. Available at: http://www.jstor.org/stable/10.2307/2346101. Accessed February 5, 2014. [ Links ]
Bhargava V, Head SR, Ordoukhanian P, Mercola M, Subramaniam S. Technical Variations in Low-Input RNAseq Methodologies. Sci Rep. 2014;4:3678. DOI:10. 1038/srep03678. [ Links ]
Bi Y, Davuluri RV. NPEBseq: nonparametric empirical bayesian -based procedure for differential expression analysis of RNA-seq data. BMC Bioinformatics. 2013;14:262. DOI:10.1186/1471-2105-14-262. [ Links ]
Bradford JR, Hey Y, Yates T, Li Y, Pepper SD, Miller CJ. A comparison of massively parallel nucleotide sequencing with oligonucleotide microarrays for global transcription profiling. BMC Genomics. 2010;11:282. DOI:10.1186/ 1471-2164-11-282. [ Links ]
Bullard JH, Purdom E, Hansen KD, Dudoit S. Evaluation of statistical methods for normalization and differential expression in mRNA-Seq experiments. BMC Bioinformatics. 2010;11:94. DOI:10.1186/1471-2105-11-94. [ Links ]
Cai G, Li H, Lu Y, Huang X, Lee J, Müller P, et al. Accuracy of RNASeq and its dependence on sequencing depth. BMC Bioinformatics. 2012;13 Suppl 1(Suppl 13):S5. DOI:10. 1186/1471-2105-13-S13-S5. [ Links ]
Chen G, Wang C, Shi T. Overview of available methods for diverse RNA-Seq data analyses. Sci China Life Sci. 2011; 54(12):1121-1128. DOI:10.1007/s11427-011-4255-x. [ Links ]
Dillies M-A, Rau A, Aubert J, Hennequet-Antier C, Jeanmougin M, Servant N, et al. A comprehensive evaluation of normalization methods for Illumina high-throughput RNA sequencing data analysis. Brief Bioinform. 2013;14 (6): 671-683. DOI:10.1093/bib/bbs046. [ Links ]
Fullwood MJ, Wei C-L, Liu ET, Ruan Y. Next-generation DNA sequencing of paired-end tags (PET) for transcriptome and genome analyses. Genome Res. 2009;19(4):521- 532. DOI:10.1101/gr.074906.107. [ Links ]
Giannoukos G, Ciulla DM, Huang K, Haas BJ, Izard J, Levin JZ, et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biol. 2012;13(3):R23. DOI:10.1186/ gb-2012-13-3-r23. [ Links ]
Hashimshony T, Wagner F, Sher N, Yanai I. CEL-Seq: singlecell RNA-Seq by multiplexed linear amplification. Cell Rep. 2012;2(3):666-673. DOI:10.1016/j.celrep.2012.08.003. [ Links ]
He Y, Vogelstein B, Velculescu VE, Papadopoulos N, Kinzler KW. The antisense transcriptomes of human cells. Science. 2008; 322(5909):1855-1857. DOI:10.1126/ science.1163853. [ Links ]
Hoen P, Ariyurek Y, Thygesen HH, Vreugdenhil E, Vossen RH, de Menezes RX, et al. Deep sequencing-based expression analysis shows major advances in robustness, resolution and inter-lab portability over five microarray platforms. Nucleic Acids Res. 2008;36(21): e141. DOI:10.1093/ nar/gkn705. [ Links ]
Kapranov P, Cheng J, Dike S, Nix DA, Duttagupta R, Willingham AT, et al. RNA maps reveal new RNA classes and a possible function for pervasive transcription. Science. 2007;316 (5830):1484-1488. DOI:10.1126/science. 1138341. [ Links ]
Kuo C-H, Ochman H. The fate of new bacterial genes. FEMS Microbiol Rev. 2009;33(1):38-43. DOI:10.1111/j.1574-6976.2008.00140.x. [ Links ]
Langmead B, Salzberg SL. Fast gapped-read alignment with Bowtie 2. Nat Methods. 2012;9(4):357-359. DOI:10.1038/nmeth.1923. [ Links ]
Langmead B, Trapnell C, Pop M, Salzberg SL. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 2009;10(3):R25. DOI:10.1186/gb-2009-10-3-r25. [ Links ]
Levin JZ, Yassour M, Adiconis X, Nusbaum Ch, Thompson D, Friedman N, et al. Comprehensive com-parative analysis of strand-specific RNA sequencing methods. 2010;7(9):709- 715. DOI:10.1038/NMETH.1491. [ Links ]
Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics. 2009;25(14): 1754-1760. DOI:10.1093/bioinformatics/ btp324. [ Links ]
Li J, Tibshirani R. Finding consistent patterns: a nonparametric approach for identifying differential expression in RNASeq data. Stat Methods Med Res. 2011;22(5):519-536. DOI:10.1177/0962280211428386. [ Links ]
Li J, Witten DM, Johnstone IM, Tibshirani R. Normalization, testing, and false discovery rate estimation for RNAsequencing data. Biostatistics. 2012;13(3):523-538. DOI:10.1093/biostatistics/kxr031. [ Links ]
Marioni JC, Mason CE, Mane SM, Stephens M, Gilad Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 2008;18(9):1509-1517. DOI:10.1101/gr.079558.108. [ Links ]
McClure R, Balasubramanian D, Sun Y, Amin V, Oberg AL, Young LJ, et al. Computational analysis of bacterial RNASeq data. Nucleic Acids Res. 2013;41(14):e140. DOI:10. 1093/nar/gkt444. [ Links ]
McIntyre LM, Lopiano KK, Morse AM, Amin V, Oberg AL, Young LJ, et al. RNA-seq: technical variability and sampling. BMC Genomics. 2011;12(1):293. DOI:10.11 86/1471-2164-12-293. [ Links ]
Metzker ML. Sequencing technologies - the next generation. Nat Rev Genet. 2010;11(1):31-46. DOI:10.1038/nrg2626. [ Links ]
Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nat Methods. 2008;5(7):621-628. DOI:10. 1038/nmeth.1226. [ Links ]
Nookaew I, Papini M, Pornputtapong N, Scalcinati G, Fagerberg L, Uhlén M, Nielsen J. A comprehensive comparison of RNA-Seq-based transcriptome analysis from reads to differential gene expression and cross-comparison with microarrays: A case study in Saccharomyces cerevisiae. Nucleic Acids Res. 2012;40(20):10084-10097. DOI:10. 1093/nar/gks804. [ Links ]
Oshlack A, Robinson MD, Young MD. From RNA-seq reads to differential expression results. Genome Biol. 2010; 11(12):220. DOI:10.1186/gb-2010-11-12-220. [ Links ]
Oshlack A, Wakefield MJ. Transcript length bias in RNA-seq data confounds systems biology. Biol Direct. 2009;4:14. DOI:10.1186/1745-6150-4-14. [ Links ]
Ozsolak F, Milos PM. High-Throughput Next Generation Sequencing. 2011;733(11):51-61. DOI:10.1007/978-1- 61779-089-8. [ Links ]
Parkhomchuk D, Borodina T, Amstislavskiy V, Banaru M, Hallen L, Krobitsch S. Transcriptome analysis by strandspecific sequencing of complementary DNA. Nucleic Acids Res. 2009;37(18):e123. DOI:10.1093/nar/gkp596. [ Links ]
Peng Y, Leung HCM, Yiu SM, Chin FYL. IDBA-UD: a de novo assembler for single-cell and metagenomic sequencing data with highly uneven depth. Bioinformatics. 2012; 28(11):1420-1428. DOI:10.1093/bioinformatics/bts174. [ Links ]
Robinson MD, McCarthy DJ, Smyth GK. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 2010;26(1): 139-140. DOI:10.1093/bioinformatics/btp616. [ Links ]
Robinson MD, Oshlack A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biol. 2010;11(3):R25. DOI:10.1186/gb-2010- 11-3-r25. [ Links ]
Robles J, Qureshi SE, Stephen SJ, Wilson SR, Burden CJ, Taylor JM. Efficient experimental design and analysis strategies for the detection of differential expression using RNA-Sequencing. BMC Genomics. 2012;13:484. DOI:10.1186/1471-2164-13-484. [ Links ]
Ruan X, Ruan Y. Transcriptional Regulation. Vancura A, ed. 2012;809. DOI:10.1007/978-1-61779-376-9. [ Links ]
Shendure J, Ji H. Next-generation DNA sequencing. Nat Biotechnol. 2008;26(10):1135-1145. DOI:10.1038/ nbt1486. [ Links ]
Smyth G. Linear Models and Empirical Bayes Methods for Assessing Differential Expression in Microarray Experiments. Stat Appl Genet Mol Biol. 2004;3(1): 1544-6115. [ Links ]
Soneson C, Delorenzi M. A comparison of methods for differential expression analysis of RNA-seq data. BMC Bioinformatics. 2013;14:91. DOI:10.1186/1471-2105- 14-91. [ Links ]
Strickler SR, Bombarely A, Mueller L a. Designing a transcriptome Next-generation sequencing project for a nonmodel plant species. Am J Bot. 2012;99(2):257-66. DOI:10.3732/ajb.1100292. [ Links ]
Tang F, Lao K, Surani M. Development and applications of single-cell transcriptome analysis. Nat Meth. 2011;8:S6-11. [ Links ]
Tarazona S, Furi P, Ferrer A, Conesa A. NOISeq : Differential Expression in RNA-seq. 2013. Available at: http://www.bioconductor.org/packages/release/bioc/vignettes/NOISeq/inst/doc/NOISeq.pdf [ Links ]
Tarazona S, García F, Ferrer A. NOIseq: a RNA-seq differential expression method robust for sequencing depth biases. EMBnet Journal. 2012;17:18-19. DOI:10.1101/ gr.124321.11.2. [ Links ]
Tarazona S, García-Alcalde F, Dopazo J, Ferrer A, Conesa A. Differential expression in RNA-seq: a matter of depth. Genome Res. 2011;21(12):2213-2223. DOI:10.1101/gr. 124321.111. [ Links ]
Trapnell C, Pachter L, Salzberg SL. TopHat: discovering splice junctions with RNA-Seq. Bioinformatics. 2009;25(9):1105- 1111. doi:10.1093/bioinformatics/btp120. [ Links ]
Trapnell C, Williams B A, Pertea G, Mortazavi A, Kwan G, van Baren MJ, et al. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation. Nat Biotechnol. 2010;28(5):511-5. DOI: 10.1038/nbt.1621. [ Links ]
Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley D, et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 2012;7(3):562-578. DOI:10.10 38/nprot.2012.016. [ Links ]
Tucker T, Marra M, Friedman JM. Massively parallel sequencing: the next big thing in genetic medicine. Am J Hum Genet. 2009;85(2):142-154. DOI:10.1016/j.ajhg. 2009.06.022. [ Links ]
Van Verk MC, Hickman R, Pieterse CMJ, Van Wees SCM. RNA-Seq: revelation of the messengers. Trends Plant Sci. 2013;18(4):175-179. DOI:10.1016/j.tplants.2013.02.001. [ Links ]
Wang Z, Gerstein M, Snyder M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 2009;10(1):57-63. [ Links ]
Wu Z, Jenkins BD, Rynearson T Empirical bayes analysis of sequencing-based transcriptional profiling without replicates. BMC Bioinformatics. 2010;11:564. DOI:10.1186/ 1471-2105-11-564. [ Links ]