Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.19 no.3 Bogotá Sept./Dec. 2014
https://doi.org/10.15446/abc.v19n3.42852
Nota Breve
http://dx.doi.org/10.15446/abc.v19n3.42852
IMPLEMENTACIÓN DE LA METODOLOGÍA DE ANÁLISIS DE ADN MITOCONDRIAL EN Rhinoclemmys nasuta (TESTUDINES:GEOEMYDIDAE)
Implementation of DNA Mitochondrial Analysis in Rhinoclemmys nasuta (Testudines: Geoemydidae)
YHERSON FRANCHESCO MOLINA HENAO1, M. Sc.; GUILLERMO BARRETO2, Ph. D.; ALAN GIRALDO1, Ph. D.
1 Universidad del Valle, Facultad de Ciencias Naturales y Exactas, Departamento de Biología, Grupo de Investigación en Ecología Animal. Calle 13 n.° 100-00. Cali, Colombia. yhersonm@univalle.edu.co; alan.giraldo@correounivalle.edu.co
2 Universidad del Valle, Facultad de Ciencias Naturales y Exactas, Departamento de Biología, Grupo de Investigación en Genética Molecular Humana. Calle 13 n.° 100-00. Cali, Colombia. guillermo.barreto@correounivalle.edu.co
Autor de correspondencia: Alan Giraldo, alan.giraldo@correounivalle.edu.co
Recibido 29 de marzo de 2014, aceptado con modificaciones 30 de abril de 2014, fecha de reenvío 15 julio de 2014.
Citation / Citar este artículo como: MOLINA HENAO YF, BARRETO G, GIRALDO A. Implementación de la metodología de análisis de ADN mitocondrial en Rhinoclemmys nasuta (Testudines:Geoemydidae). Acta biol. Colomb. 2014;19(3):507-512.
RESUMEN
Rhinoclemmys nasuta (Testudines: Geoemydidae) es considerada una especie casi endémica de Colombia y la más primitiva del género, sin embargo, se encuentra clasificada por la IUCN como deficiente de datos, ya que la información disponible no es suficiente para hacer una evaluación directa o indirecta de su riesgo de extinción. En este trabajo se describe la implementación del método para realizar la secuenciación de la región control del ADN mitocondrial de R. nasuta, con el propósito de generar herramientas técnicas para futuros estudios de evolutivos y de conservación. Se utilizó el método de desalamiento (Salting-out) para extraer ADN a muestras sanguíneas procedentes de Isla Palma y Playa Chucheros (Bahía Málaga-Pacífico Colombiano) y se utilizó una pareja de cebadores degenerados (reportada para Chrysemys picta Testudines: Emydidae) para realizar la amplificación. Se obtuvieron fragmentos de 800pb siendo exitosa la reacción de secuenciación de los amplificados, y se estableció un porcentaje de homología mayor al 92 % entre las secuencias obtenidas y las secuencias de ADNmt de Sacalia quadriocellata (Testudines:Geoemydidae) y Cuora aurocapitata (Testudines:Geoemydidae), depositadas en el GeneBank. Este resultado demuestra que el método descrito puede ser una herramienta útil para el estudio de las poblaciones de R. nasuta del Pacífico colombiano, lográndose una efectiva secuenciación de la región control del ADNmt de esta especie.
Palabras clave: ADNmt, Colombia, conservación, Neotrópico, tortugas, tortuga río chocoana.
ABSTRACT
Rhinoclemmys nasuta (Testudines:Geoemydidae) is considered an almost endemic specie to Colombia and the most primitive species of Rhynoclemmys. However, it is classified data deficient by IUCN because the available information is not enough to make a direct or indirect assessment of its extinction risk. Here, we describe the implementation of the method to analyze the mitochondrial DNA control sequence (mtDNA) of R. nasuta in order to generate tools for future studies in systematics and population conservation. Genomic mtDNA was extracted by salting-out from blood samples from Isla Palma and Playa Chucheros (Bahía Málaga-Colombian Pacific Coast) and we used a pair of degenerate primers (reported for Chrysemys picta, Testudines:Emydidae) to perform amplification. Fragments of 800pb were obtained and the sequencing reaction was effective. A homology percentage above of 92 % was established between the obtained sequences and mtDNA sequences from Sacalia quadriocellata (Testudines:Geoemydidae) ,and Cuora aurocapitata (Testudines:Geoemydidae) reported in the GenBank.
This result shows that the described method can be a useful tool for the study of R. nasuta populations in the Colombian Pacific region, achieving an effective sequencing of the mtDNA control region of this species.
Keywords: ADNmt, Colombia, conservation, large-nosed wood turtle, neotropical turtles, turtles.
Rhinoclemmys es el único género del linaje Geoemydidae restringido al Neotrópico, por lo que representa un grupo de tortugas de gran interés evolutivo (Spinks et al., 2004). Entre las especies de este género, Rhinoclemmys nasuta (conocida comúnmente como Hicotea, tortuga blanca, Chibigüi, Sabanera o tortuga de río Chocoana) ha sido catalogada como la más primitiva (Carr, 1991), siendo registrada desde la zona media del departamento del Chocó hasta el noroeste de Ecuador, razón por la cual es considerada una especie casi endémica de Colombia (Páez et al., 2012).
Teniendo en cuenta el restringido ámbito de distribución geográfico de R. nasuta y considerando que la tasa de cambio evolutivo es directamente proporcional a la varianza genética aditiva del valor adaptativo de la población (Watson et al. 1936), una reducción en la diversidad genética poblacional tenderá a reducir el potencial evolutivo, y por ende la capacidad de responder ante futuros retos ambientales ya sea de origen natural o humano. Por lo tanto, un problema crítico para el futuro inmediato de las poblaciones de R. nasuta es establecer el patrón de variación genética para poder conocer su salud genómica.
Lamentablemente, la información biológica y ecológica disponible sobre esta especie es reducida. Al punto que aún no se ha podido realizar una evaluación directa o indirecta de su riesgo de extinción (Castaño-Mora y Medem, 2002; Carr et al., 2012). En este contexto, la genética de poblaciones, y especialmente los métodos de biología molecular, puede llegar a proveer herramientas prácticas para los campos de la conservación y la evolución, fortaleciendo las acciones de conservación en este tipo de especies (Hedrick y Miller, 1992; O`Brien, 1994). Algunos de los marcadores moleculares más ampliamente utilizados para este propósito son los del ADN mitocondrial (ADNmt), los cuales proveen información acerca de la estructura poblacional, flujo génico, hibridización, biogeografía y relaciones filogenéticas (Moritz et al., 1987). Además, contiene una región con una alta tasa de evolución, denominada "región control", que es de gran utilidad en los estudios de patrones intraespecíficos de diversidad (Moritz et al., 1987).
Esta aproximación metodológica ha sido ampliamente utilizada para elucidar patrones evolutivos en tortugas y han servido de base para esfuerzos de conservación específicos (e.g. Lamb et al., 1994; Phillips et al., 1996; Caccone et al., 1999; Lenk et al., 1999; Roman et al., 1999; van der Kuyl et al., 2005; Ort y Pogson, 2007). Lamentablemente, estas aproximaciones aún no han sido utilizadas para evaluar los patrones de variación genética en las poblaciones de R. nasuta del Pacífico colombiano debido a que no han sido estandarizadas.
En el presente trabajo se describe el procedimiento para secuenciar la región control mitocondrial de R. nasuta, con el propósito de realizar un análisis genético de sus poblaciones. Para este fin se presenta el protocolo de extracción de ADN, se valida el método de PCR para la amplificación de la región control, se secuencia la región control del ADN mitocondrial de individuos de esta especie y se realiza un análisis preliminar de la diversidad nucleotídica para individuos provenientes de dos localidades del Pacífico colombiano.
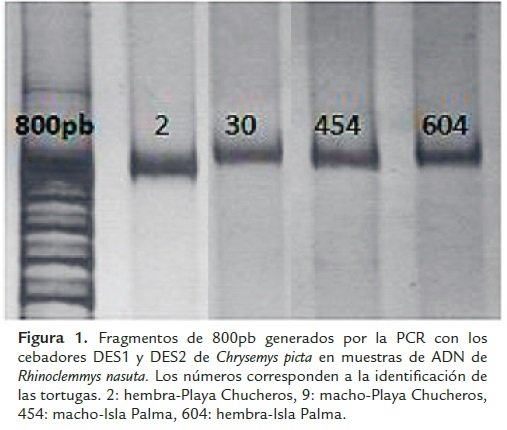
Para este fin, en el año 2007 se recolectaron muestras de sangre periférica (entre 100 μl y 200 μl) de especímenes de R. nasuta provenientes Isla Palma y Playa Chucheros, Bahía Málaga, Pacífico Colombiano (Fig. 1). La sangre se almacenó inmediatamente en tubos de 1,5 mL con una solución de EDTA 0,5M, rotuladas con el número de identidad asignado a la tortuga, el sexo y el sitio de origen, y se preservó en frío hasta su procesamiento en el laboratorio de Genética Molecular Humana de la Universidad del Valle.
Los especímenes utilizados fueron obtenidos durante el desarrollo del programa de monitoreo de las poblaciones de R. nasuta en Bahía Málaga que adelantó el grupo de investigación en Ecología Animal de la Universidad del Valle con la autorización de la Dirección General Marítima de Colombia - DIMAR para el muestreo en Isla Palma, y de la comunidad local para el muestro en Playa Chucheros. Todo el procedimiento se realizó siguiendo los protocolos estándar para la manipulación de especímenes silvestres dispuestos por la autoridad ambiental regional y considerando los lineamientos del comité de ética de la Universidad del Valle.
El ADN genómico fue extraído mediante el método estándar de Salting-out, a partir de 20μL de sangre, a los cuales se adicionó 300 μL de Buffer para lisis (Tris 40 mM, EDTA 20 mM, NaCl 100 mM a pH 7,2), 10 μL de SDS (20 %) y 10μL de Proteinasa K (10 mg/mL). Se realizaron varias inversiones y se procedió a centrifugar a 13.200 rpm durante 2 min, posteriormente se incubó a 55°C durante 16h. Luego, se adicionaron 150μL de Acetato de Amonio (7,5M) y se almacenaron a -20°C durante 30min. A continuación se centrifugó a 13.200rpm durante 20min, se transfirió el sobrenadante a nuevos tubos, a los cuales se adicionó 1mL de etanol frío (100 %), se almacenaron a -20°C durante 30min y se centrifugó a 13.200rpm durante 20 min para precipitar el ADN. En seguida, el etanol fue descartado y se aseguró de que los tubos se secaran completamente. Finalmente, el ADN precipitado fue resuspendido en 50μL de Low TE, conservado a -20°C, y cuantificado utilizando un fluorómetro Qubit®.
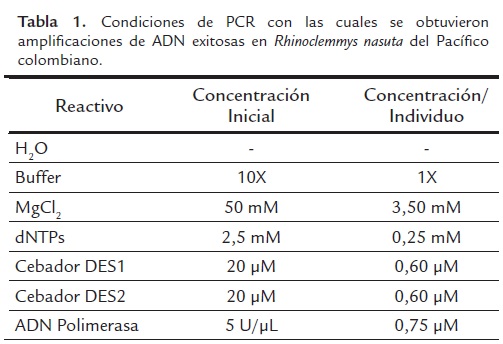
Para la reacción en cadena de la polimerasa (PCR) se tomó como punto de partida el protocolo propuesto por Kocher et al., (1989), con algunas modificaciones (Tabla 1). Se utilizaron los cebadores DES-1 (5´-GCA TTC ATC TAT TTT CCG TTA GCA-3´) y DES-2 (5´-GGA TTT AGG GGT TTG ACG AGA AT-3´) reportados por Starkey et al., (2003), los cuales corresponden a las posiciones 15876-16585 de la secuencia completa del genoma mitocondrial de Chrysemys picta. La visualización de los fragmentos generados se realizó en geles de poliacrilamida al 8 %, con nitrato de plata. La edición de las secuencias se realizó con el programa ChromasPro (Roberts et al., 2003), el alineamiento con el programa Clustal X (Thompson et al., 1997), y se comparó la secuencia consenso con la base de datos del GenBank mediante BLAST. El análisis de las secuencias se realizó con el programa estadístico DnaSP (Rozas et al., 2003) efectuando los cálculos para el número de sustituciones nucleotídicas y el número promedio de diferencias y diversidad nucleotídica. El análisis de composición nucleótica se realizó con el programa Mega 4.0 (Tamura et al., 2007) a partir de un análisis de agrupamiento basado en el método de Máxima Parsimonia (MP).
Se realizó el procedimiento de extracción de ADN por salting out en 48 muestras de R. nasuta provenientes de Isla Palma (24 machos y 24 hembras) y 15 muestras provenientes de Playa Chucheros (ocho machos y siete hembras). La concentración final de ADN estuvo entre 1,0 y 5,0 μg/mL, estableciendo una temperatura de anillamiento de 52 °C con el siguiente perfil: secuencia de temperatura de 94 °C por 3 min, seguida por 35 ciclos de 94 °C por 30 seg, 52 °C por 1 m y 72 °C por 2 min, finalizando a 72 °C por 10 min. Se consideró una amplificación exitosa de ADNmt de R. nasuta cundo se obtuvo una banda de tamaño aproximado de 720 pb, similar a la reportada para Chrysemys picta.
El rendimiento del método fue de 41,27 % siendo los fragmentos resultantes de aproximadamente 800 pb (Fig. 1). A partir de dos muestras aleatorias de cada localidad, se definieron cuatro secuencias (forward), identificando en las muestras de Isla Palma 400 nucleótidos y 470 nucleótidos respectivamente y en las de Playa Chucheros 600 nucleótidos y 700 nucleótidos respectivamente (números de acceso GenBank KJ865844-47). Desde el nucleótido 130 hasta el nucleótido 388 fue la región en la que no se encontró ambigüedad siendo la secuencia consenso resultante: GGAATTTGGACCAGTA TTGGGTTATTTCTTAATTTAGCTAATCACGAGAGATAAGCAACCCTTGTTAGTAAGATACAACATTACCAGTTTCAAGCCCATTAATTGATGGCGTACATAAGTGATCTATTCTGGCCTCTGGTTGTTTTTTCAGGCACATAACATTAATAAAGTTCATTCGTTTCTCTTTAAAAGGCCTCTGGTTAATGTGTTCTATACATTACATTTATAACCTGACATAACTTGCTCTTAAATGCATAT
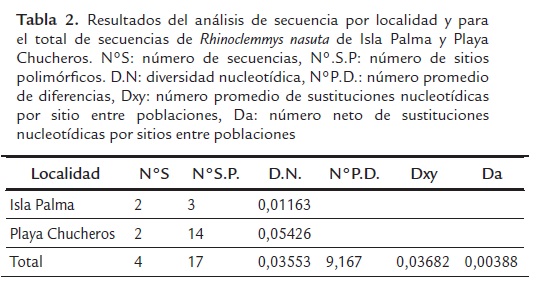
Se estableció una alta homología entre la secuencia consenso y la secuencia de la región control mitocondrial de Sacalia quadriocellata (BLAST-test, código de acceso GeneBank EF088646.1, genoma mitocondrial completo, total score: 370, Query coverage: 99 % , E-value 2e-99, identidades totales: 92 %), y Cuora aurocapitata (BLAST-test, código de acceso GeneBank AY874540.1, genoma mitocondrial completo, total score: 361, Query coverage: 97 % , E-value 1e-96, identidades totales: 93 %), especies de la familia Geoemydidae que habitan en el sur del continente asiático, lo que confirma que mediante el procedimiento desarrollado se amplificó la región control mitocondrial de R. nasuta. Además, se estableció que las secuencias procedentes de Playa Chucheros fueron más polimórficas y presentaron una diversidad nucleotídica aproximadamente cinco veces mayor (razón 14/5) que las establecidas para muestras procedentes de Isla Palma (Tabla 2). No se identificaron inserciones-delecciones, ni sitios con tres o cuatro variantes en las secuencias analizadas, identificándose la posición 110 como un sitio parsimonioso o de diferencia.
La región control en el ADNmt animal es la principal región reguladora y la única área principal no codificante. Esta contiene el origen de la replicación de la cadena pesada (Desjardins y Morais, 1990) y los promotores para la transcripción de las cadenas pesada y liviana (L'Abbé et al., 1991). A pesar de su importancia funcional, se sugiere que la región control es la parte más variable del ADNmt (Aquadro y Greenberg, 1983; Cann et al., 1984). En las tortugas, esta región tiene una longitud aproximada de 1190bp y de acuerdo con Zardoya y Meyer (1998), está localizada entre los genes tRNA-Pro y tRNA-Phe. Análisis de secuencias de la región control han permitido la identificación de un bloque conservado, un bloque conservado putativo y tres secuencias asociadas con la terminación. Además la presencia de seis repeticiones directas localizadas en tandem en el extremo 3`, estando cada repetición compuesta de una secuencia de 45pb seguida por un microsatélite (TA)n con un número variable de unidades repetidas (Zardoya y Meyer, 1998).
Durante los últimos 20 años las técnicas genéticas han sido ampliamente utilizadas para estudios de biología evolutiva y conservación de tortugas (e.g. Lamb et al., 1994; Phillips et al., 1996; Caccone et al., 1999; Lenk et al., 1999; Roman et al., 1999; van der Kuyl et al., 2005; Ort y Pogson, 2007). Aunque los cebadores utilizados en la presente investigación se diseñaron para ser utilizados en C. picta (Starkey et al., 2003), fueron eficientes para amplificar la región control del ADN mitocondrial de R. nasuta, obteniéndose una homología de secuencias superior al 90 % al comparar la secuencia obtenida con secuencias depositadas en el GeneBank para otros miembros de esta familia, como S. quadriocellata o C. aurocapitata.
El primer genoma mitocondrial de tortuga totalmente secuenciado fue el de Pelomedusa subrufa (Zardoya y Meyer, 1998). La organización de este genoma mitocondrial se ajustó al orden génico mitocondrial consenso de vertebrados. Sin embargo, presentó algunas características distintivas como una larga región no codificante separada por los genes ND5 y ND6, un solapamiento entre los genes ATPasa 6 y COIII y la existencia de nucleótidos extra en los ORFs putativos ND3 y ND4L. Aunque el reducido número de secuencias de la región control de R. nasuta establecidas en esta investigación, conllevan a asumir con precaución los resultados del análisis de secuenciación nucleotídica, la menor diversidad registrada para las muestras provenientes de Isla Palma hace necesario evaluar en un futuro cercano si este resultado es solo producto del azar o se han producido eventos demográficos importantes en esta población, como situaciones de cuellos de botella (e.g. Nei, 1987). Probablemente, sea pertinente considerar medidas especiales de manejo para esta población, toda vez que desde 2010 Isla Palma hace parte integral del PNN Uramba (MADVT, 2010) y además alberga la población más grande de esta especie reportada hasta el momento en el Pacífico Colombiano (Giraldo et al., 2012, Garcés-Restrepo et al., 2013).
En conclusión, se logró implementar la metodología de secuenciación de la región control mitocondrial para R. nasuta y se reportó por primera vez la amplificación de dicha región. Además, se confirmó la viabilidad de utilizar los cebadores DES-1 y DES-2 en este procedimiento. De otro lado, los resultados de las cuatro secuencias forward obtenidas permitirían mejorar la especificidad del cebador e incrementar el éxito de la amplificación de la región control en R. nasuta. Es importante destacar que los patrones de variación genética hallados con la utilización de la presente técnica, combinados con datos fisiológicos, ecológicos y etológicos, proveerían una perspectiva multifactorial del estado actual de las poblaciones de R. nasuta. Además, el desarrollo de una investigación filogeográfica aportaría datos valiosos para el desarrollo de planes de manejo y conservación, como también permitiría elucidar patrones evolutivos en esta especie.
AGRADECIMIENTOS
Los autores agradecen a Mario F. Garcés-Restrepo, Johnatan Loaiza, Viviana Perez y Andrés Quintero por el apoyo durante las jornadas de campo. A Brigitte Tose, Leslie Castillo y Fanny L. González por su valioso apoyo durante el trabajo de laboratorio. A Iván, Bernabé Barco y Tarsila de Barco por su hospitalidad y colaboración en Playa Chucheros. A la Dirección General Marítima de Colombia - DIMAR por permitir el ingreso a Isla Palma con el propósito de realizar el monitoreo de la población de Rhinoclemmys nasuta presente en esta localidad. A los habitantes de Playa Chucheros por permitir el ingreso a esta localidad para desarrollar los muestreos de tortugas. Este trabajo fue parcialmente financiado por el grupo de Investigación en Ecología Animal y el Laboratorio Genética Molecular Humana de la Universidad del Valle, e hizo parte de la actividad académica desarrollada por uno de los autores (YFMH) como trabajo de grado en el programa académico de Biología de la Universidad del Valle. Ninguno de los resultados expresados en este documento tiene valor comercial o se puede derivar su uso para el aprovechamiento económico de recursos genéticos.
REFERENCIAS
Aquadro C, Greenberg BD. Human Mitochondrial DNA Variation and Evolution: Analysis of Nucleotide Sequences from Seven Individuals. Genet. 1983;103(2):287-312. DOI: 10.1093/molbev/msn225 [ Links ]
Caccone A, Amato G, Gratry OC, Behler J, Powell JR. A Molecular Phylogeny of four endangered Madagascar tortoises based on mtDNA sequences. Mol Phylogenet Evol.1999;12(1):1-9. DOI: 10.1006/mpev.1998.0594 [ Links ]
Cann RL, Brown WM, Wilson AC. Polymorphic sites and mechanisms of evolution in human mitochondrial DNA. Genet. 1984;106(3):477-499. [ Links ]
Carr JL. Phylogenetic analysis of the neotropical turtle genus Rhinoclemmys Fitzinger (Testudines: Emydidae). (Dissertation). Southern Illinois University. Carbondale; 1991. p 290. [ Links ]
Carr JL, Giraldo A, Garcés-Restrepo MF. Rhinoclemmys nasuta (Boulenger 1902). In: Páez V, Morales-Betancourt MA, Lasso CA, Castaño-Mora OV, Bock B, editors. Biología y Conservación de las Tortugas Continentales de Colombia. Instituto de Investigación de los Recursos Biológicos Alexander von Humboldt (IAvH). Bogotá, Colombia; 2012. p. 315-322. [ Links ]
Castaño-Mora OV, Medem F. Rhinoclemmys nasuta. In: CASTAÑO-MORA OV, editor. Libro Rojo de Reptiles de Colombia. Serie de Libros Rojos de Especie Amenazadas de Colombia. Instituto de Ciencias Naturales-Universidad Nacional de Colombia, Ministerio de Medio Ambiente, Conservación Internacional- Colombia. Bogotá, Colombia; 2002. p. 125. [ Links ]
Desjardins P, Morais R. Sequence and gene organisation of the chicken mitochondrial genome. A novel gene order in higher vertebrates. J Mol Biol. 1990;212:599-634. DOI=10.1007/BF00178867 [ Links ]
Garcés-Restrepo MF, Giraldo A, Carr JL. Population ecology and morphometric variation of the chocoan river turtle (Rhinoclemmys nasuta) from two localities on the Colombian Pacific coast. Bol Cient Mus Hist Nat. 2013;17(2):160-171. [ Links ]
Hedrick PW, Miller PS. Conservation Genetics: Techniques and Fundamentals. Ecol Appl. 1992;2(1):30-46. DOI: 10.2307/1941887 [ Links ]
Kocher TD, Thomas WK, Meyer A. Dynamics of mitochondrial DNA evolution in animals: amplification and sequencing with conserved primers. Proc Natl Acad Sci USA. 1989;86:6196-6200. DOI: 10.1073/pnas.86.16.6196 [ Links ]
Lamb T, Lydeard C, Walker RB, Gibbons JW. Molecular Systematics of Map Turtles (Graptemys):A Comparison of Mitochondrial Restriction Site Versus Sequence Data. Syst Biol. 1994;43(4):543-559. [ Links ]
Lenk P, Fritz U, Joger U, Wink M. Mitochondrial phylogeography of the European pond turtle, Emys orbicularis (Linnaeus 1758). Mol Ecol. 1999;8(11):1911-1922. DOI: 10.1046/j.1365-294x.1999.00791.x [ Links ]
MAVDT. Resolución Número 1501 de Agosto 04 de 2010. "Por medio de la cual se declara, reserva, delimita y alindera el Parque Nacional Natural Uramba Bahía Málaga". Ministerio de Ambiente, Vivienda y Desarrollo Territorial. Bogotá; 2010. p. 17. [ Links ]
Moritz C, Dowling TE, Brown WM. Evolution of Animal Mitochondrial DNA: Relevance for Population Biology and Systematics. Annu Rev Ecol Syst. 1987;18:269-292. DOI: 10.1146/annurev.es.18.110187.001413 [ Links ]
Nei M. Molecular Evolutionary Genetics. Columbia Univ. Press, New York; 1987. p 512. [ Links ]
O'Brien SJ. A role for molecular genetics in biological conservation. Proc Natl Acad Sci USA. 1994;91:5748-5755. DOI: 10.1073/pnas.91.13.5748 [ Links ]
Ort BS, Pogson GH. Molecular Population Genetics of the Male and Female Mitochondrial DNA Molecules of the California Sea Mussel, Mytilus californianus. Genet. 2007;177:1087-1099. DOI: 10.1534/genetics.107.072934 [ Links ]
Páez V, Morales-Betancourt MA, Lasso CA, Castaño-Mora OV, Bock B. editores. Biología y Conservación de las Tortugas Continentales de Colombia. Instituto de Investigación de los Recursos Biológicos Alexander von Humboldt (IAvH), Bogotá, Colombia. 2012. p. 528. [ Links ]
PHILLIPS CA, DIMMICK WW, CARR JL. Conservation Genetics of the Common Snapping Turtle (Chelydra serpentina). Conserv Biol. 1996;10(2):397-405. [ Links ]
Roman J, Santhuff SD, Moler PE, Bowen BW. Population Structure and Cryptic Evolutionary Units in the Alligator Snapping Turtle. Conserv Biol. 1999;13(1):135-142. DOI: 10.1046/j.1523-1739.1999.98007.x [ Links ]
Rozas J, Sánchez-Delbarrio JC, Messeguer X, Rozas R. DnaSP V5, DNA polymorphism analyses by the coalescent and other methods. Bioinforma. 2003;19:2496-2497. DOI: 10.1093/bioinformatics/btp187 [ Links ]
Starkey DB, Shaffer HB, Burke RL, Forstner MRJ, Iverson JB, Janzen FJ, et al. Molecular Systematics, Phylogeography, and the Effects of Pleistocene Glaciation in the Painted Turtle (Chrysemys picta) Complex. Evol. 2003;57(1):119-128. DOI: 10.1111/j.0014-3820.2003.tb00220.x [ Links ]
Spinks PQ, Shaffer HB, Iverson JB, McCordd WP. Phylogenetic hypotheses for the turtle family Geoemydidae. Mol Phylogenet Evol. 2004;32:164-182. DOI: 10.1016/j.ympev.2003.12.015 [ Links ]
Tamura K, Dudley J, Nei M, Kumar S. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Mol Biol Evol. 2007;24:1596-1599. DOI: 10.1093/molbev/msm092 [ Links ]
Thompson JD, Gibson TG, Plewniak F, Jeanmougin F, Higgins DG. The ClustalX windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Res. 1997;24:4876-4882. DOI: 10.1093/nar/25.24.4876. [ Links ]
Van Der Kuyl AC, Ballasina DLP, Zorgdrager F. Mitochondrial haplotype diversity in the tortoise species Testudo graeca from North Africa and the Middle East. BMC Evol Biol. 2005;5:29. DOI: 10.1186/1471-2148-5-29. [ Links ]
Watson DMS, Timofeeff-Ressovsky NW, Salisbury EJ, Turrill WB, Jenkin TJ, Ruggles Gates R, et al. A Discussion on the Present State of the Theory of Natural Selection. Proc R Soc Lond B Biol Sci. 1936;121(820):43-73. [ Links ]
Zardoya R, Meyer A. Complete mitochondrial genome suggests diapsid affinities of turtles. Proc Nat Acad Sci USA. 1998;95:14226-14231. [ Links ]