Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.20 no.2 Bogotá May/Aug. 2015
https://doi.org/10.15446/abc.v20n2.42875
Doi: http://dx.doi.org/10.15446/abc.v20n2.42875
Artículo de investigación
EVALUACIÓN DE LA EXPRESIÓN DE GENES IMPLICADOS EN LA BIOSÍNTESIS DE ALMIDÓN EN DIFERENTES VARIEDADES DE YUCA
Expression Assessment of Starch Biosynthesis-Related Genes in Different Cassava Varieties
Simón CORTÉS SIERRA1, Paul CHAVARRIAGA2, Hernan CEBALLOS2, Camilo LÓPEZ CARRASCAL1.
1Grupo Manihot Biotec, Departamento de Biología, Facultad de Ciencias, Universidad Nacional de Colombia. Bogotá, Colombia.
2Plataforma de Transformación Genética, Centro Internacional de Agricultura Tropical, CIAT. Km 17 Recta Cali. Palmira, Colombia.
For correspondence. celopezc@unal.edu.co
Received: 31 March 2014; Returned for revision: 13 May 2014; Accepted: 12 October 2014.
Associate Editor: Adriana Almeida.
Citation / Citar este artículo como: Cortés Sierra S, Chavarriaga P, Ceballos H, López Carrascal C. Evaluación de la expresión de genes implicados en la biosíntesis de almidón en diferentes variedades de yuca. Acta biol. Colomb. 2015;20(2):37-46. doi: http://dx.doi.org/10.15446/abc.v20n2.42875
RESUMEN
Las raíces almacenadoras de yuca representan una fuente importante de almidón. La ruta metabólica del almidón ha sido reconstruida recientemente en yuca gracias a la liberación de la secuencia completa de su genoma. En este estudio se evaluó la expresión de los genes que codifican para las enzimas pululanasa, isoamilasa, α-amilasa, enzima desproporcionante, ADP-glucosa pirofoforilasa, almidón sintasa unida al gránulo, enzima ramificante del almidón y sintasa soluble del almidón, en las raíces almacenadoras de plantas de cinco y 11 meses de edad, en un grupo de cinco variedades de yuca. Se evidenciaron diferencias importantes en la expresión de estos genes entre las variedades evaluadas y entre los dos tiempos. Las variedades CM523-7 y SM1219-2 presentaron uno de los niveles más altos de expresión para los genes ADP-glucosa pirofoforilasa y almidón sintasa unida al gránulo mientras que el gen para α-amilasa fue el más bajo en estas dos variedades. Aunque la variedad TMS60444 presentó niveles de expresión similares en genes implicados en la síntesis de almidón, fue la que presentó el mayor nivel de expresión de la α-amilasa. Estos datos se pueden correlacionar con el relativo bajo contenido de materia seca en esta variedad. Los datos de expresión génica presentados en este trabajo permitirán complementar información sobre actividad enzimática con miras a identificar los elementos más importantes en la acumulación diferencial de almidón entre variedades de yuca.
Palabras clave: expresión diferencial, metabolismo, qRT-PCR.
ABSTRACT
Cassava storage roots represent an important starch source. Recently, the starch metabolic pathway in cassava has been reconstructed thanks to the full release of its genome. In this study gene expression was evaluated for genes coding pullulanase, isoamylase, α-amylase, deproportionating enzyme, ADP-glucose pyrophosphorylase, granule bound starch synthase, starch branching enzyme and soluble starch synthase, in cassava storage roots five and 11 months old, in five cassava varieties. Important gene expression differences were detected both at the variety and time level. CM523-7 and SM1219-2 showed one of the highest expression levels for AGPase and GBSS genes, while α-amylase showed the lowest level in these two varieties. TMS60444 variety showed similar expression levels in starch biosynthesis-related genes, but conversely also showed the highest α-amylase expression. This correlates with the relative low dry-matter content in TMS60444. Gene expression data reported here will allow complementing actual information on enzymatic activity, in order to identify the most relevant factors in differential starch accumulation between cassava varieties.
Keywords: differential expression, metabolism, qRT-PCR.
INTRODUCCIÓN
La yuca (Manihot esculenta Crantz) representa una de las fuentes más importantes de calorías para la población mundial, considerándose como un producto de seguridad alimentaria (Howeler et al., 2013). La yuca ha sido tradicionalmente considerada como un alimento básico, con calidad nutricional moderada, en especial la raíz almacenadora se resalta como fuente de almidón, pues cerca del 90 % de su peso seco lo constituyen carbohidratos (El-Sharkawy, 2004; Ospina et al., 2002). El almidón presente en las raíces de la yuca no sólo es un alimento básico para la población humana sino que también constituye la materia prima para elaborar concentrados comerciales para animales, para los fabricantes de papel y de textiles, y para la industria de alimentos procesados y la farmacéutica (Howeler et al., 2013). Actualmente las raíces de yuca se emplean también para la producción de bioetanol, con estimados de producción de hasta 280 L de etanol al 96 % a partir de una tonelada de yuca con un 30 % de almidón (Howeler et al., 2013). El porcentaje de almidón almacenado en las raíces de yuca puede variar entre el 65 % a 90 % del total de materia seca (Munyikwa et al., 1997). Para que el almidón de yuca logre un mejor posicionamiento en el mercado y un mayor impacto industrial, se requiere contar con una gama variable de almidones con características diferentes definidas. Al mismo tiempo es necesario disponer de variedades que presenten mayores contenidos de almidón en sus raíces y altos rendimientos de producción. Estas características están definidas de manera directa o indirecta por la estructura y expresión de los genes responsables de la biosíntesis de almidón.
El almidón se acumula en forma de gránulo en los amiloplastos de los tejidos almacenadores como raíces o tubérculos, como también en los cloroplastos en los tejidos fotosintéticos. El almidón es un glucano insoluble que se forma por medio de enlaces glucosídicos tipo α de monómeros de glucosa. El almidón se compone de dos polisacáridos, la amilosa y la amilopectina, los cuales representan entre el 98-99 % del peso seco del gránulo de almidón. Las propiedades y la proporción de cada uno de los polisacáridos en el gránulo de almidón determinan las características de los almidones de cada una de las fuentes vegetales. (Ballicora et al., 2004; Munyikwa et al., 1997). Los dos polisacáridos se diferencian por la longitud de la cadena y los tipos de enlaces, ya que en la amilosa son cadenas lineares de 100 a 10.000 residuos glucosil unidos por enlaces α-1,4; mientras que la amilopectina está formada por cadenas más cortas unidas por enlaces α-1,4, y adicionalmente presenta enlaces α-1,6 responsables de los puntos de ramificación. Dichos puntos se encuentran cada 15 a 30 unidades de glucosa (Baguma et al., 2003; Zeeman et al., 2010). En el almidón de yuca, como en varias especies, la amilosa sólo llega a representar entre el 15 % al 25 %, mientras que la amilopectina representa entre el 70 % al 80 % (Raemakers et al., 2005; Sánchez et al., 2009).
Las enzimas responsables de la biosíntesis del almidón no son muchas. La primera enzima que actúa en el amiloplasto, es la Adenosina difosfato glucosa pirofosforilasa (AGPasa), la cual partiendo de ATP y glucosa-1-fosfato genera ADP- glucosa, que es el substrato a partir del cual se sintetizan los dos polisacáridos. La enzima sintasa de almidón ligada al granulo de almidón (GBSS, por sus siglas en inglés Granule Bound Starch Synthase), cataliza la síntesis de amilosa por medio de la adición de ADP-glucosa al extremo no reductor de la cadena linear de glucanos preexistentes por medio de uniones α-1,4. (Zeeman et al., 2010). La amilopectina se sintetiza por medio de la acción de la enzima ramificadora de almidón (SBE por sus siglas en inglés Starch Branching Enzyme), la cual introduce puntos de ramificación sobre las cadenas lineares cada 15 a 20 unidades de glucosa del extremo no reductor, por medio de uniones α-1-6 (Zeeman et al., 2010). Adicionalmente a la SBE, las sintasas de almidón solubles (SSS, por sus siglas en inglés Soluble Starch Synthase), participan en la formación de amilopectina. Otro grupo de enzimas importantes en la biosíntesis de almidón es el de las enzimas desramificantes de almidón (DBEs por sus siglas en inglés DeBranching Enzymes). Estas enzimas son del tipo α-1,6-glucanohidrolasas, las cuales cortan puntos de ramificación de la amilopectina. En plantas se presentan dos tipos de DBEs, las isoamilasas (ISO) y las pululanasas (PUL) (Zeeman et al., 2010). Por otra parte se ha determinado que enzimas cuyo papel principal es la degradación de almidón, también juegan un papel importante en su metabolismo. Este es el caso de la enzima desproporcionante del almidón (DPE por sus siglas en inglés Deproportionating Enzyme), una α-1,4 glucanotransferasa que cataliza modificaciones en el tamaño de las cadenas de amilopectina (Bresolin et al., 2006). Finalmente la α-amilasa (AMY) también puede estar involucrada en el control de la estructura del gránulo de almidón (Bresolin et al., 2006).
Las diferentes enzimas y la ruta metabólica de síntesis de almidón ha sido bien descrita en plantas modelo como Arabidopsis y en cereales (Sonnewald y Kossmann, 2013). En yuca, hasta hace poco, sólo algunos de los genes responsables habían sido descritos. Sin embargo con la reciente liberación de la secuencia completa del genoma (Prochnik et al., 2012) fue posible reconstruir la ruta metabólica completa (Saithong et al., 2013). Este estudio mostró que existe un alto grado de conservación en los genes y, en general, en toda la ruta metabólica del almidón de yuca con otras especies como Arabidopsis, arroz, maíz, ricino y papa. Por consiguiente las propiedades contrastantes en el almidón de varias especies, así como las pequeñas diferencias presentes dentro de variedades de la misma especie pueden ser producto de la existencia de diferentes isoformas de cada una de las enzimas implicadas como también de la expresión génica diferencial (Chen et al., 2012). En yuca se ha reportado el perfil de expresión de genes durante el proceso de engrosamiento de las raíces, inicialmente mediante estrategias de ADNc-AFLP (Sojikul et al., 2010) y recientemente mediante el empleo de microarreglos (Yang et al., 2011). Si bien estos estudios han permitido comprender el proceso de tuberización y acumulación de almidón, ningún estudio se ha focalizado en la evaluación de la expresión de genes en las raíces en diferentes variedades de yuca.
La yuca posee una diversidad importante, que puede ser explotada para identificar variedades altamente productivas y adaptadas a condiciones agroecológicas particulares y con almidones diferenciales. La Colección de Germoplasma de yuca ubicada en el CIAT está representada por cerca de 6600 entradas que incluyen 5709 clones de M. esculenta en los que se encuentran clones primitivos y cultivares mejorados, y 883 accesiones correspondientes a variedades silvestres (isa.ciat.cgiar.org/urg/cassavacollection.do). Esta colección de Germoplasma ha sido evaluada, al menos parcialmente, mediante técnicas moleculares, a través del empleo de AFLP y microsatélites (Roa et al., 1997; Fregene et al., 2000) .
En este estudio se realizó un análisis comparativo de la expresión de un conjunto de genes seleccionados implicados en el metabolismo de almidón en un grupo de variedades de yuca, logrando identificar patrones de expresión génica diferencial para algunos genes en las variedades de yuca evaluadas.
MATERIALES Y MÉTODOS
Material vegetal
Plantas de yuca de las variedades listadas en la Tabla 1 fueron crecidas en condiciones de campo en la estación experimental del CIAT, a partir de estacas de variedades contenidas en el banco de germoplasma del CIAT. Se crecieron al menos cinco plantas por cada variedad. De cada planta se realizó una extracción de las raíces, en el mismo momento del día, a los cinco y 11 meses de cultivo. Las raíces se almacenaron directamente en nitrógeno líquido. A los cinco meses las raíces almacenadoras ya presentan un porte apto para su cosecha, mientras que a los 11-12 meses se suele realizar una segunda cosecha en la que las raíces presentan un incremento en peso.
Extracción de ARN, síntesis de ADNc
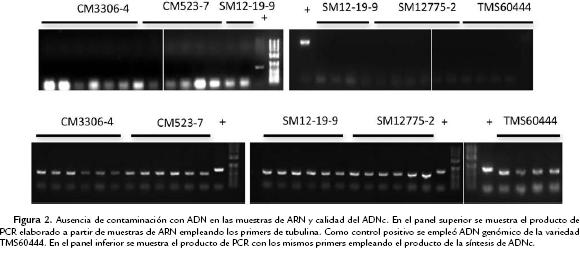
Se tomaron tres plantas de yuca por cada variedad estudiada, a los cinco y a los once meses de cultivo, y de cada planta se tomaron dos raíces almacenadoras para la extracción de ARN. El tejido macerado se resuspendió por agitación vigorosa en buffer de extracción precalentado a 65 °C (acetato de sodio 100mM (pH 6.1), EDTA 1mM, SDS 4 %). Se adicionó un volumen de fenol ácido (pH 4.3):cloroformo:alcohol isoamílico (24:24:1) y se mezcló por inversión. Posterior a la centrifugación (10500 rpm por cinco min) se recuperó el sobrenadante. Se repitió la extracción con fenol ácido dos veces. Se agregó un volumen de cloroformo:alcohol isoamílico (24:1) y se procedió a una nueva extracción. Finalmente el sobrenadante se precipitó con una solución de cloruro de litio hasta alcanzar una concentración final de 2M. Se incubó toda la noche a ‐20°C y se precipitó a 10500 rpm por diez minutos a 4 °C. El pellet se lavó con etanol al 70 %. El ARN se resuspendió en agua tratada con DEPC. Se verificó la calidad del ARN a través de electroforesis en gel de agarosa al 1,2 % teñido con bromuro de etidio y nanodrop (BioRad). Las muestras fueron tratadas con DNAsa para eliminar cualquier contaminación con ADN genómico, siguiendo las instrucciones del fabricante. Para confirmar la ausencia de ADN genómico se llevó a cabo un PCR sobre las muestras de ARN tratadas empleando los primers correspondientes al gen de tubulina. La síntesis de ADNc se llevó a cabo empleando el kit Fermentas y siguiendo las recomendaciones del fabricante.
qRT-PCR
Se evaluó la expresión de varios genes implicados en el metabolismo de almidón (Tabla 2). Los primers se diseñaron a partir de las secuencias de estos genes reportadas para el genoma de yuca (http://www.phytozome.net/cassava) empleando el programa primer3 (http://bioinfo.ut.ee/primer3-0.4.0/) las secuencias de los primers se listan en la Tabla 2. Se realizaron diluciones seriadas del ADNc para ser empleado como molde en las reacciones de qRT-PCR, según recomendaciones del fabricante. Para las reacciones de QRT-PCR se utilizó el kit SYBR GreenER™ qPCR SuperMix Universal (Life Technologies) y se siguieron las recomendaciones del fabricante. Las reacciones de qRT-PCR se llevaron a cabo en un termociclador con sistema de detección de PCR en tiempo real DNA Engine Opticon 2 de BIORAD. Se utilizó el software asociado de montaje y análisis de datos MJ Opticon Monitor analysis. A partir de las diluciones seriadas de ADNc se realizaron curvas estándar para calcular la eficiencia de los primers. En todos los casos la eficiencia de los primers usados fue cercana al valor óptimo. Se determinó la cantidad relativa de la expresión de cada gen de interés respecto al gen de referencia de la tubulina, calculando el ΔCt respectivo, en ensayos por duplicado por variedad por tiempo donde ΔCT = CT del gen blanco – CT del gen referencia. Se realizaron comparaciones pareadas entre variedades y tiempos utilizando el método de cuantificación relativa de doble delta Ct (2-ΔΔCT) donde: ΔΔCT = ΔCT muestra blanco – ΔCT muestra control, y para encontrar diferencias significativas de expresión diferencial entre muestras, se utilizaron los métodos de aleatorización y bootstrap, implementados en el software REST (http://rest.gene-quantification.info/).
Análisis de agrupamiento
Los valores de expresión de los genes en las variedades y tiempos fueron agrupados con el método de clusters jerárquicos usando la métrica de correlación no centrada, implementado en el software cluster 3.0 (de Hoon et al., 2004). Las gráficas de agrupamiento se realizaron con JTreeView (Saldanha, 2004).
RESULTADOS
Aspectos generales de la expresión de genes de almidón
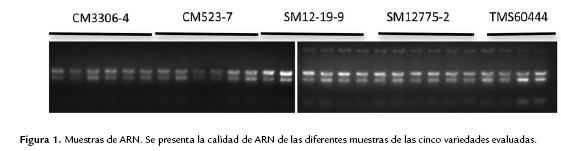
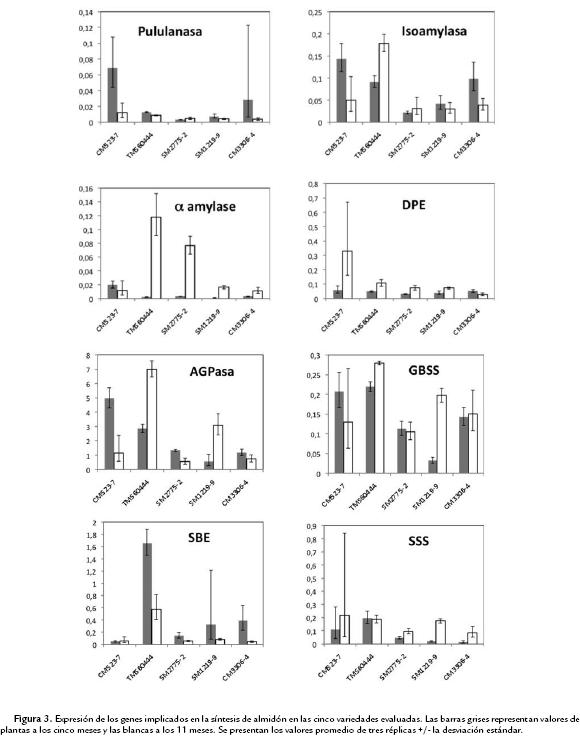
La expresión de los genes que codifican para varias enzimas implicadas en el metabolismo del almidón fue evaluada a partir de ARN extraído de raíces de cinco variedades de yuca en plantas de cinco y 11 meses. La extracción de todas las muestras fue efectiva y eficiente (Fig. 1) lo que permitió la evaluación del grupo completo de genes en todas las variedades. La calidad del ARN y ADNc fue evaluada no solamente mediante gel (Fig. 1) sino que se determinó la calidad y ausencia de contaminación de las muestras con ADN genómico mediante PCR (Fig. 2). La expresión de los genes fue normalizada empleando la expresión del gen de tubulina, por esta razón se presentan los valores como expresión relativa y son adimensionales. En algunos casos particulares se presentaron variaciones bastante grandes en los valores de expresión génica para genes particulares en ciertas variedades. Sin embargo este fenómeno no se observó de manera consistente ni para un gen o variedad en particular. Sin considerar el tiempo de las plantas e independientemente de la variedad evaluada, se puede observar que los niveles de expresión génica para los genes AGPasa y SBE fueron los que presentaron mayores valores de expresión relativa cuando se compara con los otros genes (Fig. 3). Los valores en la mayoría de los casos estuvieron por encima de 2, y alcanzaron valores de hasta 5 u 8 para AGPasa. Para SBE si bien los valores no fueron tan altos, si estuvieron cercanos a 1 en muchos casos. PUL fue el que menor expresión presentó con valores que difícilmente alcanzaban 0,1. Para ISO, AMY, DPE y SSS los valores estuvieron alrededor de 0,3 (Fig. 3).
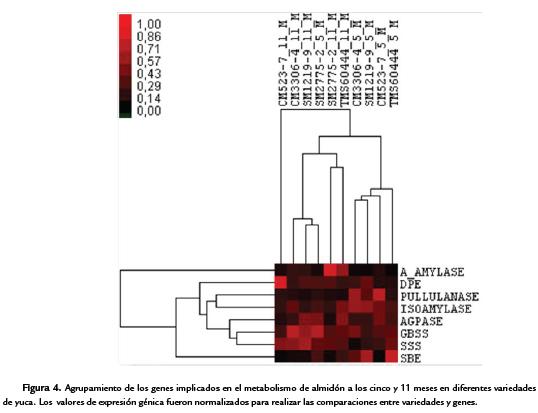
Para encontrar tendencias generales en la expresión de los genes de almidón estudiados en las diferentes variedades y tiempos, se realizó un análisis de agrupamiento mediante correlaciones jerárquicas basadas en los valores de expresión (Fig. 4). De esta forma se encontró que la expresión general de los genes analizados tiende a agruparse respecto al tiempo. Es decir, la expresión global de estos genes a los cinco meses es similar en la mayoría de las variedades y diferenciable de su expresión a los 11 meses (Fig. 4). Esto podría indicar que en el caso de las variedades estudiadas, el tiempo de madurez de las raíces tiene un mayor efecto en el perfil genético de expresión de genes del metabolismo del almidón de manera similar en todas las variedades. En cuanto al agrupamiento de genes según su perfil de expresión, se encontró que en general aquellos genes implicados en la biosíntesis de almidón como AGPasa, GBSS y SSS se agruparon, pero presentaron un perfil de expresión similar a los genes para las enzimas de degradación de almidón como la PUL e ISO (Fig. 4). Estos resultados sugieren un papel complementario de estas enzimas para la biosíntesis de almidón. Por otro lado, AMY y DPE presentaron perfiles de expresión menos parecidos al grupo anterior, mientras que el perfil de expresión de SBE no se agrupó con el de ningún otro gen, indicando un posible papel heterogéneo en la biosíntesis de almidón en las variedades estudiadas.
Expresión diferencial en el tiempo
En términos generales no se observa una tendencia bien definida a que la expresión de los genes esté afectada a lo largo del tiempo (plantas de cinco y 11 meses de cultivo) independientemente de la variedad de yuca analizada. Sin embargo sí se observa una ligera propensión a que los genes AMY, AGPasa y GBSS tengan niveles más altos de expresión a los 11 meses en diferentes variedades o al menos equivalente al que presentan a los cinco meses (Fig. 3). Es evidente que existe una expresión casi nula de AMY a los cinco meses, lo cual contrasta con niveles relativamente altos a los 11 meses. Por el contrario, PUL, ISO y SBE presentaron valores de expresión ligeramente superiores a los cinco que a los 11 meses (Fig. 3).
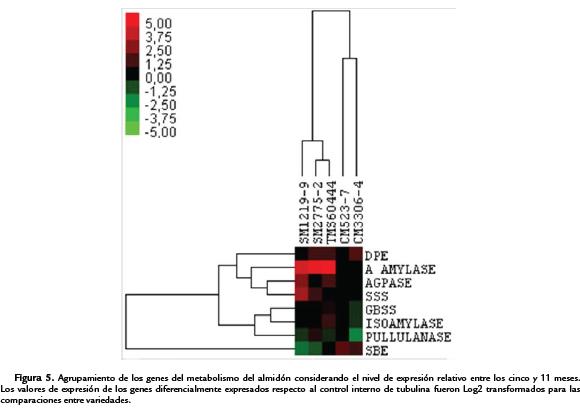
Para observar tendencias en la expresión diferencial de los genes estudiados a los 11 meses respecto a los cinco meses en las variedades empleadas, se realizó otro análisis de agrupamiento jerárquico, esta vez utilizando sólo los datos de expresión diferencial. Para este análisis los valores de expresión de los genes significativamente inducidos o reprimidos a los 11 meses respecto a los cinco meses fueron considerados, mientras que aquéllos cuyo cambio en expresión no fue estadísticamente significativo se tomó como cero (Fig. 5). A partir de este análisis de agrupamiento se encontró que las variedades SM1219-9 y SM2775-2 tienen perfiles de inducción de la expresión de los genes de biosíntesis de almidón diferenciables de los de las variedades CM523-7 y CM3306-4. Adicionalmente se puede observar que el perfil de expresión de la variedad TMS60444 es más cercano a las variedades SM (SM1219-9 y SM2775-2) que a las CM (CM523-7 y CM3306-4). Esto da indicios de que el nivel de inducción diferencial de genes de almidón puede poseer un componente dependiente de la variedad.
Respecto al agrupamiento de genes inducidos o reprimidos diferencialmente, se conservó la correlación entre la expresión de AGPase y SSS, pero esta vez estuvo más relacionada con la expresión de los genes para la α-amilasa y la DPE que con la de GBSS, PUL e ISO (Fig. 5). La expresión de SBE presenta un perfil particular pues es significativamente inducida en las variedades CM mientras que es reprimida en las SM. Dado el papel de la SBE en el contenido diferencial de amilopectina, esta diferencia en inducción entre variedades podría ser un indicador de la diferencia de la estructura del almidón de las variedades CM y SM. Igualmente es notable la fuerte inducción de AMY en el agrupamiento de las variedades SM y TMS60444.
Diferencias entre variedades
El análisis general de la expresión de los diferentes genes en las variedades de yuca empleadas permitió poner en evidencia que existe una fuerte variación entre ellas. Para PUL, las variedades CM523-7 y CM3306-4 presentaron niveles más altos que las otras variedades. ISO se expresó con mucha más fuerza en las variedades CM523-7 y TMS60444 destacándose el alto nivel de expresión para este gen en TMS60444 a los 11 meses. El nivel de expresión en esta variedad a los 11 meses fue casi tres veces mayor que en las otras variedades de yuca. Nuevamente la variedad CM523-7 junto con SM1219 y CM3306-4 se destacaron como las que presentaron los niveles más bajos de expresión para AMY (Fig. 3). Sin embargo, como se mencionó anteriormente, sí se evidenció una tendencia similar en todas las variedades a que se presentara un incremento en la expresión de este gen a los 11 meses. Las variedades de yuca evaluadas mostraron un perfil de expresión muy diferente para el gen AGPasa tanto a los cinco como a los 11 meses. Las variedades CM523-7 y TMS60444 mostraron valores muy altos a los cinco meses, sin embargo mientras que a los 11 meses el nivel de expresión se elevó en la variedad TMS60444, en CM523-7 la expresión fue apenas detectable. Un comportamiento similar se observó en las variedades SM2775-2, SM1219-2 y CM3306-4 en los dos tiempos evaluados (Fig. 3).
Todas las variedades mostraron un perfil de expresión muy similar en los genes DPE y SSS tanto a los cinco como a los 11 meses, situación que también se evidenció en la expresión del gen GBSS a los 11 meses. Sin embargo la variedad SM1219-2 se diferenció claramente de las otras por su baja expresión en el gen GBSS a los 5 meses. Por otro lado, la variedad TMS60444 se diferenció claramente de las otras por su alto nivel de expresión en el gen SBE tanto a los cinco y 11 meses, mientras que las otras variedades presentaron patrones de expresión muy similar para este gen a los dos tiempos evaluados (Fig. 3).
DISCUSIÓN
En este estudio se pudo poner en evidencia las diferencias importantes que existen en la expresión de varios genes implicados en el metabolismo de almidón entre variedades de yuca durante el ciclo del cultivo. Mediante RT-PCR en tiempo real se pudo observar que de manera general las diferentes variedades tienen diferentes patrones en los cambios de expresión de los genes estudiados respecto a la edad de las plantas (11 meses respecto a cinco meses), lo cual puede reflejar una diferencia global en el metabolismo de almidón respecto al tiempo en raíces de yuca.
En este trabajo se observó una variación importante en la expresión de ciertos genes en algunas muestras evaluadas, a pesar de las diferentes réplicas técnicas y biológicas que se emplearon encaminadas a disminuir la variabilidad biológica. Varios estudios han demostrado que la expresión de genes relacionados con el metabolismo del almidón es muy susceptible de ser modificada por leves variaciones ambientales (Martin y Smith, 1995; Baguma, 2004; Baguma et al., 2008). Es posible que la variación entre réplicas observada se explique por pequeños cambios ambientales o condiciones de suelo muy particulares en el cual se encontraban las plantas al momento de realizar la colecta del material para la extracción de ARN. Sin embargo y a pesar de esta variabilidad, en muchos de los casos se pudo observar un patrón de expresión génica diferencial en varios genes entre las variedades evaluadas.
La expresión de genes implicados en el metabolismo de almidón ha sido ampliamente estudiada en diferentes especies vegetales. En los años 90s los trabajos estuvieron dedicados a la identificación de cada uno de los genes y al estudio de su expresión de manera individual por técnicas como RT-PCR o Northern-blot (Martin y Smith, 1995; Munyikwa et al., 1997; Salehuzzaman et al., 1999). Recientemente se han llevado a cabo estudios de expresión global empleando microarreglos o ARN-seq (Yang et al., 2011; Firon et al., 2013). La suma de estos trabajos ha permitido identificar una compleja red de regulación en los genes implicados tanto en el metabolismo como catabolismo del almidón. Se ha puesto en evidencia que la regulación de la expresión de genes de almidón se ve afectada por el ritmo circadiano y la transición día/noche (Baguma et al., 2008; Sonnewald y Kossmann, 2013). Sin embargo no se evidenciaron patrones de expresión coordinada entre los genes y la actividad de las enzimas codificadas por estos genes, sugiriendo la existencia de mecanismos de regulación postranscripcional regulando no solo la transcripción sino la actividad de las enzimas (Smith et al., 2004). Algunos estudios han mostrado un incremento en la expresión de algunos genes de la síntesis de almidón durante el proceso de engrosamiento de las raíces o del ciclo de cultivo (Munyikwa et al., 1997; Baguma et al., 2003; Yang et al., 2011; Firon et al., 2013), lo cual se corresponde al incremento en la expresión de genes como AGPasa, GBSS y SSS en plantas de cinco a los 11 meses visto en la mayoría de las variedades de yuca evaluadas.
A pesar de los importantes esfuerzos que se han dado por comprender la regulación de la expresión y actividad de las enzimas implicadas en el metabolismo del almidón, ningún estudio ha realizado un análisis comparativo de la expresión de genes en variedades que presentan características diferenciales en el contenido y/o estructura de almidón. En este trabajo se logró identificar patrones de expresión génica diferencial entre variedades de yuca para algunos de los genes de almidón, los cuales se pueden correlacionar al menos parcialmente con el contenido de materia seca. De manera interesante las variedades CM523-7 y SM1219-2 fueron las que presentaron los más bajos niveles de expresión para algunos de los genes implicados en la degradación de almidón (α-amilasa e isoamilasa) y niveles relativamente similares en los genes de biosíntesis de almidón, lo cual permitiría explicar parcialmente los relativos altos niveles de materia seca que presentan estas variedades, dado que un bajo nivel de degradación de almidón por la α-amilasa y la isoamilasa, contrastado con la producción constante de almidón por las enzimas biosintéticas, estaría generando un incremento constante de materia seca. Sin embargo la situación puede ser un poco más compleja, ya que una variedad contrastante con bajo contenido de materia seca como TMS60444 presentó niveles relativamente altos o al menos muy similares a los de estas variedades en la expresión de genes implicados en la síntesis de almidón. La diferencia más importante se presentó en la expresión de la α-amilasa para el cual la variedad 60444 obtuvo los mayores niveles de expresión.
Nosotros logramos observar un comportamiento general en el que las variedades SM fueron agrupadas y separadas de las variedades CM según su perfil de expresión de genes implicados en el metabolismo del almidón. Esta información podría ser útil en posteriores estudios encaminados a identificar variedades promisorias para la industria del almidón o en la selección de variedades y su fondo genético para ser incluidas dentro de programas de mejoramiento genético.
CONCLUSIONES
El metabolismo del almidón presenta una compleja regulación que implica no solamente el control transcripcional de los genes implicados a través de mecanismos tales como el ritmo circadiano, hormonas y el contenido de azúcar (Munyikwa et al., 1997; Baguma et al., 2003; Smith et al., 2004). De igual manera, la regulación en la actividad enzimática mediante modificaciones post-transcripcionales e inhibición alostérica también ha sido reportada (Martin y Smith, 1995). La presencia de múltiples isoformas es un elemento adicional que incrementa la complejidad en la regulación en la acumulación de almidón en las raíces almacenadoras (Ball y Morell, 2003). Todos estos elementos deben considerarse en un futuro en diferentes variedades de yuca con miras a ganar una mejor comprensión de los factores que determinan sus características diferenciales en el contenido de materia seca y en consecuencia de almidón. En este estudio se ha contribuido parcialmente a este conocimiento a través de la evaluación de la expresión de varios genes implicados en el metabolismo de almidón. Estos datos podrán correlacionarse con actividad enzimática y contenido de almidón en futuros estudios.
AGRADECIMIENTOS
A los integrantes de la plataforma de transformación genética del CIAT. Este trabajo fue financiado por el Ministerio de Agricultura y Desarrollo Rural de Colombia, proyecto No 2007D7499-155.
REFERENCIAS
Baguma Y. Regulation of starch synthesis in cassava (Tesis Doctoral). Uppsala: Department of Plant Biology and Forest Genetics, Swedish University of Agricultural Sciences; 2004; 11-25. [ Links ]
Baguma Y, Sun C, Ahlandsberg S, Mutisya J, Palmqvist S, et al. Expression patterns of the gene encoding starch branching enzyme II in the storage roots of cassava (Manihot esculenta Crantz). Plant Sci. 2003;164:833-839. Doi: http://dx.doi.org/10.1016/S0168-9452(03)00072-4. [ Links ]
Baguma Y, Sun C, Borén M, Olsson H, Rosenqvist S, et al. Sugar-mediated semidian oscillation of gene expression in the cassava storage root regulates starch synthesis. Plant Signal Behav. 2008;3:439-445. Doi: http://dx.doi.org/10.4161/psb.3.7.5715. [ Links ]
Ball SG, Morell MK. From bacterial glycogen to starch: understanding the biogenesis of the plant starch granule. Annu Rev Plant Biol. 2003;54:207-233. Doi: http://dx.doi.org/10.1146/annurev.arplant.54.031902.134927. [ Links ]
Ballicora MA, Iglesias AA, Preiss J. ADP-Glucose Pyrophosphorylase: A Regulatory Enzyme for Plant Starch Synthesis. Photosyn Res. 2004;79:1-24. Doi: http://dx.doi.org/10.1023/B:PRES.0000011916.67519.58. [ Links ]
Bresolin NS, Li Z, Kosar-Hashemi B, Tetlow IJ, Chatterjee M, et al. Characterisation of disproportionating enzyme from wheat endosperm. Planta. 2006;224:20-31. Doi: http://dx.doi.org/10.1007/s00425-005-0187-7. [ Links ]
Chen J, Zhang J, Liu H, Hu Y, Huang Y. Molecular strategies in manipulation of the starch synthesis pathway for improving storage starch content in plants. Plant Physiol Biochem. 2012;61C:1-8. Doi: 10.1016/j.plaphy.2012.08.013. [ Links ]
De Hoon MJ, Imoto S, Nolan J, Miyano S. Open source clustering software. Bioinformatics. 2004;20:1453-1454. Doi: 10.1093/bioinformatics/bth078. [ Links ]
El-Sharkawy MA. Cassava biology and physiology. Plant Mol Biol. 2004;56:481-501. Doi: 10.1007/s11103-005-2270-7. [ Links ]
Firon N, Labonte D, Villordon A, Kfir Y, Solis J, et al. Transcriptional profiling of sweetpotato (Ipomoea batatas) roots indicates down-regulation of lignin biosynthesis and up-regulation of starch biosynthesis at an early stage of storage root formation. BMC genomics. 2013;14:460. Doi: 10.1186/1471-2164-14-460. [ Links ]
Fregene M, Bernal A, Duque M, Dixon A, Tohme J. AFLP analysis of African cassava (Manihot esculenta Crantz) germplasm resistant to the cassava mosaic disease (CMD). Theor Appl Genet. 2000;100:678-685. Doi: 10.1007/s001220051339. [ Links ]
Howeler R, Lutaladio N, Thomas G. Save and Grow: Cassava. A guide to sustainable production intensification. 1 ed. FAO. Roma; 2013; 87-97 [ Links ]
Martin C, Smith AM. Starch biosynthesis. Plant Cell. 1995;7:971-985. Doi: http://dx.doi.org/10.1105/tpc.7.7.971. [ Links ]
Munyikwa TRI, Langeveld S, Salehuzzaman SNIM, Jacobsen E, Visser RGF. Cassava starch biosynthesis: new avenues for modifying starch quantity and quality. Euphytica. 1997;96:65-75. Doi: 10.1023/A:1002935603412. [ Links ]
Ospina PB, Ceballos H, Alvarez E, Bellotti AC, Calvert LA, et al. La yuca en el Tercer Milenio. Sistemas modernos de producción, procesamiento, utilización y comercialización. 1 ed. CIAT. Cali; 2002;34-49. [ Links ]
Prochnik S, Marri PR, Desany B, Rabinowicz PD, Kodira C, et al. The Cassava Genome: Current Progress, Future Directions. Trop Plant Biol. 2012;5:88-94. Doi: 10.1007/s12042-011-9088-z. [ Links ]
Raemakers K, Schreuder M, Suurs L, Furrer-Verhorst H, Vincken JP, et al. Improved Cassava Starch by Antisense Inhibition of Granule-bound Starch Synthase I. Mol Breed. 2005;16:163-172. Doi: 10.1007/s11032-005-7874-8. [ Links ]
Roa AC, Maya MM, Duque MC, Tohme J, Allem AC, et al. AFLP analysis of relationships among cassava and other Manihot species. Theor Appl Genet. 1997;95:741-750. Doi: 10.1007/s001220050620. [ Links ]
Saithong T, Rongsirikul O, Kalapanulak S, Chiewchankaset P, Siriwat W, et al. Starch biosynthesis in cassava: a genome-based pathway reconstruction and its exploitation in data integration. BMC Syst Biol. 2013;7:75. Doi: 10.1186/1752-0509-7-75. [ Links ]
Saldanha AJ. Java Treeview-extensible visualization of microarray data. Bioinformatics. 2004;20:3246-3248. Doi: 10.1093/bioinformatics/bth349. [ Links ]
Salehuzzaman S, Vincken JP, De Wal MV, Jacobsen E, Visser RGF. Expression of a cassava granule-bound starch synthase gene in the amylose-free potato only partially restores amylose content. Plant Cell Environ. 1999;22:1311-1318. Doi: 10.1046/j.1365-3040.1999.00493.x. [ Links ]
Sánchez T, Salcedo E, Ceballos H, Dufour D, Mafla G, et al. Screening of Starch Quality Traits in Cassava (Manihot esculenta Crantz). Starke. 2009;61:12-19. Doi: 10.1002/star.200800058. [ Links ]
Smith SM, Fulton DC, Chia T, Thorneycroft D, Chapple A, et al. Diurnal changes in the transcriptome encoding enzymes of starch metabolism provide evidence for both transcriptional and posttranscriptional regulation of starch metabolism in Arabidopsis leaves. Plant Physiol. 2004;136:2687-2699. Doi: http://dx.doi.org/10.1104/pp.104.044347. [ Links ]
Sojikul P, Kongsawadworakul P, Viboonjun U, Thaiprasit J, Intawong B, et al. AFLP-based transcript profiling for cassava genome-wide expression analysis in the onset of storage root formation. Physiol Plant. 2010;140:189-298. Doi: 10.1111/j.1399-3054.2010.01389.x. [ Links ]
Sonnewald U, Kossmann J. Starches-from current models to genetic engineering. Plant Biotechnol J. 2013;11:223-232. Doi: 10.1111/pbi.12029. [ Links ]
Yang J, An D, Zhang P. Expression Profiling of Cassava Storage Roots Reveals an Active Process of Glycolysis/Gluconeogenesis. J Integr Plant Biol. 2011;53:193-211. Doi: 10.1111/j.1744-7909.2010.01018.x. [ Links ]
Zeeman SC, Kossmann J, Smith AM. Starch: its metabolism, evolution, and biotechnological modification in plants. Annu Rev Plant Biol. 2010;61:209-234. Doi: 10.1146/annurev-arplant-042809-112301. [ Links ]