Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Acta Biológica Colombiana
versão impressa ISSN 0120-548X
Acta biol.Colomb. vol.21 no.3 Bogotá set./dez. 2016
https://doi.org/10.15446/abc.v21n3.54046
DOI: http://dx.doi.org/10.15446/abc.v21n3.54046
ANÁLISIS PROTEÓMICO DEL VENENO DE LA ABEJA AFRICANIZADA: COMPARACIÓN DE MÉTODOS DE EXTRACCIÓN
Proteomic Analysis of Africanized Bee Venom: a Comparison of Protein Extraction Methods
Yessica PINEDA-GUERRA1; Johana BETANCUR-ECHEVERRI1; Johanna PEDROZA-DÍAZ1; Edilson DELGADO-TREJOS2; Sarah ROTHLISBERGER1.
1 Facultad de Ciencias Exactas y Aplicadas, Instituto Tecnológico Metropolitano (ITM), Campus Robledo, bloque I, segundo piso, Calle 73 n°. 76A-354, Vía al Volador. Medellín, Colombia.
2 Facultad de Ciencias Económicas y Administrativas, Instituto Tecnológico Metropolitano (ITM). Calle 73 n° 76A-354. Medellín, Colombia.
For correspondence. sarahrothlisberger@itm.edu.co
Received: 9th November 2015, Returned for revision: 22nd February 2016, Accepted: 18th March 2016.
Associate Editor: Geraldo Mäder.
Citation/Citar este artículo como: Pineda Guerra Y, Betancur Echeverri J, Pedroza-Díaz J, Delgado-Trejos E, Rothlisberger S. Análisis proteómico del veneno de la abeja africanizada: comparación de métodos de extracción. Acta biol. Colomb. 2016;21(3):619-626. DOI: http://dx.doi.org/10.15446/abc.v21n3.54046
RESUMEN
La abeja africanizada es la más común en la apicultura colombiana y a su veneno (apitoxina) se le han atribuido propiedades terapéuticas para diferentes enfermedades, sin mayor soporte científico. Al revisar en la literatura los reportes publicados sobre el análisis proteómico de la apitoxina, se encontraron cuatro métodos distintos para la extracción de proteínas de la apitoxina. El primer método consiste en resuspender la apitoxina en Urea 7 M, precipitar con acetona y finalmente resuspender en Urea 7 M y CHAPS 4 %. Para el segundo método se resuspende la apitoxina en buffer de lisis, se precipita con ácido tricloroacético, y luego se resuspende en Urea 7 M y CHAPS 4 %. El tercer método es igual al anterior, excepto que la precipitación se realiza con acetona en vez de ácido tricloroacético. Finalmente, el cuarto método consiste en resuspender la apitoxina en agua destilada, precipitar con acetona y resuspender en Urea 7 M y CHAPS 4 %. Este trabajo se enfocó en comparar el desempeño de estos cuatro métodos de extracción y determinar el método con el mejor resultado en cuanto a la concentración e integridad obtenida de las proteínas. De los distintos métodos evaluados, se encontró que los mejores resultados en cuanto a concentración de proteínas se obtuvieron con la resuspensión de apitoxina en buffer de lisis y precipitación con acetona (método 3) y con el método de resuspensión de apitoxina en agua destilada y precipitación con acetona (método 4). De estos, el mejor método de extracción en cuanto a integridad de las proteínas y perfil proteómico fue el de resuspensión de apitoxina en buffer de lisis seguido de precipitación con acetona (método 3).
Palabras clave: acetona, apitoxina, electroforesis en gel bidimensional,perfil proteico.
ABSTRACT
The Africanised bee is the most common type of bee in Colombia, and therapeutic properties for different diseases have been attributed to its venom, without much scientific support. A literature search of reports on the proteomic analysis of honeybee venom yielded four different methods for extracting proteins from bee venom. The first method consists in resuspending the venom in 7 M Urea, followed by precipitation with acetone and finally resuspending the pellet in 7 M Urea and 4 % CHAPS. For the second method, the venom is resuspended in lysis buffer, precipitated with trichloroacetic acid, and then resuspended in 7 M Urea and 4 % CHAPS. The third method is similar to the previous one, except that the precipitation step is performed with acetone instead of trichloroacetic acid. Finally, the fourth method is to resuspend the venom in distilled water, precipitate with acetone and resuspend in 7 M Urea and 4 % CHAPS. This work focused on comparing the performance of these four extraction methods, in order to determine the method with the best results in terms of concentration and integrity of the proteins obtained. Of the four methods evaluated, the best results in terms of protein concentration and yield were obtained by resuspending the bee venom in lysis buffer followed by precipitation with acetone (method 3), and by resuspending in distilled water followed by precipitation with acetone (method 4). Of these, the method that maintained protein integrity and yielded the best proteomic profile was that in which the bee venom was resuspended in lysis buffer followed by precipitation with acetone (method 3).
Keywords: acetone, bee venom, proteomic profile, two-dimensional gel electrophoresis.
INTRODUCCIÓN
Hay diversos protocolos en la literatura acerca de la extracción de proteínas en general, pero no existen muchos orientados a la extracción de proteínas del veneno de la abeja africanizada, siendo esta especie de abeja la más común en la apicultura colombiana. El veneno secretado por las abejas obreras se conoce como apitoxina, éstas lo emplean como mecanismo de defensa ante sus depredadores naturales, al igual que contra otras especies de abejas (Nates-Parra, 2011). La apitoxina está compuesta por diversos tipos de aminas biogénicas (histamina, dopamina, serotonina), oligopéptido (melitina) y proteínas con actividad enzimática (fosfolipasas) (Foreman-Wykert et al., 1999; Leandro et al., 2015).
Es de importancia el estudio de la apitoxina ya que contribuye a una mejor comprensión de los procesos de envenenamiento. Los efectos tóxicos del veneno de la abeja africanizada se han asociado principalmente a las reacciones alérgicas, pero un ataque masivo de abejas puede resultar incluso mortal para la víctima. Los síntomas iniciales de envenenamiento masivo incluyen edema, fatiga, mareos, náuseas, vómitos, fiebre y pérdida del conocimiento (Vetter et al., 1999; Francese et al., 2009). El desarrollo de anti-venómicos, en general, representa un campo de investigación que impacta en áreas como la toxicología, fisiología de emergencias, inmunología y neurotoxicología; sin embargo, los avances en esta área son muy reducidos debido a la complejidad molecular de los venenos (Isbister 2002; Calvete et al., 2012). Otra gran aplicación del estudio de la apitoxina se centra en el desarrollo de fármacos. Gracias a las acciones analgésicas, neurotóxicas, vasoactivas, hemolíticas y citotóxicas de la apitoxina (Peiren et al., 2005) se le han atribuido efectos terapéuticos, aunque en su mayoría no han sido plenamente formalizados por métodos científicos. La acción analgésica y anti-inflamatoria de la apitoxina ha impulsado su uso como terapia alternativa en casos de artritis (Lee et al., 2005), además de sugerirse el poder de este veneno para inhibir la infección por VIH (Hood et al., 2013). Recientes estudios realizados en modelos animales, han demostrado el efecto de la apitoxina sobre el sistema dopaminérgico que podría beneficiar el desarrollo de nuevas estrategias para el tratamiento de trastornos mentales, ya que su principal componente es la melitina, la cual presenta propiedades antipsicóticas sin los efectos secundarios clásicos de los fármacos neurolépticos similares (Dantas et al., 2014).
Es importante resaltar que gran parte de los estudios sobre apitoxina reportados en la literatura han tomado como objeto de estudio poblaciones de abejas europeas de miel común (Apis mellifera) (El-Kik et al., 2013; Li et al., 2013; Van Vaerenbergh et al., 2014; Leandro et al., 2015; Lee et al., 2015) y existen muy pocos reportes con técnicas moleculares modernas sobre apitoxina de abejas africanizadas (Ferreira Junior et al., 2010; Sciani et al., 2010; Resende et al., 2013). Lo anterior genera un vacío importante en el conocimiento científico, ya que las abejas africanizadas son la especie de abeja más común en el continente Americano y en la apicultura colombiana. En los pocos trabajos publicados, donde se ha abordado a la apitoxina a nivel molecular, encontramos que cada uno emplea un método distinto de extracción de proteínas, por lo tanto, no existe un protocolo unificado para este fin, ni se ha comparado cuál método es el más óptimo para estudios proteómicos.
Tomando como base los reportes publicados sobre extracción y análisis de proteínas de apitoxina de las abejas de miel común, el presente estudio tiene como objetivo comparar la efectividad de los distintos métodos de extracción usados en esos reportes y aplicarlos a la extracción de proteínas de la apitoxina de las abejas africanizadas del territorio Colombiano. Con este trabajo se busca proponer el método más óptimo y contribuir así a futuros estudios proteómicos de la apitoxina de abejas africanizadas en Colombia.
MATERIALES Y MÉTODOS
Recolección de apitoxina
Se recolectaron muestras de apitoxina de abejas africanizadas (obreras de exterior) por apicultores con experiencia, usando el método de estimulación eléctrica (Li et al., 2013) con el uso de un colector trampa 4G (Ketitlen, Colombia) para extracción de apitoxina. En este proceso de extracción, las abejas son expuestas a un voltaje muy bajo que busca la estimulación de las glándulas del animal para obtener el veneno sin necesidad de picaduras o ejecuciones que signifiquen algún atentado a la persona o al animal. Esta metodología fue revisada y avalada por el Comité de Ética para la Investigación Científica del Instituto Tecnológico Metropolitano, en sesión realizada el día 14 de Agosto de 2014 como consta en el Acta No. 07.
Equipos y reactivos
En la precipitación de proteínas se utilizó ácido tricloroacético (TCA) y acetona (Panreac AppliChem, EE.UU). Se determinó la concentración de proteínas mediante el kit comercial de ácido bicinconínico (BCA) (Sigma-Aldrich, EE.UU) y se midió la absorbancia con el GloMax®-Multi Detection System (Promega, EE.UU). Para evaluar de manera cualitativa la integridad de las proteínas se realizó electroforesis unidimensional en gel poliacrilamida-dodecilsulfato de sodio (SDS-PAGE), utilizando el marcador de peso molecular Novex® Sharp Pre-stained Protein Standard (Thermo Fisher Scientific, EE.UU) y el equipo PowerPac™ Basic Power Supply (BioRad, EEUU). Para la segunda dimensión, se utilizó el equipo PowerPac™ HV High-Voltage Power Supply (BioRad, EE.UU). La tinción se hizo con Azul Brillante de Coomassie G-250 (AMRESCO, EE.UU).
Métodos de extracción
Se realizó una búsqueda en la literatura en cuanto a la extracción de proteínas de la apitoxina y se encontraron cuatro métodos distintos para este fin (Fujita et al., 2010; Matysiak et al., 2011; Zhang et al., 2012; Li et al., 2013; Yiou et al., 2013), los cuales se resumen en la Fig. 1 y se describen a continuación.
Método 1
Este método consiste en resuspender la apitoxina en Urea 7 M. Para esto, se pesaron 20 mg de apitoxina los cuales fueron resuspendidos en 200 µl de Urea 7 M con agitación con vortex (1 min), sonicación en frío (10 min) y nuevamente agitación con vortex (1 min). Se centrifugó durante cinco minutos a 14000 rpm y 4 °C, y se conservó el sobrenadante. Seguido de esto, se precipitaron las proteínas en el sobrenadante con una concentración final de 80 % (v/v) acetona a -20 °C durante toda la noche. Al finalizar la incubación se centrifugó por 30 minutos a 14000 rpm y 4 °C. El pellet resultante se lavó tres veces con acetona y se secó en placa de calentamiento a 95 °C por 30 segundos con el fin de remover la acetona residual. Posteriormente, las proteínas del pellet fueron resuspendidas en buffer de hidratación (Urea 7 M y CHAPS al 4 %) y almacenadas a -20 °C.
Método 2
Este método, adaptado de (Fujita et al., 2010; Yiou et al., 2013) consiste en resuspender la apitoxina en buffer de lisis, precipitar con TCA y resuspender en Urea 7 M y CHAPS 4 %. Para esto se pesaron 20 mg de apitoxina los cuales fueron resuspendidos en 200 µl de buffer de lísis (Urea 8 M, Tiourea 2 M, CHAPS (3-[(3-Cholamidopropyl)dimethylammonio]-1-propanesulfonate) al 4 %, Tris-base 20 mM, DTT (Dithiothreitol) 30 mM, coctel inhibidor de proteasa), seguido por agitación con vortex (1 min), sonicación en frío (10 min) y nuevamente agitación con vortex (1 min). Se centrifugó durante cinco minutos a 14000 rpm y 4 °C, y se conservó el sobrenadante. Al sobrenadante obtenido, se le adicionó ácido tricloroacético (TCA) al 25 % y se incubó por 10 minutos sobre hielo con agitación en vortex. Al finalizar la incubación se centrifugó durante cinco minutos a 14000 rpm y 4 °C, el sobrenadante se descartó y se lavó el pellet tres veces con acetona -20 °C. Después de los lavados se secó el pellet en placa de calentamiento a 95 °C por 30 segundos, con el fin de remover la acetona residual, y fue resuspendido en Urea 7 M y CHAPS al 4 %. La muestra fue almacenada a -20 °C.
Método 3
Este método, adaptado de (Zhang et al., 2012; Li et al., 2013), consiste en resuspender la apitoxina en buffer de lisis, precipitar con acetona y resuspender en Urea 7 M y CHAPS 4 %. Para esto, se pesaron 20 mg de apitoxina los cuales fueron resuspendidos en 200 µl de buffer de lísis (Urea 8 M, Tiourea 2 M, CHAPS (3-[(3-Cholamidopropyl)dimethylammonio]-1-propanesulfonate) al 4 %, Tris-base 20 mM, DTT (Dithiothreitol) 30 mM, coctel inhibidor de proteasa), seguido por agitación con vortex (1 min), sonicación en frío (10 min) y nuevamente agitación con vortex (1 min). Se centrifugó durante cinco minutos a 14000 rpm y 4 °C, y se conservó el sobrenadante. Al sobrenadante obtenido, se le adicionó acetona a -20 °C a una concentración final de 80 % (v/v), seguido de agitación con vórtex, y se incubó durante toda la noche a -20 °C. Al finalizar la incubación se centrifugó por 30 minutos a 14000 rpm y 4 °C. El pellet resultante se lavó tres veces con acetona y se secó en placa de calentamiento a 95 °C por 30 segundos con el fin de remover la acetona residual. Posteriormente, el pellet fue resuspendido en buffer de hidratación (Urea 7 M y CHAPS al 4 %) y almacenado a -20 °C.
Método 4
Para este método, descrito en (Matysiak et al., 2011), se resuspendieron 2 mg de apitoxina en 200 µl de agua destilada con agitación con vortex (1 min), sonicación en frío (10 min) y nuevamente agitación con vortex (1 min). Se centrifugó durante cinco minutos a 14000 rpm y 4 °C, y se conservó el sobrenadante. Seguido de esto, se precipitaron las proteínas en el sobrenadante con una concentración final de 80 % (v/v) acetona a -20 °C durante toda la noche. Al finalizar la incubación se centrifugó por 30 minutos a 14000 rpm y 4 °C. El pellet resultante se lavó tres veces con acetona y se secó en placa de calentamiento a 95 °C por 30 segundos con el fin de remover la acetona residual. Posteriormente, las proteínas del pellet fueron resuspendidas en buffer de hidratación (Urea 7 M y CHAPS al 4 %) y almacenada a -20 °C.
Electroforesis unidimensional (SDS-PAGE)
Para este tipo de electroforesis se cargaron 5 µg y 10 µg de las muestras obtenidas por cada método descrito anteriormente en el gel de poliacrilamida al 10 % y fueron corridas a 120 v evaluando el frente de corrido, esta metodología fue adaptada de (Li et al., 2013). El revelado de los geles fue realizado usando tinción con azul de Coomassie.
Electroforesis bidimensional (2D-PAGE)
Para la obtención del perfil proteico mediante electroforesis bidimensional se resuspendieron 50 µg de proteína en buffer de hidratación (Urea 7 M, Tiourea 2 M, CHAPS al 4 %, DTT 50 mM y anfolitos pH 3-10 (ZOOM® Carrier Ampholytes 3-10 de Thermo Fisher Scientific)), con lo cual se rehidrató la tirilla IPG (Immobilized pH gradient) con rango pH 3-10 de 7 cm (ZOOM® IPG Runner™ System). El isoelectroenfoque se hizo mediante un protocolo de aumento progresivo de voltaje: 200-450-600-750-950 v durante 25 minutos, luego 1200-1400-1600 v durante 30 minutos y finalmente, 2000 v durante 45 minutos. Se sumergió la tirilla en buffer de equilibrio 1 (NuPAGE® LDS Sample Buffer de Thermo Fisher Scientific con DTT al 2 %) durante 15 minutos y luego se sumergió en buffer de equilibrio 2 (LDS Sample Buffer con Iodoacetamida al 2,5 %) por 15 minutos adicionales. Para la segunda dimensión se utilizaron geles preformados (NuPAGE™ Novex™ 4-12 % Bis-Tris Protein Gels, 1,5 mm de Thermo Fisher Scientific). La separación fue realizada usando NuPAGE® MOPS SDS Running Buffer (20X) en el equipo XCell SureLock® Mini-Cell de Invitrogen a 200 v durante 40 minutos. Esta metodología fue adaptada de (Li et al., 2013). Los geles fueron teñidos con azul de Coomassie.
RESULTADOS
Rendimiento y concentración de las proteínas obtenidas
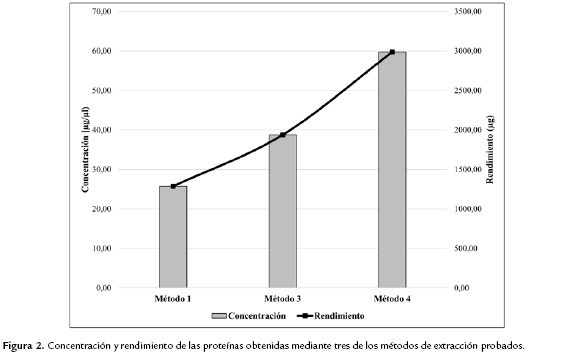
La obtención de proteínas de la apitoxina fue exitosa en tres de los métodos descritos. En el caso del método 2 (precipitación con ácido tricloroacético), las proteínas no precipitaron y no se obtuvo un pellet visible. Por esta razón, se descartó el método 2 y no se incluyó en los experimentos posteriores. Se evaluó la concentración y el rendimiento de las proteínas obtenidas a partir de los métodos 1, 3, y 4. Como se observa en la Fig. 2, por el método 1 (apitoxina resuspendida inicialmente en Urea 7 M) se obtuvo una concentración de 25,73 µg/µl y un rendimiento de 1286,5 µg. El método 3 (apitoxina resuspendida en buffer de lisis y precipitada con acetona) dio como resultado una concentración de 38,77 µg/µl y un rendimiento de 1938,3 µg. El método que mejores resultados dio en términos de concentración y rendimiento fue el método 4 (apitoxina resuspendida en agua destilada). Para este método la concentración obtenida fue de 59,74 µg/µl y el rendimiento de 2987 µg.
Calidad de las proteínas obtenidas
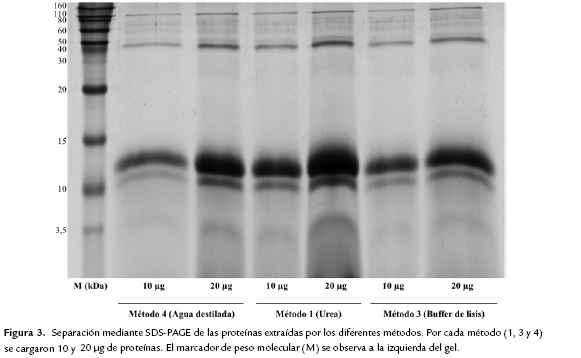
En la Fig. 3, se presenta la imagen de electroforesis unidimensional en gel de poliacrilamida (SDS-PAGE) al 10 % y se puede evidenciar el mismo número de bandas al comparar los tres métodos. Entre los 110 y 40 kDa se reconocen aproximadamente seis delgadas bandas de proteína, dos bandas bastante acentuadas entre 15 y 10 kDa, y finalmente una banda a 3,5 kDa.
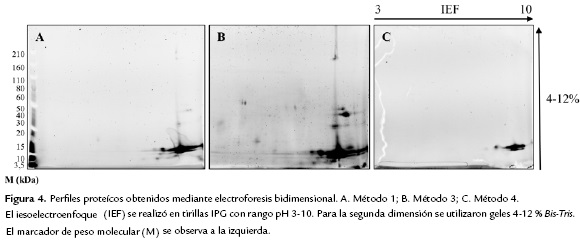
En la Fig. 4, se encuentran los perfiles proteícos obtenidos mediante electroforesis bidimensional. Se observa que el número de spots es bajo, sin embargo la resolución da cuenta de que no hay interferentes ni degradación proteíca. El método que proporcionó el mayor número de spots en el gel fue el método 3 (apitoxina resuspendida en buffer de lísis), seguido del método 1 (apitoxina resuspendida en Urea 7 M). La mayor concentración de proteínas se encuentran ubicadas en la parte inferior derecha del gel, entre los 3,5 - 15 kDa, lo cual es concordante con las dos bandas acentuadas que se observaron en el SDS-PAGE. También se perciben aproximadamente cuatro spots entre los 20 - 50 kDa, y dos spots entre los 160 - 210 kDa. El método que proporcionó el menor número de spots en el gel 2D fue el método 4 (apitoxina resuspendida en agua destilada), con solo la agrupación de spots entre los 3,5 - 15 kDa siendo visible.
DISCUSIÓN
Al realizar la extracción de proteínas se descartó el método 2 (precipitación con ácido tricloroacético), ya que no fue posible obtener un pellet de proteína precipitada con este método, a pesar de extender el tiempo de precipitación. En cuanto a concentración y rendimiento de proteína obtenida, cualquiera de los tres métodos restantes (1, 3 y 4) es efectivo. Sin embargo, el mejor resultado se observó con el método 4 (apitoxina resuspendida inicialmente en agua destilada), con el cual se obtuvo más del doble de proteína que con el método 1 (apitoxina resuspendida inicialmente en Urea 7 M), además de ser el método más sencillo y económico. Los métodos de extracción de proteínas aquí probados fueron adaptados de trabajos publicados sobre extracción y análisis de proteínas de apitoxina, de jalea real, y de las glándulas salivales de las abejas de miel común (Fujita et al., 2010; Matysiak et al., 2011; Zhang et al., 2012; Li et al., 2013; Yiou et al., 2013). No es posible hacer la comparación de concentración y rendimiento de los cuatro métodos aquí descritos con los encontrados en la literatura, ya que no se reportan valores de concentración y rendimiento en esos artículos.
En el análisis de la Fig. 3, obtenida a partir de electroforesis unidimensional, se puede observar que en los tres métodos evaluados se obtuvo el mismo número de bandas, y las proteínas presentan buena integridad, sin estrías ni interferentes en el gel. Las bandas obtenidas son las esperadas de acuerdo con lo reportado en literatura. En el trabajo publicado por Li et al., (2013), se analizó la apitoxina extraída por el método manual y por estimulación eléctrica. En la apitoxina extraída por estimulación eléctrica detectaron 13 bandas de proteínas en gel unidimensional (SDS-PAGE). De estas 13 bandas de proteína aproximadamente seis se encuentran entre los 95 y 40 kDa, y la mayor concentración de proteína, tres bandas muy pronunciadas, se halló entre los 17 y 10 kDa; lo cual se ve reflejado en nuestros resultados. El método de extracción de proteínas de la apitoxina empleado por Li et al., 2013 es igual al método tres probado en este trabajo (apitoxina resuspendida en buffer de lisis seguido de precipitación con 80 % acetona).
Los resultados obtenidos aquí también son comparables a los publicados por (Van Vaerenbergh et al., 2014), quienes realizaron enriquecimiento de proteínas con un kit comercial (ProteoMiner protein enrichment small-capacity kit, Bio-Rad) y la separación de proteínas en geles tris-tricina. En el gel se presentan bandas entre los 110-80 kDa donde se podría encontrar la dipeptidildipeptidasa IV (Api m 5) que tiene un peso molecular de 100 kDa, la cual es capaz de activar los basófilos humanos de pacientes alérgicos al veneno (Blank et al., 2010). En el rango de los 30-50 KDa, es probable hallar la hialuronidasa (Api m 2) que tiene una abundancia de 1,5-2 % en el veneno y con peso de 39 kDa, que se conoce como el factor de expansión, ya que degrada el ácido hialurónico a segmentos no viscosos, lo que permite la rápida propagación de los compuestos de veneno a través del espacio intersticial (de Lima and Brochetto-Braga, 2003) y la fosfatasa ácida (Api m 3) con aproximadamente un 1 % de abundancia en el veneno y con un peso de 43 kDa que conduce a reacciones de hipersensibilidad en personas susceptibles al veneno (Ali, 2012). Se observan también, bandas definidas con una buena cantidad de proteína entre los 15-20 kDa y se infiere que se podría encontrar la Fosfolipasa A2 (Api m 1) de peso molecular 16 kDa, con una abundancia de 10-12 %, que tiene como función generar poros en las membranas, y por lo tanto, lisis celular (de Lima and Brochetto-Braga, 2003). También se presentan bandas a los 3,5 kDa, lo cual podría corresponder a péptidos como la melitina (Api m 4) con 3 kDa de peso molecular y abundancia del 50-52 %, que es la encargada de causar la destrucción de fibras musculares, generando hipercontracción y ruptura de las microfibrillas. Asimismo, otros péptidos presentan peso molecular parecido pero en menor abundancia como la apamina que se comporta como una neurotoxina de acción motora y presenta efectos cardioestimulantes parecidos al de las drogas adrenérgicas (Ali, 2012).
Se puede observar en los perfiles proteicos obtenidos por electroforesis bidimensional (ver Fig. 4) que las proteínas poseen buena calidad y sin interferentes presentes en el gel. El método que proporcionó el mejor perfil proteico fue el método 3 (apitoxina resuspendida en buffer de lísis seguido de precipitación con acetona), posiblemente porque las proteínas están mejor solubilizadas. Este es el mismo método empleado en dos importantes trabajos proteómicos con apitoxina de abejas europeas (Peiren et al., 2005; Li et al., 2013). En el perfil proteico obtenido por el método 3 se presentan baja cantidad de spots a comparación con los trabajos ya publicados con este método. Esto podría explicarse por la cantidad de proteína que se cargó en el gel. En el trabajo por (Li et al., 2013) se cargaron 500 µg de proteína, y en (Peiren et al., 2005) se cargaron 1000 µg en el gel usando tirillas IPG de 18 cm, por lo cual se observaron muchos más spots que en nuestro estudio, en el cual se cargaron 50 µg de proteína en el gel usando tirillas de 7 cm.
CONCLUSIONES
De acuerdo con el análisis de los resultados experimentales de este trabajo se concluye que, de los métodos evaluados, el método 4 proporcionó los mejores resultados en cuanto a concentración y rendimiento, seguido del método 3. Mediante SDS-PAGE se observó el mismo número de bandas de proteína por los tres métodos evaluados, con buena integridad y sin estrías ni interferentes en el gel. Sin embargo, si lo que se quiere es hacer estudios proteómicos con electroforesis bidimensional la metodología más óptima es el método 3 (apitoxina resuspendida en buffer de lisis y precipitada con acetona). Esta metodología ha sido implementada exitosamente en otros estudios proteómicos de la apitoxina de las abejas europeas, usando electroforesis bidimensional (Peiren et al., 2005; Li et al., 2013), y es de fácil ejecución y de bajo costo. Para mejorar el perfil proteico se puede cargar mayor concentración de proteína (500-1000 µg) en un gel de mayor tamaño (tirilla IPG de 18 cm) con el fin de visualizar las proteínas de baja abundancia, además de usar geles tris-tricina que permiten la separación de proteínas y péptidos de bajo peso molecular, que son las que se encuentran en mayor abundancia en la apitoxina.
AGRADECIMIENTOS
Este trabajo es derivado del proyecto de investigación P14227, financiado por el Instituto Tecnológico Metropolitano -ITM de Medellín.
REFERENCIAS
Ali M. Studies on Bee Venom and Its Medical Uses. Int J Adv Res Technol. 2012;1(2):69-83. [ Links ]
Blank S, Seismann H, Bockisch B, Braren I, Cifuentes L, McIntyre M, et al. Identification, recombinant expression, and characterization of the 100 kDa high molecular weight Hymenoptera venom allergens Api m 5 and Ves v 3. J Immunol. 2010;184(9):5403-5413. Doi: http://dx.doi.org/10.4049/jimmunol.0803709. [ Links ]
Calvete JJ, Pérez A, Lomonte B, Sánchez EE, Sanz L. Snake venomics of Crotalus tigris: the minimalist toxin arsenal of the deadliest Nearctic rattlesnake venom. Evolutionary Clues for generating a pan-specific antivenom against crotalid type II venoms [corrected]. J Proteome Res. 2012;11(2):1382-1390. Doi: http://dx.doi.org/10.1021/pr201021d. [ Links ]
Dantas CG, Nunes T, Nunes T, Paixão A, Reis FP, Júnior W de L, et al. Pharmacological evaluation of bee venom and melittin. Rev Bras Farmacogn. 2014;24(1):67-72. Doi: http://dx.doi.org/10.1590/0102-695X20142413365. [ Links ]
El-Kik CZ, Fernandes FFA, Tomaz MA, Gaban GA, Fonseca TF, Calil-Elias S, et al. Neutralization of Apis mellifera bee venom activities by suramin. Toxicon. 2013;67:55-62. Doi: http://dx.doi.org/10.1016/j.toxicon.2013.02.007. [ Links ]
Ferreira Junior R, Sciani JM, Marques-Porto R, Junior AL, Orsi RDO, Barraviera B, et al. Africanized honey bee (Apis mellifera) venom profiling: Seasonal variation of melittin and phospholipase A2 levels. Toxicon. 2010;56(3):355-362. Doi: http://doi.org/10.1016/j.toxicon.2010.03.023. [ Links ]
Foreman-Wykert AK, Weinrauch Y, Elsbach P, Weiss J. Cell-wall determinants of the bactericidal action of group IIA phospholipase A2 against Gram-positive bacteria. J Clin Invest. 1999;103(5):715-721. Doi: http://dx.doi.org/10.1172/JCI5468. [ Links ]
Francese S, Lambardi D, Mastrobuoni G, la Marca G, Moneti G, Turillazzi S. Detection of honeybee venom in envenomed tissues by direct MALDI MSI. J Am Soc Mass Spectrom. 2009;20(1):112-123. Doi: http://dx.doi.org/10.1016/j.jasms.2008.09.006. [ Links ]
Fujita T, Kozuka-Hata H, Uno Y, Nishikori K, Morioka M, Oyama M, et al. Functional analysis of the honeybee (Apis mellifera L.) salivary system using proteomics. Biochem Biophys Res Commun. 2010;397(4):740-744. Doi: http://dx.doi.org/10.1016/j.bbrc.2010.06.023. [ Links ]
Hood JL, Jallouk AP, Campbell N, Ratner L, Wickline SA. Cytolytic nanoparticles attenuate HIV-1 infectivity. Antivir Ther. 2013;18(1):95-103. Doi: http://dx.doi.org/10.3851/IMP2346. [ Links ]
Isbister GK. Failure of intramuscular antivenom in Red-back spider envenoming. Emerg Med Fremantle WA. 2002;14(4):436-439. Doi: http://dx.doi.org/10.1046/j.1442-2026.2002.00356.x. [ Links ]
Leandro LF, Mendes CA, Casemiro LA, Vinholis AHC, Cunha WR, Almeida R, et al. Antimicrobial activity of apitoxin, melittin and phospholipase A of honey bee (Apis mellifera) venom against oral pathogens. An Acad Bras Ciênc. 2015;87(1):147-155. Doi: http://dx.doi.org/10.1590/0001-3765201520130511. [ Links ]
Lee HL, Park SH, Kim TM, Jung YY, Park MH, Oh SH, et al. Bee venom inhibits growth of human cervical tumors in mice. Oncotarget. 2015;6(9):7280-7292. Doi: http://dx.doi.org/10.18632/oncotarget.3110. [ Links ]
Lee J-D, Park H-J, Chae Y, Lim S. An overview of bee venom acupuncture in the treatment of arthritis. Evid-Based Complement Altern Med. 2005;2(1):79-84. Doi: http://dx.doi.org/10.1093/ecam/neh070. [ Links ]
de Lima PR, Brochetto-Braga MR. Hymenoptera venom review focusing on Apis mellifera. J Venom Anim Toxins Trop Dis. 2003;9(2):149-162. Doi: http://dx.doi.org/10.1590/S1678-91992003000200002. [ Links ]
Li R, Zhang L, Fang Y, Han B, Lu X, Zhou T, et al. Proteome and phosphoproteome analysis of honeybee (Apis mellifera) venom collected from electrical stimulation and manual extraction of the venom gland. Genomics. 2013;14(1):766. Doi: http://dx.doi.org/10.1186/1471-2164-14-766. [ Links ]
Matysiak J, Schmelzer CEH, Neubert RHH, Kokot ZJ. Characterization of honeybee venom by MALDI-TOF and nanoESI-QqTOF mass spectrometry. J Pharm Biomed Anal. 2011;54(2):273-278. Doi: http://dx.doi.org/10.1016/j.jpba.2010.08.020. [ Links ]
Nates-Parra G. Genética del comportmaiento: abejas como modelo. Acta biol Colomb. 2011;16(3):213-220. [ Links ]
Peiren N, Vanrobaeys F, de Graaf DC, Devreese B, Van Beeumen J, Jacobs FJ. The protein composition of honeybee venom reconsidered by a proteomic approach. Biochim Biophys Acta. 2005;1752(1):1-5. Doi: http://dx.doi.org/10.1016/j.bbapap.2005.07.017. [ Links ]
Resende VMF, Vasilj A, Santos KS, Palma MS, Shevchenko A. Proteome and phosphoproteome of Africanized and European honeybee venoms. Proteomics. 2013;13(17):2638-2648. Doi: http://doi.org/10.1002/pmic.201300038. [ Links ]
Sciani JM, Marques-Porto R, Lourenço Junior A, Orsi R, Ferreira Junior RS, Barraviera B, et al. Identification of a novel melittin isoform from Africanized Apis mellifera venom. Peptides. 2010;31(8):1473-1479. Doi: http://doi.org/10.1016/j.peptides.2010.05.001. [ Links ]
Van Vaerenbergh M, Debyser G, Devreese B, de Graaf DC. Exploring the hidden honeybee (Apis mellifera) venom proteome by integrating a combinatorial peptide ligand library approach with FTMS. J Proteomics. 2014;99:169-178. Doi: http://dx.doi.org/10.1016/j.jprot.2013.04.039. [ Links ]
Vetter RS, Visscher PK, Camazine S. Mass envenomations by honey bees and wasps. West J Med. 1999;170(4):223-227. [ Links ]
Yiou P, Shaoli A, Kebin L, Tao W, Kui F, Hua Z, et al. Evaluation of extraction procedures for 2-DE analysis of aphid proteins. J Sep Sci. 2013;36(3):532-539. Doi: http://dx.doi.org/10.1002/jssc.201200642. [ Links ]
Zhang L, Fang Y, Li R, Feng M, Han B, Zhou T, et al. Towards posttranslational modification proteome of royal jelly. J Proteomics. 2012;75(17):5327-5341. Doi: http://dx.doi.org/10.1016/j.jprot.2012.06.008. [ Links ]