INTRODUCCIÓN
La población colombiana es producto de un proceso de mestizaje que ha sido ampliamente documentado tanto histórica como biológicamente (Ossa et al., 2016). El mestizaje es un proceso evolutivo que incorpora variabilidad genética muy rápidamente y además puede hacerlo en varios loci a la vez (Templeton, 2006). Recientes eventos de mestizaje pueden tener efectos sobre la identificación de regiones asociadas con la susceptibilidad a enfermedades, ya que pueden ser una causa de alteración de los patrones de desequilibrio de ligamiento, lo cual afecta la reproducibilidad de los estudios de asociación entre distintas poblaciones (Pritchard y Rosenberg, 1999; Cardon y Palmer, 2003).
La anemia falciforme es una enfermedad causada por la presencia de una hemoglobina anormal llamada hemoglobina S (HbS). La HbS es producto de una mutación en el sexto codón del gen de Beta globina (Ingram, 1956). La presencia de HbS genera protección en contra de la infección con malaria. Es por ello que se cree que esta mutación ocurrió en cuatro eventos independientes en Africa y un quinto en la India como una respuesta selectiva en contra de esta patología (Haldane, 1949). La identificación de estos cuatro eventos mutacionales independientes se debió a la presencia en pacientes africanos de cuatro estructuras genéticas diferentes determinadas a partir de sitios de restricción dentro del cluster de Beta globina. Los haplotipos más frecuentes dentro de los afectados se denominaron típicos (Benín, Bantú, Senegal, Camerún, y Árabe/Hindú) mientras que los que presentaban bajas frecuencias fueron denominados atípicos (Pagnier et al., 1984; Lapouméroulie et al., 1992; Zago et al., 2000). Estos haplotipos típicos ligados a la mutación para HbS presentaban una marcada distribución geográfica en África y han servido tanto para la identificación de poblaciones africanas en otros continentes, así como como predictores de la severidad de la enfermedad (Nagel et al., 1987; Thein, 2008).
En Colombia la caracterización de esta enfermedad se ha centrado en la determinación de la frecuencia de la mutación en algunas poblaciones en alto riesgo (Silva et al., 1998; De Bernal et al., 2010; Satizabal Soto et al., 2013; Ortega et al., 2015), en la identificación de los haplotipos ligados a HbS y en el estudio de variantes moduladores de hemoglobina fetal (Cuellar-Ambrosi et al., 2000; Fong et al., 2013; Fong et al., 2015). Sin embargo, continúa siendo desconocido el efecto del mestizaje de la población colombiana sobre las características genéticas de los afectados con anemia falciforme. Por ejemplo, ¿los haplotipos atípicos que se reportan en esta población son producto de recombinación de haplotipos típicos africanos o fueron adquiridos por medio de cruzamiento de individuos africanos con personas de otros grupos étnicos presentes en Colombia? Este trabajo tuvo como objeto identificar los haplotipos presentes en una muestra de personas con anemia falciforme, evaluar el grado de desequilibrio de ligamiento que hay entre los marcadores que definen estos haplotipos, y comparar esta población con otras poblaciones de afectados como de no afectados para evidenciar relaciones genéticas.
MATERIALES Y MÉTODOS
Muestra
Se tomaron muestras de sangre a 83 personas no emparentadas diagnosticadas previamente con anemia falciforme (homocigotos para hemoglobina S) en las ciudades de Buenaventura (siete individuos), Cali (29 individuos), Cartagena (39 individuos) y Montería (ocho individuos). Todos los participantes firmaron un consentimiento informado previo al enrolamiento en el estudio. Esta investigación contó con la aprobación del comité de ética de la Universidad del Valle (acta 09-09). A cada participante se le tomó una muestra de sangre de 4ml, la cual se almacenó en tubos con EDTA como anticoagulante. Los tubos se almacenaron a 4 °C hasta el momento de la extracción de ADN.
Extracción de ADN
A partir de las muestras de sangre se extrajo ADN por medio de "salting out" (Miller et al., 1988) con modificaciones. A 1 ml de sangre se adicionó 4,5 ml de solución de lisis de células rojas frío (NH4Cl 15,5mM, NaHCO3 10 mM, EDTA 0,1 mM, pH 7,4). Se incubó en hielo por 20 minutos, mezclando suavemente durante diez minutos. Después se centrifugó por diez minutos a 7000 rpm. Se eliminó el sobrenadante y se resuspendió la pastilla de leucocitos en 3,5ml de solución de lisis de células rojas se incubó en hielo por 15 minutos y se centrifugó a 7000 rpm. Se eliminó el sobrenadante y se añadió 1ml de solución de lisis de células rojas, se centrifugó cuatro minutos a 7000 rpm y se eliminó el sobrenadante. El botón se resuspendió en 300ul de solución lisis de leucocitos (10mM Tris-HCl, 400mM NaCl, 2mM EDTA ph 8,2) más 20ul de dodecilsulfato de sodio (SDS) al 10 % y 20ul de proteinasa K (20ng/ml). Esta solución se incubó por 20 horas a 37 °C. Pasado la incubación a la solución se agregó 300ul de NaCl (6M) y se agitó por 15 segundos, posteriormente se centrifugó a 7000 rpm. El sobrenadante se transirió a un tubo limpio y se adicionó 1ml de etanol absoluto frío. El tubo se invirtió entre cuatro a cinco veces hasta observar ilamentos de ADN. El ovillo de ilamentos de ADN se transfirió a un tubo de 1,5ml limpio que contenía 150ul de low TE 1x (10Mm Tris HCl). El ADN se dejó a 37 °C por 24 horas.
Amplificación de sitios de restricción y digestión enzimática
La determinación del genotipo para el locus para hemoglobina S se hizo amplificando la región 5' del gen Beta globina siguiendo las indicaciones de Attila et al. (2004). Los productos de PCR fueron digeridos usando la enzima de restricción Ddel (Fermentas, Vilnius, Lituania) de acuerdo a las indicaciones del fabricante y se incubó por 16 horas a 37 °C. Los fragmentos de digestión se visualizaron en geles de poliacrilamida (PAGE) al 8 % con tinción de nitrato de plata.
Para la determinación de los haplotipos se usaron cinco sitios de restricción dentro del cluster de Beta globina: 5'Épsilon globina, IVSII Gamma globina G, IVSII Gamma globina A, pseudo gen Beta, 3' pseudo gen Beta. Los sitios de restricción fueron ampliicados usando los primers y condiciones de PCR publicadas por Varawalla et al. (1992). Para la digestión enzimática se utilizaron las enzimas de restricción HincII (Fermentas, Vilnius, Lituania) para los sitios de restricción 5' Épsilon, pseudogen Beta, y 3' pseudogen Beta y la enzima HindIII (Fermentas, Vilnius, Lituania) para los sitios de restricción IVSII Gamma G globina e IVSII Gamma A globina. Las condiciones de la digestión se hicieron siguiendo las indicaciones del fabricante y se incubó por 16 horas a 37 °C. Los fragmentos de restricción se separaron en geles de acrilamida (PAGE) al 8 %, la visualización de los fragmentos se hizo con tinción por nitrato de plata.
Construcción de los haplotipos
Debido a que la muestra poblacional estaba formada por individuos no emparentados la construcción de los haplotipos se hizo estimando la fase gamética. La formación de estos haplotipos y la estimación de su frecuencia alélica y genotípica se hizo por medio de la función haplotype del paquete haplo.stats v1,7,7 (Sinnwell y Schaid, 2016) para la plataforma R.
Análisis estadisticos
La heterocigosidad de los haplotipos se calculó de la forma señalada por Rao (Rao, 1982). Se calculó la distancia entre los haplotipos (distancia de Hamming), con esta información se construyó una red de haplotipos usando la función haploNet del paquete pegas v 0,10 (Paradis, 2010). La estimación del grado de desequilibrio de ligamiento (LD) entre los sitios de restricción se hizo por medio del programa haploview 4,1 (Barrett et al., 2005). Para comparar la similitud o diferencias entre la población de estudio y otras poblaciones de afectados con anemia falciforme se calculó Fst's pareados (Wright, 1951) entre estas poblaciones por medio del programa arlequín 3,5 (Excoffier y Lischer, 2010). Estos valores de Fst's se utilizaron para graicar la distancia entre las poblaciones por medio de la función table.paint del paquete ade4 para la plataforma R (Dray y Dufour , 2007). Este mismo procedimiento se hizo para comparar la similitud entre la población de estudio y poblaciones indígenas colombianas, europeas, y africanas que no presentaban hemoglobina S. Las frecuencias de los haplotipos en las poblaciones de afectados americanos fueron tomados de diferentes estudios publicados (Tabla 1), mientras que las frecuencias en las poblaciones sin hemoglobina S fueron tomadas de la base de datos ALFRED (Kidd et al., 2003).
RESULTADOS
En la población de estudio se observaron 33 Genotipos, y de estos solamente 11 presentaron frecuencia igual o superior a dos por ciento. Los genotipos más frecuentes presentaron los haplotipos II y VII. El heterocigoto II/VII fue el más común (16,9 %), seguido por II/II (10,8 %). Algunos genotipos con haplotipos atípicos tuvieron frecuencias superiores a dos por ciento como es el caso de I/II (4,8 %), e incluso se lograron observar en estado homocigoto como es el caso del I/I. La heterocigosidad observada en esta población fue de 0,79.
En la población estudiada se inirieron 17 haplotipos (Fig. 1). 58 (35,1 %) cromosomas presentaron el haplotipo II o haplotipo Benín, haciéndolo el más frecuente, con el haplotipo VII (Bantú) se observaron 43 cromosomas (26,5 %). Los otros dos haplotipos típicos de origen africano también fueron observados, cinco cromosomas (2,6 %) tuvieron el haplotipos IX (Senegal) y tres cromosomas (1,7 %) contenían el haplotipo XI (Camerún). Dentro del grupo de haplotipos atípicos, el más predominante fue el haplotipo I (-----), 16 cromosomas (10 %) presentaban este haplotipo. El segundo haplotipo atípico más frecuente en nuestro estudio fue VIII (- +-- +), 10 cromosomas (6,4 %) presentaban este haplotipo. Este alelo presenta una amplia distribución en diferentes poblaciones del mundo, en pacientes con anemia falciforme en Colombia su frecuencia es incluso más alta que haplotipos típicos como Senegal o Camerún.
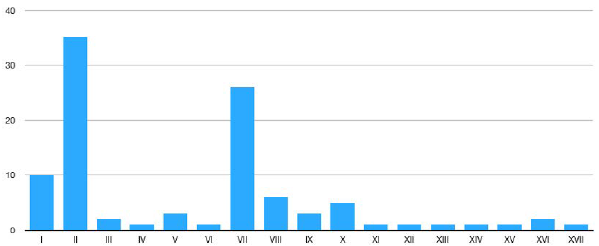
Figura 1 Frecuencia de haplotipos en pacientes con anemia falciforme en Colombia. Los haplotipos típicos en la población con anemia falciforme son II (Benín), VII (Bantú), IX (Senegal), y XI (Camerún). Los haplotipos más comunes fueron el II y VII. Un haplotipo no africano con frecuencia apreciable (10 %) es el haplotipo I, típico de población europea.
Dentro de nuestra muestra de pacientes se observaron haplotipos que no estaban presentes en la base de datos Alfred o reportados en otros estudios. Estos son los haplotipos III (--- + -), V (--+-+), VI (--+ + +), XV (+-+-+), y XVII (+ +-+ -). En general estos haplotipos tuvieron una frecuencia muy baja en nuestra población.
Relación entre haplotipos
Para conocer qué tipo de relación evolutiva podrían tener los haplotipos observados en la población de estudio se realizó un análisis de networks (Fig. 2). La red resultante muestra al haplotipo I como un haplotipo central de donde derivan dos subredes. Long et al. (1990) clasificaron los haplotipos en el cluster de Beta globina en dos tipos: primer y segundo orden. Los de primer orden difieren en un solo paso de mutación o conversión génica entre ellos; los de segundo orden son productos de eventos de recombinación (Long et al., 1990). El haplotipo I (-----) se encuentra central en la red de haplotipos resultante mostrando su posición como haplotipo ancestral (Chen et al., 1990; Long et al., 1990;). El haplotipo II (-----+) es un haplotipo de primer orden, de este se derivan los haplotipos VIII (- +--+) y XIII (+-+) los cuales son haplotipos de segundo orden. A partir del haplotipo IV (--- + +) se forma una pequeña red con el haplotipo VI (--+ + +) y el haplotipo IX (- +-+ +). El haplotipo VI no ha sido reportado y puede ser producto de recombinación entre haplotipos II y IV. Otra red formada a partir del haplotipo II es la conformada por los haplotipos V, XV, XI. Los dos primeros no han sido reportados, el haplotipo XI, es el haplotipo Camerún, es un haplotipo presente en poblaciones del viejo mundo (Africa, Europa, Asia).
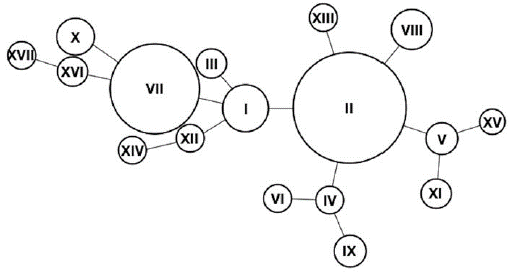
Figura 2 Red de haplotipos observados. Se puede ver como los haplotipos derivan del haplotipo I o del haplotipo II.
A partir del haplotipo I se forman dos redes, mientras que el haplotipo III (--- + -) se deriva de I en solitario. El haplotipo VII (Bantú) deriva del haplotipo I por un paso mutacional, del haplotipo VII se deriva el haplotipo X (un haplotipo de segundo orden) por un paso mutacional. Los haplotipos XVI (+ +-) y XVII (+ +-+ -) forman una pequeña subred derivada del haplotipo VII. El haplotipo XVI ha sido reportado en otros estudios y es clasificado como haplotipo de orden dos, por el contrario, el haplotipo XVII no ha sido reportado. Los haplotipos XII (+ -----) y XIV (+-+--) forman otra subred. El haplotipo XII junto al haplotipo I son considerados como haplotipos ancestrales ambos se diferencian en nuestra red por un paso mutacional. El haplotipo XIV se diferencia del XII por un paso mutacional.
Desequilibrio de ligamiento entre los sitios de restricción
El desequilibrio de ligamiento (LD) entre los sitios de restricción fue bajo para algunos sitios y en otros inexistente. En la Figura 3A se observa los valores de D' y se puede ver que son bajos en general. Solamente 3' pseudogen Beta y Gamma G presentan LD, aunque su valor de D' no es muy alto. Para comprobar si la presencia de haplotipos diferentes a los típicos puede ser un factor en la reducción del desequilibrio de ligamiento entre los sitios de restricción estudiados se hizo el cálculo de desequilibrio de ligamiento únicamente a los 34 individuos de la muestra que presentaron solo haplotipos "típico" en sus genotipos. El resultado se puede ver en la Figura 3B. Existe un aumento en el grado de desequilibrio de ligamiento entre los sitios de restricción. De nuevo el LD entre 3'pseudogen Beta y Gamma G es el más fuerte, los otros sitios de restricción pasan de no tener ningún grado de LD a poseer niveles medios. Esto demostraría que existe una influencia de alelos introducidos a la población de afectados desde poblaciones europeas y amerindias en el grado de LD entre los sitios de restricción.
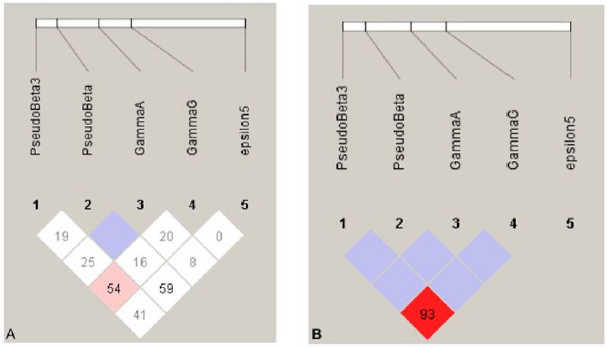
Figura 3 Desequilibrio de ligamiento entre sitios de restricción. La intensidad del color indica la fuerza del LD, es decir los cuadros blancos indican ausencia de LD, los cuadros violetas indican un LD intermedio (LOD < 2), los cuadros rojos brillantes indican un fuerte valor de LD (LOD> 2). La barra de arriba muestra la posición relativa de los sitios de restricción. La figura A muestra el grado de desequilibrio entre los sitios de restricción evaluados en los 83 individuos estudiados. Se puede observar el bajo o nulo nivel de desequilibrio. La figura B muestra el grado de desequilibrio de ligamiento solamente tomando en cuenta los 34 individuos que tenían haplotipos típicos para HbS.
Relación entre la población con anemia falciforme y otras poblaciones
Las frecuencias alélicas de la población de estudio se compararon con las observadas en otras poblaciones americanas de afectados con anemia falciforme. En la tabla 1 se muestra la frecuencia de haplotipos típicos y atípicos en estas poblaciones americanas . Los valores de Fst estuvieron dentro de un rango entre 0,06 y 0,23. Las poblaciones con las que se presentó una mayor similitud fueron dos poblaciones de afectados venezolanos, mientras que la mayor diferencia se evidenció con la población mexicana (Fig. 4). Se comparó también las frecuencias haplotípicas de la población de estudio con poblaciones africanas, europeas y de indígenas colombianos sin la enfermedad. Los valores de Fst estuvieron entre 0,05 y 0,48. En este caso la mayor similitud se observó con las poblaciones africanas Lao y Mandenka (0,05 y 0,09 respectivamente), mientras que el mayor grado de diferencia fue visto con los indígenas colombianos de las tribus Kamsa y Wayuu (0,48 para ambas poblaciones).

Figura 4 Similitud genética entre la población de afectados de Colombia (estudio actual) y otras poblaciones de afectados de América. Las frecuencias son extraídas de los estudios reportados en la tabla 1. Los cuadros blancos indican menores diferencias genéticas, los cuadros oscuros representan mayores diferencias. Se puede ver como la mayor similitud genética se presenta con las poblaciones de Venezuela y la mayor diferencia se presentó con la población mexicana. Cp= Ciudad de panamá, Ba y Bah= Bahía, Am=Amazonas, Ar= Aragua, Su= Sucre, Be= Belém, Cc= Costa Chica, Ha= La Habana, To= Toronto.
DISCUSIÓN
La frecuencia de los haplotipos típicos encontrados en la población de estudio no diiere de lo reportado en otras poblaciones de afectados en América, en donde los haplotipos Benín y Bantú son los más frecuentes (Moreno et al., 2002; Lemos Cardoso y Farias Guerreiro, 2006; Okumura et al., 2013). Este resultado mostraría la procedencia de la población africana llegada a Colombia, que sería principalmente del golfo de Guinea y del África centro-oriental (Pagnier et al., 1984). Sin embargo, hay que señalar que la gran mayoría de los estudios presentan un porcentaje bajo de haplotipos atípicos (Tabla 1), mientras que en nuestra población el 35 % son haplotipos de este tipo. Este resultado puede ser explicado por un extensivo proceso de mestizaje entre grupos africanos, amerindios, y europeos. Datos genéticos muestran que la población afrodescendiente en Colombia presenta un mestizaje de tres vías, con un alto porcentaje de ancestría africana (76 %), el resto del componente se divide en europeo (13 %) y amerindio (11 %). El tiempo para el inicio de este evento de mestizaje se calcula en aproximadamente diez generaciones atrás desde el presente (Conley et al., 2017).
Tabla 1 Frecuencia relativa de haplotipos típicos y atípicos para hemoglobina S en diferentes poblaciones de América.
| Estudio/País | N°. de cromosomas | Benin | Bantú | Senegal | Camerún | Árabe | Atípicos |
| (Rusanova et al., 2011)/Panamá (Ciudad de Panamá) | 200 | 30,0 | 51,0 | 8,5 | 4,0 | 1,0 | 5,5 |
| (Goncalves et al., 2003)/Brasil (Bahía) | 80 | 45,6 | 48,1 | 0,6 | 0,0 | 0,0 | 5,6 |
| (Pante-de-Sousa et al., 1999)/Brasil (Amazonas) | 20 | 10,0 | 60,0 | 30,0 | 0,0 | 0,0 | 0,0 |
| (Moreno et al., 2002)/Venezuela (Aragua) | 96 | 40,6 | 47,9 | 3,1 | 0,0 | 2,1 | 6,3 |
| (Adorno et al., 2008)/Brasil (Bahía) | 160 | 55,2 | 41,6 | 0,4 | 1,2 | 0,4 | 1,2 |
| (Vívenes de Lugo et al., 2003)/Venezuela (Sucre) | 42 | 35,7 | 50,0 | 2,4 | 0,0 | 0,0 | 11,9 |
| (Lemos Cardoso y Farias Guerreiro, 2006)/Brasil (Belém) | 260 | 66,0 | 21,8 | 10,9 | 1,3 | 0,0 | 0,0 |
| (Magaña et al., 2002)/México (Costa Chica) | 33 | 18,2 | 78,8 | 0,0 | 0,0 | 0,0 | 3,0 |
| (Muniz et al., 1995)/Cuba (Habana) | 169 | 52,7 | 40,2 | 7,1 | 0,0 | 0,0 | 0,0 |
| (Goncalves et al., 1994)/Brasil (San Pablo) | 74 | 34,0 | 62,0 | 0,0 | 0,0 | 0,0 | 4,0 |
| (Oner et al., 1992)/Canadá (Toronto) | 61 | 49,2 | 11,5 | 13,1 | 13,1 | 0,0 | 13,1 |
| Colombia (estudio actual) | 166 | 35,0 | 26,0 | 3,0 | 1,0 | 0,0 | 35,0 |
Ahora, ¿cómo estos haplotipos pudieron alcanzar un porcentaje como el observado en la población de afectados con anemia falciforme en Colombia?. Esto pudo haber sucedido a través de deriva genética. En poblaciones pequeñas y aisladas como la de los palenques la deriva génica puede aumentar la frecuencia alélica de variantes raras (haplotipos no africanos) si estas poblaciones hubiesen mantenido un crecimiento constante después de la fundación de las mismas (Templeton, 2006). Los haplotipos no africanos pudieron llegar a la población por mestizaje con europeos durante el periodo de servidumbre, así como por mestizaje con grupos indígenas ubicados cerca al palenque (Navarrete, 2001). Un dato que provee evidencia a un fuerte efecto de deriva sobre la frecuencia de haplotipos en esta población es el caso de los haplotipos XII (+-----) y I (-----). El primero de estos haplotipos es muy abundante en poblaciones indígenas colombianas (superior al 80 %) (Shimizu et al., 2004), pero a pesar de ello en la población de estudio este haplotipo alcanzó 1 %. Por el contrario, el haplotipo I (-----) que no ha sido reportado en poblaciones africanas, que es poco frecuente en indígenas suramericanos (Kaufman et al., 1998) y que tiene una frecuencia entre 3-6 % en poblaciones del Mediterráneo (Latini et al., 2003) alcanzó en la población de estudio un 10 %. Es importante mencionar que el haplotipo I (-----) no ha sido observado anteriormente en población indígena colombiana, lo que podría indicar que este tiene un origen europeo.
Cinco de los haplotipos observados en los afectados con anemia falciforme no se encontraron en bases de datos (III, V, VI, XV, y XVII). Estos haplotipos fueron encontrados en baja frecuencia 1-3 %. Dado su baja frecuencia es posible que estos haplotipos puedan ser producto de recientes eventos mutacionales o de recombinación. Por ejemplo, el haplotipo XVII (+ +-+ -) es muy similar al haplotipo Arabe/ Hindú (+ +-+ +), el cual es muy frecuente en afectados con anemia falciforme en estas dos regiones (Kulozik et al., 1987). Similar situación tiene el haplotipo XV que se diferencia por un sitio de restricción con un haplotipo ya observado en población indígena colombiana (+-+--), también presente en baja frecuencia (Shimizu et al., 2004).
A pesar de que la población de estudio eran todos individuos homocigotos para la hemoglobina S, esta población presentó un número de haplotipos bastante similar a los encontrados en poblaciones sanas del mundo. La heterocigosidad en la población de estudio fue de 0,79, la cual es mayor a la reportada para varias poblaciones a nivel mundial (0,593) (Chen et al., 1990; Long et al., 1990). Una explicación para este gran número de haplotipos puede estar relacionado a los procesos de recombinación presente dentro del cluster de Beta globina que incrementan el número de haplotipos. También, el número de haplotipos pudo haber sido influenciado por el hecho de las diferencias en la frecuencia de haplotipos presentes entre poblaciones europeas, amerindias y africanas (Chen et al., 1990; Long et al., 1990).
Un fuerte desequilibrio de ligamiento se ha observado dentro del cluster de Beta globina en estudios previos. Chakrevarty etal. (1984) encontraron dos agrupaciones de sitios de restricción que presentaban asociaciones altamente no aleatorias. Estas dos agrupaciones están separadas por una región ubicada 5' al gen Beta globina con un alto nivel de recombinación, lo cual lo califica como un hot spot para recombinación meiótica (Chakravarti et al., 1984). En población de afectados con anemia falciforme se ha encontrado un fuerte y extenso ligamiento (>400kb) en el cluster de Beta globina sobretodo en poblaciones de África y del Caribe (Hanchard et al., 2007). Liu et al (2009) utilizando los cinco sitios de restricción usados en el presente estudio encontraron en pacientes con anemia falciforme afroamericanos un bloque de ligamiento que va desde 5' Épsilon hasta Gamma A (21kb). Este bloque de ligamiento es de menor tamaño al observado en individuos sanos el cual se extendía hasta pseudo gen Beta (27kb), sugiriendo esto un menor nivel de ligamiento en los pacientes con anemia falciforme (Liu et al., 2009). En nuestros resultados los sitios de restricción en general no presentan ligamiento (cuadros blancos Fig. 4A). Este resultado puede ser debido al mestizaje. Aunque el mestizaje puede crear ligamiento entre sitios, esto sucederá si los alelos provienen de la misma población ancestral (Pfaff et al., 2001). Dado que sitios de restricción como 5' Épsilon, Gamma G, y pseudo gen Beta presentan diferencias en las frecuencias alélicas entre poblaciones (1000 Genomes Project Consortium, 2015), es posible que alelos provenientes de poblaciones distintas hayan influenciado en la caída de LD entre los sitios de restricción observados en la población de estudio. Esto se evidencia al analizar los individuos con haplotipos típicos donde se observa un aumento del LD entre los sitios de restricción (Fig. 4B).
A partir de sus diferencias genéticas los haplotipos observados formaron dos grandes grupos, y todos son derivados de los haplotipos, I (-----) y II ( ----+), el primero al parecer es el haplotipo ancestral en los humanos (Chen et al., 1990) el segundo es un haplotipo de primer orden, es decir haplotipos presentes en casi todas las poblaciones humanas que provienen de mutación o recombinación entre haplotipos de primer orden (Chen et al., 1990; Long et al., 1990). Otros haplotipos definidos como de primer orden (VII, VIII, IX, XI), en nuestra red se encuentran separados por un evento mutacional a excepción de los haplotipos IX y XI. Estos, parecen derivar del haplotipo IV, el cual es un haplotipo de segundo orden y del haplotipo V, que es un haplotipo no observado en otras poblaciones. Es posible que los haplotipos IX y XI sean producto tanto de eventos mutacionales de haplotipos de primer orden como de aquellos menos frecuentes, hay que tener en cuenta que los eventos de recombinación pueden producir haplotipos idénticos en estado por diferentes modos (Long et al., 1990).
Cuando se comparó la población de afectados de Colombia con los de otras regiones de América se encontró que los valores de Fst rondaban entre valores de baja a moderada estructuración. Esto puede ser producto del tráfico de esclavos durante el período colonial en América. Los esclavos llegados a los actuales países americanos venían a través de un comercio instalado por potencias europeas (Portugal, Holanda, Inglaterra) que transportaba individuos desde diferentes partes de África, los cuales eran luego distribuidos en las colonias en América (Pavy, 1967). Por lo tanto, es muy factible que individuos provenientes de zonas en África de alta frecuencia de haplotipos Benín, Bantú, Senegal, Camerún fueran distribuidos en distintas colonias de América y por ello mostrar estos niveles bajos a medios de estructuración. La similitud de nuestra población con las dos poblaciones de Venezuela puede ser debido a que estas comparten con la población colombiana una distribución intermedia de los haplotipos Benin y Bantú. Del mismo modo la gran diferencia observada con la población de México puede ser por la alta frecuencia del haplotipo Bantú en esta población. Este fenómeno ya fue observado por Lindenau et al.(2016) ellos observaron que poblaciones con predominio del haplotipo Benín se agrupaban entre ellas, lo mismo sucedía con las poblaciones con predominio Bantú o con poblaciones con frecuencia intermedia de ambos haplotipos (Lindenau et al., 2016).
La comparación entre la población de estudio y poblaciones sin la presencia de hemoglobina S arrojó Fsts entre 0,05 y 0,48, mostrando una alta similitud con poblaciones africanas (Lao y Mandenka), mientras que la mayor diferencia se dio con el grupo indígena Wayuu (Fst=0,48). La similitud observada con poblaciones africanas puede ser debido al predomino de haplotipos africanos en nuestra población de estudio. Lo mismo puede explicar la gran diferencia con las poblaciones indígenas, los Kamsas y Wayuu presentan una frecuencia baja del haplotipo II, un haplotipo típico africano.
CONCLUSIONES
Nuestro estudio mostró que la variabilidad genética de los pacientes con anemia falciforme en Colombia va más allá de su herencia africana y que esta población también ha sido influenciada por el proceso de mestizaje tri-étnico de la totalidad de la población colombiana, generando una apreciable variabilidad genética dentro de los afectados con anemia falciforme. La alteración en el patrón de desequilibrio de ligamiento dentro del cluster de Beta globina en nuestra población con respecto a otras poblaciones de afectados, puede ser un ejemplo del efecto del mestizaje como una fuerza que cambia las relaciones entre loci, lo cual puede tener un efecto en estudios de asociación, sobretodo en los casos en que se trata de verificar la asociación de variantes genéticas que han sido establecidas en poblaciones distintas a la colombiana.














