INTRODUCCIÓN
Los microorganismos potencialmente fitopatógenos, deben superar varios obstáculos para acceder a los nutrientes de las plantas. En primera instancia se encuentran las barreras físicas que poseen las plantas, como la cutícula, la pared celular, o las capas de cera. Además, deben hacer frente a la presencia de barreras químicas, algunas de las cuales pueden producirse de manera constitutiva como los metabolitos secundarios (Mysore y Ryu, 2004). Una vez superados estos obstáculos las plantas son capaces de detectar patógenos potenciales a través de receptores que identifican patrones moleculares asociados a microorganismos/patógenos o MAMPs/PAMPs (por sus siglas en inglés, Microbial/Pathogen Associated Molecular Patterns) (Boutrot y Zipfel, 2017). Este reconocimiento depende de receptores transmembranales conocidos como PRRs (Pattern Recognition Receptors) (Boutrot y Zipfel, 2017). La mayoría de los PRRs se caracterizan por poseer un dominio kinasa citoplasmático y pueden tener ya sea un dominio LRR (Leucine Rich Repeat) extracelular recibiendo el nombre genérico de RLKs (Receptor Like Kinases) o pueden tener un dominio extracelular formado por motivos LysM (Tang et al., 2017). Los PRRs establecen interacciones moleculares con otras proteínas que pueden ser también RLKs o RLKs citoplasmáticos, RLCK (Receptor Like Citoplasmatic Kinases) (Monaghan y Zipfel, 2012). Esta primera línea de defensa es eficiente para controlar microorganismos potencialmente patógenos y recibe el nombre de PTI (del inglés PAMP Trigered Immunity) (Yu et al., 2017). La intricada red de interacciones de PRRs con otras proteínas conduce a una serie de fosforilaciones y transfosforilaciones las cuales son necesarias para activar vías de señalización mediadas por MAP kinasas y traen consigo la producción de especies reactivas de oxígeno, refuerzo de la pared celular a través de depósitos de calosa, cambios a nivel de canales iónicos, entrada de Ca+2 y finalmente la activación de factores de transcripción que producen una reprogramación de la expresión génica llevando a la restricción del patógeno (Bigeard et al., 2015). Con el objetivo de limitar esta respuesta inmune desencadenada por el reconocimiento de PAMPs, el patógeno ha desarrollado una serie de mecanismos que conllevan a la degradación controlada de PRRs mediante su interacción con proteínas de la familia E3 ubiquitin ligasas (Trujillo y Shirasu, 2010).
Durante el proceso co-evolutivo, algunas especies de microorganismos desarrollaron un grupo especial de proteínas denominadas efectores, las cuales son introducidas al interior del citoplasma de la célula hospedera (Toruño et al., 2016). Para el caso particular de bacterias patógenas los efectores son translocados a través del sistema de secreción tipo tres TTSS (Type Three Secretion System) (Deslandes y Rivas, 2012). La función de los efectores es bloquear la respuesta inmune de tipo PTI de la planta a través de diferentes mecanismos (Toruño et al., 2016). Las plantas a su vez desarrollaron otro grupo de receptores, los cuales en su mayoría son citoplasmáticos, denominados proteínas de Resistencia (R), y que reconocen de manera altamente específica los efectores de los patógenos adaptados (Jones et al., 2016). Cuando un efector determinado es reconocido por una proteína R recibe el nombre de proteína Avr. Este reconocimiento desencadena diferentes tipos de respuestas algunas sobrelapantes con la PTI pero la más conspicua es la respuesta hipersensible o HR (Hypersensitive Response), que consiste en una muerte celular programada que permite restringir la colonización del patógeno a tejidos circundantes del punto de infección (Zhang et al., 2017). El reconocimiento de la proteína de Avr por medio de la proteína R puede ser de manera directa o indirecta. En el primer caso la proteína R actúa como receptor y la proteína Avr como ligando. En el caso de la interacción indirecta, la proteína efectora se une a una proteína de la planta que no es la proteína R, denominada blanco de patogenicidad, generando algún tipo de modificación (fosforilación, uridilación, clivaje, etc). La proteína R reconoce por su interacción con la proteína blanco de patogenicidad la modificación que el efector ha hecho de ella, activando así la señalización que conduce a la resistencia (Baggs et al., 2017). La mayoría de proteínas R conocidas también como NB-LRRs o NLRs se caracterizan por poseer un dominio TIR (Toll Interleukin Related) o CC (Coiled-coil) en el extremo N-terminal, seguido de un dominio NB-ARC (Nucleotide Binding ) y de un dominio LRR (Leucine Rich Repeat) en su extremo C-terminal (Jones et al., 2016; Baggs et al., 2017).
La yuca, Manihot esculenta, es una planta de la familia Euphorbiaceae, originaria del sur de la cuenca amazónica (Olsen y Shaal, 1999). Inicialmente fue cultivada en el neotrópico americano, pero hoy su cultivo se extiende a lo largo de toda la franja ecuatorial, incluyendo países de Africa y Asia. Se estima que anualmente se emplean 96 millones de toneladas de la raíz en alimentación, subproductos industriales derivados del almidón y recientemente biocombustibles (FAO, 2017). La yuca representa la principal fuente de calorías para más de un billón de personas y presenta varias características excepcionales como su capacidad de adaptación a suelos pobres y ácidos, un rango amplio de crecimiento en cuanto temperaturas (entre los 10 oC y 40 oC), alturas (entre los 0 y 1800 m.s.n.m) y de precipitación (Ceballos y De la Cruz, 2012). Todas estas características hacen que la yuca se constituya en el cuarto cultivo más importante en el mundo. Dentro de las limitaciones más importantes para el desarrollo de la yuca, se encuentra la bacteriosis vascular, enfermedad que puede alcanzar a ocasionar pérdidas de hasta el 100 % de la producción (Lozano, 1986). El agente causal de esta enfermedad es Xanthomonas axonopodis pv. manihotis (Xam), una bacteria gram negativa (Lozano y Sequeira, 1974). La enfermedad tiene un primer ciclo de infección epifítico en donde se reproduce para después penetrar durante la época lluviosa a través de estomas y heridas al tejido vascular. Es al comenzar esta segunda fase de infección que empiezan a presentarse síntomas como manchas angulares, seguidos por marchitamiento, exudados en los tallos y finalmente la muerte de la planta (Verdier et al., 2012). Si bien existen variedades que presentan cierto grado de resistencia a algunas cepas de Xam (Trujillo et al., 2014), estas no poseen buenos atributos agroculinarios y en consecuencia no han sido adoptadas por los productores. Si bien en los últimos años se han logrado grandes avances por conocer las bases moleculares de la resistencia de la yuca a la bacteriosis vascular (Muñoz et al., 2014; Soto et al., 2015; 2017), hasta ahora no se han identificado proteínas PRR y/o R en yuca a ninguno de sus patógenos. Recientemente se reportó la identificación del gen RXam1 el cual codifica para una proteína de tipo RLK, la cual colocaliza con un QTL (del inglés Quantitative Trait Locus) que explica el 16 % de la resistencia a la cepa Xam CIO136 (Jorge et al., 2000). La sobreexpresión de este gen incrementó de manera significativa la resistencia de la variedad de yuca susceptible 60444 a esta cepa, por lo cual se considera que es un gen realmente implicado en la resistencia a la bacteriosis vascular (Diaz et al., 2018). La identificación de proteínas que interactúan con RXam1 se constituye en el primer paso hacía determinar su mecanismo de acción y así mismo las posibles vías de regulación de su actividad. En este trabajo se buscó identificar las proteínas de yuca que interactúan con el dominio Ser/Tre kinasa (STK) citoplasmático de esta proteína empleando la estrategia de doble híbrido.
MATERIALES Y MÉTODOS
Amplificación y clonación del dominio STK de RXam1
A partir de la secuencia de RXam1 se diseñaron los cebadores STKF 5' agtcgcaaacccagttcatc 3' y Cassa 5'tcaggggccattaacttgtcgacaagttaatggcccctga 3'que cubrieran la región del dominio kinasa citoplasmático permitiendo la amplificación de un fragmento de aproximadamente 1000 pb. Para la reacción de PCR se empleó el ADN de la variedad resistente MBRA685 el cual fue extraído a partir de 150 mg de tejido foliar almacenado en nitrógeno líquido basado en el protocolo de Dellaporta et al., (1983). La reacción de PCR se llevó a cabo en un volumen final de 10 μl, con una concentración de 1 X de Buffer de PCR (DreamTaq Buffer, Fermentas), 0,2 mM de dNTPs, 0,1 μM de cada primer, 20 mM MgCl2. y 0,25 U de Taq polimerasa (DreamTaq, Fermentas). Las reacciones de PCR se efectuaron en un termociclador MyCyclerTM BioRad® utilizando los siguientes ciclos de temperatura: denaturación a 95 °C por 2 minutos, 30 ciclos de amplificación con 30 segundos de apertura a 95 °C, 30 segundos de alineamiento a 54 °C y 1 minuto de extensión a 72 °C, con una extensión final a 72 °C por 5 minutos. El producto de amplificación se verificó por electroforesis en gel de agarosa al 1 % teñido con bromuro de etidio en buffer TAE 0.5 X. El tamaño del fragmento amplificado se confirmó empleando el marcador de peso molecular 1 Kb Plus Ladder ® (Invitrogen, Carlsbad, CA, U.S.A.). El amplicón obtenido fue eluido a partir del gel de agarosa empleando el kit Gel DNA extraction (Qiagen) y el producto se clonó en el vector de entrada al sistema Gateway pENTR/SD/D-TOPO siguiendo las instrucciones del fabricante (Invitrogen). El producto de clonación fue transformado en células competentes de Escherichia coli DH10B mediante electroporación empleando el equipo MicroPulserTM BioRad. Las células se incubaron una hora a 37 °C en 1 mL de medio 2 XYT (triptona 16 g/L, extracto de levadura 10 g/L, cloruro de sodio 5 g/L) con agitación constante. Las bacterias se sembraron en medio LB sólido (extracto de levadura 5 g/L, Triptona 10 g/L, cloruro de sodio 5 g/L y agar 15 g/L) con selección para ampicilina a 100 μg/μL y se incubaron a 37 °C por 12 h. Se confirmó la presencia de los clones positivos elegidos al azar mediante PCR de colonia, bajo las condiciones descritas anteriormente. A partir de los clones positivos se realizó el aislamiento del ADN plasmídico, empleando el kit GeneJET Plasmid Miniprep (Fermentas) utilizando 10 mL de un cultivo en medio LB líquido con ampicilina 100 μg/μL, el cual fue empleado para transferir el dominio STK al vector pLAW11 mediante una reacción BP siguiendo las recomendaciones del fabricante (Invitrogen). Se realizaron los mismos protocolos de transformación y selección de colonias descritos previamente.
Transferencia de la librería de ADNc al vector pLAW10
A partir de la librería de ADNc de yuca, la cual se encontraba en el vector Gateway compatible pENTRY222 (González y López et al., 2008), se realizaron extracciones de ADN plasmídico a partir de tres alícuotas de la librería empleando el kit GeneJ ET Plasmid Miniprep (Fermentas). El producto total de estos plásmidos fue precipitado con polietilenglicol (PEG) y empleado para transferir los insertos al vector pLAW10 mediante una reacción LR (Invitrogen). El producto total de la reacción fue precipitado con etanol y empleado para transformar células de E.coli DH10B como se describió en la sección anterior. El total de bacterias obtenidas se recuperó para realizar nuevamente una nueva extracción de ADN plasmídico, el cual fue empleado para la transformación de levadura.
Transformación de levadura y ensayo de doble híbrido
Los ensayos de doble híbrido se llevaron a cabo utilizando las cepas de Saccharomyces cerevisiae Y187 y AH109, proporcionadas por el Dr R. Michelmore, UC Davis. Para su crecimiento se empleó el medio YPDA (extracto Levadura 1 %, Peptona 2 %, Dextrosa 2 %, Adenina 0,04 % y Agar 2 %). Para la elaboración de cepas competentes de levadura se empleó el protocolo descrito por Clontech, (2009) con leves modificaciones. Las cepas se crecieron en medio YPD líquido durante 16 horas a 30 °C con agitación constante (250 rpm). El cultivo fue transferido a medio YPD fresco y se incubó bajo las mismas condiciones durante seis horas. Cuando el cultivo alcanzó una densidad óptica a λ = 600 nm de 1,0 se centrifugó a 2000 rpm por 5 minutos. El precipitado fue lavado varias veces con agua destilada y centrifugado a 2000 rpm por 5 minutos. Después se resuspendieron en una solución de TE/LiAc (LiAC 110 mM, TE 1.1X) y posteriormente se realizaron dos lavados con la misma solución.
La cepa de levadura Y187 se empleó para transformar la librería de ADNc en el vector pLAW10, mientras que la cepa AH109 se transformó con el vector pLAW11 conteniendo el dominio STK. El método de transformación de levadura se basó en el protocolo descrito por Clontech, (2009) utilizando 50 μL de levadura competente, 100-300 ng del vector correspondiente en cada caso y 50 μg de esperma de salmón denaturado (Invitrogen, Carlsbad, CA, USA). A la reacción de transformación se adicionaron 500 μL de la solución PEG/LiAc (50 μL de LiAC 1 M, 50 μL de TE 10 X y 400 μL de PEG 3350 50 %) y 20 μL de DMSO 100 % (Sigma®). Las levaduras transformadas fueron seleccionadas en medio SD (extracto de levadura sin aminoácidos 0,67 %, adenina 0,004 %, dextrosa 2 %, agar 2 % y suplemento de aminoácidos). Para seleccionar levaduras con el vector pLAW10 se utilizó el mismo medio SD pero sin triptófano (suplemento de aminoácidos sin triptófano 0,074 %) y para seleccionar el vector pLAW11 se utilizó el medio SD sin leucina (suplemento de aminoácidos sin leucina 0,069 %).
Para el tamizaje de los interactores de STK, se realizó el apareamiento entre las levaduras transformadas como se describió anteriormente, la Y187 conteniendo la librería de ADNc y la AH109 conteniendo el dominio STK. Para el apareamiento, se empleó 7 mL del cultivo con la cepa Y187, 200 ml de YPDA con cloranfenicol (100 μg/mL) y 5 mL de la cepa AH109. Este procedimiento se realizó por duplicado y se incubó a 75 rpm durante 24 horas a 30 °C. Las levaduras se lavaron con agua destilada tres veces para eliminar el medio de cultivo. Para la selección de los posibles interactores, el producto del apareamiento se plaqueó en el medio SD/QDO (extracto de levadura sin aminoácidos 0,67 %, dextrosa 2 %, agar 2 % y 1 X solución DropOut, Clontech). Se incubó a 30 °C por varios días hasta identificar crecimiento de levaduras las cuales se consideraron contener los posibles interactores.
Confirmación de los interactores y secuenciación
El miniprep de levadura se llevó a cabo mediante lisis alcalina y utilizando 3 U de ZymoliaseTM (Zymo Research, CA, USA) (Clontech, 2009). Con el plásmido producto del miniprep de levadura se transformó E. coli DH10BTM como se mencionó anteriormente. Para seleccionar las bacterias que presentaban el plásmido pLAW10, se plaqueó en medio LB con ampicilina (100 μg/mL). Las colonias que crecieron de la transformación se evaluaron mediante PCR de colonia empleando los primers del vector. De las colonias seleccionadas se extrajo el plásmido por lisis alcalina y se transformó nuevamente en la cepa Y187 de levadura. La confirmación de los interactores se realizó mediante apareamiento entre las levaduras Y187 obtenidas anteriormente con la levadura AH109 conteniendo pLAW11 con el dominio STK siguiendo el protocolo descrito precedentemente. El producto del apareamiento se plaqueó en medio SD/QDO e incubó a 30 °C. Los interactores evaluados se confirmaron si se presentaba crecimiento. De cada interactor se realizó miniprep con el kit GeneJET™ Plasmid Miniprep Kit (Fermentas, Glen Burnie, MD, USA) el cual fue secuenciado por la compañía MacrogenTM, Corea, utilizando los primers del vector. Las secuencias se editaron y analizaron con el programa Sequencher® versión 4.1.4. Las búsquedas de similitud se realizaron mediante el algoritmo BLAST en las bases de datos de yuca (www.phytozome.net) y del NCBI (http://blast.ncbi.nlm.nih.gov), con los parámetros por defecto.
Análisis Bioinformático
Para establecer la presencia de dominios en la proteína que interactúa con el dominio STK de RXam1 se empleó el programa CDART (https://www.ncbi.nlm.nih.gov/Structure/lexington/lexington.cgi). Para determinar la especificidad de la interacción entre RXam1 y la proteína CERF3-1, en primera instancia se identificaron potenciales ortólogos más cercanos de estas dos proteínas en la colección de doble híbrido "CHARGE" (http://charge.ucdavis.edu/charge_db/chargedb_index.php). Las secuencias fueron alineadas con MAFFT (Katoh y Standley, 2008) y los alineamentos fueron sometidos a análisis filogenético con RAxML (Stamatakis, 2014), usando máxima verosimilitud (ML), el modelo PROTGAMMAWAG de sustitución de aminoácidos y 1000 iteraciones. Los dendrogramas fueron vusializados en iTOL (http://itol.embl.de/). A partir de los dendrogramas se seleccionaron los genes más similares a RXam1 y CERF-3 para conducir los nuevos ensayos de doble hibrido.
RESULTADOS
Clonación del dominio STK de RXam1 en pLAW11
Empleando como molde el ADN de la variedad de yuca MBRA685 fue posible amplificar el fragmento correspondiente al dominio Ser/Tre kinasa (STK) de RXam1 el cual fue posteriormente clonado en el vector Gateway compatible S-D TOPO y posteriormente transferido al vector para doble híbrido pLAW11 el cual contiene el dominio de unión al ADN (BD, Binding Domain). La transformación de bacterias permitió obtener un alto número de colonias, de las cuales se seleccionaron ocho para determinar la presencia de STK mediante PCR de colonia. Como se observa en la figura 1, todas ellas presentaron un amplicón del tamaño esperado (Fig. 1). La secuenciación de dos de estos clones permitió corroborar la identidad del dominio STK de RXam1. Este mismo ADN plasmídico fue empleado para transformar la cepa de levadura AH109. Los primeros ensayos mostraron autoactivación del gen reportero, por lo cual fue necesario llevar a cabo una prueba con diferentes concentraciones de 3AT encontrando que a una concentración de 5 mM se eliminaba el fenómeno de autoactivación. Esta concentración fue empleada en el tamizaje de la librería.
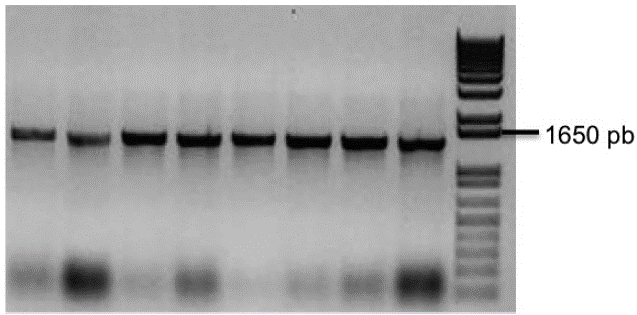
Figura 1 Confirmación de la clonación del dominio STK en el vector pLAW11. Producto de amplificación de 1100 pb a partir de ocho colonias tomadas aleatoriamente de la transformación de bacterias con el plásmido de doble hibrido pLAW11 conteniendo el dominio STK de RXam1. Para la amplificación se emplearon los primers del vector. Carril 1-8 clones bacterianos. Carril 9, marcador de peso molecular 1 kb+.
Transferencia de la librería de ADNc al vector pLAW10
Se partió de una librería de ADNc de yuca obtenida a partir de plantas in vitro de la variedad SG107-35 inoculada con XamCIO151. Esta librería se encontraba en el vector Gateway compatible pDONR222 y contenía insertos que variaban entre 600 -1500 pb (González y López, 2008). La librería fue transferida al vector de doble híbrido pLAW10 que contenía el dominio de activación (AD, Activation Domain) obteniéndose un título de 7x105 unidades formadoras de colonia (ufc). La calidad de la librería se evaluó por PCR de un grupo de colonias y como se observa en la figura 2, el tamaño de los insertos osciló entre 800 y 2000 pb (Fig. 2). También se secuenciaron 10 insertos de manera aleatoria encontrando que correspondían a transcritos diferentes y que poseían la cola polyA en el extremo 3' (datos no mostrados).
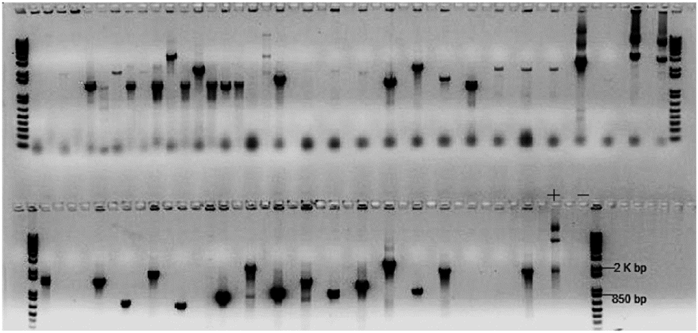
Figura 2 Amplificación mediante PCR de los insertos de la librería de ADNc en el vector pLAW10. A partir de colonias de la librería de ADNc obtenidas de la transformación con el plásmido pLAW10 con los insertos de ADNc en bacterias E. coli se realizó la reacción de PCR empleando los cebadores del vector. En los carriles de los extremos izquierdo y derecho se encuentra el marcador de peso molecular 1 kb+. El carril (+) indica PCR de un miniprep de una colonia conteniendo el dominio STK. Carril (-) indica PCR de una mezcla sin ADN molde. En los restantes carriles se representa la amplificación de cada uno de los clones de la librería de ADNc.
Dado que se buscó mantener la representatividad de la librería, todas las bacterias que crecieron en medio sólido fueron recuperadas con el objeto de evitar la amplificación logarítmica de los clones más comunes y se mantuviera la representación de los transcritos menos comunes. A partir de estas colonias se realizó la extracción del ADN plasmídico, el cual fue empleado para transformar las levaduras AH109, obteniéndose un título de 2x105 ufc.
Tamizaje de interactores del dominio STK de RXam1
Para identificar las posibles proteínas de yuca que interactúan con el dominio STK de RXam1 se procedió a realizar el ensayo de doble híbrido de levadura a través del apareamiento entre las levaduras AH109 conteniendo el dominio STK en el vector pLAW11 y las Y187 que contenía la librería de ADNc de yuca en el vector pLAW10. El producto del apareamiento se plaqueó e incubó por varios días (2-6 días) hasta observar crecimiento de colonias. La titulación del apareamiento permitió obtener en promedio 1,9 X 106 clones, por lo cual se realizaron seis apareamientos independientes, hasta lograr un tamizaje de 3x108 clones. Tras la primera selección después del apareamiento, fue posible identificar 200 colonias de levadura, las cuales fueron re-plaqueadas a un nuevo medio de selección. De éstas, 150 volvieron a presentar crecimiento. Se llevó a cabo miniprep de ellas y se re-transformaron células de E.coli. A las bacterias obtenidas se les realizó extracción de ADN plasmídico el cual fue utilizado para retransformar nuevamente la cepa de levadura AH109 conteniendo el vector pLAW11 con STK. Finalmente se obtuvieron cinco colonias de levaduras a las cuales se les extrajo el ADN plasmídico para llevar a cabo la secuenciación del inserto. La secuencia de estos plásmidos permitió determinar que el inserto de los cinco clones correspondía al mismo gen, el cual codifica para una pequeña proteína de 350 aminoácidos conteniendo un dominio central de dedos de zinc CHY (Valor E = 1 x10-9), seguido por un dominio C-terminal "Ring finger" y un "Zinc ribbon" (Valor E = 4x10-4) (Figura 3). Este gen fue denominado CRFE3-1 (Cassava RING Finger E3 ligase).

Figura 3 a. Secuencia del gen que codifica para CRFE3-1, proteína que interactúa con el dominio STK de RXam1. b. Representación esquemática de la proteína CRFE3-1. Se representan los tres dominios que presenta la proteína, identificados a través del programa CDART (https://www.ncbi.nlm.nih.gov/Structure/lexington/lexington.cgi).
Determinación de la especificidad de la interacción STK-CRFE3-1
Con el objeto de determinar la especificidad de la interacción entre STK y CRFE3-1 se procedió a realizar la prueba de la interacción recíproca, es decir evaluar la interacción al clonar el dominio STK en el vector pLAW10 y CRFE3-1 en pLAW11. Como resultado se encontró que CRFE3-1 es una proteína autoactiva cuando se encuentra en el plásmido que contiene el BD (Segunda línea Fig. 4b). Esto es de esperarse teniendo en cuenta que los dominios de dedos de zinc son superficies que fácilmente interactúan con el ADN.
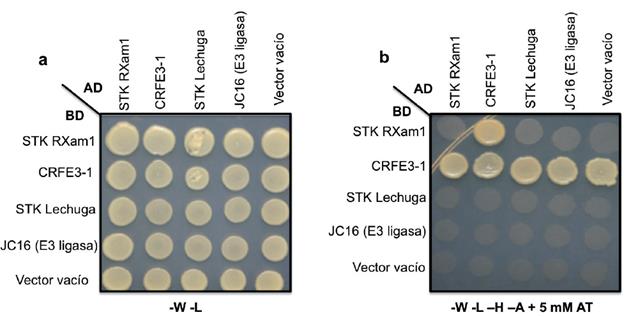
Figura 4 Determinación de la especificidad de la interacción entre STK y la proteína CRFE3-1 de yuca. a. Crecimiento de levaduras en medio sin triptófano ni leucina (-W -L) posterior al apareamiento entre cepas de levadura que contienen el vector con el dominio de unión al ADN (BD) y los insertos STK, CRFE3-1, RLK de lechuga y JC16 de Arabidopsis y el vector con el dominio de activación (AD) con los mismos insertos. Esta prueba confirma la presencia de los dos vectores en las levaduras. b. Crecimiento de levaduras en medio sin triptófano, leucina, histidina y alanina (-W -L -H -A) posterior al apareamiento entre cepas de levadura que se describen en A. Las colonias que sobreviven en el medio son aquellas en donde hay interacción. Se observa el fenómeno de autoactivación de CRFE3-1.
Como alternativa para determinar la especificidad se evaluó la interacción entre el STK de RXam1 con otro tipo de proteínas E3 ligasas. Para ello se identificaron posibles proteínas ortologas en otras especies y a partir de las secuencias obtenidas se construyó un dendrograma el cual permitió identificar a JC16, una proteína que corresponde a una ubiquitin E3 ligasa de tipo H2 de Arabidopsis (At1g19680) como la más cercana a CRFE3-1 de yuca (Fig. 5a). CRFE3-1 y JC16 presentan 30 % de identidad y 55 % de similitud. Al realizar el ensayo de doble hibrido entre STK de RXam1 y JC16 no se evidenció una interacción, demostrando que STK no interactúa indiscriminadamente con E3 ligasas (Fig. 4b). De igual manera se procedió a evaluar la especificidad de la interacción entre CRFE3-1 con el dominio STK. La búsqueda de posibles ortólogos de STK permitió identificar varias proteínas, dentro de las cuales, y a partir del dendrograma obtenido (Fig. 5b), se seleccionó el dominio STK del RLK de Lactuca sativa (LsAviH8TMK1), el cual presenta una alta similitud con el dominio kinasa de RXam1 (40 % identidad y60 % similitud). Por ensayos de doble hibrido se pudo demostrar que CRFE3-1 no interactuó con el dominio kinasa de LsAviH8TMK1 (Fig. 4b), dejando de manifiesto que la interacción encontrada entre CRFE3-1 y STK tiene alto grado de especificidad.
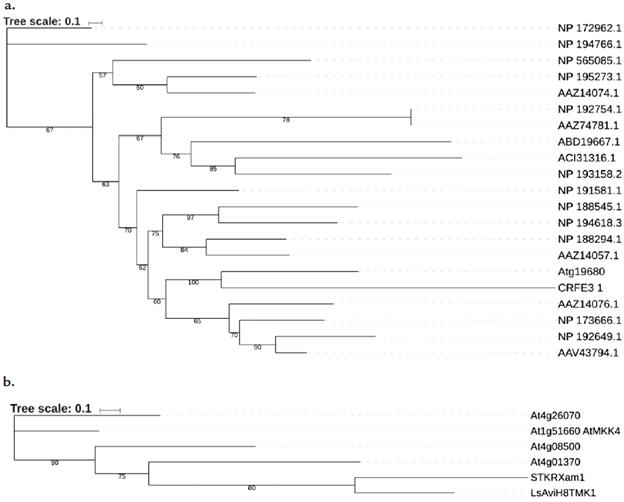
Figura 5 a. Dendrograma entre diferentes proteínas E3 Ubiquitin ligasas y el interactor CRFE3-1 de yuca. El empleado en los experimentos de especificidad por doble híbrido corresponde a Atg19680. b. Dendrograma entre diferentes dominios STK de RLKs y el dominio STK de RXam1. El empleado en los experimentos de especificidad por doble híbrido corresponde a TMK1. Los números en los clados corresponden al valor de iteración que soportan cada clado.
DISCUSIÓN
El gen RXam1 codifica para una proteína tipo RLK, la cual recientemente ha demostrado conferir resistencia en yuca a la bacteriosis vascular (Diaz et al., 2018). En este trabajo fue posible identificar mediante doble híbrido una proteína de la familia E3 ubiquitina ligasa como un interactor del dominio STK de RXam1.
Las proteínas de tipo RLK son clásicamente receptores extracelulares capaces de reconocer moléculas de patógenos para activar una respuesta inmune (Tang et al., 2017). Este tipo de receptores interactúan con otro tipo de proteínas dentro de las cuales se encuentran RLCKs así como proteínas kinasas dependientes de calcio (Kadota et al., 2018). También se ha demostrado la interacción directa entre RLKs y RLCKs para activar proteínas del complejo NADPH oxidasa (Li et al., 2014) y con MAP kinasas (Yamada et al., 2016) permitiendo establecer un puente entre la percepción del patógeno y la activación de las respuestas inmunes. Se podría haber esperado encontrar que proteínas de tipo RLCKs interactuasen con el dominio STK de RXam1 como se ha reportado para varios PRRs (Li et al., 2014; Kadota et al., 2018). El tamizaje de un mayor número de clones de la librería de ADNc podría permitir encontrar este tipo de interacciones o con otras proteínas implicadas en la vía de señalización. La activación de una respuesta de resistencia debe ser estrictamente controlada por cuanto si ésta se mantiene en el tiempo conlleva a una expansión de la muerte celular y a un desbalance celular en detrimento de la planta. Se ha establecido que pocos minutos después de la activación de la inmunidad por PRRs, estos receptores transmembranales son blanco de proteínas E3 ligasas, las cuales interactúan físicamente y de manera directa con los PRRs (Craig et al., 2009). La E3 ligasas introducen ubiquitinas a los PRRs y de esta forma son etiquetados para que sean degradados por el proteosoma y así detener la activación de la inmunidad vegetal (Couto y Zipfel, 2016). En el caso particular de los PRRs se ha demostrado que estos interactúan con PUBs (Plant U-box type E3 ubiquitin ligases), una familia de E3 ligasas, para dirigirlos a la maquinaria del proteosoma (Trujillo et al., 2008). Así mismo se ha establecido que este sistema de regulación también se emplea para controlar las respuestas mediadas por receptores tipo NLR (Cheng et al., 2012; Trujillo, 2018).
El gen RXam1 de yuca inicialmente fue identificado a través de una aproximación gen candidato (Jorge et al., 2000). Para ello se emplearon primers diseñados a partir del gen Xa21 de arroz, el cual confiere resistencia a cepas de X. oryzae pv. oryzae (Song et al., 1995). Tanto RXam1 como Xa21 codifican para proteínas de tipo RLK (Song et al., 1995; Diaz et al., 2018). De manera interesante el dominio STK de Xa21 de arroz interactúa con una E3 ubiquitin ligasa denominada XB3, la cual regula su actividad (Wang et al., 2006). En el presente estudio se logró identificar que uno de los interactores del dominio STK de RXam1 es una proteína que posee un dominio dedos de zinc CHY central, seguido de un dominio "RING finger" y un "Zincribbon. Las proteínas con "RING finger" son un tipo común de Ubiquitin E3 ligasas y están implicadas en distintos procesos en plantas (Stone et al., 2005; Mazzucotelli et al., 2006). Este hecho sugiere que a lo largo de la evolución entre monocotiledoneas y dicotiledoneas se ha conservado un mecanismo de regulación de receptores tipo RLKs, implicando particularmente procesos de degradación mediados por el proteosoma.
La red de interactores de Xa21 ha sido establecida recientemente, así como la propia red de interactores de XB3 en arroz (Cantu et al., 2013). Este tipo de estudios de interactomica ha permitido identificar nuevos genes candidatos implicados en inmunidad, algunos de los cuales han sido validados experimentalmente. A través de nuevas estrategias a gran escala de búsqueda de interactores (Suter et al., 2015), será posible continuar con el mapeo de proteínas de yuca implicadas en la respuesta durante la infección con Xam.
Las proteínas de tipo RLK como RXam1, poseen un dominio STK el cual puede ser fácilmente empleado para identificar las proteínas que interactúan con él mediante la técnica de doble híbrido clásica como la que se empleó en este estudio. Sin embargo, resulta interesante determinar los interactores del dominio LRR extracelular, el cual en principio estaría detectando los determinantes claves del patógeno para activar la respuesta inmune. Para ello se hace necesario recurrir a otro tipo de aproximaciones o modificaciones del sistema de doble híbrido que apelan al sistema Ubiquitinsplit Y2H el cual es empleado para identificar interacciones empleando proteínas de membrana (Li et al., 2016).
La identificación de la proteína CRFE3-1 de yuca abre nuevas perspectivas de estudio. Será interesante determinar por ejemplo la actividad E3 ligasa de CRFE3-1, así como su patrón de expresión en tejidos y en el tiempo durante la infección. A través de estudios de edición genómica o de silenciamiento génico se podrá determinar la función de esta nueva proteína en la inmunidad de yuca.
CONCLUSIONES
El dominio STK de la proteína RXam1 interactúa con una proteína denominada CRFE3-1 la cual posee dominios propios de la familia de las E3 ligasas indicando que la degradación proteica es un mecanismo de regulación de RXam1 en yuca como se ha reportado para PRRs y NLRs en otras especies.















