INTRODUCCIÓN
Actualmente las microalgas son consideradas una buena alternativa para satisfacer la creciente demanda de biocombustibles. Esto se atribuye a su alto contenido y productividad de triglicéridos (Shrivastav et al., 2015). Asimismo, estos microorganismos fotosintéticos sintetizan diversas biomoléculas que pueden servir de materia prima para producir varios tipos de biocombustibles (Demirbas, 2010). Es decir, representan una fuente energética segura, sustentable, renovable y amigable con el ambiente (Garibay et al., 2009) que no ponen en riesgo la seguridad alimentaria (Amaro et al., 2011).
Diversos estudios han demostrado que el contenido de lípidos totales depende de las cepas de microalgas y de las condiciones de cultivo. De acuerdo con Chisti, (2007) el contenido de lípidos en microalgas oscila de 1 a 75 % de su peso seco, excepcionalmente algunas especies pueden acumular hasta el 90 % de su peso seco. Por otra parte, las microalgas acumulan lípidos, específicamente los triglicéridos (TG), cuando son cultivadas en condiciones estresantes debido a fluctuaciones extremas de los parámetros físicos (elevadas temperaturas, altas intensidades de iluminación, fotoperiodo, etc) y químicos (deficiencia de nutrientes, pH ácidos o alcalinos, medios hipersalinos, etc) que limitan el crecimiento microalgal (Anand y Arumugam, 2015; Kamalanathan et al., 2015; Cobos et al., 2017).
Aunque existen avances importantes sobre el aislamiento y caracterización de diversas especies y cepas de microalgas (Karnataka, 2011; Pan et al., 2011; Duong et al., 2012; Cobos et al., 2017) y han sido optimizadas las condiciones de cultivo para mejorar sus productividades de triglicéridos para muchos de estos aislados (Isleten-Hosoglu et al., 2012; El-Sheekh et al., 2013; Karpagam et al., 2015). Sin embargo, hasta la fecha los estudios de caracterización a nivel molecular de los procesos metabólicos y por ende de las enzimas que controlan la producción de los triglicéridos son inexistentes o sólo están limitados para algunas especies de microalgas modelo como Chlamydomonas reinhardtii (Riekhof et al., 2005; Fan et al., 2011; Gargouri et al., 2015).
Al respecto, investigaciones realizadas en plantas y microalgas sugieren que la biosíntesis de novo de ácidos grasos es controlada por la Acetil-CoA carboxilasa. Esta enzima cataliza la carboxilación de acetil-CoA para sintetizar malonil-CoA, la molécula precursora para la elongación enzimática de los ácidos grasos (Thelen y Ohlrogge, 2002). Varios reportes indican que en la naturaleza existen dos formas de Acetil-CoA carboxilasa, la homomérica y la heteromérica (Sasaki y Nagano, 2004; Huerlimann y Heimann, 2013). Estructuralmente, la enzima heteromérica está formada por cuatro subunidades polipeptídicas: la biotina carboxilasa (BC), la proteína portadora de biotina-carboxilo (BCCP) y dos carboxiltransferasas (alfa [αCT] y beta [βCT]). Las tres primeras cadenas polipeptídicas son codificadas por genes nucleares, mientras que la cuarta (βCT) es codificada por un gen cloroplástico (Cronan y Waldrop, 2002; Huerlimann y Heimann, 2013).
Por tanto, con el propósito de ampliar nuestros conocimientos a nivel molecular de la biosíntesis de lípidos en las microalgas Chlorella sp. y Scenedesmus sp., dos especies aisladas de la Amazonía peruana, en este estudio hemos realizado la caracterización in silico y analizado la expresión de la subunidad alfa de la acetil-Coenzima A carboxilasa heteromérica (αACCasa) de ambas especies. Entre las principales razones que hacen interesante investigar la αACCasa son: a) esta subunidad como componente del dominio carboxiltransferasa de la enzima participa en un mecanismo regulador inusual, porque regula su propia traducción al unirse a las regiones codantes de los ARNms que codifican sus subunidades, esta unión a su vez inhibe recíprocamente la actividad catalítica de transferencia de grupos carboxilo (Meades et al., 2010), b) la determinación de su estructura tridimensional in silico nos proporcionaría nuevos conocimientos para delinear las relaciones de estructura-función, particularmente los mecanismos moleculares que gobiernan la catálisis de la subunidad carboxiltransferasa y el mecanismo regulatorio inusual previamente indicado. También, la estructura 3D podrá ser comparada con estructuras proteicas similares de bacterianas (Bilder et al., 2006) a fin de poder resaltar las diferencias clave entre las isoenzimas bacterianas y microalgales. Asimismo, como la subunidad carboxiltransferasa bacteriana es un blanco para antibióticos (Wu y Seyedsayamdost, 2018; Zhang et al., 2018), se puede determinar si esos antibióticos también inhiben a la carboxiltransferasa microalgal y nos puede servir de guía para el diseño de pequeñas moléculas inhibitorias útiles para la selección de cepas microalgales modificadas genéticamente con fines biotecnológicos, c) la caracterización in silico de la proteína codificada nos proporcionará información de sus relaciones filogenéticas con otros grupos de organismos para comprender mejor los procesos evolutivos implicados y d) al conocer si el nivel de expresión del gen que codifica la αACCasa influye en la producción de triglicéridos en las dos especies de microalgas de interés, se puede realizar la manipulación genética para sobreexpresar este gen y los otros genes que codifican las demás subunidades de la acetil-Coenzima A carboxilasa heteromérica con una apropiada estequiometría y así dispondríamos de cepas microalgales hiperproductoras de triglicéridos potencialmente útiles para la producción industrial de biodiesel.
MATERIALES Y MÉTODOS
Identificación y caracterización in silico de la αACCasa
Se hizo una búsqueda Blast local con BlastStation 2 (TM Software, Arcadia, CA, USA) empleando la secuencia proteíca correspondiente de Chlamydomonas reinhardtii (XP_001696945.1) como un query para buscar en nuestra base de datos de los transcriptomas anotados de Chlorella sp. y Scenedesmus sp. y seleccionó las secuencias con E-valor < 1x10-5. Las secuencias nucleotídicas candidatas fueron extraídas de las bases de datos y corroboradas su identidad mediante análisis Blastp (Altschul et al., 1997) del GenBank (Benson et al., 2013).
Posteriormente, la herramienta ExPASy translate (Artimo et al., 2012) fue empleada para determinar las secuencias de las proteínas de αACCasa de ambas especies de microalgas. Plant-mPLoc (Chou y Shen, 2010) y TargetP (Emanuelsson et al., 2007) fueron utilizados para predecir la localización intracelular de las proteínas. Con el programa ProtParam (Artimo et al., 2012) se calculó el peso molecular, número de aminoácidos, el pI teórico, entre otros parámetros. También, con el servidor PredictProtein (Yachdav et al., 2014) se hicieron anotaciones estructurales (estructuras secundarias, accesibilidad al solvent, etc) y funcionales (términos de ontología de genes, sitios de unión, etc). Adicionamente, se han realizado predicciones de las estructuras tridimensionales de las proteínas con los servidores Phyre2 (Kelley et al., 2015) y SWISS-MODEL (Arnold et al., 2006). Las estructuras obtenidas fueron refinadas con el programa GalaxyRefine (Heo et al., 2013) y la calidad de las estructuras obtenidas fueron analizadas con RAMPAGE (Lovell et al., 2003) y ProSA-web (Wiederstein y Sippl, 2007). Finalmente, para analizar si existe conservación estructural de las aACCasa de ambas especies microalgales se hizo un alineamiento de las estructuras tridimensionales con el servidor TM-align (Zhang y Skolnick, 2005).
Análisis filogenético
Secuencias homólogas de las proteínas fueron investigadas con el algoritmo Blastp (Altschul et al., 1997) y descargadas del GenBank. Se hizo el alineamiento multiple de las secuencias con Clustal Omega (Sievers y Higgins, 2014), editados con el programa BioEdit (Hall, 1999) y los árboles filogenéticos fueron construídos con el programa MEGA 7.0 (Kumar et al., 2016) empleando el método Neighbor-Joining (Saitou y Nei, 1987) con distancias derivadas del modelo evolutivo Jones-Taylor-Thornton (Jones et al., 1992) y se hicieron 1000 réplicas bootstrap.
Cultivo de las cepas en medio CHU-10 con y sin nitrógeno
Todos los ensayos se hicieron como previamente lo describió Cobos et al., (2017) con las siguientes condiciones de cultivo: temperatura a 25 ± 1 °C con ciclos de 12:12-h luz-oscuridad, empleando lámparas de fluorescente de luz blanca de 100 μE·m-2·s-1 de intensidad y agitación continua a 150 rpm. La tasa de crecimiento específico de los cultivos microalgales se determinó de acuerdo con Cobos et al., (2017). Al cabo de 7 días de cultivo las microalgas fueron cosechadas por centrifugación a 4000 rpm por 10 min a 4 °C.
Análisis de la expresión de genes, productividad lipídica y contenido de lípidos totales
El ARN fue purificado como ha sido descrito previamente (Castro et al., 2017). A partir del ARN purificado se realizó la síntesis del ADN complementario siguiendo las instrucciones del Applied Biosystems™ GeneAmp™ RNA PCR Core Kit (ThermoFisher Scientific, Waltham, MA, USA), empleando Oligo dT(16) como cebador en un volumen final de 20 μl. El nivel de expresión de los genes fue determinado por PCR en Tiempo Real en base a tres réplicas biológicas y tres réplicas técnicas. Las reacciones de amplificación se realizaron en un termociclador Mastercycler ep Gradient realplex S (Eppendorf, NY, USA) empleando los cebadores específicos (Chl_α ACCasa_f:5'-GCGAGTACGAGCACATGAA-3', Chl_ α ACCasa_r:5'-GTTTGAGGAGTGGGTGGTC-3'; Sce_ αACCasa_f:5'-CAGGCACTACGAGAACATGAG-3', Sce_ α ACCasa_r:5'-GTTTGAGGAGTGGGTGGTG-3'; Chl_GFDH_ f:5'-GGCACCATGGTCAAGATCTAC-3', Chl_ GFDH_r:5'-AAGATGGTGGCGGACAAG-3'; Sce_GFDH_ f:5'-GTGGTGTCCACGGACTTT-3', Sce_GFDH_r:5'-TTTGTGAAGCTGGTCTCCTG-3'), y reactivos de la tecnología SYBR Green I. La eficiencia de las reacciones se determinó con el programa LinRegPCR (Ruijter et al., 2009) y la expresión relativa se estimó con el método comparativo del CT (Simon, 2003). Todos los datos de expresión se normalizaron con respecto al nivel de expresión del gen gliceraldehido-3-fosfato deshidrogenasa (GFDH), que en ensayos previos demostró mayor estabilidad de expresión con respecto a otros genes evaluados.
Los lípidos totales fueron extraídos con el método estándar de Bligh y Dyer, (1959). El contenido de lípidos totales se determinó gravimétricamente y tanto la productividad lipídica como el contenido de lípidos totales se calcularon según Yu et al., (2012).
Análisis estadísticos
Los cálculos para el test de normalidad de Shapiro-Wilk, prueba F de varianzas iguales, prueba t de Student, promedio, desviación estándar, ANOVA de una vía y la prueba HSD de Tukey del nivel de expresión relativa del gen αACCasa y el contenido de lípidos totales fueron realizados con el programa R version 3.4.4. Las diferencias con p < 0,05 fueron considerados estadísticamente significativos.
RESULTADOS
Identificación y caracterización in silico de la αACCasa
Mediante los análisis blast locales se ha identificado en nuestra base de datos de transcriptomas los genes que codifican la αACCasa de ambas especies de microalgas.
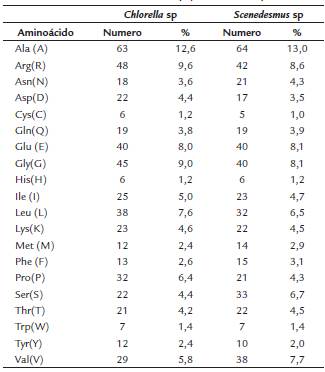
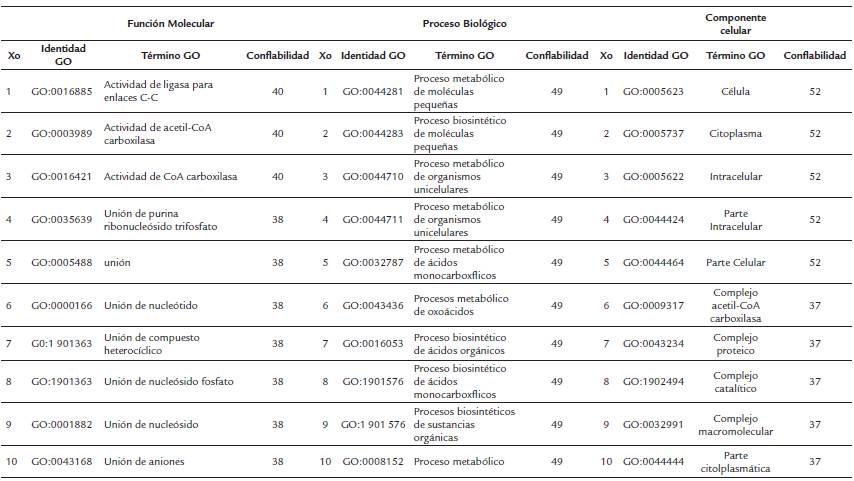
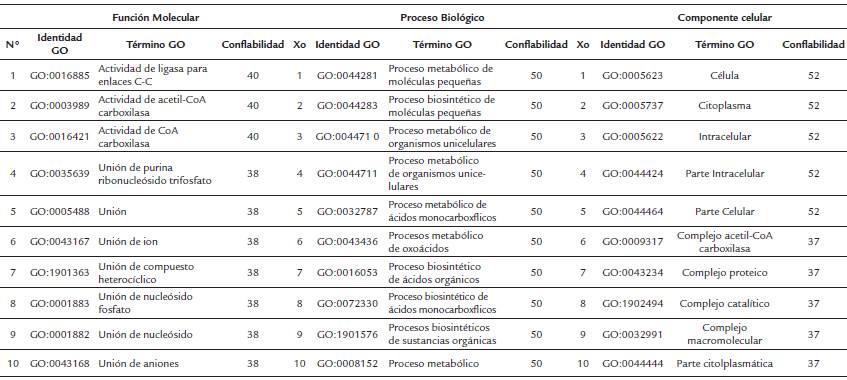
Los parámetros fisicoquímicos indican que las proteínas deducidas tienen pesos moleculares entre 53 y 55 kDa y muestran liegras diferencias con respecto a otros parámetros evaluados como el pI, resíduos con carga positiva, etc. (Tabla 1). Además, presentaron algunas diferencias en la composición de aminoácidos (Tabla S1). Los análisis blastp indican que ambas secuencias se tratan de la subunidad alfa de la acetil-CoA carboxilasa heteromérica, debido a que presentaron porcentajes de identifdad mayores al 60 % con proteínas de otras especies de microalgas y plantas depositadas en el GenBank (Tablas S2 y S3). Adicionalmente, los análisis de ontología genética corroboran que se trata de esta proteína (Tablas S4 y S5). Finalmente, los análisis bioinformáticos de localización intracelular indican que es una proteína que se localiza en los cloroplastos.
Tabla 1 Comparación de los parámetros fisicoquímicos de la secuencia de aminoácidos deducidas de las subunidades alfa de la Acetil-CoA carboxilasa heteromérica de las microalgas Chlorella sp. y Scenedesmus sp.
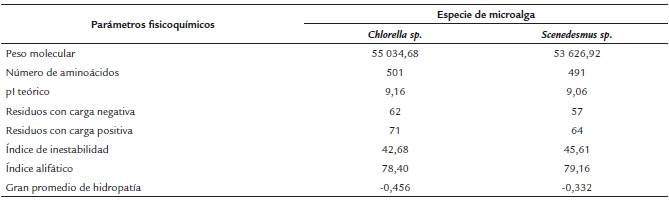
Tabla S2 Resultados del análisis Blastp para la secuencia aminoacídica de la αACCasa de Chlorella sp.
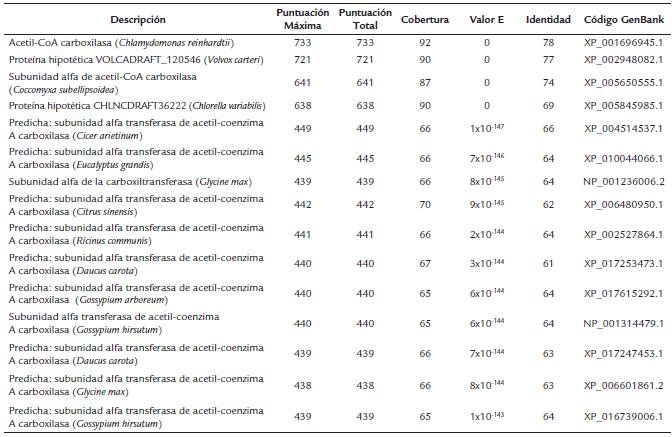
Tabla S3 Resultados del análisis Blastp para la secuencia aminoacídica de la αACCasa de Scenedesmus sp.
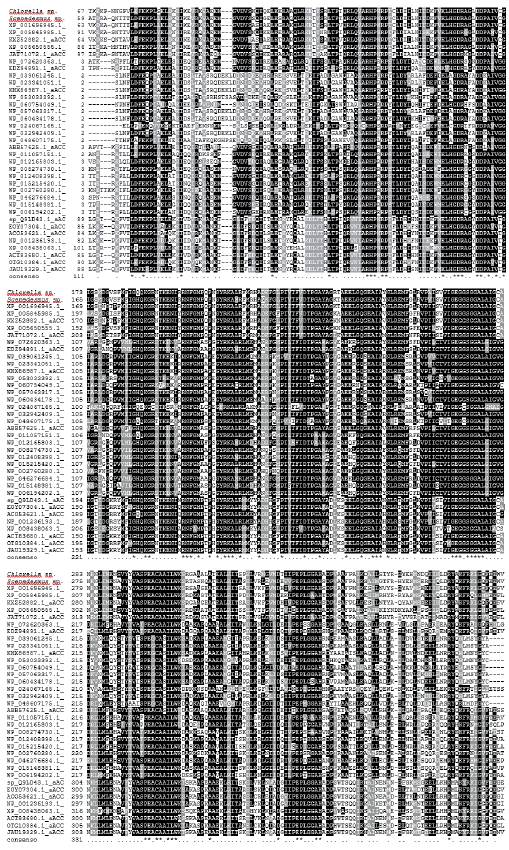
Por otra parte, con base a las secuencias deducidas de las proteínas se ha mostrado que se trata de proteínas mixtas, por la composición de estructuras secundarias que presentan, es decir, estuvieron constituídas por helices a, hojas β y bucles (Fig. 1, Fig. S1).
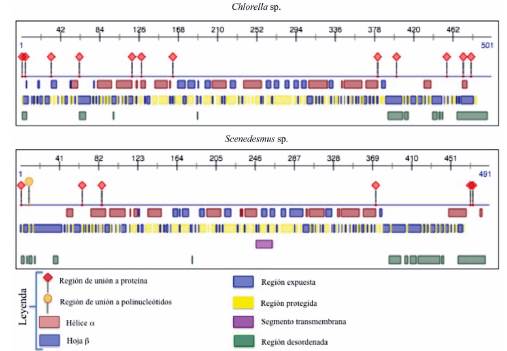
Fig. 1 Anotación estructural de las αACCasa de Chlorella sp. y Scenedesmus sp. donde se muestran las regiones que corresponden a las estructuras secundarias típicas (hélices a y hojas β) presentes en estas proteínas. Además, se presentan regiones implicadas en la interacción con proteínas, polinucleótidos y regiones que están embebidas en el núcleo hidrofóbico o si se encuentran en la superficie de las αACCasa de ambas especies microalgales.
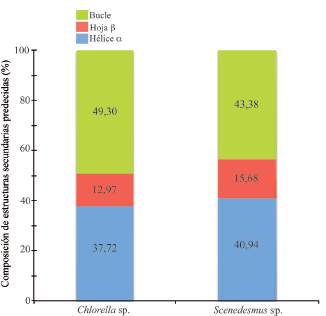
Fig. S1 Composición de estructuras secundarias predecidas en la proteína aACCasa de Chlorella sp. y Scenedesmus sp.
También con base a las secuencias protéicas deducidas de ambos genes se ha podido predecir las estructuras tridimensionales (Fig. 2) empleando plantillas de proteínas que tienen sus estructuras 3D determinadas por cristalografía de rayos X. Los modelos predecidos fueron refinados y analizados para verificar la calidad de los modelos obtenidos. Los resultados indican que estos modelos fueron de alta calidad porque en los plots de Ramachandran y las puntuaciones obtenidas con ProSA-web no mostraron aberraciones estructurales (Fig. 3).
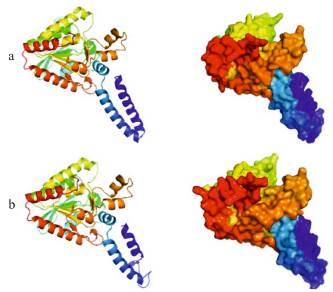
Fig. 2 Comparación de las estructuras tridimensionales de las proteínas αACCasa de Chlorella sp. (a) y Scenedesmus sp. (b) generadas por predicción con base a estructuras 3D de proteínas homólogas que perviamente han sido obtenidas por cristalografía de rayos X. Ambas proteínas presentan el mismo patron estructural corroborando su conservación funcional en la catálisis de la transferencia de grupos carboxilo.
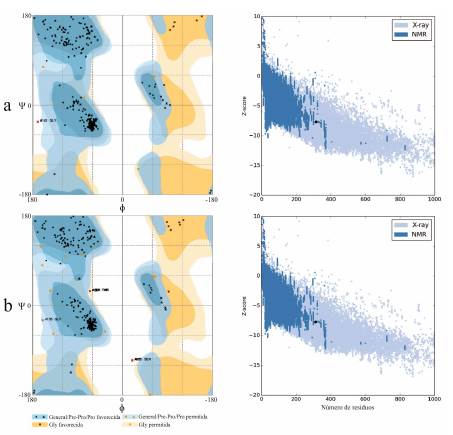
Fig. 3 Análisis de la calidad de las estructuras tridimensionales predecidas de las proteínas αACCasa de Chlorella sp. (a) y Scenedesmus sp. (b) con los plots de Ramachandran y ProSA-web. Los patrones de distribución de los enlaces peptídicos de los aminoácidos y el Z-score obtenidos por las αACCasa de ambas microalgas está de acuerdo con los patrones comunmente encontrados en las proteínas cuyas estructuras 3D han sido determinadas mediante cristalografía de rayos X y resonancia magnética nuclear.
Adicionalmente, se ha demostrado que ambas proteínas presentan el mismo plegamiento estructural (TM-Score= 0,98), lo que sugiere la gran conservación tridimensional de las mismas en estas especies de microalgas (Fig. S2).
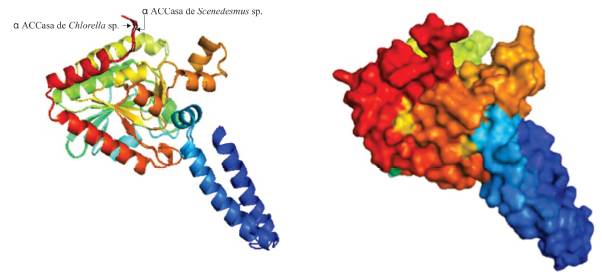
Fig. S2 Alineamiento estructural de las estructuras tridimensionales predecidas de las proteínas aACCasa de Chlorella sp. y Scenedesmus sp.
Análisis filogenético
Existen relaciones filogenéticas bien establecidas entre los difentes grupos de organismos evaluados a nivel de las secuencias aminoacídicas de la αACCasa, porque ambas secuencias forman un clado junto con otras especies de microalgas. Asimismo, el clado que agrupa a las plantas superiores tiene mayor similitud genética con las microalgas. En contraste, las cianobacterias y las bacterias forman clados que están más distanciados genéticamente de las microalgas (Fig. 4, Fig. S3).
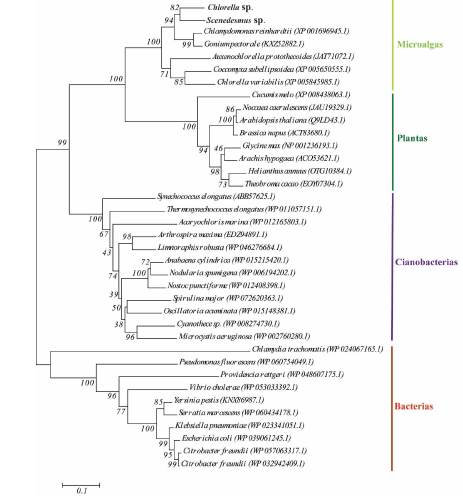
Fig. 4 Análisis filogenético de las αACCasa con base a las secuencias deducidas de las proteínas homólogas de microalgas, plantas, cianobacterias y bacterias. El árbol filogenético fue construido empleando el método Neighbor-Joining y las distancias de las ramas son las proporciones de las sustituciones de aminoácidos derivadas del modelo evolutivo Jones-Taylor-Thornton. Los valores de bootstrap corresponden a los porcentajes obtenidos a partir de 1000 réplicas.
Tasa de crecimiento, análisis de la expresión de genes y contenido de lípidos
Los resultados experimentales muestran que las tasas de crecimiento de las dos especies de microalgas difieren significativamente (t = 11,7; gl = 8; p < 0,001) al ser cultivadas en medio con nitrógeno (0,142 ± 0,008 d-1 vs 0,216 ± 0,011 d-1 para Chlorella sp. y Scenedesmus sp., respectivamente) y presentan tasas de crecimiento menores (~ 4 veces menos) y con diferencias estadísticas significativas (t = 16,1; gl = 8; p < 0,001) al ser cultivadas en medio sin nitrógeno (0,034 ± 0,002 d-1 vs 0,060 ± 0,002 d-1 para Chlorella sp. y Scenedesmus sp., respectivamente). Asimismo, es evidente que bajo condiciones de cultivo en ausencia de nitrógeno existe una significativa inducción en el nivel de expresión del gen que codifica las αACCasa tanto de Chlorella sp. como de Scenedesmus sp. Esta inducción de la expresión génica está relacionada con un aumento significativo (t = 11,4; gl = 8; p < 0,001) en las productividades lipídicas de ambas especies microalgales (3,35 ± 0,10 mg-L-1-d-1 vs 4,55 ± 0,23 mg-L-1-d-1 para Chlorella sp. y Scenedesmus sp., respectivamente) al ser cultivadas en medio sin nitrógeno. También, el aumento en la expresión de estos genes se relaciona con un incremento significativo en el contenido de lípidos totales en ambas especies de microalgas (Fig. 5), lo que sugiere que la actividad catalítica de la acetil-CoA carboxilasa heteromérica se ha incrementado.
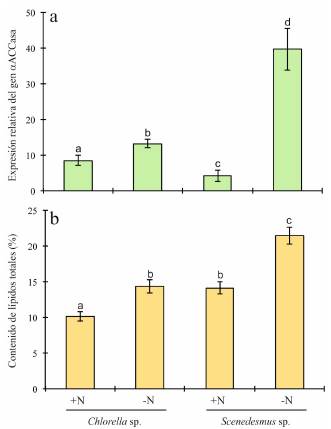
Fig. 5 Expresión relativa del gen αACCasa (a) y contenido de lípidos totales (b) en las microalgas Chlorella sp. y Scenedesmus sp. sometidas a estrés nutritivo por deficiencia de nitrógeno. Las columnas representan los promedios obtenidos de tres réplicas biológicas y tres réplicas técnicas. Las barras de error indican valores de desviación estándar y letras diferentes (p.ej., a, b, c, etc.) sobre las columnas significa que existen diferencias estadísticas significativas (p < 0,05)
DISCUSIÓN
Identificación y caracterización in silico de la αACCasa
Los resultados muestran que ambas αACCasa presentan patrones de estructuras secundarias que son típicos en este tipo de proteínas, tal como han sido reportados en otros estudios (Cronan y Waldrop, 2002; Huerlimann y Heimann, 2013; Salie y Thelen, 2016). Respecto a la estructura tridimensional predecida se evidencia que tiene una gran similitud estructural con las correspondientes subunidades de acetil-CoA carboxilasa heteromérica de Staphylococcus aureus y Escherichia coli, las que fueron previamente determinadas mediante cristalografía de rayos X a resoluciones de 2,0 y 3,0 A, respectivamente (Bilder et al., 2006). Además, cabe indicar que mediante análisis de filtración en gel, equilibrio de sedimentación y cristalografía de rayos X ha sido demostrado que las subunidades alfa y beta se ensamblan como heterotretámeros (α2β2) funcionales, sin que exista un cambio estructural en las estructuras tridimensionales de cada subunidad (Guchhait et al., 1974; Bilder et al., 2006). Por tanto, la predicción de la estructura tridimensional realizada es una aproximación apropiada para conocer la estructura de estas proteínas, que posteriormente deberán ser corroboradas experimentalmente.
Análisis filogenético
El análisis filogenético nos revela que entre microalgas existe una alta similitud genética a nivel de secuencias aminoacídicas de la αACCasa y diferentes niveles de distancia genética con respecto a las secuencias correspondientes de otros organismos analizados (plantas, cianobacterias y bacterias). De acuerdo con Huerlimann et al., (2015), el entendimiento de la filogenia de las microlagas se complica por el desconocimiento del número de eventos de transferencia genética horizontal y procesos endosimbióticos primarios, secundarios y terciarios. Adicionalmente, estos investigadores manifiestan que los plástidos de las microalgas que surgieron por eventos endosimbióticos primarios contienen las acetil-CoA carboxilasas heteroméricas, las que consisten en tres subunidades codificadas en el núcleo y una subunidad codificada en el cloroplasto. Este origen común de los cloroplastos microalgales y de las plantas superiores explicaría la gran similitud genética entre estos grupos de organismos (Fig. 4, Fig. S3). Aunque a nivel de secuencias aminoacídicas se evidencia distanciamiento genético de las microalgas con otros grupos de organismos analizados (Fig. 4), sin embargo, es preciso destacar que existe una alta conservación estructural y funcional de esta subunidad en los dominios de union (Huerlimann y Heimann, 2013), toda vez que por acoplamiento con la subunidad beta forman un sitio activo con actividad carboxiltransferasa que reconoce ésteres de coenzima A y por ende cataliza la semireacción que consiste en la transferencia del grupo carboxilo activado a la acetil-Coenzima A para producir malonil-Coenzima A (Bilder et al., 2006; Tong, 2013).
Tasa de crecimiento, análisis de la expresión de genes y contenido de lípidos totales
Los resultados muestran que al cultivar ambas especies de microalgas en medio deficiente de nitrógeno disminuyen significativamente sus tasas de crecimiento. Este comportamiento ha sido evidenciado en estudios similares con varias especies de microalgas (Lin y Lin, 2011; Do Nascimento et al., 2012; Cobos et al., 2017). De acuerdo con algunas investigaciones la disminución en la tasa de crecimiento de las microalgas en cultivos deficientes de nitrógeno se debe a una detención del ciclo celular en su etapa inicial, particularmente en la Fase G1 (Olson y Chisholm, 1986; Vaulot et al., 1987). Además, recientemente ha sido demostrado que la disminución en la tasa de crecimiento de las microalgas se asocia con un descenso en los niveles de expresión de genes que regulan el crecimiento de las células y el ciclo celular (Li et al., 2012). Por otra parte los resultados indican que existe correlación positiva entre el nivel de expresión de los genes analizados y la productividad y la acumulación de lípidos totales bajo deficiencia de nitrógeno. Similares resultados han sido reportados en otras especies de microalgas, que muestran sobreexpresión de los genes involucrados en el metabolismo lipídico cuando son sometidos a deficiencia de nitrógeno (Guarnieri et al., 2011; Li et al., 2012; Rismani-Yazdi et al., 2012). También, varios estudios han demostrado que tanto en plantas como en microalgas existe una correlación positiva entre los niveles de expresión de las cuatro subunidades de la acetil-CoA carboxilasa heteromérica y la acumulación de lípidos (Fan et al., 2014; Cui et al., 2017; Kumar et al., 2017).
Adicionalmente, es preciso señalar que el cultivo de las microalgas en un medio deficiente de nitrógeno causa cambios significativos en el contenido de sus principales biomoleculas, es decir, además de incrementarse el contenido lipídico, también hay aumento de los carbohidratos, pero una disminución marcada de las proteínas (Do Nascimento et al., 2012; Singh et al., 2015; Cobos et al., 2017). Esto sugiere que bajo estas condiciones de cultivo las microalgas redireccionan su metabolismo hacia la biosíntesis de ácidos grasos y triglicéridos, procesos que están determinados por una sobre expresión de los genes involucrados en la glucólisis, degradación de almidón, biosíntesis de ácidos grasos y de triglicéridos. Simultáneamente, existe una disminución significativa en la expresión de genes que participan en la gluconeogénesis, síntesis de almidón, degradación de triglicéridos y /?-oxidación de los ácidos grasos (Lim et al., 2017; Wang et al., 2017; Sirikhachornkit et al., 2018).
En contraste a nuestros hallazgos, Li et al., (2014) revelaron que la microalga oleaginosa Nannochloropsis oceanica al ser sometida a condiciones de estrés por deficiencia de nitrógeno disminuye el nivel de expresión de los genes de la acetil-CoA carboxilasa heteromérica. Asimismo, Huerlimann et al., (2014) en base a experimentos realizados con Chromera velia e Isochrysis aff. galbana, concluyen que los niveles de expresión de la Acetil-CoA carboxilasa homomérica y heteromérica cambian con la fase de crecimiento de las microalgas y el contenido de nutrientes de una manera especie-específica y que la limitación del nitrógeno en los medios de cultivo no siempre induce la acumulación de lípidos.
Consecuentemente, aún existen grandes vacíos para comprender por completo varios procesos claves, tales como los componentes moleculares de las vías de señalización intracelular por el cual el estrés nutritivo estimula la expresión de genes involucrados directa e indirectamente con la acumulación de lípidos, la magnitud y el tiempo de la deficiencia de nutrientes requerida para la inducción y las interacciones entre crecimiento celular y contenido de lípidos (Goncalves et al., 2016). Por tanto, es necesario realizar más investigaciones integrando diversas aproximaciones tales como análisis transcriptómicos, proteómicos y metabolómicos para comprender la interdependencia y regulación de las vías anabólicas y catabólicas de lípidos y de otros compuestos (p.ej., carbohidratos y proteínas) bajo condiciones de estrés nutritivo, que nos permita tener el conocimiento básico suficiente para desarrollar nuevas cepas microalgales con capacidades mejoradas para canalizar los metabolitos hacia precursores para la producción de biocombustibles (Msanne et al., 2012).
CONCLUSIONES
El análisis in silico de la αACCasa de Chlorella sp. y Scenedesmus sp. presentan características estructurales, funcionales y evolutivas muy similares al de otras especies de microalgas y plantas. Asimismo, el estudio revela que en ambas especies el gen se sobreexpresa cuando las microalgas son sometidas a estrés por deficiencia de nitrógeno, el cual se relaciona significativamente con la acumulación de lípidos totales en estas células.