El Potato virus Y (PVY) (Potyvirus, Potyviridae) es uno de los virus más limitantes para la producción de papa en todo el mundo al causar reducciones del 40-70 % en los rendimientos de los cultivos (Nolte et al., 2004; Kerlan, 2008). Algunas de sus variantes (ej. PVYNTN) pueden causar la enfermedad de los anillos necróticos de los tubérculos (PNTRD), afectando su calidad comercial y su utilidad como material de siembra (Halterman et al., 2012; Karasev y Gray, 2013). La infección de PVY en papa y en otros hospedantes de la familia Solanaceae (ej. tomate, lulo, pimentón) se manifiesta por una diversidad de síntomas que incluyen mosaicos, rugosidad foliar, necrosis de nervaduras y PTNRD (Halterman et al., 2012; Karasev y Gray, 2013). El PVY es transmitido por diferentes especies de áfidos, por herramientas de trabajo y por material de siembra vegetativo (Fageria et al., 2013). Hasta el momento se han encontrado al menos nueve variantes de PVY que difieren en sus características biológicas, serológicas y moleculares, pero que pueden ser dividas en dos serotipos: Ordinario (O) y Necrótico (N); el primero incluye las cepas PVYO, PVYN:O, PVYN-Wi y PVYC; y el segundo a PVYN, PVYNTN, PVYE, PVYZ, PVYNA-N y PVYNE11 (Karasev y Gray, 2013; Treder et al., 2018). La identificación del PVY tanto en tejido foliar como en brotes de tubérculos se realiza mediante pruebas de ELISA (Fageria et al., 2013; Stammler et al., 2018), así como con pruebas moleculares como RT-PCR convencional (Singh et al., 2000; 2013; Lorenzen et al., 2006); RT-qPCR (Agindotan et al., 2007; Hühnlein et al., 2013; Singh et al., 2013) y LAMP (Treder et al., 2018). En Colombia, se han identificado en cultivos de papa común (Solanum tuberosum) y de papa criolla (S. phureja), las cepas PVYN, PVYNTN y PVY0 (Gil et al., 2011; Villamil et al., 2014; Muñoz et al., 2016 a, b; Riascos et al., 2018), y adicionalmente dos variantes divergentes (I-Col y IV-Col) encontradas en papa y tomate de árbol (Henao et al., 2013). Recientemente, en un estudio tendiente a identificar con pruebas de RT-PCR convencional y secuenciación los potyvirus que afectan los cultivos de papa común y criolla en el oriente de Antioquia (Colombia), no se encontró la presencia del Potato virus A (PVA), pero sí de PVY y del Potato virus V (PVV); aunque sorpresivamente el primero sólo se detectó en S. tuberosum y el segundo en S. phureja (Riascos et al., 2018). Con el fin de corroborar dicha situación, en el presente estudio se evaluó la infección de estos virus en diez muestras de tejido foliar de papa común cv. Diacol-Capiro y diez muestras de papa criolla cv. Criolla-Colombia obtenidas en cultivos del municipio de Marinilla (Antioquia), pero en este caso utilizando pruebas de ELISA y de RT-qPCR. Para la detección serológica de PVY se utilizaron pruebas de TAS-ELISA (SRA 20001), mientras que para PVV se empleó el formato de DAS-ELISA (SRA 10100) (Agdia, EEUU). Para la prueba de RT-qPCR, se procedió a la extracción del ARN total con el kit GeneJET Gel Extraction (Thermo Fisher Scientific, EEUU) y posteriormente la síntesis de cDNA se realizó en un volumen de 20 μL con 200 U de la retro-transcriptasa (Maxima RT), 1X de buffer RT, 0,5 mM de los cuatro dNTPs, 100 pmol del cebador Oligo-(dT)18 y 20 U de inhibidor de ARNAsas (Thermo Fisher Scientific, Waltham, MA). Las reacciones se incubaron a 65 °C por 5 min, seguidos de 50 °C por 30 min y finalmente a 85 °C por 5 min en un equipo T3 thermal cycler (Biometra, Germany) (Muñoz et al., 2016b). Las reacciones de qPCR se realizaron con el kit Maxima SYBR Green/ROX Master Mix (2X) (Thermo Fisher Scientific, EEUU), con los cebadores PVY-1 FP (5'CCAATCGTTGAGAATGCAAAAC3') y PVY-1 RP (5'ATATACGCTTCTGCAACATCTGAGA3') diseñados por Singh et al., (2013) para amplificar 74 pb de la cápside viral (CP) de PVY y con los cebadores PVV_phu_F (5'ATG CTGGAAAAGATCCAGC3') y qPVV_phu_R (5'CATCCCGC TCCTCAAC3') diseñados por Alvarez et al., (2016) para amplificar un fragmento de 89 pb de CP. Los resultados de las pruebas de ELISA detectaron al PVY en seis de las muestras de papa común cv. Diacol-Capira, pero no en ninguna de las muestras de papa criolla; mientras que PVV se detectó en cuatro de las muestras de papa criolla y en ninguna de papa común. Estos resultados, además de llamar la atención de los altos niveles de infección de las muestras bajo análisis, apoyaban los hallazgos antes mencionados de Riascos et al., (2018). Sin embargo, al realizar las pruebas de RT-qPCR, el PVY se encontró en ocho de las diez muestras de papa criolla y en la totalidad de las muestras de papa común; y dicho resultado se mantuvo prácticamente invariable aún después de repetir las reacciones con aumentos sucesivos de temperatura de anillamiento desde 52 °C hasta 62 °C. Los valores de ciclo umbral (CT) y de temperatura de desnaturalización (Tm) para las muestras positivas se ubicaron en el rango de 29,41 - 35 y 77,3 - 78 °C, respectivamente, confirmándose los valores previos de Tm (77,5 ± 0,5 °C) encontrados por Medina et al., (2015). Para el caso de PVV, cinco de las muestras de papa criolla resultaron positivas para este virus, y ninguna de las muestras de papa común. Los valores de Ct encontrados se presentaron en el rango de 16,41 a 21,11 y los de Tm entre 79,5 °C y 80,5 °C, lo que concuerda con el reporte original de Alvarez et al., (2016) para estos cebadores (Tm=78,7 °C -80,2 °C). La clara discrepancia entre los resultados encontrados para PVY con las pruebas de ELISA y RT-qPCR, nos condujeron a evaluar in silico la especificidad de los cebadores PVY-1 FP/RP utilizando como base de comparación los genomas de aislamientos colombianos de PVV y PVY recientemente publicados (Alvarez et al., 2016; Gutiérrez et al., 2016; Muñoz et al., 2016a, b), además de otros genomas de referencia disponibles en GenBank. En dichos análisis, efectivamente se encontró una región de CP de PVV compatible con la amplificación por estos cebadores de PVY y por tanto se decidió diseñar un nuevo par de cebadores para la detección de PVY sin reactividad cruzada con PVV (Figura 1). De esta forma, se repitió el experimento de detección bajo las mismas condiciones antes descritas, pero en este caso con los cebadores PVY_Col_F (5'CTATCACGTCCAAAATGAGAATGC3') y PVY_Col_qR (5'TCAATTTGYTGTGGAGCATACTC3') que amplifican 92 pb de CP. Adicionalmente, se diseñó el cebador PVY_Col_R (5'GCATACTTGGAGARACATCCTCG3') para su utilización con PVY_Col_F en reacciones de RT-PCR convencional para amplificar una región de 630 pb de CP. Estos cebadores se evaluaron en cuatro muestras de papa y una de lulo (S. quitoense) (control positivo), siguiendo las condiciones descritas por Riascos et al., (2018). Los resultados con los nuevos cebadores diseñados para RT-qPCR indicaron la presencia de PVY en una de las diez muestras de papa criolla evaluadas (Ct = 23,64; Tm = 76,5 °C), mientras que en papa común el virus se detectó en ocho de las muestras con valores de Ct en el rango de 8,56 a 15,66 y de Tm= 77-77,8 °C. Para el caso de las reacciones de RT-PCR convencional, el uso de los nuevos cebadores permitió obtener los amplicones del tamaño esperado en las cinco muestras evaluadas para este fin (Figura 1), confirmándose su naturaleza viral por secuenciación Sanger, al presentar 99 % de identidad (cobertura = 100%; valor e = 0,0) con secuencias de la región CP de cepas de PVYNTN previamente depositadas en GenBank (ej. MF176820, MF176827, MF176821) por Riascos et al., (2018). Los hallazgos de este trabajo sobre la reactividad cruzada de los cebadores PVY-1 FP/RP con el genoma de PVV, hacen necesario reevaluar su uso para la detección de PVY en sistemas de RT-qPCR con SYBR-Green; aunque es probable que su utilización con la sonda PVY-1 (5'-FAM TTA GGC AAA TCA TGG CAC AT MGB-NFQ-3') que originalmente reportaron Singh et al., (2013) ofrezca la especificidad requerida para PVY. El diseño de los cebadores PVY_Col amplía las herramientas disponibles para la detección molecular de este virus (Lorenzen et al., 2006; Agindotan et al., 2007; Hühnlein et al., 2013; Treder et al., 2018), no sólo en Colombia sino en otras partes del mundo donde se presenta la coinfección de varios potyvirus en los cultivos de papa y muy especialmente en países como Perú (Spetz et al., 2003), Estados Unidos (Shiel et al., 2004), Irán (Shamsadden-Saeed et al., 2014) Holanda, Reino Unido y Noruega (Oruetxebarria et al., 2000) donde PVV y PVY se han registrado afectando diferentes variedades de S. tuberosum. Se espera que los cebadores PVY_Col en conjunto con los PVV_phu_F/R diseñados por Alvarez et al., (2016) para PVV, sean utilizados en programas de certificación de tubérculo-semilla, estudios epidemiológicos y manejo fitosanitario de estos potyvirus en cultivos de papa y de otras solanáceas susceptibles a su infección.
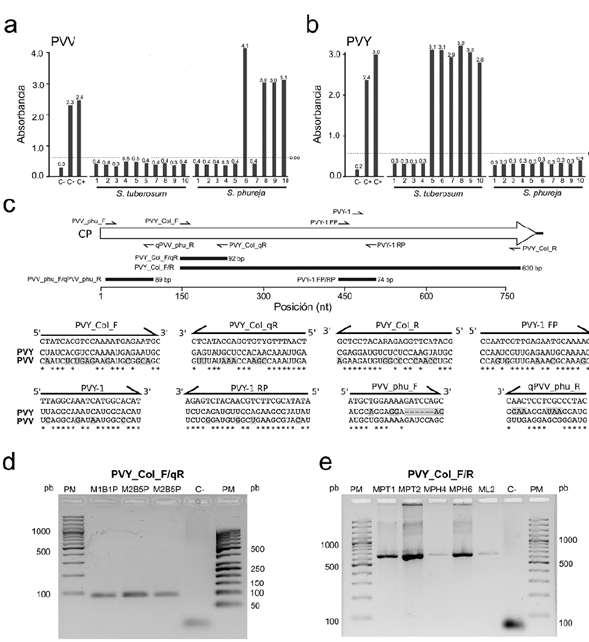
Figura 1. a y b) Detección por pruebas de ELISA de los virus PVV y PVY en diez muestras de papa común cv. Diacol-Capiro y de papa criolla cv. Criolla-Colombia del Oriente de Antioquia. Las muestras positivas son aquellas en las que las barras sobrepasan la línea punteada. Esta línea indica el punto de corte de valores de absorbancia obtenidos en el revelado de la prueba. c) Secuencias y ubicación de los cebadores diseñados en este trabajo (PVY_Col) y en otros reportes para la detección de PVY y PVV. Los nucleótidos sombreados en los genomas de PVY y/o PVV corresponden a los sitios no complementarios para cada cebador. d y e) Corridos electroforéticos de los amplicones obtenidos a partir de reacciones de RT-qPCR y RT-PCR convencional con los cebadores diseñados en este trabajo. PM: Marcador de peso molecular de 100 pb y/o 50 pb. C-: Control negativo.















