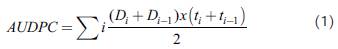INTRODUCCIÓN
La yuca (Manihot esculenta Crantz) es un arbusto perenne perteneciente a la familia Euphorbriaceae. Morfológicamente se caracteriza por presentar un sistema radical que comprende aproximadamente el 85 % del peso total de la planta, con una prominente capacidad de almacenamiento de almidón y eficiencia en la producción de carbohidratos (Ceballos y de la Cruz, 2012). La yuca es un alimento digestivo rico en carbohidratos (85 % del total de compuestos orgánicos que produce), proteínas (18-22 % del peso seco), vitaminas (carotenos y vitamina C) y minerales (calcio, hierro, magnesio y potasio) (García et al., 2014). Entre 800 y 1000 millones de personas en el mundo dependen del consumo de yuca como fuente de calorías (García et al., 2014). Actualmente se estima que 70 millones de personas en el mundo dependen de esta planta como fuente primaria de alimento, aportando en promedio 43 kilocalorías por persona al día (kcal/p/d) (FAO, 2017). El almidón de yuca también tiene una amplia utilización en diferente tipo de industrias (Ceballos y de la Cruz, 2012). Las plantas de yuca toleran largos periodos de sequía debido a su capacidad de almacenar cantidades considerables de CO2 respiratorio en presencia de luz (El-Sharkawy, 1990).
Existen factores limitantes en la productividad del cultivo, uno de los cuales es la bacteriosis vascular, enfermedad causada por la bacteria Gram negativa Xanthomonas axonopodis pv. manihotis (Xam) (Lozano y Sequeira, 1974). La bacteria presenta un hábito epífito, foliar y vascular (Verdier et al., 2012). El proceso infeccioso comienza con la multiplicación epífita en la hoja y la penetración a través de estomas y heridas. Una vez al interior pasa por una fase de desarrollo intercelular en el mesófilo de la hoja y posteriormente invade el xilema, tornándose la infección sistémica (Verdier et al., 2012). La invasión afecta los tejidos vasculares y foliares, genera daños como exudado y necrosis vascular y a nivel foliar, manchas angulares, añublo (quemazón) y muerte progresiva (Lozano, 1986). Aunque se han implementado medidas de control del patógeno, éstas han mostrado ser ineficientes en muchos de los casos por falta de identificación de fuentes de resistencia, alto nivel de diversidad del patógeno y desconocimiento de estrategias para controlar la enfermedad (López y Bernal, 2012).
Recientemente se identificó el gen RXAM1, el cual colocaliza con un QTL (Quantitative Trait Loci) que explica el 13 % de la resistencia a la cepa de XamCIO136 (Jorge et al., 2000; Díaz et al., 2018). El gen RXAM1 codifica para una proteína que posee un dominio LRR (Leucine Rich Repeats) extracelular y un dominio STK (Serine/treonine kinase) intracitoplasmatico. Este tipo de proteínas son conocidas como RLKs (Receptor Like Kinases). Dentro de la familia de RLKs se encuentran las proteínas PRRs (Pattern Recognition Receptors), las cuales reconocen PAMPs (Pathogen Associated Molecular Patterns) y activan una inmunidad que es efectiva contra patógenos no adaptados conocida como PTI (PAMP Triggered Immunity) (Boutrot y Zipfel, 2017). Las plantas de yuca que sobre expresan el gen RXAM1 permiten un menor crecimiento de la cepa XamCIO136 (Diaz et al., 2018). De esta manera, diferentes proteínas dentro de la familia RLKs parecen estar implicadas tanto en la activación de la resistencia de tipo cuantitativo (Vásquez et al., 2018) como en la resistencia no hospedero o PTI.
En este trabajo se determinó por un lado el nivel de diversidad alélica del gen RXAM1 en una colección de variedades de yuca, y por otro la respuesta de estas variedades a la infección con XamCIO136. A partir de esta información se buscó establecer una asociación entre las variantes alélicas y la respuesta a la infección.
MATERIALES Y MÉTODOS
Material vegetal e inoculación
Las variedades de yuca MCOL1505, MCOL1522, MCOL 2215, MBRA902, CM523-7, CM6438-14, MBRA685, TMS60444, SG10735 y CM2177-2 fueron obtenidas a partir de la colección de germoplasma del CIAT. Las estacas se sembraron en materas plásticas bajo condiciones de invernadero (28 °C, fotoperíodo de 12 horas) en tierra. Para los experimentos se emplearon plantas de aproximadamente 45 días de sembradas.
La cepa de Xam empleada para las inoculaciones fue XamCIO136. La bacteria se activó en medio LPGA sólido (0,5 % extracto de levadura, 0,5 % peptona, 0,5 % glucosa y 1,5 % agar) durante 48 horas a 28 °C proveniente de un stock de la cepa a - 80 °C. Transcurrido este tiempo, la bacteria se incubó en medio LPG líquido (0,5 % extracto de levadura, 0,5 % peptona y 0,5 % glucosa) por 16 horas. Las bacterias fueron recuperadas por centrifugación y calibradas a una densidad óptica de OD600nm= 0,002 (1*106UFC/ml) con una solución 10 mM de MgCl2. Con un palillo estéril se realizó una punción en el tallo hacia la parte apical de la planta, a una distancia de 3 cm debajo del ápice. Una vez realizada la herida se introdujo una punta con 5 µl de la suspensión bacteriana, la cual se dejó por 24-48 horas hasta que la bacteria entrará totalmente al tejido de la planta. Se inocularon diez plantas por variedad y se evaluaron los síntomas de enfermedad a los siete, 14, 21 y 28 días después de inoculadas (DDI) y el registro de los síntomas se llevó a cabo como ha sido previamente reportado (Trujillo et al., 2014), siguiendo la siguiente escala de síntomas de cero a cinco, así: cero: plantas sin síntomas, uno: necrosis alrededor del punto de inoculación, dos: exudado alrededor del punto de inoculación, tres: necrosis, exudado, marchitamiento de una o dos hojas, cuatro: necrosis, exudado y marchitamiento de tres o más hojas, cinco: muerte celular de la planta. A partir de los síntomas registrados para diez individuos (diez réplicas) de cada variedad, se calculó el área bajo la curva del progreso de la enfermedad AUDPC (Area Under Disease Progression Curve) de acuerdo con Shaner y Finney (1977):
Donde D corresponde al síntoma y t al tiempo postinoculación.
Para el análisis de los datos de AUDPC, se realizó una prueba de homogeneidad y normalidad de los datos registrados mediante la prueba de Kolmogorov-Smirnov y un análisis de varianza de comparación múltiple de medias aplicando los tests de Kruskal-Wallis (H) y de Games-Howell, con un intervalo de confianza de 95 % (p ≤ 0,05), mediante el programa SPSS 19®.
Extracción de ADN, amplificación del gen RXAM1, clonación y secuenciación
La extracción de ADN de las diez variedades de yuca se realizó a partir de 150 mg de tejido foliar almacenado en nitrógeno líquido basado en el protocolo de Dellaporta et al. (1983). A partir de la secuencia del gen RXAM1 se diseñaron los primers RX1FSmaI 5'-GCAGGATCCTCTCTGAATCATTACCTGCTGTC-3' y RX1RBamH1 5'- GCACCCGGGATGGGGTGTGGATGCTTC-3'. Los primers permitieron amplificar 3182pb desde el inicio de la transcripción del gen, sin incluir la región promotora. La reacción de PCR se preparó en un volumen final de 10 µl, con una concentración de 1X de Buffer de PCR (DreamTaq Buffer, Fermentas), 0,2 mM de dNTPs, 0,1 µM de cada primer, 20 mM de MgCl2 y 0,05 U de Taq polimerasa (DreamTaq, Fermentas) y utilizando 100 ng de ADN molde.
Las reacciones de PCR se efectuaron en un termociclador MyCyclerTM BioRad® utilizando los siguientes ciclos de temperatura: denaturación inicial a 95 °C por 2 minutos, seguido de 30 ciclos de amplificación con 30 segundos de apertura a 95 °C, 30 segundos de anillamiento a 54 °C y 3 minutos y 30 segundos de extensión a 72 °C, con un ciclo final de extensión a 72 °C por 5 minutos y tiempo de enfriamiento a 20 °C por 5 minutos. El producto de amplificación se verificó por electroforesis en gel de agarosa al 1 % teñido con bromuro de etidio en buffer TAE 0,5X. El tamaño del fragmento amplificado se confirmó empleando el marcador de peso molecular 1Kb Plus Ladder ® (Invitrogen, Carlsbad, CA, U.S.A.). El producto de PCR se purificó mediante el kit Wizard SV Gel and PCR Cleanup System (Promega, Madison, WI, USA). Cada amplicón eluído se clonó en el vector pGEM-T-easy (Promega, Madison, WI, USA) de acuerdo con las instrucciones de la casa comercial. El producto de la ligación se transformó en células competentes de Escherichia coli DH10B mediante electroporación empleando el equipo MicroPulserTM BioRad®, siguiendo las condiciones de voltaje, resistencia y capacitancia recomendadas por la casa comercial. Las células se incubaron una hora a 37 °C en 1 ml de medio 2XYT (triptona 16 g, extracto de levadura 10 g, cloruro de sodio 5 g) con agitación constante. Las bacterias se sembraron en medio LB sólido (extracto de levadura 0,5 %, Triptona 1 %, NaCl 0,5 % y agar 1,5 %) con selección para ampicilina a 100 µg/µl -IPTG (0,5 mM X-Gal (50 mg/µl) y se incubaron a 37 °C por 12 h. Se confirmó la presencia de los clones positivos elegidos al azar mediante PCR de colonia. A partir de los clones positivos se realizó el aislamiento del ADN plasmídico empleando el kit GeneJET Plasmid Miniprep (Fermentas). El inserto fue secuenciado en el centro de servicios certificado MACROGEN, utilizando varios primers que abarcaban la región completa de RXAM1 (Fig. 1 y Tabla 1). Posteriormente, se realizaron los alineamientos usando el software Sequencher y ClustalW teniendo en cuenta la secuencia de referencia del gen previamente obtenida por Diaz et al. (2018). A partir de los alineamientos, se revisaron manualmente los cromatogramas con el fin de comprobar su calidad y revisar la posición correcta de cada polimorfismo. Con el fin de establecer posibles efectos de selección sobre el gen RXAM1, se determinó si los polimorfismos encontrados en los dominios LRR y STK son sinónimos o no-sinónimos y se calcularon sus tasas de sustitución (dN/dS) empleando el programa DnaSP (Librado y Rozas, 2009).
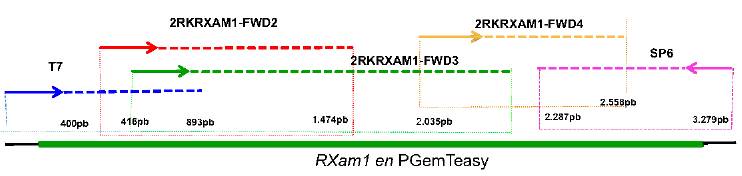
Figura 1 Posición de los primers en el gen RXAM1 empleados para la secuenciación. Se muestra el esquema del gen RXAM1 clonado en el vector pGEMT-easy y los primers indicados con su posición y el tamaño promedio de las lecturas de secuenciación obtenidas para cada caso.
Asociación polimorfismos y fenotipo
Se construyó una matriz de polimorfismos y para identificar asociación con el fenotipo (resistente/ susceptible) de las variedades evaluadas. Posteriormente se determinaron el alelo común y el alelo raro. El genotipo de las variedades se codificó como homocigotas o heterocigotas, asignándoles un valor de cero, uno o dos, donde cero es el genotipo homocigoto común, uno el genotipo heterocigoto y dos el genotipo homocigoto raro (poco frecuente). Para catalogar como R o S se consideró el criterio previamente reportado por Trujillo et al. (2014), en el cual una variedad es considerada como resistente si el valor de logAUDPC es inferior a 1,59 y susceptible si es mayor de este valor. Finalmente, se realizó una prueba de chi-cuadrado(X2) asignándole un p-valor correspondiente mediante el programa estadístico R® y para evitar errores Tipo I (probabilidad de rechazar la hipótesis nula cuando es cierta), se realizó una corrección de Bonferroni (α* = α /n), donde α = 0,05. Los análisis se llevaron a cabo en el paquete estadístico R®.
RESULTADOS
Evaluación fenotípica de diez variedades de yuca
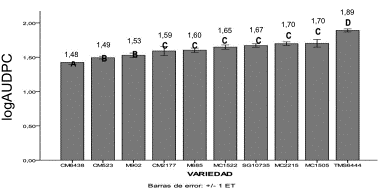
Con el objetivo de evaluar la respuesta de las diez variedades de yuca a la inoculación con la cepa XamCIO136 se llevó a cabo un seguimiento de la respuesta durante 28 días con evaluaciones periódicas cada siete días. A los primeros siete DDI, se observó que pese a ser una fase temprana para la manifestación de síntomas, el 40 % de las variedades mostraron una leve necrosis y exudado de color amarillo claro en el punto de inoculación. Para el segundo tiempo de evaluación, aquellas variedades que inicialmente no habían presentado signos de la enfermedad (por ejemplo, CM523-7 y CM6438-14), comenzaron a presentar algunos síntomas tales como exudado de color amarillo claro y necrosis en el punto de inoculación. Por otro lado, las variedades que mostraron síntomas tempranos de la enfermedad en el registro anterior presentaron un aumento en el nivel de infección evidenciado por doblez y decoloración en los ápices foliares, aumento del área necrosada, cambio de tonalidad del exudado de amarillo claro a marrón y un leve desplazamiento del mismo hacia la parte superior del tallo. Para la evaluación a los 21 DDI se encontró un comportamiento progresivo de la enfermedad para todas las variedades evaluadas, manifestando marchitamiento foliar de dos a tres hojas, perdida de turgencia del tallo, expansión del exudado y prolongación del área necrosada en las variedades más susceptibles (TMS60444, SG10735, MCOL1505, MCOL1522 y MCOL2215). Finalmente, a los 28 DDI se encontró que las variedades SG10735, MCOL1505, MCOL2215 y MCOL1522 alcanzaron un alto nivel de decoloración y marchitamiento foliar de tres o más hojas. Además, presentaron un deterioro vascular, evidenciado como marchitamiento de tallos y hojas, destacándose la variedad TMS60444 por presentar muerte total de la planta al final de la evaluación, alcanzando el mayor valor de logAUDPC con 1,89. Por otro lado, las variedades CM6438-14, CM523-7 y MBRA902 evidenciaron síntomas moderados o reducidos, lo cual se vio reflejado por bajos valores de logAUDPC, entre 1,43 y 1,53 (Fig. 2). La prueba de Kruskal Wallis (H = 53,142; gl = 9; p < 0,05) indicó diferencias significativas entre variedades y entre periodos de evaluación (Fig. 2).
Identificación de SNPs en el gen RXAM1
El gen RXAM1 logró ser amplificado de manera satisfactoria a pesar de su gran tamaño en cada una de diez las variedades de yuca evaluadas. Tras la amplificación, clonación y secuenciación de los clones obtenidos por cada variedad se logró obtener secuencias de calidad aptas para el análisis de todas las variedades, excepto para los clones obtenidos de la variedad MCOL2215, cuyas secuencias mostraron una muy baja calidad y no pudieron ser analizadas.
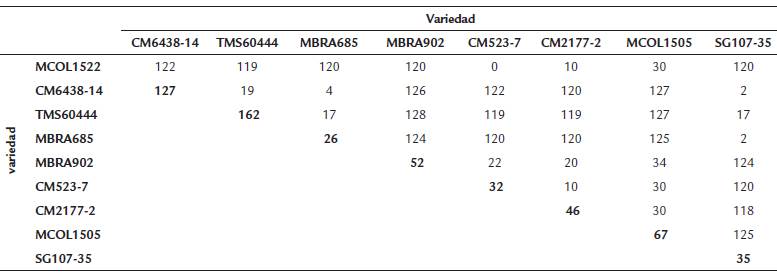
Teniendo en cuenta que la yuca es considerada una especie altamente heterocigota, es posible identificar SNPs dentro de la misma variedad (intravariedad), así como SNPs presentes entre dos o más variedades (entrevariedades). Por esta razón los análisis se realizaron según esta clasificación. En la Tabla 2 se presenta el número de SNPs intravariedades identificados, en donde se observa que las variedades CM643814, TMS60444 y MCOL1505 presentaron el mayor número de SNPs, mientras que las variedades CM523-7, CM2177-2 y MBRA685 mostraron un bajo número de polimorfismos. Con respecto a los SNPs entre variedades, se determinaron mediante una comparación pareada para precisar el número total de polimorfismos entre variedades (Tabla 2). El número de SNPs varió de manera considerable entre variedades. Un caso extremo lo constituyen CM523-7 y MCOL1522, en las que no se identificaron polimorfismos en el gen. En unos casos el número de SNPs fue muy bajo (entre 2 y 4, por ejemplo, SG107-35 Vs. CM6438-14 o MBRA685 Vs. CM6438-14), mientras que en otros el número estuvo alrededor de 120 (CM6438 Vs. MCOL1522, MBRA902 Vs. MBRA685, por ejemplo) (Tabla 2).
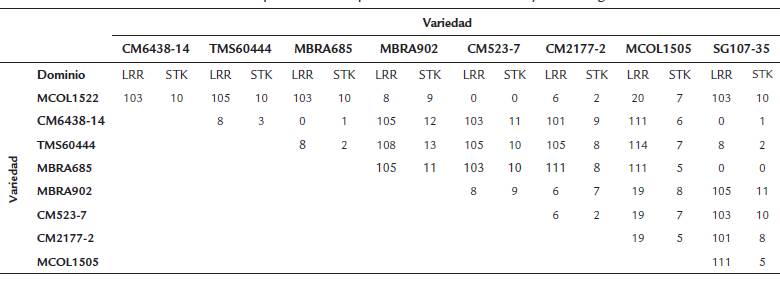
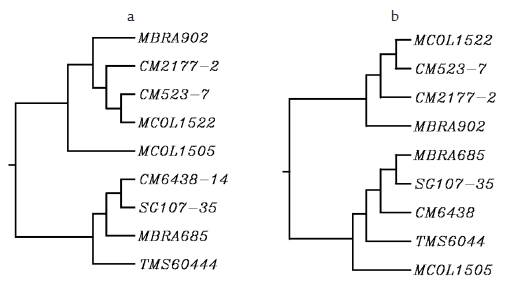
Posteriormente, se consideraron las regiones correspondientes a los dos dominios funcionales de la proteína: el dominio LRR y el STK. En la Figura 3 se observa que el dominio LRR se encuentra entre el aminoácido 75 y el 584, el TM (transmembranal) está delimitado entre los aminoácidos 585 y 703, mientras que el STK comprende los aminoácidos 704 a 1005. A partir de los resultados obtenidos se encontró que la región correspondiente al dominio LRR fue más polimórfica con respecto al dominio STK (Tabla 3). En algunos casos se detectaron más de 100 SNPs en el dominio LRR al comparar dos variedades (MBRA902 Vs. TMS60444, MBRA902 Vs. MBRA685, Tabla 3), pero en otros el nivel de polimorfismo en este mismo dominio fue muy bajo (CM2177-2 Vs. MBRA902, CM2177-2 Vs. CM523-7, TMS60444 Vs. CM6438-14, Tabla 3). Por otro lado, para el dominio STK el número de SNPs fue relativamente constante y bajo, oscilando entre 0 y 13 (Tabla 3). A partir del alineamiento de las secuencias de los dominios LRR y STK se construyeron árboles filogenéticos para determinar la relación entre las variedades. Como se observa en las figuras 4ay 4b, los agrupamientos en lo esencial se mantienen tanto para el análisis del dominio LRR como para el STK, respectivamente. La variedad MCOL1505, de acuerdo con los polimorfismos en el LRR, aparece cercana al grupo de MBRA902, CM2177-2, CM523-7 y MCOL1522 (Figura 4a), mientras que de acuerdo con el dominio STK se agrupa con el otro grupo de variedades. La relación Ka/ Ks (dN/dS) promedio para el dominio LRR fue de 0,79 (X 2 , p< 0,001), indicando que se encuentra bajo selección estabilizadora o purificadora. Los cambios no-sinónimos sobre el dominio STK se ven favorecidos (Ka/Ks = 1,19 (X2, p< 0,001), por lo que se puede establecer que este dominio se encuentra bajo selección positiva.
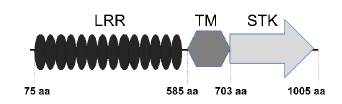
Figura 3 Esquema de la estructura de la proteína RXAM1. Se muestran las posiciones de los dominios LRR, TM (transmembranal) y STK. El esquema no se encuentra a escala.
Correlación entre fenotipo y SNPs
A partir de las secuencias de los cinco clones de cada variedad de yuca evaluados, se construyó una matriz de los SNPs identificados con el fin de observar solo las posiciones polimórficas. En este estudio se analizaron 203 SNPs, lo cual corresponde un α* = 0,05/203 =0,00024631 (ver métodos). De acuerdo con la prueba de chi-cuadrado (X2), no se presentó una asociación estadísticamente significativa entre las variantes alélicas determinadas y el fenotipo de resistencia/susceptibilidad para las variedades evaluadas.
DISCUSIÓN
En este trabajo se estudió la posible correlación entre polimorfismos tipo SNPs en el gen RXAM1 y el fenotipo de resistencia/susceptibilidad a la bacteriosis vascular en variedades de yuca. La evaluación fenotípica permitió identificar respuestas diferenciales entre las variedades. Así mismo, a partir del análisis de la secuencia del gen RXAM1 se identificaron polimorfismos tipo SNPs intra y entre variedades. Los análisis determinaron una gran variación en el número de polimorfismos, tanto entre variedades como dentro de ellas. Sin embargo, ninguno de los polimorfismos identificados mostró asociación con el fenotipo de respuesta a la enfermedad.
Los resultados obtenidos a partir de la evaluación fenotípica de diez variedades de yuca con la cepa CIO136 de Xam, indican diferencias entre variedades frente a esta cepa. La variedad TMS60444 se destacó como la más susceptible. Los síntomas observados pueden ser producto de procesos tales como la degradación de las paredes celulares, defoliación y pérdida de turgencia hasta la muerte celular, tal como ha sido reportado en estudios previos (Boher et al., 1995), y se relaciona con altos valores de AUDPC observados. Por otra parte, las variedades MCOL2215, MCOL1505, SG10735 y MCOL1522 se consideran también susceptibles, pero en una escala menor que TMS60444, porque a pesar de presentar síntomas de susceptibilidad y altos valores de AUDPC, no presentaron muerte total. Resultados similares han sido reportados previamente (Trujillo et al., 2014), en los cuales las variedades TMS60444, SG10735, MCOL1522 y MCOL2215 también presentaron los mayores niveles de susceptibilidad. En este estudio, también se pudo evidenciar que las variedades CM523-7, CM6438-14 y MBRA902 son resistentes a la cepa evaluada; estas observaciones concuerdan con los trabajos previos, en los que estas variedades también fueron catalogadas como resistentes frente a otras cepas de Xam (Trujillo et al., 2014).
Los resultados obtenidos en este estudio muestran que las variedades SG10735, TMS60444, CM6438-14, MBRA685 y MBRA902 presentan el mayor número de SNPs dentro de ellas y entre las demás variedades evaluadas. Estos resultados coinciden con los análisis realizados por Contreras y López (2011), según los cuales las variedades TMS60444, SG10735 y MCOL2246 también se caracterizan por presentar un mayor número de SNPs en otro gen de resistencia. Los resultados obtenidos en estos estudios se pueden explicar considerando varios trabajos en los que se presenta evidencia de que este grupo de variedades provienen de diferentes orígenes geográficos y de especies silvestres distintas (Roa et al., 1997; Olsen y Schaal, 1999). En particular, se ha establecido que la variedad TMS60444 es originaria de África (Nigeria) y producto de un programa de mejoramiento en el que se empleó como fuente genética la especie Manihot glaziovii. Por otro lado, la variedad MBRA685 es nativa de Brasil y su silvestre ancestral es Manihot flabellifolia. Los reportes previos de Castelblanco et al. (2004) han demostrado que las accesiones de yuca procedentes de diferentes países presentan una marcada divergencia genética que evidencian un alto nivel de polimorfismo y heterocigocidad. En contraste, Roa et al. (1997; 2000) reportan una estrecha relación genética entre las variedades CM2177-2, MCOL1505, CM3306-9 y MNGA-2 porque comparten un mismo origen geográfico (Brasil) y un ancestro silvestre (Manihot esculenta subsp. peruviana). En consecuencia, es coherente encontrar una estrecha relación genética entre las variedades evaluadas CM2177-2, CM523-7 y MCOL1522, dado su origen (Roa et al., 1997), tal como se encontró en este estudio (Figs. 4a y b). En este sentido, los resultados obtenidos muestran que las variedades analizadas presentan un bajo número de SNPs, posiblemente porque los genotipos parentales de las variedades pertenecen al mismo acervo genético, lo cual indica una estrecha relación filogenética. Es posible que los programas de mejoramiento genético, al buscar materiales con buenas características agronómicas, han utilizado un limitado grupo de progenitores en sus cruzamientos, reduciendo la base genética de los mismos (Bredeson et al., 2016).
En cuanto al análisis de SNPs de las regiones correspondientes a los dominios LRR y STK de RXAM1, se evidencia que la región correspondiente al dominio STK presenta un número de SNPs relativamente menor a aquella que codifica para el dominio LRR. Considerando que los dominios LRR en las proteínas de resistencia generalmente están implicados en la especificidad del reconocimiento de proteínas del patógeno, éstos deben presentar una alta variabilidad para adaptarse a las nuevas variantes de las proteínas del patógeno (Ellis et al., 1999). A pesar del mayor número de SNPs en el dominio LRR, los análisis de Ka/Ks demostraron que este dominio se encuentra bajo selección purificadora. Es posible que esta región de la proteína reconozca moléculas conservadas del patógeno y por ello sus variantes estén asociadas con sustituciones sinónimas. Por otro lado, los bajos niveles de polimorfismos en el dominio STK estuvieron asociados con sustituciones no sinónimas, sugiriendo la posibilidad de que es necesaria cierta variación en la acción ejecutora del dominio responsable de la activación de la señalización de la inmunidad. La estructura general de RXAM1, al ser del tipo RLK, apunta a que este podría ser un PRR que reconociera un PAMP particular de Xanthomonas y que podría existir un mecanismo molecular conservado entre la PTI y la resistencia cuantitativa (Vásquez et al., 2018).
A partir del análisis de asociación no se observó una relación significativa entre las variantes alélicas identificadas del gen RXAM1 en las diferentes variedades y su respuesta fenotípica a la cepa CIO136 de Xam. Este resultado puede deberse a que se trata de un QTL que explica un bajo porcentaje de resistencia (13 %) y por lo tanto es difícil de identificar una asociación.
A pesar de que en el presente estudio no se observa una relación significativa entre los SNPs y el fenotipo, varios estudios en plantas reportan SNPs en genes candidatos de resistencia asociados significativamente con el fenotipo de resistencia para diferentes agentes patógenos (Hart y Griffiths, 2014; Chang et al., 2016). Es posible también que el gen RXAM1 pueda tener una función en resistencia, pero a otras cepas o patógenos. Al respecto, es conveniente monitorear la resistencia con otras cepas utilizando las mismas variedades analizadas. Adicionalmente, es importante destacar que en este estudio se trabajó con un bajo número de variedades. Los estudios que han encontrado asociación genotipo-fenotipo evalúan entre 60 a 100 o incluso más genotipos (Hart y Griffiths, 2014; Chang et al., 2016). A pesar de que existe una importante colección de germoplasma en CIAT, la evaluación fenotípica, la cual requiere un alto número de réplicas, representa demasiado trabajo y espacio en el invernadero. En este sentido sería importante desarrollar nuevas metodologías de fenotipaje de resistencia a la bacteriosis a gran escala.
Otra de las limitaciones de este trabajo fue la obtención de la secuencia completa del gen candidato RXAM1 , lo cual se logró para nueve variedades, pero sería importante expandirlo a un mayor número de variedades. La obtención de la secuencia como se realizó en este estudio fue costosa y dispendiosa. Dado que la yuca es una especie heterocigota, la secuenciación de los productos directamente de PCR generan secuencias ilegibles. Por esta razón fue necesario hacer clonación para capturar las variantes. Con la reducción en los costos de secuenciación y el desarrollo de nuevas estrategias de secuenciación, tales como GBS (Genotyping by Sequencing) o exómica, es posible obtener la secuencia parcial del genoma de un gran número de variedades, lo que facilitaría la identificación de polimorfismos de manera simultánea en varios genes candidatos (Xiao et al., 2017; Cockram y Mackay, 2018).
El presente estudio representa una aproximación inicial que complementa otros estudios relacionados con la búsqueda de fuentes de resistencia a diferentes cepas de Xam. A futuro, las variedades que muestran un fenotipo contrastante identificadas en este estudio podrían cruzarse para obtener poblaciones y desarrollar mapas genéticos y nuevos análisis de QTLs. Así mismo, algunas de las variedades estudiadas en este trabajo podrán cruzarse con otras variedades comerciales susceptibles a la bacteriosis y así obtener progenies con rasgos comerciales superiores. Finalmente, el resultado de esta investigación es promisorio para continuar con otros análisis que validen los efectos de estos genes candidatos en yuca y sus variantes como herramienta útil para la búsqueda de asociaciones con el fenotipo de resistencia. Este trabajo puede considerarse como un paso preliminar a los estudios de asociación a gran escala (GWAS). Este tipo de estrategias permiten no solo la identificación de nuevos genes candidatos, sino que determinan su distribución y frecuencia en diferentes subpoblaciones de yuca. Esta información es vital en la identificación de una amplia gama de fuentes de resistencia a Xam para ser incorporadas en los programas regionales, aumentando así la eficiencia de mejoramiento con base en la buena calidad de los datos fenotípicos y genotípicos.
CONCLUSIONES
La evaluación fenotípica de la resistencia en diez variedades de yuca a la cepa CIO136 de Xam permitió identificar respuestas diferenciales en la velocidad e intensidad de los síntomas entre las variedades. TMS60444, SG10735, MCOL1505, MCOL2215 y MCOL1522 fueron las variedades más susceptibles a la cepa CIO136 de Xam, mientras que CM6438-14, CM523 y BRA902 fueron resistentes. La identificación de SNPs en el gen RXAM1 para estas variedades mostró una gran variabilidad en el número de SNPs, tanto entre variedades como dentro de las mismas. El dominio STK presentó menor número de SNPs pero estos estuvieron asociados con selección diversificadora. Aunque el dominio LRR presentó en algunos casos un alto número de SNPs, la mayoría de ellos estuvieron asociados con cambios sinónimos. Es posible que esto se deba a que reconoce moléculas conservadas del patógeno. En este estudio no se observó una correlación significativa entre los SNPs y el fenotipo. Es necesario expandir este tipo de estudio a un repertorio mayor de variedades de yuca y frente a otras cepas de Xam.