INTRODUCCIÓN
Talinum Adans. incluye aproximadamente 50 especies de plantas semi-suculentas, herbáceas o arbustivas, que habitan regiones tropicales y subtropicales de África, América y Asia (Veselova et al., 2012; Mendoza y Wood, 2013). Taxonómicamente, el género Talinum se ubicaba dentro de la familia Portulacaceae Juss. Estudios recientes (Nyffeler y Eggli, 2010), referidos a análisis filogenéticos basados en caracteres morfológicos y moleculares permitieron excluir a los géneros Talinum, Amphipetalum Bacigalupo y Talinella Baill. de las Portulacaceae, reubicándolos en una nueva familia denominada Talinaceae.
Para Argentina Talinum cuenta con tres especies: Talinum fruticosum (L.) Juss., Talinum. paniculatum (Jacq.) Gaertn. y Talinum polygaloides Guillies ex. Arn.; las dos primeras adventicias y la última nativa. T. paniculatum es una hierba perenne, con flores purpúreas (Moench, 1794) típica de lugares disturbados que crece entre los 0-2000 m. s. n. m. Habita en las provincias argentinas de Buenos Aires, Catamarca, Córdoba, Corrientes, Entre Ríos, Formosa, Jujuy, La Rioja, Mendoza, Misiones, Salta, Santa Fé, San Juan, San Luis, Tucumán y en países limítrofes como Brasil, Paraguay y Uruguay (Anton y Zuloaga, 2018). T. paniculatum es polimórfico y se reconocen dos variedades: T. paniculatum var. paniculatum y T. paniculatum var. sarmentosum (Engelm.) Poelln. (Von Poellnitz, 1933).
Talinum paniculatum es una especie económicamente importante como maleza resistente a herbicidas, ocasionando daños en cultivos de café y palma (Bellavita et al., 1967; Salazar-Gutiérrez e Hincapié-Gómez, 2007; Ariza y Almaza-Merchán, 2012; Calderón, 2013). Entre sus propiedades útiles Rapoport et. al. (2009) mencionaron que las hojas y tallos de esta especie se utilizan como verdura comestible, mientras que Kumar y Prasad (2010) hicieron alusión a su potencial como planta ornamental y a sus propiedades medicinales.
Los escasos antecedentes anatómicos para el género Talinum son citados por Metcalfe y Chalk (1950); ellos hacen referencia a la presencia de epidermis papilosa y cristales en hoja. No se registra información anatómica para T. paniculatum. Entre los antecedentes citogenéticos se puede citar a Darlington y Wylie (1955), quienes le atribuyeron a Talinum el número básico x = 6, criterio compartido por Black y Mordy (1972) y Andrada-Mansilla et al. (2007). Lewis (1980) mencionó un número básico diferente al citado por estos autores, indicando que Talinum presenta únicamente x = 12. Recuentos cromosómicos llevados a cabo por Xu et al. (1992) en poblaciones de T. paniculatum provenientes de Shangái indicaron que el taxón es diploide (2n = 24).
En la provincia de Tucumán se observaron variaciones morfológicas en órganos vegetativos y reproductivos entre individuos de distintas poblaciones naturalizadas de T. paniculatum. Las variaciones fueron principalmente en lo referente al porte de las plantas, la disposición de sus hojas y el color de sus flores y frutos. Esto llevó a plantear la posible existencia de dos morfotipos. El objetivo de este trabajo fue determinar las características morfo-anatómicas de hoja y tallo, citogenéticas y moleculares en dos morfotipos de T. paniculatum de la provincia de Tucumán, y establecer diferencias entre ellos.
MATERIALES Y MÉTODOS
Estudios morfológicos
Se seleccionaron al azar cinco individuos completos de material fresco. El estudio comparativo se basó en las siguientes variables: altura de la planta; disposición, forma, color y margen de la hoja; color de la flor; color y tamaño del fruto.
Estudios anatómicos
Se seleccionaron al azar cinco plantas por cada población y se fijaron en FAA diez hojas por cada individuo (D'Ambrogio de Argüeso, 1986). Para el análisis de la epidermis se utilizaron cinco hojas y se sometieron a la técnica de diafanizado (Dizeo de Strittmatter, 1973). Con la técnica "mano alzada", se realizaron cortes transversales en lámina y tallo; en este último los cortes se hicieron en la parte inferior, media y superior (D'Ambrogio de Argüeso, 1986). Las tinciones se llevaron a cabo con cristal violeta y la doble tinción azul astra-safranina. Se utilizaron técnicas histoquímicas con lugol para evaluar la presencia de almidón y nitrato de plata para proteínas (D'Ambrogio de Argüeso, 1986). Los preparados transitorios se montaron en agua glicerina (1:1). Los tipos de aparatos estomáticos se clasificaron de acuerdo con Dilcher (1974) y se estimó el tamaño y la densidad promedio de estomas (estomas/mm2).
Estudios citogenéticos
Se pretrataron raíces con 8 hidroxiquinoleina al 0,002 M durante 24 horas a 0 °C, con posterior lavado en agua destilada y fijación en alcohol etílico-ácido acético (3:1) por 24 horas, para luego conservarse en alcohol etílico al 70° a 4 °C hasta su utilización. La coloración empleada fue hematoxilina propiónica al 2 %, previa hidrólisis ácida en HCl 1 N a 60 °C durante 20 minutos. Para la elaboración del cariotipo se utilizaron siete placas metafásicas. Las mediciones se efectuaron mediante el software MicroMeasure 3.3 (Reeves, 2001). Las variables consideradas fueron: c (longitud total), s (longitud de brazo corto), l (longitud de brazo largo), Ic (índice centromérico) y longitud total del complemento cromosómico haploide (LTC). Los cromosomas se clasificaron de acuerdo con Levan et al. (1964). Para el cálculo de las asimetrías cromosómicas A1 y A2 se siguió la metodología propuesta por Romero-Zarco (1986).
Las observaciones se realizaron en un microscopio óptico (Axiostar Plus, Carl Zeiss, Göttingen, Alemania) y en microscopio óptico de luz polarizada (AXIO Lab. A1, Carl Zeiss, Göttingen, Alemania). Las fotomicrografías se tomaron con cámara digital Canon, Power Shot A620, 7.1 M.P. La confección del idiograma y la edición de imágenes se efectuaron con el programa Corel Draw X3.
Análisis moleculares
La extracción de ADN se realizó a partir del método de Saghai-Marrof et al. (1984) en hojas frescas, con posterior uso de kit de extracción Quiagen DNeasy Blood & Tissue (Quiagen GMBH, Germany). Las secuencias ITS2 se amplificaron mediante un par de primers universales (White et al., 1990; Chen et al., 2010): ITS-2F, 5'-(ATGCGATACTTGGTGTGAAT)-3' and ITS-4R, 5'-(TCCTCCGCTTATTGATATGC)-3'. Los primers se sintetizaron en Invitrogen Argentina. La técnica de PCR consistió en 25 μL de volumen final, conteniendo Buffer de reacción (10X Highway 2,5 μ!; 0,75 μ! de MgCl2 25 mM Thermo Scientific); 0,5 μL de Primer ITS2F (200 ng^L); 0,5 μL de Primer ITS4R (200 ng^L); 0,5 μL de dNTPs (0,5 mM); 0,15 μL de TAQ T-plus Highway polimerasa 2U; agua bidestilada (18,1 μL) y 2 μL de ADN total (200 ng^L). La amplificación se realizó en un Termociclador Nyxtechnik ATC 201 con el siguiente programa de PCR: 94 °C por cinco minutos; seguido de un ciclo de 95 °C por 45 seg, 47 °C por 45 seg, 72°C por un minutos; un ciclo de 95 °C por 45 seg, 48 °C por 45 seg, 72 °C por un minuto; tres ciclo de 95 °C por 45 seg, 49 °C por 45 seg, 72 °C por un minuto; 40 ciclos a 95 °C por 45 seg, 50 °C por 45 seg, 72 °C por un minuto; y por último 72 °C por diez minutos; finalmente se mantuvo a 4 °C hasta su utilización. Los productos de PCR se secuenciaron en los laboratorios de Macrogen (Seúl, Corea del Sur).
Análisis de datos: las secuencias obtenidas se editaron manualmente, se alinearon automáticamente mediante la función Clustal W (Thompson et al., 1994) y se corrigieron de forma manual empleando el programa MEGA 6 (Tamura et al., 2013). Tomando en cuenta el criterio de Hidden Markov Model (HMM) (Keller et al., 2009), se eliminaron de los amplicones las secuencias ribosomales con el fin de obtener fragmentos ITS2 puros. Para confirmar la identidad de los ITS2 obtenidos, los fragmentos se compararon con la base de datos del GenBank mediante la función Basic Local Alignment Search Tool (BLAST, http://blast.ncbi. nlm.nih.gov/Blast.cgi). Se seleccionaron las secuencias correspondientes con el género Talinum, además de tres secuencias del género Portulaca como grupo externo (Tabla 2). Se llevaron a cabo análisis de máxima parsimonia (MP) y máximo "likelihood" (ML) en el programa MEGA 6. Para el análisis de MP se emplearon opciones heurísticas CNI que incluyeron la selección de 10 000 réplicas para capturar toda la variación topológica (cf. Sanderson y Doyle, 1993).
La prueba de estabilidad de los clados se efectuó mediante bootstrap (100 réplicas) y se estableció el árbol de consenso correspondiente, tomando como valores para el establecimiento de los nodos aquellos superiores al 50 % de soporte. El análisis filogenético por el método de ML se efectuó a través del modelo (GTR), con tasa de sustitución GAMMA y sitios invariables (G+I) y la prueba de bootstrap con 1 000 réplicas.
Material estudiado
MATERIAL ESTUDIADO: Talinum paniculatum. ARGENTINA. Prov. Tucumán. Dpto. San Miguel de Tucumán, Loc. Ciudadela, 15 V 2016, 26°49'57"S, 65°13'21"W, A. R. Andrada (615229 LIL). 20 II 2018, 26°49'53"S, 65°13'21"W, A. R. Andrada 163; Dpto. Tafí Viejo, Loc. Barrio Orlandi, 10 XII 2018, 26°43'23"S, 65°15'10"W, A. R. Andrada 162; Loc. Reserva Aguas Chiquitas, 12 XII 18, 26°36'58"S, 65°11'07"W, A. R. Andrada 164.
MATERIAL ADICIONAL EXAMINADO: Talinun paniculatum (Jacq) Gaertn (flores rosada-purpurea). ARGENTINA. Prov. Buenos Aires: Dpto. Baradero, 23 II 1974, Krapovickas 3427A-C (LIL). Prov. Corrientes: Dpto. Santo Tomé, Loc. Timbó, 28°21'00"S, 55°47'00"W, 4 X 1982, 570621 LIL. Prov. Formosa: Dpto. Pilcomayo, Loc. Ruta 86. Puente de la Gobernación, 16 IV 1947, Ismael Morel 2590 (LIL); Loc. Ruta 11-Clorinda a Formosa, 2 XII 1946, Ismael Morel 1826 (LIL). Prov. Misiones: Dpto. Cainguás, Loc. Salto Encantado, V 1950, 230 m, Schimidth 4669; Dpto. Candelaria, 7 XI 1984, 230 m, Legname , López y Serrano 9377; Loc. Ruta 12, orillas del arroyo San Juan, 15 XI 1980, 85 m, Legname, D. Serrano 7673; Dpto. Iguazú, Loc. Parque Nacional Iguazú, 6 VIII 1991, 230 m, R. Vanni, S. Ferrari, S. Chiquisola s. n. (602290 LIL); Dpto. Oberá, Loc. Panambi, 28 XI 1975, S.N. Pires y L.A. Nroginaki 324; Dpto. Posadas, 3 XI 1957, 379369 LIL; Dpto. San Ignacio, 27 I 1947, 11706 LIL.; Loc. Corpus- puerto, 5 XI 1988, 26166 LIL; Dpto. San Pedro, Loc. Arroyo Piray camino de Eldorado a San Pedro, 18 XI 1980, Legname, J. López D Serrano 7695. Prov. Tucumán: Dpto. Juan Bautista Alberdi, Loc. Dique Escaba, a 64 km de la hostería, 13 XI 2002, 610 m, M.R. Figueroa Romero, A. Slanis. N. Muruaga 1524; Dpto. San Miguel de Tucumán (Capital), Loc. Ciudadela, 3 IX 2007, A. R. Andrada s. n. (6109612 LIL).
Talinun paniculatum (Jacq) Gaertn (flores blanco-amarillentas). ARGENTINA. Prov. Buenos Aires: III 1973, T. Meyer 2252 (LIL). Prov. Catamarca: Dpto. Andalgalá, 9 XII 1915, 1238 m, 11538 (LIL). Prov. Chaco: Dpto. Resistencia, Loc. Río Araza, 2 II 1947, T. Meyer 11873 (LIL). Prov. Córdoba: Dpto. Colón, Loc. Arroyo Pampa, I 1965, Villa Caranzo 2139 (LIL); Dpto. Capital, Loc. Cerca del observatorio, I 1950, J. Huziker 2646; Dpto. San Javier, Loc. Villa Dolores, 25 III 1943, M. Zerribile 695; Dpto. Colón, Loc. Agua Dulce, II 1952, 436763 LIL. Prov. Corrientes: Dpto. Empedrado, Loc. Empedrado, 3 V 1950, G. J. Silmorkz 9981; Loc. Manuel Derqui, 11 III 1950, Q. J. Schvar 10146; Dpto. Santo Tomé, Loc. Colonia Garabi, 3 XI 1970, 555497 LIL. Prov. Formosa: Dpto. Pilcomayo, Loc. Ruta 86. KM 83, 13 I 1949, 332736 LIL; Loc. Ruta 11 - de Clorinda a Formosa, 2 XII 1946, Ismael Morel 1826 (LIL); 1918, 11543 LIL. Prov. Jujuy: Dpto. Santa Bárbara, Loc. Vinalito, 30 III 1983, 445810 LIL; Dpto. Tumbaya, Loc. Volcán, 14 I 1927, Castillón 102; Misma localidad, II 1927, 2300 m, 11544 LIL; 12 I 1927, 2100 m, Castillón 42. Prov. Misiones: Dpto. Candelaria, Loc. Candelaria, 23 V 1944, 98520 LIL; Dpto. San Ignacio, Loc. Gobernador Roca, II 1945, 226 m, 128761 LIL. Prov. Salta: Dpto. Anta, Loc. Coronel Olleros, 3 III 1980, 300 m, Legname, J. López y D, Serrano 7229; Dpto. Rosario de la Frontera, Loc. Rosario de la frontera, 17 II 1947, 790 m, A. V. de la Zota s. n., 195577 LIL; misma localidad, 17 II 1947, 790 m, A. V. de la Sota 189; 9 IV 1945, 12876 LIL. Prov. Tucumán: Dpto. Juan Bautista Alberdi, Loc. Dique Escaba, alrededores, 13 XI 2002, 725 m, M.R. Figueroa Romero, A. Slanis. N. Muruaga 1563a-b; Loc. Próximo Dique Escaba, a 64km de la hostería, 13 XI 2002, 610 m, M.R. Figueroa Romero, A. Slanis. N. Muruaga 1524; Dpto. Río Chico, Loc. La Calera, 30 XI 1913, 500 m, León Monetti 1567; Dpto. San Miguel de Tucumán (Capital), Loc. Villa Luján, 1918, 460 m, 11541 LIL; Dpto. Tafí, 22 XI 1908, 2050 m, 11536 LIL; Dpto. Tafí del Valle, Loc. Cerro el Pelado, 11 I 1978, 2000 m, A. M. Tiurpe 3017; Dpto. Tafí Viejo, Loc. Orillas de la ruta Camino al Cadillal, 4 XI 1945, 127662 LIL; Dpto. Trancas, Loc. Camino a Hualinchay, 15 XII 1975, Judith Lotti N°88 (LIL); Prov. Santiago del Estero: Dpto. Quebrachos, Loc. Taco Pozo, XI 1932, 250 m, S/C 11622 LIL; misma localidad, XI 2017, 500 m, 1 1620 LIL; XI 1932, 11622 LIL.
RESULTADOS
Estudios morfológicos
Morfotipo 1 (MT1): planta de hasta 0,60 m de altura; hoja elíptica, ovada u obovada con lámina suavemente ondulada y en ocasiones ápice acuminado (Fig. 1a), de color verde oscuro en el haz y verde claro en el envés; hojas opuestas-subopuestas (Fig. 1c), excepto en la porción superior del tallo próxima a la inflorescencia con dos a tres hojas pequeñas alternas. Flores rosada-purpúrea y frutos capsulares de color amarillo-rojizo de 21-25 mm (Fig. 1b). Material: A. R. Andrada (615229 LIL), A. R. Andrada 162. Morfotipo 2 (MT2): planta de hasta 1,20 m de altura, hojas elípticas oblongo-obovada (Fig. 1d), alternas de disposición helicoidal a lo largo del tallo (Fig. 1f), de color verde claro en haz y envés, con lámina sin ondulación. Flor blanco-amarillenta; fruto cápsula de color verde claro de 30-34 mm (Fig. 1e). Material: A. R. Andrada 163, 164 (LIL).

Figura 1 Talinum paniculatum (Jacq.) Gaertn. a-c Morfotipo 1 (MT1). a) Aspecto morfológico general de la planta. b) Detalle de flor y fruto. c) Hojas basales opuestas. d-f Morfotipo 2 (MT2). d) Aspecto morfológico general de la planta. e) Detalle de flor y fruto. f) Hojas basales alternas helicoidalmente.
Estudios anatómicos
ANATOMÍA DE LA LÁMINA. Los morfotipos analizados comparten los siguientes caracteres: en vista paradermal las células epidérmicas son poligonales con paredes rectas a curvas, lámina anfistomática y estomas de tipo paracítico y hemiparacítico (Figs. 2a-b, respectivamente). En corte transversal, se observa la lámina dorsiventral y el nervio principal colateral con casquete colenquimático en floema (Figs. 2g, h). Presencia de mucílago, rafidios (Fig. 2e) y drusas en mesofilo (Figs. 2i, j); idioblastos cristalíferos proteicos en epidermis y mesofilo (Fig. 2k, l).
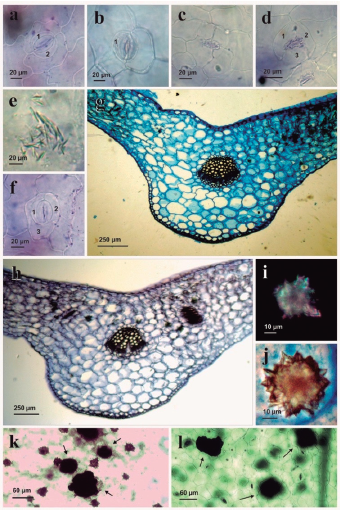
Figura 2: Anatomía foliar de Talinum paniculatum (Jacq.) Gaertn. MT1: a, c, d, e, f, i, k, l. MT2: b, g, h, j. a) Estoma paracítico. b) Estoma hemiparacítico. c) Estoma anomocítico. d) Estoma anisocítico. e) Rafidios. f) Estoma hemi-anfiparacítico. g, h) Corte transversal de la lámina a nivel de la nervadura central. i, j) Drusas con luz polarizada y luz blanca respectivamente. k, l) Proteínas teñidas con nitrato de plata. Abreviaciones: 1, 2, 3=células subsidiarias. Tinciones: cristal violeta a-f, azul astra-safranina g-h y J, nitrato de plata k-l.
Los morfotipos analizados se diferencian por los siguientes caracteres:
MT1: estomas de tipo anfiparacíticos y hemianfiparacíticos (Fig. 2f); su densidad fue 5,4 estomas/mm2 y 13,6 estomas/ mm2 en superficie adaxial y abaxial respectivamente.
MT2: estomas de tipo anomocíticos (Fig. 2c) y anisocítico (Fig. 2d), con una densidad de 11,1 estomas/mm2 en superficie adaxial y 8,8 estomas/mm2 en abaxial.
ANATOMÍA DEL TALLO: los morfotipos analizados presentaron crecimiento primario y distintos estadios de crecimiento secundario; se observaron mucílago, drusas, proteínas cristalinas y almidón en parénquima cortical y medular. El crecimiento primario presentó una epidermis unistrata, colénquima angular subepidérmico con tres a cuatro capas de células y parénquima cortical con cuatro a diez estratos. El sistema vascular es una sifonostela ectoflioca con un anillo de esclerénquima hacia el floema (Fig. 3a, b, e, f). El crecimiento secundario del tallo evidenció peridermis con lenticelas en diferentes grados de desarrollo, floema y xilema continuo, esclerénquima discontinuo hacia el floema, además del colénquima subepidérmico y el parénquima cortical con cinco a seis estratos (Fig. 3c, d, g, h).
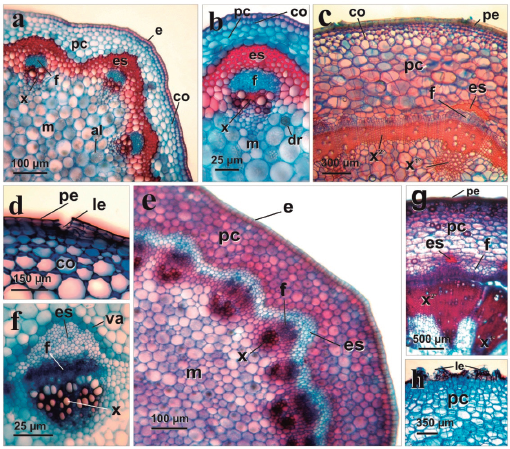
Figura 3: Anatomía caulinar de Talinum paniculatum (Jacq.) Gaertn. MT1: a-d. MT2: e-h. a) Corte transversal de tallo con crecimiento primario. b) Detalle del haz vascular. c) Corte transversal de tallo con crecimiento secundario. d) Detalle de peridermis con lenticela y colénquima angular. e) Corte transversal de tallo con crecimiento primario. f) Detalle del haz vascular. g) Corte transversal de tallo con crecimiento secundario. h) Detalle de peridermis con lenticelas. Abreviaciones: al=almidón; co=colénquima; dr=drusa; e=epidermis; es=esclerénquima; f=floema; le=lenticela; m=médula; pe=peridermis; pc=parénquima cortical; va=vaina amilífera; x=xilema; x1°=xilema primario; x2°=xilema secundario. Tinción: azul astra-safranina.
Las diferencias observadas en el tallo se limitan a la forma que presenta en corte transversal en crecimiento primario y a la presencia o no de vaina endodermoide amilífera que rodea los haces vasculares. MT1: el corte transversal es de forma subtriangular, sin vaina endodermoide. MT2: el corte transversal es de forma subcircular; con una vaina endodermoide amilífera (Fig. 3f).
Estudios citogenéticos
Ambos morfotipos presentaron un número cromosómico diploide de 2n = 24 cromosomas y fórmula cariotípica de 24 m (Fig. 4). En la Tabla 1 se registran los parámetros analizados para cada morfotipo.

Figura 4 Talinum paniculatum (Jacq.) Gaertn. a) Cariograma de MT1. b) Cariograma de MT2. Sat = satélite. Escala = 5 μm.
Tabla 1 Parámetros morfométricos de los cromosomas de Talinum paniculatum (Jacq.) Gaertn. Longitud cromosomica total media (c). Long/itud media brazo largo (l). Longitud media brazo corto (s). Índice centromérico (Ic). Metacéntrico (m). Desviación estándar (DE).
| Par cromosómico |
C (μ) |
L (μ) |
S (μ) |
Lc% | Tipo cromosómico |
|---|---|---|---|---|---|
| MT1 | |||||
| 1 | 2,24±0,57 | 1,37±0,46 | 0,87±0,12 | 38 | m |
| 2 | 2,09±0,42 | 1,03±0,12 | 1,06±0,31 | 50 | m |
| 3 | 1,83±0,16 | 1,00±0,09 | 0,83±0,08 | 45 | m |
| 4 | 1,86±0,19 | 1,02±0,11 | 0,84±0,09 | 45 | m |
| 5 | 1,78±0,11 | 0,98±0,07 | 0,80±0,05 | 44 | m |
| 6 | 1,65±0,02 | 0,92±0,01 | 0,73±0,02 | 44 | m |
| 7 | 1,61±0,06 | 0,83±0,08 | 0,78±0,03 | 48 | m |
| 8 | 1,57±0,10 | 0,86±0,05 | 0,71±0,04 | 45 | m |
| 9 | 1,49±0,18 | 0,79±0,12 | 0,70±0,05 | 46 | m |
| 10 | 1,42±0,25 | 0,76±0,15 | 0,66±0,09 | 46 | m |
| 11 | 1,37±0,30 | 0,81±0,1 | 0,56±0,19 | 40 | m |
| 12 | 1,19±0,48 | 0,66±0,25 | 0,53±0,22 | 44 | m |
| MT2 | |||||
| 1 | 2,60±0,52 | 1,51±0,34 | 1,09±0,29 | 42 | m sat |
| 2 | 2,42±0,35 | 1,26±0,09 | 1,16±0,36 | 47 | m |
| 3 | 2,32±0,25 | 1,30±0,13 | 1,02±0,22 | 43 | m |
| 4 | 2,16±0,09 | 1,22±0,05 | 0,94±0,14 | 43 | m |
| 5 | 2,10±0,03 | 1,15±0,02 | 0,95±0,15 | 45 | m |
| 6 | 2,04±0,03 | 1,18±0,01 | 0,86±0,06 | 42 | m |
| 7 | 2,30±0,23 | 1,39±0,22 | 0,91±0,11 | 39 | m |
| 8 | 1,94±0,13 | 1,11±0,06 | 0,83±0,03 | 42 | m |
| 9 | 1,88±0,19 | 1,05±0,12 | 0,83±0,03 | 44 | m |
| 10 | 1,82±0,25 | 1,02±0,15 | 0,80±0,005 | 43 | m |
| 11 | 1,74±0,33 | 098±0,19 | 0,76±0,04 | 43 | m |
| 12 | 1,54±0,53 | 0,87±0,3 | 0,67±0,13 | 43 | m |
MT1: la longitud total del complemento cromosómico haploide fue de 20,10 μιη y el rango de las longitudes cromosómicas medias fue de 1,19 a 2,24 μπη (Tabla 1). Su índice centromérico medio fue Ic = 44,10 con categoría de metacéntrico. El cariotipo es unimodal debido a la uniformidad en el tamaño cromosómico, justificado por los valores de su índice de asimetría intercromosómica A2 = 0,20. El índice de asimetría itracromosómica A1 = 0,10 evidencia que el cariotipo de este taxón es simétrico (Fig. 4a).
MT2: el complemento cromosómico calculado fue de 24,86 μΓη y la variación de longitud estuvo entre 1,82 y 2,60 μΓη (Tabla 1). El índice centromérico medio fue Ic = 44,97 con categoría metacéntrico. El índice de asimetría intercromosómica fue de A2 = 0,15, lo que denota un cariotipo unimodal, mientras que el índice intracromosómico de A1 = 0,18 evidencia un cariotipo simétrico. Las observaciones en las células metafásicas muestran en este morfotipo la presencia de satélite terminal en el brazo corto del primer par cromosómico (Fig. 4b).
Análisis moleculares
IDENTIDAD DE LAS REGIONES ITS2. Al comparar las secuencias ITS2 halladas con los datos del GenBank se confirma que se corresponden con las del género Talinun. Del listado de secuencia ITS2 producto del BLAST, 16 pertenecieron a Talinum: 12 de T. paniculatum, uno de T. caffrum (Thunb.) Eckl. & Zeyh., uno de T. fruticosum, uno de T. paraguayense Speg. y uno de T. portulacifolium (Forssk.) Asch. ex Schweinf. Entre los taxones cercanos se tomó como grupo externo las secuencias correspondientes a Portulaca psammotropha Hance, P. yecorensis Henrickson & T. Van Devender y P. oleracea L. (números accesiones en Tabla 2).
Tabla 2 Material del que se obtuvieron las secuencias ITS2, incluyendo las accesiones de las bases de datos GenBank (G) y Boldsystems (B) y las referencias bibliográficas en el caso de los trabajos publicados. El símbolo (*) indica secuencias inéditas.
| Especie | País de origen | Seudónimo utilizado | Herbario: N° de colección | N° de Acceso al GenBank | Referencia |
|---|---|---|---|---|---|
| Talinum paniculatum | Argentina | MT1 Ciudadela | 615248 (LIL) | BENAR040-16 B | * |
| T. paniculatum | Argentina | MT1 Tafí Viejo | Andrada A. R. 158 | MK830707 G | * |
| T. paniculatum | Argentina | MT2 Ciudadela | Andrada A. R. 159 | MK836043G | * |
| T. paniculatum | Argentina | MT2 El Cadillal | Andrada A. R. 160 | MK828557 G | * |
| T. paniculatum | China | - | - | KC878579.1 G | No publicado |
| T. paniculatum | Estados Unidos | USA 1 | - | EU410357.1 G | Brockington S. F., Mavrodiev E., Ramdial J., Dhingra A., Soltis P. S. y Soltis D. E. 2008 |
| T. paniculatum | Estados Unidos | USA 2 | - | JF508608.1 G | Ocampo G. y Columbus J. T. 2012 |
| T. paniculatum | Estados Unidos | USA 3 | - | L78094.1 G | Hershkovitz M. A. y Zimmer E. A. 2000 |
| T. paniculatum | Vietnam | Vietnam 1 | - | LT852526.1 G | No publicado |
| T. paniculatum | Vietnam | Vietnam 2 | - | LT852525.1 G | No publicado |
| T. paniculatum | Vietnam | Vietnam 3 | - | LT853590.1 G | No publicado |
| T. paniculatum | Vietnam | Vietnam 4 | - | LT853591.1 G | No publicado |
| T. paniculatum | Vietnam | Vietnam 5 | - | LT852524.1 G | No publicado |
| T. paniculatum | Vietnam | Vietnam 6 | - | LT852523.1 G | No publicado |
| T. paniculatum | Vietnam | Vietnam 7 | - | LT852522.1 G | No publicado |
| T. caffrum | Estados Unidos | - | - | L78055.1 G | Hershkovitz M. A. y Zimmer E. A. 1997 |
| T. fruticosum | India | - | - | KJ380908.1 G | Swarna J., Ravindhran R. y Lokeswari T. S. 2015 |
| T. paraguayense | Estados Unidos | - | - | L78056.1 G | Hershkovitz M. A. y Zimmer E. A. 1997 |
| T. portulacifolium | Estados Unidos | - | - | L78057.1 G | Hershkovitz M. A. y Zimmer E. A. 1997 |
| Portulaca oleracea | Japón | - | - | AB823847.1 G | Kokubugata G., Nakamura K., Hirayama Y. y Yokota M. 2013 |
| P. psammotropha | Japón | - | - | LC021400.1 G | No publicado |
| P. yecorensis | Estados Unidos | - | - | JF508606.1 G | Ocampo G. y Columbus J. T. 2012 |
ANÁLISIS FILOGENÉTICO. Las longitudes de las secuencias ITS2 utilizadas en el análisis tuvieron un rango de 208-222 nucleótidos, con un promedio de 220,19.
Máxima parsimonia (MP): el análisis de MP produjo como resultado 100 000 árboles informativos. El árbol de consenso de mayoría estableció dos clados principales con soporte de 100%, el primer clado, denominado (Tp), incluye a T. paniculatum y el segundo clado (Tl) comprende al resto de las especies. En el cladograma puede observarse que un fragmento de ITS2 de T. paniculatum (proveniente de China), se ubicó en el clado Tl, correspondiente al subclado de T. fruticosum, con soporte de 100 % de bootstrap; esto puede deberse a un error en la identidad del taxón. Dentro del clado Tp, los taxones de flores rosada-purpúrea MT1 conforman un clado irresuelto con dos de los tres ejemplares de Estados Unidos (USA1 y USA3), los que a su vez forman un grupo hermano con dos clados secundarios bien definidos, el clado Tv que agrupa T. paniculatum de Vietnam (soportado por bootstrap = 100), y el clado Tfb que agrupa a MT2 de flores blancas con el taxón de USA2 (bootstrap = 92) (Fig. 5).
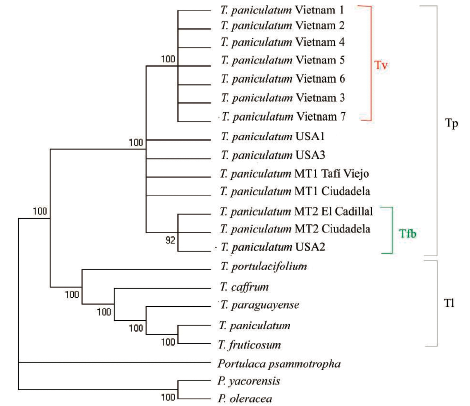
Figura 5 Árbol consenso de Talinum paniculatum (Jacq.) Gaertn. Análisis de MP del fragmento ITS2, obtenido a partir de los 100 000 árboles más parsimoniosos. Longitud: 84 pasos. Indice de consistencia: 0,854167. Indice de retención: 0,914634. El árbol inicial fue derivado de un total de 10 000 réplicas. Los valores de bootstrap se muestran en los nodos, con un valor de corte con soporte igual o superior al 50%. Clados: Tp, incluye los taxones cuyos datos de recolección se corresponden con T. paniculatum, Tl, incluye otras especies de Talinum; Tfb, incluye el MT2 (blanco-amarillentas) y Tv, incluye a T. paniculatum provenientes de Vietnam.
Máximo likelihood (ML): el análisis de ML produjo un árbol con topología similar al de MP. Se mantienen los clados principales Tp y Tl (soportados por bootstrap = 84 %) y los subclados Tv y Tfb (ambos con bootstrap = 63 %). T. paniculatum de China se ubica junto con T. fruticosum de la India (bootstrap = 99 %). MT1 y los taxones USA1 y USA3 continúan irresueltos dentro de Tp (Fig. 6).
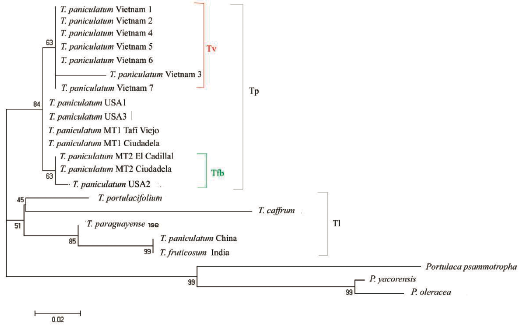
Figura 6 Árbol filogenético Talinum paniculatum (Jacq.) Gaertn. Análisis de ML Máximo likelihood obtenido mediante el uso de ITS2 del ADN ribosómico y modelo GTR+G+I. Distribución gamma (a = 2,1), con cero sitios de variación. Los valores de bootstrap se muestran en los nodos (1000 réplicas). Clados: Tp, incluye todos los taxones cuyos datos de recolección se corresponden T. paniculatum; Tl, incluye otras especies de Talinum; Tfb, incluye el MT2 (flores blanco-amarillentas) y Tv, incluye T. paniculatum provenientes de Vietnam.
DISCUSIÓN
Si bien Mendoza y Word (2013) mencionan que el género Talinum está integrado por 50 especies, la base de datos Trópicos (http://www.tropicos.org, Julio de 2018) cita 97 especies. En esta última Talinum paniculatum está representada por dos variedades: T. paniculatum var. paniculatum y T. paniculatum var. sarmentosum.
En taxones que presentan un patrón de distribución extenso, como T. paniculatum, es frecuente observar variaciones morfológicas dentro de una misma especie en ambientes naturales, lo cual se relaciona con la capacidad de adaptación a diferentes climas y ambientes que generan así cierta variación morfogenética (Svensson, 1983; Spaniel et al., 2008; Kumar, 2010). Los ejemplares aquí estudiados de T. paniculatum pertenecientes a distintas poblaciones presentaron variaciones morfológicas que permitieron diferenciar dos morfotipos: MT1 y MT2. Los morfotipos mencionados se diferenciaron por el porte de las plantas, disposición de sus hojas, color de sus flores, tamaño y color del fruto, tipos y densidad de estomas, presencia de cristales de oxalato de calcio en forma de drusa, forma del corte transversal de tallo con crecimiento primario y la presencia de vaina amilífera. Cabe destacar que las descripciones realizadas por Moench (1794), Von Poellnitz (1933) y Rapoport et al. (2009) en T. paniculatum hacen referencia al color de las flores rosado-purpúrea o blanco-amarillentas con hojas opuestas-subopuestas o alternas, en coincidencia con los morfotipos MT1 o MT2 de este trabajo, sin que se diferencien dichos morfotipos.
En coincidencia con lo descripto por Metcalfe y Chalk (1950) para el género Talinum, en este trabajo se describe la forma y ubicación de cristales para T. paniculatum. Se citan por primera vez la presencia de mucílago y proteínas cristalinas en hoja y se describe la anatomía foliar y caulinar de la especie estudiada.
Los análisis citogenéticos realizados por Lewis (1980) establecieron para Talinum un único número básico x = 12. Esto se sustenta por los números cromosómicos observados por Goldblatt (1979+) (El signo + indica base de datos que se actualiza constantemente (www.tropicos.org/project/ipcn) en su Index to Plant Chromosome Numbers (IPCN), quien confirma 12 o múltiplo de este número para los recuentos cromosómicos del género. Otros autores proponen para Talinum el número básico x = 6 (Darlington y Wylie, 1955; Black y Mordy, 1972; Andrada-Mansilla et al., 2007). La determinación de este último número básico se fundamenta en inferencias hipotéticas ya que no cuenta con pruebas que lo sustenten. Black y Mordy (1972) hacen referencia que el género no cuenta con números cromosómicos diploides menores a 2n = 24.
Actualmente se conocen antecedentes de recuentos cromosómicos para 14 especies del género (Steiner, 1944; Nyananyo y Olowokudejo, 1986; Murdy y Brown-Carter, 2001; Molero et al., 2006; Andrada-Mansilla et al., 2007), con variaciones numéricas que generalmente derivan por poliploidía interespecíficas, y números cromosómicos diploides variables entre 24 y 96 cromosomas.
Además, se observaron autopoliploides intraespecíficos como T. mengesii W. Wolf, T. calycinum Engelm., T. triangulare (= T. fruticosum) y T. parviflorum Nutt. con 2n = 24 y 2n = 48 (Black y Mordy, 1972; Nyananyo y Olowokudejo, 1986; Murdy y Brown-Carter, 2001).
Se ha sugerido que T. variegata es una variedad hexaploide (2n = 72) de T. paniculatun (Steiner, 1944). Lombello y Pinto-Maglio (2009) observaron en T. triangulare, en poblaciones del Sudeste de Brasil, la presencia de aneuploides con n = 22 y 2n = 44. Los recuentos efectuados en los morfotipos de T. paniculatum analizados en este trabajo, afianzan el número básico x = 12, en concordancia con lo postulado por Lewis (1980).
Los cariotipos obtenidos en este trabajo son los primeros estadísticamente confiables para el género, ya que cuentan con soporte estadístico como lo establecen Levan et al. (1964), así como Li y Chen (1985). La presencia de constricciones secundarias similares a las observadas en el morfotipo MT2 se citaron en Steiner (1944) para especies de la familia Portulacaceae.
En cuanto a la filogenia molecular esta investigación aporta el primer árbol con base en marcadores ITS2 para el género y revela que Talinum es un grupo monofilético, en el que T. paniculatum se separó tempranamente, constituyendo un grupo hermano relacionado al resto de las especies que integran el género. Los linajes Tv y Tfb dentro de los árboles de MP y ML se separan de T. paniculatum.
CONCLUSIONES
La presente contribución documenta las características morfológicas, anatómicas, citogenéticas y moleculares de Talinum paniculatum, lo que permitió diferenciar los morfotipos MT1 y MT2. Los caracteres morfológicos de valor diagnóstico para distinguirlos son: tamaño de la planta, disposición, forma y color de la hoja, color de la flor, tamaño y color del fruto, tipos y densidad de estomas, presencia de cristales de oxalato de calcio en forma de drusa, forma del corte transversal de tallo con crecimiento primario y la presencia de vaina amilífera.
Talinum paniculatum es un taxón polimórfico y los morfotipos analizados son diploides con 2n = 24 cromosomas. Los cariotipos son unimodales y se presentan por primera vez para la especie. Ambos morfotipos fueron similares, con fórmula cariotípica de 12 m. El morfotipo MT2 se distingue por la presencia de constricciones secundarias a modo de satélites.
Los análisis moleculares permitieron establecer que Talinum es un grupo monofi lético. El morfotipo MT2 se halla ampliamente distribuido en Argentina, es un grupo hermano de T. paniculatum y presenta diferencias moleculares que lo caracterizan.
Con base en los resultados obtenidos en esta investigación, se concluye que MT2 podría considerarse como una especie o variedad aún no descrita para la flora de Argentina.















