INTRODUCCIÓN
Desde su descubrimiento y hasta la fecha los microorganismos han sido de gran interés ya que colonizan todo tipo de ambiente, lo que les permite participar en procesos metabólicos, ecológicos y biotecnológicos importantes para el mantenimiento de la vida (Abreu y Taga, 2016). Sin embargo, hoy en día sabemos que solo conocemos las condiciones idóneas para cultivar muy pocos microorganismos del total de los existentes en nuestro planeta, lo cual nos ha obligado a replantearnos la siguiente pregunta ¿realmente cuánto conocemos sobre la diversidad microbiana? En la actualidad, la microbiología ha tomado un nuevo enfoque basado en tratar de conocer y comprender el gran número de microorganismos no cultivables a través del uso de nuevas herramientas como la metagenómica (Solden etal., 2016).
La metagenómica es una herramienta que ha generado un gran avance científico, al permitir la obtención de nuevos genes y conocer la interrelación entre comunidades de microorganismos. En la búsqueda de nuevos microorganismos no cultivables, se desarrollaron nuevas técnicas de biología molecular que permiten la clonación de genes y su secuenciación (Jiang et al., 2016). Esto ha permitido en conjunto con otras áreas como la bioinformática y la genética, estudiar los genomas de toda una comunidad ambiental sin la necesidad de un cultivo previo o del aislamiento de los microorganismos, proporcionándonos mayor información sobre la composición microbiana de un nicho ecológico, su historia evolutiva, las interacciones de los microorganismos, así como, el potencial metabólico que toda la información que se podría obtener de las poblaciones cultivables (Mirete et al., 2016; Faner et al., 2017).
Como menciona el National Research Council (US) Committee on Metagenomics: la metagenómica nunca estará delimitada por una definición, pero algo muy parecido a la conceptualización de la metagenómica sería "la ciencia de descubrir, modelar, comprender y finalmente gestionar a nivel molecular las relaciones dinámicas entre las moléculas que definen micro-comunidades y la biosfera" (National Research Council (US) Committee on Metagenomics: Challenges and Functional Applications, 2007).
Por consiguiente, la metagenómica se divide en dos categorías: la metagenómica funcional, la cual se basa en buscar genes con una función determinada apoyándose en el tamizado de una biblioteca metagenómica y la metagenómica estructural, la cual a partir de la secuenciación de ADN metagenómico se realizan análisis bioinformáticos para tratar de resolver dos preguntas clave ¿Qué microorganismos están presentes? y ¿Qué hace cada uno de ellos? (Ravin et al., 2015).
Sin importar el ambiente o ecosistema a estudiar existen tres pasos importantes en la realización de un estudio metagenómico; el primero es la colecta de la muestra de interés, el segundo es la extracción del ADN total de la muestra y el tercero son las tres diferentes opciones que existen para analizar la muestra. A) El ADN metagenómico puede ser sometido a un proceso de digestión y clonación en vectores de expresión como los denominados cromosomas artificiales de bacterias (BAC), cromosomas artificiales de levadura (YACs), Cósmidos, o Fósmidos, que nos permiten construir librerías metagenómicas recuperando insertos de ADN mayores a 40 Kb (Jung y Kim, 2018; Piel et al., 2018; Tocchetti et al., 2018; Buckley y Ettensohn, 2019). B) amplificación por PCR de los genes que codifican para los ARN's ribosomales (ARN's) 16S o 18S, lo cual permite conocer la diversidad de microorganismos en la muestra (bacterias, arqueas o eucariontes) para realizar un análisis filogenético. C) secuenciación directa de la muestra, usando la plataforma de Biosistems SOLID system o las de nueva generación (NGS) como las de Illumina sequencing technology o Roche Genome Secuencer (Dabdoub et al., 2016; Roeh et al., 2017; Ravi et al., 2018). En cualquiera de las opciones anteriores el paso final es el análisis de secuencias de los genes buscando alguna actividad de interés.
Actualmente se han realizado análisis metagenómicos de diversos ambientes, por ejemplo, los estudios en la cavidad oral nos han proporcionado una mejor comprensión sobre los microorganismos presentes en infecciones endodónticas que afectan a humanos o los microorganismos presentes en enfermedades como la periodontitis (Dabdoub et al., 2016; Sánchez-Sanhueza et al., 2018). Un estudio realizado al aire hospitalario nos ha permitido conocer la diversidad de microorganismos y en un futuro ayudará a tener una mejor comprensión de cómo estos pueden influir en la propagación de infecciones asociadas a la atención médica (King et al., 2016). Los análisis metagenómicos realizados en lodos activados usados para tratar aguas residuales contaminadas con compuestos fenólicos permitieron detectar nuevos genes de resistencia a bleomicina, el cual es un agente antitumoral de uso clínico (Mori et al., 2008). El estudio enfocado al intestino de las vacas permitió conocer los microorganismos presentes en este ambiente y como estos influyen en la producción de leche (Pitta et al., 2016). Un análisis metagenómico de las aguas subterráneas de Yucatán permitió el descubrimiento de dos nuevos genes que codifican para proteasas (Apolinar-Hernández et al., 2016). El proyecto metagenómico realizado a la producción del vino permitió elucidar el consorcio de microorganismos asociados con la fermentación maloláctica (Berbegal et al., 2019). Además, los estudios metagenómicos de suelo que también han permitido el aislamiento de nuevos compuestos orgánicos como la Turbomicina A y Turbomicina B que exhiben actividad antibiótica contra microorganismos tanto Gram-positivos como Gram-negativos (Gillespie et al., 2002). Cada uno de los estudios mencionados, más los que se pueden observar en la tabla suplementaria 1, en donde se muestran las diferentes metodologías usadas, el tipo de ambiente de donde proviene la muestra, el método para la generación de librerías y la plataforma elegida para la secuenciación, reafirman que la metagenómica es una herramienta muy poderosa que se puede aplicar a diversos ambientes generando un impacto a nivel de la salud, en química, biotecnología o a nivel farmacéutico. En este artículo se realizó una revisión de trabajos científicos que emplearon metagenómica, usando las bases de datos de PudMed y Google Schoolar, con el objetivo de examinar los beneficios y limitaciones de las diversas metodologías empleadas, específicamente en el tratamiento del ADN metagenómico de suelo y el impacto de la metagenómica en la agricultura.
MATERIALES Y MÉTODOS
Se realizó una revisión bibliográfica de artículos científicos sobre análisis metagenómicos publicados entre los años 2015 a 2020. Los criterios de inclusión en la búsqueda fueron: metagenónica de agua, metagenónica de aire, metagenómica de suelo, metagenómica en alimentos, metagenómica en la agricultura, análisis metagenómicos en salud humana, métodos de extracción de ADN metagenómico y tipo de cepas receptoras de la banca metagenómica.
La revisión se realizó a través de los buscadores PudMed y Google Shoolar, los idiomas de los artículos revisados estaban en el idioma inglés seguido del español y los criterios de selección de los artículos hallados se basó en el tipo de ambiente que se usa para el análisis metagenómico, tipo de secuenciación, metodología para la realización de librerias genómicas, área geográfica y metodologías empleadas para el tratamiento del ADN metagenómico.
METAGENÓMICA DE SUELOS
El suelo es uno de los ambientes más complejos a estudiar debido a su composición y enorme diversidad microbiológica; ambos elementos influyen en procesos importantes como los ciclos biogeoquímicos que contribuyen a la nutrición y al buen estado de la tierra para diferentes ambientes tanto agrícolas como ambientes extremos.
Si bien se han realizado diversos trabajos metagenómicos enfocados en el suelo, cada uno de ellos ha presentado un reto diferente y esto se debe a que el suelo puede presentar diversas variantes que podrían convertirse en limitaciones si no se elige la metodología correcta para su estudio. Dos de los factores que podrían convertirse en una limitante son la composición y la diversidad microbiana del suelo por ejemplo en el trabajo de Llacsa et al. (2019), se buscó identificar los microorganismos presentes en la rizosfera de los árboles de Tabebuia chrysantha y Tabebuia billbergii, los cuales son nativos de la región de Tumbes en Perú y se encuentran en peligro de extinción, con la finalidad de implementar programas de reforestación. El estudio mostró que para el árbol T. billbergii los filos más abundantes fueron Glomeromycota, Ascomycota, Firmicutes, Actinobacteriasy Proteobacterias, en cuanto a T. chrysantha los filos presentes fueron Ascomycota, proteobacterias y actinobacterias, los autores concluyen que se observó una gran diversidad de géneros y de especies de hongos y bacterias, información que forma parte de la base para generar un plan estratégico que les permita mantener la diversidad genética para futuros programas de reforestación.
Otro estudio metagenómico es el realizado por Mendes y Tsai (2018) en una región de bosque y manglar en el sureste de Brasil con la finalidad de conocer la composición taxonómica y funcional de tres regiones: bosque-descanso-manglar que se encuentran muy cercanos entre sí, los resultados obtenidos demostraron que el 94 % de los microorganismos detectados correspondían a bacterias, 3,5 % correspondían a eucariotas y el 1,8 % correspondió a arqueas. Aunque estos microorganismos estaban presentes en las tres áreas, hubo diferencia en su abundancia, la cual está correlacionada con las características específicas de cada suelo, por lo que los autores concluyen que el estudio permitió una mejor comprensión de la relación que existe entre la diversidad microbiana, las funciones y los parámetros ambientales que se podrían utilizar para una mejor conservación de los ambientes.
El análisis metagenómico del suelo de la sabana neotropical ubicado al centro de Brasil compuesto por una combinación de ambientes que abarcan desde los pastizales hasta las formaciones forestales que combinan suelos arcillosos, incluyó ARN ribosomales de alto rendimiento para explorar la composición taxonómica y las funciones potenciales de las comunidades microbianas de cuatro diferentes tipos de suelo: suelos de cerrado denso, cerrado sensu estricto, campo sujo y bosque, bajo las condiciones de dos estaciones diferentes, temporada seca y temporada de lluvias. Como resultado se encontró que durante la temporada de lluvias aumentaban los filos de Proteobacterias y Ascomicetos, mientras que durante la temporada seca existía abundancia de los filos Planctomycetes, Thermoprotei y Glomeromycota. Este estudio permitió ampliar la comprensión de cómo las comunidades microbianas del suelo de la sabana tropical pueden verse inluenciadas por la cubierta vegetal y las variaciones temporales de la humedad del suelo (de Castro et al., 2016).
La diversidad y estructura de comunidades fúngicas en suelos neotropicales también ha sido importante, por tal motivo se ha realizado un análisis metagenómico para lograr comprender la distribución y posibles interacciones fúngicas con los árboles endomicorrícicos entre dos diferentes suelos: una plantación y un bosque natural. El análisis mostró 42 ordenes y 14 clases fúngicas diferentes, existiendo heterogeneidad espacial entre los dos tipos de suelos; los autores mencionan que no se observó una fuerte variación en la riqueza de especies, ni en la uniformidad entre las parcelas de árboles. Sin embargo, las especies arbóreas dieron forma a los ensamblajes fúngicos de suelo y cubierta vegetal, explicando hasta el 18 % de la variación entre las comunidades en el bosque natural (Schimann et al., 2017).
Finalmente, Fonseca et al. (2018) Realizo un estudio para conocer la diversidad y función de los microbiomas presentes en la reserva forestal amazónica de Snakewood y un suelo a granel, teniendo en cuenta la importancia de los microorganismos en el mantenimiento de los ecosistemas forestales a través de la descomposición del tejido vegetal y liberación nutrientes hacia las plantas. Los resultados indican que existen cambios significativos entre los dos ambientes, los taxones bacterianos y fúngicos son más abundantes en la muestra de la rizosfera forestal, mientras que las arqueobacterias fueron más abundantes en el suelo a granel, sin embargo, se consideró que se requería un mayor número de muestreos para poder comprender que factores son los que impulsan los cambios entre los dos diferentes perfiles microbianos.
Todos los estudios mencionados anteriormente fueron realizados en suelos del trópico y neotrópico cuya finalidad era conocer la composición taxonómica y funcional de diversos ambientes para tener una mejor comprensión de la relación que existe entre la diversidad microbiana y con ello poder resolver problemas como la reforestación. Cada uno de los autores tuvo el reto de elegir un método de extracción de ADN metagenómico que les permitiera conocer la gran diversidad de microorganismos presentes bajo las características específicas de cada suelo, tomaron en cuenta el pH del suelo, sus propiedades químicas y la cantidad de muestra a evaluar, evitando el sesgo de los resultados hacia un grupo específico de microorganismos, hecho en el cual radica el éxito de sus resultados.
Otro factor que podría representar un obstáculo al momento de realizar estudios metagenómicos es la alta concentración de metales o la contaminación del suelo. Por ejemplo, Smulek et al. (2020) realizaron un estudio para caracterizar la degradación de hidrocarburos aromáticos policíclicos (HAD) por parte de consorcios nativos de bacterias, este estudio fue realizado en muestras de suelo sujetas a contaminación por más de 40 años. Este estudio reveló las bacterias nativas Pseudomonas mendocina y Brevundimonas olei estaba presentes y eran capaces de degradar cerca del 60 % de los HAP, además de que el contacto a largo plazo con los compuestos aromáticos indujo a las cepas bacterianas a utilizar los HAP como fuente de carbono y energía.
Otro ejemplo es el estudio del suelo de las aguas termales de Topovan ubicadas en la India, que tuvo la finalidad de conocer la comunidad bacteriana en el suelo con características extremas como alta concentración de azufre y variación de metales dependiendo de la temporada. Se encontraron 14 filos bacterianos diferentes para el periodo de primavera, siendo el filo más representativo el de los Firmicutes y el menos representativo el de Chlorobi (Rawat y Joshi, 2019). Para ambos trabajos el reto consistió en realizar análisis fisicoquímicos del suelo que les permitiera conocer el contenido de HAP o de nitrógeno, fósforo, carbono, azufre y otros metales, así como determinar la humedad del suelo, el nivel de pH, la temperatura e incluso la elección de la distribución del tamaño del grano del suelo. Todos se consideraron factores importantes para que se realizara una correcta extracción de ADN metagenómico y una correcta amplificación de los genes ARN'r 16S.
El uso de sustratos como indicadores para la identificación de enzimas o la selección de cepas receptoras de las librerías metagenómicas pueden ayudar al desarrollo experimental de algunos estudios. Por ejemplo, el análisis en donde se descubrieron nueve carbapenemasas de clase B, conocidas como metalo-beta-lactamasas (MLB), las cuales no tenían ninguna relación con las MLB anteriormente reportadas y que permitió ampliar la comprensión de la diversidad de MLB en la microbiota del suelo (Gudeta et al., 2016). Para este análisis utilizaron cultivos de selección con antibióticos para seleccionar a las bacterias productoras de carbapenemasas.
Un ejemplo similar es el caso de identificación de microorganismos que usan como fuente de carbono y energía al DMS (sulfato de dimetilo) en ambientes terrestres, el cual permitió detectar bacterias de la familia Methylophilaceae que desempeñan una función importante como metilótrofos, los cuales probablemente utilizan enzimas para degradar el DMS; que aún se desconocen ya que estas bacterias tampoco se han logrado cultivar (Eyice et al., 2015). Sin embargo, para detectar esas bacterias utilizaron como sustrato de identificación al propio DMS.
Por último, el análisis de unas muestras provenientes de la Reserva Natural de Kogelberg en Sudáfrica, presentó el reto de realizar una correcta selección tanto del vector en donde se generarían las librerías como de la cepa receptora y la elección del tamaño de las librerías metagenómicas a tamizar. Con esta cuidadosa metodología se lograron identificar a varios genes, la mayoría cuyas secuencias eran de identidad y función desconocida pero uno de ellos codificando para una proteína con actividad de endonucleasa no específica, abriendo la posibilidad de realizar nuevas caracterizaciones de enzimas que ayuden en el diagnóstico de laboratorio (Mtimka et al., 2020).
Como se puede observar varios son los factores que influyen en el desarrollo del análisis del ADN metagenómico, estos factores se relacionan directamente con el rendimiento y la pureza del ADN extraído del suelo a analizar, la poca diversidad de secuencias reportadas o el tipo de cepa bacteriana receptora de la biblioteca metagenómica, pero ¿Por qué la composición y diversidad microbiana, la contaminación del suelo, el uso de sustratos indicadores para identificación de enzimas o el mismo pH pueden afectar en el análisis del ADN metagenómico? (Katz et al., 2016; Makhalanyane et al., 2016; Holland et al., 2018; Gobbi et al., 2019), a continuación abordaremos estas preguntas.
Rendimiento y pureza
La obtención de ADN metagenómico de suelo es un paso crítico ya que debe obtenerse en cantidades suficientes y ser de alta calidad. El ADN metagenómico puede obtenerse mediante dos métodos de extracción: directa e indirecta (Gobbi et al., 2019). La extracción de ADN directa consiste, como su nombre lo dice, en extraer el ADN directamente de la muestra ambiental y todos los protocolos reportados se caracterizan por dos pasos, la disrupción de la pared celular que permite liberar a los ácidos nucleicos y la purificación para separar los ácidos nucleicos de los restos celulares y partículas del suelo (Gobbi et al., 2019). Esta metodología presenta ciertas dificultades debido a las propiedades del suelo como el contenido de ácido húmico, la dispersión del suelo, el valor del pH, el contenido celular y otros contaminantes que se encuentran en algunos sedimentos como hidrocarburos aromáticos policíclicos o metales pesados, los cuales pueden dificultar la extracción, cuantificación y amplificación del ADN (Zhou et al., 1996; Wagner et al., 2015; Shen et al., 2016; Yan et al., 2017; Sáenz et al., 2019).
Por otra parte, la extracción de ADN indirecta consiste en separar las células de la matriz en la cual se encuentran, para después lisarlas, con este procedimiento se separan las células bacterianas y arqueas de las células eucariotas a través de un gradiente de densidad (Högfors-Rönnholm et al., 2018). Este paso de exclusión genera ciertas ventajas, si se quiere trabajar únicamente con bacterias y arqueas ya que incrementa la cantidad de secuencias que se pueden encontrar en una muestra y el grado de pureza que se obtiene del ADN es mayor, así como el tamaño del ADN que se recupera; lo cual es importante cuando el objetivo del estudio es generar librerías con insertos grandes de ADN. Sin embargo, las desventajas de esta metodología son que el proceso de exclusión impide realizar estudios de interacciones entre diversos microorganismos y que los rendimientos que se obtienen de ADN son bajos en comparación con la extracción directa (Högfors-Rönnholm et al., 2018).
La diversidad de métodos de extracción también genera variación en el rendimiento, calidad y tamaño del ADN obtenido ya que durante cada paso del procedimiento existen variantes. Tan solo en la lisis celular se puede incluir uno o los tres elementos siguientes: disrupción física, lisis química o lisis enzimática, a esto le sumamos que cada uno de los elementos anteriores puede realizarse de diversas maneras. Para la disrupción física se han descrito cuatro técnicas, congelación-descongelación, homogeneización de molinos de bolas, ultrasonido y molienda bajo nitrógeno líquido. En cuanto a las mezclas utilizadas en lisis química, se pueden categorizar en mezclas que contienen detergentes y cloruro de sodio (NaCl) y mezclas que contienen diversos tampones. En el caso de lisis enzimática se ha utilizado lizosima, proteinasa K, acromopeptidasa y pronasa E (Bruner et al., 2015; Wagner et al., 2015; Kashi, 2016; Mazziotti et al., 2018). Otro punto importante es el pH del suelo, ya que en suelos arcillosos que poseen un pH bajo, durante el paso de lisis celular se favorece la absorción del ADN en la superficie de la arcilla provocando bajos rendimientos de ADN, ahora se sabe que en algunos casos se pueden obtener mejores rendimientos de ADN utilizando nuevas formulaciones de buffer (solución tampón) (Narayan et al., 2016).
La capacidad de dispersión de los suelos puede variar cuando están en líquido debido a sus propiedades, en algunos no se genera una mezcla homogénea entre el suelo y el buffer, mientras que en otros las muestras absorben gran cantidad de buffer, por lo que es importante garantizar una buena dispersión del suelo para obtener mejores rendimientos de ADN (Ettenauer et al., 2012). Se ha observado que se obtiene mejor rendimiento de ADN al utilizar cantidades de muestras más grandes, lo cual puede ser explicado por la relación entre la cantidad de muestra utilizada y el volumen de buffer utilizado para la extracción, si la dispersión del suelo durante la lisis es problemática, una opción sería reducir el tamaño de la muestra y hacer múltiples extracciones de ADN para el análisis (Ettenauer et al., 2012). La composición de la comunidad bacteriana también puede influir en los rendimientos de ADN, Zhou et al. (1996) proponen que la baja lisis celular de algunos suelos puede deberse a un mayor contenido de células Gram-positivas y de estructuras más resistentes como endoesporas.
La extracción del ADN directamente de la muestra ambiental, también puede resultar en una coextracción de otros componentes como los ácidos húmicos y metales pesados, los cuales pueden interferir con las subsiguientes reacciones analíticas, tales como la modificación enzimática del ADN, el análisis por PCR, puede reducir la eficacia de transformación así como la especificidad en un proceso de hibridación (Técher et al., 2010; Hemmat-Jou et al., 2018). Además, no se recomienda la estimación de la concentración del ADN mediante análisis densitométrico en gel de agarosa teñido con bromuro de etidio (EtBr) debido a que los valores de absorbancia a 260 nm pueden no concordar entre si debido a la presencia de ácidos húmicos (Kuhn et al., 2017). Se han reportado algunos métodos para eliminar compuestos aromáticos como sustancias húmicas y polifenoles de las muestras del suelo utilizando resinas de intercambio catiónico y detergentes, polivinilpirrolidona (PVP), hidroxiapatita y carbono activado, pero la purificación compromete el rendimiento de ADN (Hemmat-Jou et al., 2018). Otros contaminantes como el cobre pueden estar presentes en las extracciones de ADN de suelos contaminados como lo demostró Sabrina R. Muller-Spitz y colaboradores (Mueller-Spitz et al., 2006), los cuales también interfieren con las modificaciones enzimáticas.
¿Cantidad o diversidad?
La evaluación de muchos protocolos para la extracción de ADN se basa frecuentemente en la cantidad y pureza, pero no debemos olvidar que una mayor cantidad de ADN no necesariamente equivale a una mayor diversidad de especies. Stach y colaboradores (2001) evaluaron distintos procedimientos de extracción de ADN observando que los métodos que produjeron las mayores cantidades de ADN no necesariamente presentaban la mayor diversidad de secuencias. En estudios donde se ha usado electroforésis en gel con gradiente de temperatura (TGGE) y electroforésis en gel con gradiente desnaturalizante (DGGE) de fragmentos de ARNr 16S se ha comparado la eficacia y la reproducibilidad de diferentes protocolos de extracción de ADN y han mostrado que los patrones de TGGE y DGGE varían dependiendo del método de lisis (Liu et al., 2016). Se recomienda el uso de varios métodos de extracción y purificación de ADN en el suelo, para estudiar la ecología de un hábitat, debido a que la evaluación de la calidad en la extracción de ADN basadas solo en la cantidad y la pureza son engañosas.
Tamizado de la biblioteca
La metagenómica funcional tiene dos limitaciones importantes: la primera de ellas es que la expresión del ADN metagenómico se debe llevar a cabo en un huésped diferente del cual proviene, lo que puede generar que no se detecten clones que posean secuencias de nuestro interés. Este sesgo provocado durante el tamizado de la biblioteca metagenómica por la cepa receptora se debe a diversos factores entre los que se incluyen: la preferencia del uso codones, los substratos faltantes y la incapacidad de reconocer elementos reguladores externos, incluidos los promotores y los sitios de unión ribosómica. Debido a estas consideraciones se ha propuesto que el tamizado de las bibliotecas metagenómicas se realice en diferentes hospederos (Bouhajja et al., 2016). Una suposición general es que el mejor huésped heterólogo probablemente sea un organismo similar o estrechamente relacionado con la fuente donadora del material genético, esta hipótesis no ha sido rigurosamente probada y existe evidencia empírica limitada para respaldar esta afirmación (Zhang et al., 2017). En varios estudios se ha informado que ninguna especie huésped revela la función de todos los genes en una biblioteca metagenómica, por lo que, para la selección de la cepa receptora se recomienda el uso de múltiples especies y tomar en consideración otras características que tengan que ver más con la facilidad de realizar el tamizado de la biblioteca metagenómica, por ejemplo utilizar una cepa receptora en la cual los genes clonados corrijan algún tipo de mutación que no existe en cepas utilizadas convencionalmente como cepas receptoras (Katzke et al., 2017).
La segunda limitante tiene que ver con los sustratos indicadores que se utilizan para la identificación de enzimas, los cuales pueden favorecer a las enzimas más similares al indicador. La utilización de más de un indicador para el tamizado de bibliotecas metagenómicas se ha utilizado con éxito como lo reporta Terrón (Terrón-González et al., 2016), quien obtuvo una gran diversidad de genes que codifican a la enzima dioxigenasa extradiol (Edos); los cuales no se atribuyeron a las subfamilias de Edos previamente definidas. Además, se encontró una relación entre el tipo de sustrato utilizado para el tamizado y el tipo de familia a la cual pertenecen estas Edos, por lo que se sugiere que existe un fuerte sesgo que depende del sustrato utilizado para el tamizado.
La gran diversidad de microorganismos desconocidos que habitan en el suelo representa un gran recurso inexplorado para ser usado tanto en la biotecnología como en la agricultura, por tal motivo desde el 2016 algunos países han desarrollado proyectos metagenómicos enfocados en el estudio del microbioma del suelo como Earth Microbiome Project (http://www.earthmicrobiome.org), TerraGenome (https://www.terragenome.org), the Brazilian Microbiome Project (https://www.brmicrobiome.org) y China soil Microbiome (http://english.issas.cas.cn/), con el fin de obtener respuestas a las preguntas: ¿Qué organismos están unidos en un hábitat de suelo? ¿Cómo la abundancia de los microorganismos cambia con el cambio de condiciones edáficas? y ¿Cómo los conjuntos microbianos interactúan e influyen unos con otros? (Nesme et al., 2016; Shao y Gao, 2018; Lacerda-Júnior et al., 2019).
METAGENÓMICA EN LA AGRICULTURA
Como sabemos, la mayoría de los alimentos que comemos a diario provienen del suelo, por lo que éste también desempeña una función importante en el bienestar de los humanos. En la búsqueda por satisfacer las necesidades alimenticias de una población en crecimiento, en los años 1950-1960 se implementó una alternativa denominada revolución verde la cual implicaba la producción masiva de productos agrícolas a través del uso de nuevas variedades de cultivos, grandes cantidades de fertilizantes químicos, pesticidas y mayor irrigación (Davis et al., 2019; Karunarathne et al., 2020). Con esta medida los rendimientos de cultivos se elevaron, sin embargo, con el tiempo estas técnicas provocaron daños al ambiente como la pérdida significativa de la biodiversidad microbiana del suelo y contaminación del aire y del agua (Vassilev et al., 2015). Este hecho a su vez ha generado preocupación sobre el impacto que pueden tener los fertilizantes químicos y los pesticidas en la salud humana, ya que algunos estudios reportan que el exceso de nitrógeno utilizado por el uso continuo de fertilizantes, conduce a la lixiviación del nitrato en el agua subterránea o superficial. A su vez, el nitrato al ser suministrado a las personas a través del agua y entrar en contacto con la flora intestinal se transforma en un compuesto tóxico, ya que la microflora intestinal convierte el nitrato en nitrito y el nitrito a su vez reacciona con la hemoglobina formando metahemoglobina, lo que evita que el oxígeno sea transportado a todo el cuerpo generando, la enfermedad de metahemoglobinemia y en algunos niños la de cianosis (Bryan y Ivy, 2015; Steffan et al. 2018). En cuanto a los pesticidas, también se ha reportado que su uso excesivo en la agricultura puede afectar a los humanos, ya que éstos al ingresar al cuerpo, viajan a través del torrente sanguíneo acumulándose en algunos órganos como el hígado produciendo la enfermedad del hígado graso, también pueden alterar el sistema inmune o pueden llegar hasta el sistema nervioso acumulándose en las neuronas desencadenando enfermedades neuronales como Alzheimer o Parkinson (Pellacani y Costa, 2018). Además de lo anterior, en el sistema reproductivo se ha observado que los pesticidas también pueden producir un desorden hormonal tanto en hombres como mujeres (Liu et al., 2016; Pellacani y Costa, 2018; He et al., 2020).
Una alternativa a la aplicación de fertilizantes y pesticidas es el uso de inoculantes, los cuales están hechos a base de bacterias promotoras del crecimiento de plantas (PGPR's), que en asociación con otros organismos benéficos promueven el crecimiento de las plantas y les confieren protección contra presiones ambientales como las heladas, la salinidad y el estrés por presencia de contaminantes xenobióticos (Baez-Rogelio et al., 2017), lo que se traduce en elevados rendimientos agrícolas, mejor salud y preservación del medio ambiente, ya que con esta alternativa se disminuye el uso de fertilizantes y pesticidas. Hasta el momento se ha observado que la inoculación de plantas con más de tres diferentes bacterias del tipo PGPR's proporcionan mayor beneficio a las plantas en comparación con las inoculadas con una o dos bacterias de este tipo, si bien algunas bacterias de los géneros Pseudomonas, Bacillus, Enterobacter, Klebsiella, Azobacter, Variovorax, Azospirillum y Serratiahis han sido comercializadas para ser usadas en el campo aún hace falta tener mayor conocimiento sobre microorganismos que sean benéficos al inocularlos con las plantas y que puedan coexistir en consorcio con otras bacterias (Vejan et al., 2016; Morales-García et al., 2019).
Es por esto que la metagenómica también se ha utlizado para analizar los suelos agrícolas con la finalidad de conocer las comunidades bacteriales endófitas que les permita mejorar la producción y salud de las plantas. Ejemplo de ello, es el estudio de la remolacha azucarera (Beta vulgaris L), en el cual se identificaron tanto las bacterias como los genes involucrados en la promoción del crecimiento de la remolacha, siendo Mesorhizobium, Bradyrhyzobium y Streptomices los géneros bacterianos más abundantes. Mientras que los genes mayormente expresados fueron el gen B-1,3-gluconasa, genes que codifican para quitinasa, el gen que codifica para el isocrorismato piruvato liasa y el gen que codifica para la quinoproteína glucosa deshidrogenasa. Los genes mencionados están involucrados en la supresión de enfermedades de las plantas ya que actúan como antifúngicos y antibióticos contra diversos microorganismos (Tsurumaru et al., 2015). Por otro lado, en Crocus sativus (azafrán) se determinó la diversidad fúngica asociada a la rizósfera y a la cormósfera en dos etapas diferentes de floración y durante la etapa latente, mostrando que el filo dominante en la rizósfera fue el Zigomycota mientras que en la cormósfera durante la etapa de floración el filo predominante fue Basidiomycota y en la etapa latente Zygomycota y Rhizopus fueron los filos más representativos. Sin embargo, tanto Ascomycota como Fusarium son filos presentes en ambas etapas siendo este último el hongo responsable de la pudrición del cormo en C. sativus (Ambardar et al., 2016).
Otro estudio metagenómico fue el realizado a la rizosfera de plantas de cebada y alfalfa sembradas en suelos contaminados con petróleo con la finalidad de conocer los microorganismos que desempeñan una función importante en la fitorremediación. De este estudio se determinó que tanto en la planta de alfalfa como en la de cebada los microorganismos más abundantes fueron Proteobacterias, Bacteroidetes, Nitrosomonas, Thermi y Cloroflexi, mientras que los microorganismos conocidos como degradadores fueron del género Alcanivorax y Aequorivita reafirmando su importancia como fitorremediadores presentes en estos dos diferentes tipos de plantas (Kumar et al., 2018). Por otra parte, en el 2015 se conformó el Consorcio Internacional de Microbiomas de Cítricos con la finalidad de conocer el microbioma de la rizósfera de los cítricos. Para este estudio se recolectaron 23 muestras de suelo y de la rizósfera de cítricos de ocho países productores que abarcan a seis continentes, estas muestras incluyeron siete tipos diferentes de suelo con variaciones en el pH que iban desde el rango de 5,2 a 8,8, seis diferentes tipos de clima y suelos con contenidos orgánicos variables en carbono, nitrógeno y fósforo. El estudio reveló que pese a las diferencias geográficas el microbioma central de los cítricos está conformado por Pseudomonas, Agrobacterium, Cupriavidus, Bradirhizobium, Rhizobium, Mesorhizobium, Burkholderia, Cellvibrio, Sphingomonas, Variovorax y Paraburkholderia. Los autores mencionan que este resultado podría sugerir una selección dirigida por la misma planta para rasgos particulares, además de que con este estudio se logra proporcionar información importante que permitirá aislar microorganismos para mejorar la producción y salud de las plantas (Xu et al., 2018). De igual modo el estudio realizado a la rizósfera de la planta Paspalum scrobiculatum L (comúnmente llamada kodo millet), del cual se obtiene un grano con valor nutricional importante en los países de Nepal, India, Filipinas, Indonesia, Vietnam, Tailandia y África reveló que el filo bacteriano más abundante en la rizosfera es el de las Actinobacterias y Firmicutes. Sin embargo, también hay predominancia de otro grupo de bacterias que no se pudieron clasificar pese a la gran cantidad de secuencias analizadas (Prabha et al., 2018).
Un estudio más, fue el realizado a la rizósfera de la planta del cacahuate (Arachis hypogaea L) cuya productividad se ve limitada por la sequía, el análisis se realizó bajo las condiciones de sequía y sin ella, para lo cual los resultados obtenidos demostraron que los filos de Actinobacterias, Proteobacterias, Saccharibacterias, Cloroflexi, Acidobacterias y Cianobacterias eran los dominantes en la rizosfera del cacahuate; sin embargo bajo las condiciones de sequía los filos de Actinobacterias y Acidobacterias aumentaron tanto en la etapa de plántulas como en la de vainas. En la etapa de floración los filos con mayor presencia fueron Cyanobacterias y Gemmatimonadetes concluyendo que el estudio aportó información importante sobre la relación que existe entre la sequía y los microorganismos presentes en la rizosfera por lo que en un futuro se podrían hacer mejoras a la resistencia ante la sequía modificando los microorganismos presentes en el suelo (Dai et al., 2019).
Muchos suelos agrícolas se encuentran contaminados con aceite de motor, es por ello que Salam et al. (2017) realizaron un análisis metagenómico de un suelo agrícola tropical en Nigeria con la finalidad de conocer la composición y función de la microbiota presente. El análisis demostró una abundancia de miembros del filo de Proteobacterias y Actinobacterias, en particular de la especie Geodermatophilus, cuya importancia radica en que cada uno de estos microorganismos poseen enzimas que son capaces de degradar hidrocarburos por lo que este estudio podría servir para diseñar mejores estrategias para la biorremediación de suelos contaminados con aceite de motor.
Por último el estudio realizado al cultivo del camote con diferentes tipos de suelo y condiciones climáticas, permitió conocer que en general no existe variación en la comunidad bacteriana con respecto a las condiciones usadas, sin embargo, si existe una predominancia del género bacteriano de Pseudomonas, por lo que los autores mencionan que si las bacterias predominantes encontradas tienen propiedades promotoras del crecimiento representarían un enorme potencial para ser utilizadas en los sistemas de producción agrícola (Puri et al., 2019).
En cuanto a la agricultura, la metagenómica nos permite utilizar el conocimiento adquirido en beneficio de la salud y economía, ya que cada análisis anteriormente mencionado nos proporciona información valiosa sobre microorganismos que podrían utilizarse para realizar inoculantes que incluyan más de tres microorganismos capaces de coexistir en consorcio con otras bacterias y potenciar tanto el rendimiento de las cosechas como la protección contra patógenos de plantas disminuyendo o eliminando, en un futuro, el uso de contaminantes.
DISCUSIÓN Y CONCLUSIONES
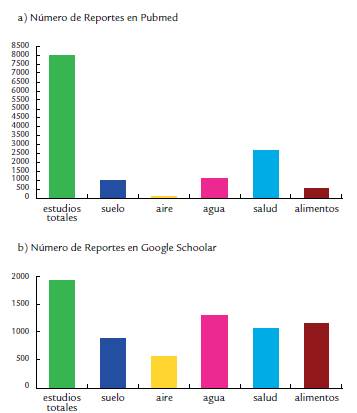
La Metagenómica surge gracias al desarrollo de nuevas técnicas de biología molecular y al avance tecnológico en los métodos de secuenciación, con esta herramienta se ha logrado dar un paso importante en la ciencia debido a que se pueden estudiar comunidades enteras de microorganismos en diversos ambientes como podemos observar en la Tabla suplementaria 1, hasta el día 23 de enero del 2020, de acuerdo a las referencias reportadas en PubMed existen 7956 análisis metagenómicos, de los cuales 78 son estudios realizados en aire, 962 son análisis metagenómicos realizados en alimentos, 1011 son estudios metagenómicos realizados a partir de muestras de suelo, 1114 corresponden a estudios metagenómicos realizados en agua y 2667 corresponden a estudios metagenómicos enfocados en la salud humana (Fig. 1a). Mientras que en el buscador Google Schoolar se reportan 1950 estudios metagenómicos, de los cuales, 876 son estudios realizados a partir de muestras de suelo, 534 son estudios realizados en aire, 1310 estudios corresponden a trabajos realizados en agua, 1060, son estudios enfocados en la salud humana y1160 son estudios realizados en alimentos (Fig. 1b), si bien existe una variación en cuanto a los número de trabajos reportados por los dos diferentes buscadores cada uno de los estudios se ha apoyado en el uso de diversas metodologías de secuenciación y han generado grandes aportes como el descubrimiento de nuevos genes, también nos han permitido tener un mejor conocimiento sobre las comunidades microbianas presentes en espacios físicos, alimentos o enfermedades específicas. Sin embargo, el suelo representa uno de los ambientes más complejos a estudiar, no solo por su composición y diversidad microbiológica, si no por ser la base de la agricultura para la obtención de alimentos. Aunado al desarrollo industrial, el suelo ha sido contaminado con el uso excesivo de pesticidas, fertilizantes y diversos desechos químicos como el petróleo, solventes o metales pesados; los cuales han generado mayor dificultad al momento de realizar estudios metagenómicos. Por lo que debemos tomar en cuenta el tipo de extracción de ADN que realizaremos si pretendemos conocer la diversidad microbiológica para no generar un sesgo en la identificación de estos microorganismos, o que se extraigan otros componentes como ácidos húmicos y metales pesados que nos interfieran con la modificación enzimática del ADN, el análisis de PCR o la eficacia de transformación. Si requerimos identificar enzimas, lo recomendable es usar diversos sustratos indicadores de la presencia de la o las enzimas a identificar y posteriormente amplificar los genes correspondientes, en caso de realizar librerías debemos cerciorarnos de que las cepas receptoras estén relacionadas con la fuente donadora del material genético o usar múltiples especies receptoras para poder detectar clonas con secuencias de nuestro de interés. A pesar de los problemas técnicos, con la metagenómica de suelos ha sido posible conocer genes que participan en procesos importantes para la salud de los humanos. Por ejemplo, la metagenómica aplicada al área de la agricultura no solo nos ha permitido descubrir y comprender la interacción de microorganismos en un nicho específico, sino también ha permitido usar ese conocimiento para mejorar nuestra calidad de vida, ya que si logramos generar inoculantes con más de tres microorganismos del tipo PGPR's, en un futuro no tan lejano se podrían aplicar para potenciar el crecimiento de cultivos agrícolas, hacer un mejor uso de los recursos naturales y evitar el desgaste del medio ambiente.