INTRODUCCIÓN
Las tecnologías en Secuenciación de Nueva Generación (NGS, por sus siglas en inglés), permiten estudiar regiones genómicas en muestras de ADN de diversos organismos, muchas de las cuales, para lograr suficiente fuerza en la señal, requieren amplificar el segmento de ADN mediante la Reacción en Cadena de la Polimerasa (PCR, por sus siglas en inglés) (Andreson et al., 2015). Esta técnica utiliza cebadores, secuencias cortas de nucleótidos que deben cumplir ciertas condiciones que permitan la obtención de producto confiable en el procedimiento de PCR a partir de un extracto genómico (Palumbi et al., 2002; Degen et al., 2006; Andreson et al., 2008).
El principal objetivo en el diseño de los cebadores es la especificidad que guardan (Andreson et al., 2008). Según Palumbi et al. (2002), se pueden construir cebadores degenerados, es decir, aquellos que en cierta posición pueden presentar distintos nucleótidos como resultado de la variación de los genomas a medida que se aumenta en el nivel taxonómico (Hebert et al., 2003), estos mejoran la probabilidad de amplificación cuando se desconoce la secuencia molde y pueden afectar la especificidad al hibridar en sitios distintos al deseado. Lo anterior, incrementa la posibilidad de fallar o generar falsos positivos (Andreson et al., 2008; Kõressaar et al., 2018) como consecuencia de la falta de fidelidad que presentan estas secuencias con respecto al ADN genómico de interés (Sambrook y Rusell, 2001). Esta situación se puede verificar mediante la utilidad de programas bioinformáticos (Andreson et al., 2015) como BLAST (Basic Local Alignament Search Tool) (Altschul et al., 1990), el cual permite establecer el grado de similitud de una secuencia de nucleótidos con respecto al genoma, además de identificar sus potenciales sitios de enlace (Johnson et al., 2008; Ye et al., 2012).
Por otro lado, en el Plan Nacional de Acción para la Conservación de Tiburones, Rayas y Quimeras de Colombia (PAN Tiburones-Colombia), se cuenta con representantes de la familia Potamotrygonidae (Peces elasmobranquios restringidos a ambientes dulceacuícolas) (Caldas et al., 2010). Dentro de esta, Potamotrygon magdalenae (Duméril, 1865) está catalogada como de alta importancia; por tanto, es relevante estudiar diferentes aspectos de su biología (Caldas et al., 2010; Lasso et al., 2016), incluidos aquellos que permitan profundizar en la genética con fines de conservación de estos organismos. Aunque se han realizado algunos estudios de diferente índole (Ramos-Socha y Grijalba-Bendeck, 2011; Ramírez y Davenport, 2013; Lasso et al., 2016), son escasos aquellos que comprenden aspectos de la biología molecular de esta especie.
En el sentido expuesto Garcia et al. (2015) y Renza-Millán et al. (2019), reportaron la obtención de secuencias de los genes mitocondriales (ADNmt) Citocromo C Oxidasa subunidad 1 (MT-CO1) y Citocromo b (MT-CYB), en muestras de diferentes especies de la familia Potamotrygonidae. En el primero de estos estudios, se presentaron reconstrucciones filéticas de representantes de la familia en tres países sudamericanos y evidenció la agrupación del clado Potamotrygon, entre los cuales se encuentran las especies P. magdalenae y P. motoro (Müller & Henle, 1841) (Garcia et al., 2015). Por su parte, el segundo estudio estuvo orientado al análisis de la estructura poblacional de P. motoro (Renza-Millán et al., 2019). Pese a lo anterior, estos estudios presentan información incongruente con respecto a la secuencia de los cebadores descritos, falta de homología con secuencias reportadas en GenBank, o la generación de distintos sitios de enlace potenciales dentro del genoma mitocondrial de P. magdalenae (datos no mostrados).
Debido a que poco se conoce sobre aspectos de la biología molecular de P. magdalenae y que puede resultar poco confiable la información de algunos cebadores descritos en la literatura que permitirían acceder al polimorfismo nucleotídico presente en el ADNmt, el principal objetivo del presente estudio fue amplificar regiones de genes mitocondriales en esta especie de Raya endémica de Colombia, a partir de cebadores diseñados bajo el Modelo de Pérdida de ADN (DNA-LM, por sus siglas en inglés) propuesto por Martínez- Pérez et al. (2007). En este modelo se plantea que, secuencias homologas pueden producir híbridos virtuales de genes, que por rearreglos y pérdidas de nucleótidos eliminables de la función final del neuropéptido, generan nuevos marcos de lectura abiertos virtuales (ORFv, por sus siglas en inglés) codificantes para estos genes.
MATERIALES Y MÉTODOS
Obtención de muestras y extracción de ADN genómico
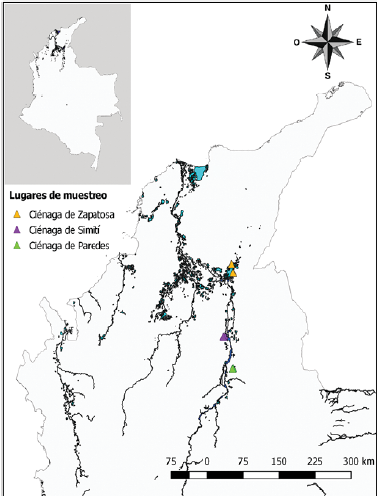
Obtuvimos muestras de tejido proveniente del disco de 20 individuos de P. magdalenae recolectadas entre febrero de 2017 y enero de 2018, después de faenas incidentales realizadas por pescadores de diferentes ciénagas dentro de la cuenca media del río Magdalena (Fig. 1). Extrajimos el ADN de los tejidos, previamente individualizados en etanol absoluto a 4 °C, acorde al protocolo que requiere Fenol-Cloroformo-Alcohol Isoamílico propuesto por Green y Sambrook (2012).
Diseño de cebadores
Realizamos búsquedas de secuencias disponibles en las bases de datos del National Center for Biotecnology Information (NCBI, por sus siglas en ingles), para los genes MT-CO1 y MT-CYB de organismos relacionados taxonómicamente con P. magdalenae hasta el nivel de la superfamilia Dayastoidea. A partir de las secuencias de genes completos y parciales (Apéndice 1), organizamos para cada nivel taxonómico alineamientos de múltiples secuencias (MSA) con el programa Kaling (Lassmann y Sonnhammer, 2005), de la siguiente manera: (a) la especie con mayor número de secuencias disponibles en NCBI; b) género; c) familia; y d) superfamilia. En todos los casos implementamos los parámetros: Gap open penalti (5), Gap extension penalty (0,2) y Terminal gaps penalties (0,1); que acorde a los preceptos del DNA-LM, permiten establecer las regiones conservadas de las especies pertenecientes a la superfamilia Dayastoidea. Las características físico-químicas de las secuencias cebadoras obtenidas fueron determinadas con OligoAnalyzer Tool 3.1 (https://eu.idtdna.com/calc/analyzer).
Estandarización y visualización de productos de PCR
Para la obtención de productos amplificados de los genes MT-CO1 y MT-CYB de P. magdalenae, estandarizamos la PCR en el equipo Eppendorf™ Mastercycler™ Nexus Thermal Cycler, a partir de los parámetros descritos por Degen et al. (2006). Los amplicones fueron separados por electroforesis en gel de agarosa al 1 %, comparados con marcador de peso molecular (MPM) de 1000 pb (Thermo Scientific), visualizados con el agente intercalante EZ-Vision (Amresco) y detectados con un transiluminador de luz ultravioleta UV TFX-35M (Life Tecnologies). Las imágenes fueron capturadas en un equipo foto-documentador CLS-MDOCBASIC (Cleaver Scientific).
Secuenciación de amplicones y análisis de correspondencia
Los amplicones generados fueron secuenciados, con los cebadores diseñados en el presente estudio y el kit Big Dye terminator v3.1 (Applied Biosystems®), en un analizador genético ABI PRISM™ 3130xl (Applied Biosystems®). Alineamos y editamos las secuencias obtenidas en el software BioEdit (Hall, 1999). Establecimos la correspondencia de las secuencias de los genes MT-CO1 y MT-CYB amplificados con BLAST (Altschul et al., 1990).
RESULTADOS
Diseñamos los cebadores con la información de 363 secuencias del gen MT-CO1 y 174 del gen MT-CYB, presentes en el NCBI para diferentes miembros de la superfamilia Dayastoidea. Del total de secuencias, utilizamos 75 Marcos de Lectura Abierto (ORF, por sus siglas en inglés) para el gen MT-CO1 y 41 ORF para el gen MT-CYB.
Los alineamientos obtenidos mostraron secuencias consenso en cada nivel taxonómico abordado para ambos genes de manera independiente. A partir de las secuencias consenso, seleccionamos dos regiones en cada uno de los genes considerados para P. magdalenae. Estas regiones fueron constantes entre los diferentes grupos taxonómicos y presentaron la mayor variación observada dentro de estos (Fig. 2). Con la información de los codones constantes y variables de las regiones seleccionadas, diseñamos dos cebadores, uno Forward y uno Reverse, para los genes MT-CO1 y MT-CYB de P. magdalenae, todos ellos con identidad superior al 92 % y valores variables del contenido GC entre el 40 y el 56 % (Tabla 1).

Figura 2 Alineamientos obtenidos en Kaling para el diseño de los cebadores. a. FPmagCOI531; b. RPmagCOI1432; c. FPmagCytb182; y d. RPmagCytb932. En todos los casos, destacado en amarillo: región donde se ubicó cada cebador; con: secuencia consenso; sp: especie; gepot: género Potamotrygon; fmpot: familia Potamotrygonidae; pmo: Potamotrygon motoro; ad-s: rayas de agua dulce y salobre; coi, cytb o cytbsn: genes CoI y Cytb; y lo números se refieren al número de secuencias usadas para la construcción de la secuencia consenso.
Tabla 1 Secuencias de cebadores propuestos para amplificar una región del gen MT-CO1 y MT-CYB de P. magdalenae.
| Cebador* | Secuencia | Identidad (%)** | Posición DNAmt*** | %G/C | Tm °C | Th °C |
|---|---|---|---|---|---|---|
| FPmagCOI531 | 5'-GCAATCTCTCAATACCAAACACCAC-3' | 96 | 6023 | 44 | 67,1 | 60,1 |
| RPmagCOI1432 | 5'-CGTTTT GATGCAAATGCTTCTCAGAG-3' | 96 | 6939 | 40 | 68,0 | 60,1 |
| FPmagCytb182 | 5'-CATCAGCCTTCTCCTCMATCGCAC-3' | 96 | 14584 | 54 | 69.3 | 58,0 |
| RPmagCytb932 | 5'-CGGAAGGTGAGGCTTCGTTGTTTGG -3' | 92 | 15358 | 56 | 71.2 | 58,0 |
Tm: Temperatura de hibridación calculada con OligoAnalyzer Tool; Th: temperatura de hibridación. * En todos los casos la nomenclatura está dada como sigue: F o R, dirección de síntesis del cebador; Pmag, Potamotrygon magdalenae; COI o CYTB, gen mitocondrial; y los números se refieren al primer nucleótido del cebador en el sentido 5' que se sintetiza dentro del gen. ** Porcentaje de identidad de cada cebador obtenido con BLAST. *** Corresponde al primer nucleótido sintetizado en sentido 5' del genoma mitocondrial de P. magdalenae.
La estandarización de productos amplificados por PCR mostró diferencias en el valor de las concentraciones de MgCl2, dNTPs, cebadores y Taq Polimerasa (Tabla 2), respecto a lo descrito por Degen et al. (2006). La reacción de PCR fue óptima a 94 °C por 5 minutos de denaturación inicial; 25 ciclos que incluyen denaturación a 94 °C, anheling (60.1 °C para MT-CO1 y 58 °C para MT-CYB) y extensión a 72 °C, por 30 segundos cada paso; y la extensión final a 72 °C por 7 minutos. El producto amplificado del gen MT-CO1 de P. magdalenae se estimó en 916 pb y 774 pb el del gen MT-CYB.
Tabla 2 Concentración de reactivos para amplificar por PCR fragmento de los genes MT-CO1 y MT-CYB de P. magdalenae a partir de los cebadores diseñados.
| Reactivos | Degen et al., (2006) | Presente estudio |
|---|---|---|
| H2O Milliq | vv | vv |
| Buffer | 1X | 1X |
| MgCl2 | 1.5mM | 2.25mM |
| dNTPs | 0.2mM | 0.8mM |
| Cebadores | 0.2μM | 1.25μM |
| Taq DNA Polymera-se (Recombinant) | 2U | 1U |
vv: volumen variable.
En promedio, el contenido nucleotídico de las secuencias MT-CO1 equivale a 26,2 % T, 26,8 % C, 28,5 % A y 15,5 % G, siendo más frecuente G (0,292), T (0,396) y C (0,444) en la primer, segunda y tercera posición de los codones, respectivamente. Para el caso de las secuencias MT-CYB, el contenido nucleotídico se estimó en 26,8 % T, 32,7 % C, 27,9 % A y 12,6 % G, siendo más frecuente C (0,281), T (0,423) y C (0,465) en la primera, segunda y tercera posición de los codones, respectivamente.
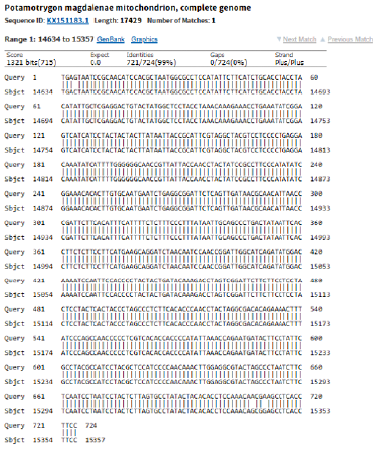
Los alineamientos en BLAST de las secuencias obtenidas a partir de los productos amplificados de los genes mitocondriales considerados, comparados con la secuencia del genoma mitocondrial completo KX151183 de P. magdalenae, evidenció identidad del 99 % (Fig. 3) para cada gen. Adicionalmente, dentro de la región de cobertura que presentan las secuencias obtenidas del gen MT-CO1, observamos entre dos y 22 sitios variantes, lo que posibilita el uso de este gen en futuros estudios genéticos poblacionales de la especie de raya P. magdalenae.
DISCUSIÓN
Las secuencias de cebadores diseñadas en este estudio para los genes MT-CO1 y MT-CYB aplicando DNA-LM, constituyen una herramienta que puede responder preguntas que exploren aspectos de la biología molecular de P. magdalenae. El modelo de pérdida de ADN permite comparar secuencias conservadas de genes presentes en diferentes taxa que guardan relación filética (Martínez et al., 2007), como ha sido demostrado en la evolución de neuropéptido de moluscos y otros metazoa (Jékely, 2013; De Oliveira et al., 2019). Este modelo fue propuesto y probado, inicialmente, en organismos invertebrados y plantea que la evolución de genes neuroendocrinos ocurrió a partir de la duplicación de un gen ancestral para un precursor neuropeptídico con distinto número de copias. Además, del movimiento de intrones y la pérdida de codones generan nuevos genes en especies filogenéticamente cercanas (Martínez et al., 2007); esto implica considerar que, las sustituciones aminoacídicas se conservan (Illegard et al., 2009; Cocco et al., 2018; Contreras-Moreira, 2018) y, por tanto, la incertidumbre disminuye (Lassman et al., 2008; Kõressaar et al., 2018). Bajo estos supuestos, la información de secuencias disponibles en bases de datos constituye un enfoque para el diseño de cebadores con sitios de enlace específicos que permitan incrementar la fidelidad de los resultados (Ye et al., 2012). Lo anterior fue demostrado con el diseño de sistemas de diagnóstico por RT-PCR en tiempo real para el virus de influenza A H1N1 (González-Barrios et al., 2017).
El alineamiento que realizamos con la estrategia MSA, permitió visualizar y establecer las posiciones variables y conservadas en secuencias que guardan identidad (Higgs y Attwood, 2005; Donkor et al., 2014). Lo anterior, posibilita un mejor entendimiento de la estructura, función y el cambio que tienen dichas secuencias en diferentes organismos, aspectos que se pueden aprovechar considerando diferentes enfoques bioinformáticos (Khodakov et al., 2016).
Precisamente, como parte de aprovechar los datos y las herramientas bioinformáticas disponibles, sumado a la aplicación de DNA-LM, planteamos una estrategia orientada al diseño de cebadores siguiendo las recomendaciones de rigor (Sambrook y Green, 2001; Degen et al., 2006; Contreras-Moreira, 2018), que disminuyeron la probabilidad de generar múltiples sitios de enlace durante la PCR (Andreson et al., 2015), lo que redundó en la amplificación de las regiones deseadas y evitó la presencia de amplicones inespecíficos.
La eficiencia y especificidad de la reacción de PCR se debe en principio al diseño de los cebadores, que, en conjunto con otros aditivos y cosolventes, constituyen el éxito de la reacción (Sambrook y Green, 2001). Dado que todas las secuencias de cebadores difieren, las variables físicas y químicas asociadas a estos también lo hacen y, en consecuencia, las condiciones de amplificación. De hecho, todos los aditivos y cosolventes que se utilizan en la reacción de PCR constituyen agentes inhibidores de esta (Sambrook y Green, 2001). Como consecuencia de esta situación, establecer las condiciones mínimas para obtener productos amplificados es un buen punto de partida, cuyo ajuste mejorará la calidad de las bandas y la confiabilidad de los resultados (Khodakov et al., 2016), en especial si llegase a requerir el uso de secuencias para analizar sitios variantes.
La herramienta BLAST (Altschul et al., 1990) encuentra la relación más alta entre la secuencia de consulta y los registros disponibles en las bases de datos del GenBank (Selzer et al., 2018). El uso de esta herramienta arrojó un valor de identidad > 99 % para las secuencias de los genes amplificados con los cebadores que diseñamos para MT-CO1 y MT-CYB de P. magdalenae, con respecto a las secuencias de esos genes inmersas en la secuencia genómica KX151183. Lo anterior permite establecer una alta relación entre secuencias de esta especie de Raya, que posibilita la cercanía u homología en términos biológicos (Donkor et al., 2014; Selzer et al., 2018). De hecho, análisis adicionales siguiendo la estrategia UPGMA implementada en el software MEGA X (Kumar et al., 2018), sugieren que la secuencia genómica KX151183 presenta más similitud con P. magdalenae muestreada en las Ciénaga de Paredes y Cimití, que con muestras obtenidas en la Ciénaga de Zapatosa (datos no mostrados), lo que permite hipotetizar que dicha secuencia guarda estrecha relación filética con las halladas a partir de los cebadores diseñados.
Cuando analizamos con BLAST las secuencias de cebadores aquí diseñadas, identificamos regiones que permiten la obtención de un único producto amplificable por PCR en cada gen de interés (Fig. 3). En los estudios realizados por Garcia et al. (2015) y Renza-Millan et al. (2019) se reportó la obtención de secuencias en organismos pertenecientes a la familia Potamotrygonidae. No obstante, al analizar con BLAST las secuencias de cebadores descritos en esos trabajos, encontramos que se forman múltiples fragmentos de tamaño variable que no guardan correspondencia con la secuencia de cada gen considerado. Este hallazgo genera incertidumbre frente a los amplicones obtenidos en P. magdalenae con los cebadores descritos por Garcia et al. (2015) y Renza-Millan et al. (2019), más si se tiene en cuenta que el mejor predictor en los fallos y falsos positivos de la PCR, es el incremento en el número de potenciales sitios de enlace que genere un cebador (Andreson et al., 2008; Kõressaar et al., 2018).
La identificación de diferentes sitios variables en secuencias del gen MT-CO1 en muestras de individuos de P. magdalenae, posibilita el uso de los cebadores aquí diseñados en posteriores estudios orientados a resolver preguntas de índole microevolutivo a nivel genético poblacional. Adicionalmente, destacamos que utilizamos cebadores heterólogos diseñados para amplificar el gen mitocondrial NADH deshidrogenasa 2 (ND2) de Aves (Hackett, 1996; Johnson y Sorenson, 1998), en experimentos cuyos resultados mostraron productos amplificados secuenciables o susceptibles de ser digeridos con endonucleasas de restricción en muestras de P. magdalenae (datos no mostrados). Lo anterior, se puede explicar ya que los cebadores diseñados para amplificar el gen ND2 incluyen secuencias de genes de tRNA (Metiona y Triptofano, respectivamente), estructuras altamente conservadas (López y Montoya, 2012) que deben reconocer los codones de los aminoácidos involucrados, por tanto, su secuencia de nucleótidos puede ser similar (Lang et al., 2012) entre distintos taxa.
CONCLUSIONES
Concluimos que siguiendo los preceptos de DNA-LM se diseñaron cebadores que generan productos amplificados de los genes mitocondriales Citocromo C Oxidasa 1 y Citocromo b de Potamotrygon magdalenae. Lo anterior posibilita la aplicación de DNA-LM en el diseño de cebadores de genes mitocondriales presentes en otros vertebrados distintos a Elasmobranquios. Además, los cebadores diseñados permitirán responder preguntas relacionadas con aspectos genéticos, filogenéticos y biogeográficos en esta especie de Raya de agua dulce.