INTRODUCCIÓN
La guanábana (Annona muricata L.) pertenece a la familia de las anonáceas (Linnaeus, 1753), cuyo centro de origen algunos autores lo presentan en América tropical (Nugraha et al., 2021). Agustín y Segura Ledesma (2014) realizaron un análisis sobre la conservación de anonáceas en México, reportando que la conservación de guanábana es in situ (huertas de traspatio) y ex situ (bancos de germoplasma) mediante la conservación de semillas.
Escobedo-López (2019) realizó un estudio de las principales zonas productoras de annonas en México, reportando que el germoplasma del género Annona está distribuido en áreas tropicales y subtropicales. En este sentido, Nayarit es el principal productor a nivel nacional con el 73 % de la producción total, concentrándose en los municipios de Compostela, Tepic y San Blas (SIAP, 2019). Sin embargo, existe escasa información respecto a la variabilidad genética de la guanábana debido principalmente al poco conocimiento de su genoma (Berumen-Varela et al., 2019). Por tanto, se necesita establecer un programa de mejoramiento genético para analizar la diversidad genética de guanábana mediante el uso marcadores moleculares, ya que permiten evaluar variaciones y polimorfismos en las secuencias del ADN.
Los marcadores moleculares se basan en el análisis de las diferencias en pequeñas secuencias de ADN entre individuos, siendo diversos, y de carácter dominante, favoreciendo la generación de datos multiloci o codominante que permite la identificación de homocigotos y heterocigotos (Canales-Delgadillo et al., 2015). Uno de los principales marcadores moleculares son los microsatélites (SSR), los cuales son de carácter codominante y permiten determinar las variaciones entre poblaciones e incluso entre especies (Vázquez et al., 2014). Presentan ventajas sobre otros marcadores debido a su elevado grado de polimorfismo, el tipo de segregación carácter codominante y una interpretación rápida (Carneiro et al., 2016). Otra de las ventajas que muestran los microsatélites, es que son marcadores homólogos para diferentes especies e incluso géneros, lo que permite hacer estudios comparativos entre éstos (Lassois et al., 2016). Asimismo, los marcadores conocidos como polimorfismo amplificado relacionado a la secuencia (SRAP) se basan en dos amplificaciones específicas que nos permiten obtener datos de carácter dominante (Li y Quiroz, 2001).
En una investigación sobre el análisis de la diversidad genética de algunas especies de Annonas, se analizaron 11 genotipos de saramuyo (Annona squamosa L.) mediante marcadores RAPD (Bharad et al., 2009), donde se encontró que cinco oligonucleótidos produjeron bandas reproducibles; además, se generaron un total de 26 marcadores, de los cuales 19 fueron polimórficos. El análisis de RAPD demostró que tres genotipos fueron difererentes y el resto fueron similares, agrupándose en tres grupos diferentes. Asimismo, Guimarães et al. (2013) analizaron 64 accesiones de Annona squamosa L. provenientes del norte de Minas Gerais, Brasil utilizando marcadores RAPD. Los autores reportaron 167 fragmentos, de los cuales 48 fueron polimórficos (28,7 %) encontrándose bajo porcentaje de polimorfismo, lo que indica un bajo nivel de variación genética entre las accesiones evaluadas.
Por otro lado, se han realizado estudios de diversidad genética de guanábana en diferentes países, como Indonesia, Venezuela, Brasil y la India, encontrando baja variabilidad genética entre cada una de las accesiones del mismo país (Brown et al., 2003; Suratman et al., 2015; Anuragi et al., 2016; Hasan et al., 2017; Berumen-Varela et al., 2019). En México, Jiménez-Zurita et al. (2016) realizaron una caracterización morfológica de tres selecciones de frutos de guanábana en Tepic, Nayarit. Los resultados de esta investigación, mostraron variabilidad entre las selecciones evaluadas, agrupándose en cuatro grupos a partir de los datos de la masa de la cáscara, pulpa y semilla, diámetro longitudinal y ecuatorial, luminosidad y cromaticidad, entre otras. Por otro lado, Talamantes et al. (2019) evaluaron la diversidad genética de 88 genotipos de guanábana de huertos comerciales de Compostela, Nayarit usando SRAP, concluyendo que existen niveles bajos a moderados de diversidad genética en guanábana, indicando vulnerabilidad a factores ambientales.
Como puede observarse, existen escasos estudios a nivel genético de los árboles de guanábana en México. Debido a la importancia económica que posee el fruto de éste en Nayarit, es importante generar el estudio y conocimiento de la diversidad genética para identificar ecotipos o fenotipos y así poder establecer un programa de mejoramiento genético para obtener variedades con características deseables para su comercialización. Por tanto, el objetivo de la presente investigación fue analizar la diversidad genética de guanábana de las principales zonas productoras de Nayarit, México.
MATERIALES Y MÉTODOS
Material vegetal
Se recolectó tejido foliar de 219 árboles de guanábana durante la temporada otoño-invierno 2016 y 2017 a partir de huertos ubicados en Compostela (Tonino, Las Varas uno, Las Varas dos, Chacala uno y Chacala dos), San Blas (El Cocodrilario, La Palma y Tecuitata) y Tepic (Trapichillo, V. Carranza y El Limón) sin daños físicos, libre de plagas y enfermedades.
Los árboles de guanábana de los huertos de Compostela, fueron propagados por semilla, y durante el estudio éstos tenían una edad entre los diez y 12 años (poblaciones de Las Varas y Chacala) y de 31 años para El Tonino. Por otro lado, se desconoce la edad de los árboles de guanábana de San Blas y Tepic, ya que éstos se encuentran a campo abierto o de traspatio, considerándolos como plantas silvestres; con excepción de los árboles establecidos en el ejido de Venustiano Carranza (Tepic), que fueron propagados por semilla, y tienen una edad de 20 años.
En seguida, el tejido foliar fue almacenado a -80 °C durante 48 h en un ultracongelador (Thermo Scientific Revco Value Series ULT1386-3-a41, USA) y después fue liofilizado (Free Zone 2.5, LABCONCO, Houston, Texas, USA), a 420 mBar, -46 °C, durante 48 h. Porteriormente, el tejido fue pulverizado por medio de un molino (KRUPS GX4100, Israel) y finalmente almacenado a temperatura ambiente hasta su uso.
Extracción de ADN
El ADN genómico de guanábana se extrajo a partir de 50 mg de tejido foliar de acuerdo a la metodología reportada por Lira-Ortiz et al. (2020). La concentración y calidad del ADN se determinó mediante un nanoespectrofotómetro Nanodrop 2000® (Thermo Scientific, 17 Waltham, MA, USA), a longitudes de onda de A260/280 nm y A260/230 nm así como por electroforesis en gel de agarosa al 1,0 %.
Marcadores microsatélites (SSR)
Se seleccionaron 22 marcadores microsatélites para el género Annona (Annona crassiflora y Annona cherimola Mill.) a partir de trabajos previamente reportados (Pereira , 2007; Escribano et al., 2008; Zonneveld et al., 2012) los cuales se muestran en el Anexo 1. La reacción de PCR consistió en 4 μL de 0.8x REDTaq ready MixTM PCR Reaction Mix R2523 (Sigma Aldrich Spruce Street, St. Louis, USA), 0.5 μL de cada marcador (0.5 mM), 2 |xL de ADN (12.5 ng/ μL) y 3 μL de agua libre de ADNasa para un volumen final de 10 μL. Las condiciones de PCR fueron las siguientes: a 94 °C por 5 min, seguido de 35 ciclos de 94 °C por 40 seg (desnaturalización), 55 °C por 40 seg y 72 °C por 1 min (extensión) con una extensión final de 72 °C por 10 min en un equipo Termociclador Prime Thermal Cycle (5PRIME/02, Cole-Parmer, Stone, Staffordshire, OSA, UK).
Marcadores SRAP
Se seleccionaron tres marcadores SRAP a partir de lo previamente reportado (Li y Quiroz, 2001; Ferriol et al., 2003; Alghamdi et al., 2012; Aneja et al., 2013) como se observa en el Cuadro 1. La reacción de PCR consistió en 2 μL de 0.8x REDTaq ready MixTM PCR Reaction Mix R2523 (Sigma Aldrich Spruce Street, St. Louis, USA), 0,25 μL de cada marcador (0,5 mM), 1 μL de ADN (12,5 ng/ μL) y 1,5 μL de agua libre de ADNasa para un volumen final de 5 μL. Las condiciones de la PCR fueron las siguientes: 94 °C por 5 min, seguido por cinco ciclos a 94 °C por 1 min, 35 °C 1 min y 72 °C durante 1 min, para desnaturalización, alineamiento y extensión, respectivamente. Posteriormente, se utilizaron 35 ciclos de 94 °C por 1 min para desnaturalización, 50 °C por 1 min para alineamiento y 72 °C por 1 min para extensión. La extensión final se llevó a cabo a 72 °C durante 10 min.
Geles de poliacrilamida por tinción de plata
El producto amplificado porPCR mediante los marcadores SSR y SRAP fue analizado en geles de poliacrilamida al 8 % a 250 V por 2 h. El revelado de los geles se llevó a cabo por tinción de plata mediante el método reportado por Sanguinetti et al. (1994).
Análisis de resultados
Los resultados obtenidos fueron evaluados mediante la elaboración de una matriz de datos binarios de ausencia (cero) y presencia (uno) de bandas. Se determinaron los parámetros de diversidad genética: Indice de Shannon (/) (PLA, 2006), Porcentaje de loci polimórfico (P), y Heterocigocidad esperada (He) (Nei, 1978), y análisis de componentes principales (PCA) mediante programa GenAlEx 6.5. La estructura genética fue evaluada mediante: análisis gráfico de agrupamiento por medio de la Agrupación promedio con media aritmética no ponderada (UPGMA), Distancias genéticas de Nei (D) (Nei y Feldman, 1972), Prueba de exactitud (Raymod et al., 1995), mediante el software TFPGA version 1.3 desarrollado por Miller (1997). El índice de diferenciación FST (Wright, 1965) se realizó mediante el software Arlequin version 3.5 (Excoffier y Lischer, 2010). Las fórmulas de los paramétros de diversidad genética así como los valores de identidad/distancia genética se encuentran en el Anexo 3 y 4, respectivamente. Además, se llevó a cabo un análisis bayesiano de asignación de individuos mediante el software STRUCTURE version 2.3.4 (Evanno et al., 2005).
RESULTADOS
SSR
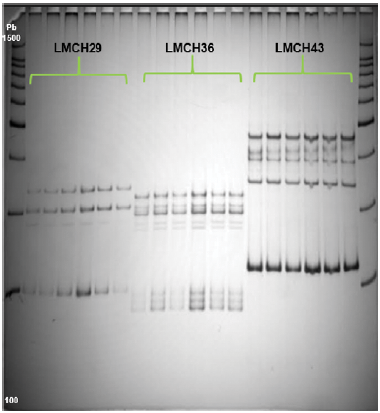
Con el fin de analizar la diversidad genética de guanábana en el estado de Nayarit, se utilizaron diferentes tipos de marcadores moleculares. Los marcadores SSR no mostraron polimorfismo en ninguna de las regiones analizadas (Fig. 1).
SRAP
En los marcadores SRAP se visualizó un patrón de bandas de116 loci polimórficos en un rango de diez a 500 pb. Además, se realizó una matriz de datos binomial, mediante cero (ausencia) y uno (presencia) de acuerdo a la presencia de bandas en el gel para obtener los parámetros de diversidad genética. En ese sentido, se obtuvo un promedio de porcentaje de loci polimórfico (P) de las tres zonas analizadas de 29,55 % Compostela presentó el promedio de P más alto (43,5 %), seguido de San Blas (20,7 %) y Tepic (15,2 %) (Cuadro 2).
Tabla 1 Marcadores SRAP utilizados para la identificación de polimorfismos en guanábana
| No. Marcador | Tipo de marcador | Secuencia del Primer |
|---|---|---|
| SRAP 1 | Forward (5'3') me3 | TGAGTCCAAACCGGAAT |
| Reverse (5'3') em12 | GACTGCGTACGAATTGTC | |
| SRAP 2 | Forward (5'3') me3 | TGAGTCCAAACCGGAAT |
| Reverse (5'3') em13 | GACTGCGTACGAATTGGT | |
| SRAP 3 | Forward (5'3') me4 | TGAGTCCAAACCGGACC |
| Reverse (5'3') em15 | GACTGCGTACGAATTCTG |
El índice de Shannon (/), refleja la heterogeneidad de una población sobre dos factores: el número de especies presentes y su abundancia relativa. De las tres zonas evaluadas, Compostela presentó los valores más altos de índice de Shannon (/), con un valor promedio de 0,127. Este valor es considerado bajo dentro de las poblaciones. Asimismo, San Blas y Tepic también presentaron bajos valores de / de 0,061 y 0,043, respectivamente. Por tanto, los resultados obtenidos en este estudio sugieren que la población es poco abundante en especies.
El coeficiente de heterocigocidad esperada (He) oscila entre valores de -1 a 1, valores negativos muestran heterocigocidad y positivos homocigocidad (Eguiarte, 2007). El promedio de He de las tres zonas evaluadas fue de 0,050, Compostela obtuvo un valor de He promedio de 0,074, seguido de San Blas con un valor de 0,036 y Tepic con un valor de 0,024. Por consiguiente, estos resultados indican que la guanábana de las tres zonas evaluadas son materiales clonados y por lo tanto, la variabilidad tiende a ser cero.
Como puede apreciarse, se obtuvieron bajos índices de diversidad genética (P, /, y He) en todas las zonas analizadas (Tabla 2). Compostela presentó los valores más altos en todos los índices analizados. Por otro lado, en esta investigación se realizó un análisis de varianza molecular AMOVA (Anexo 2) en el cual se observa que el mayor porcentaje de varianza se ubica dentro de las poblaciones al presentar un valor del 100 %. Esto indica que dentro de las poblaciones existe una alta diferencia genética. Asimismo, no se encontraron diferencias entre las zonas, así como entre poblaciones dentro de las zonas.
Tabla 2 Índices de diversidad genética de guanábana
| Zona | Población | P | I | He |
|---|---|---|---|---|
| Compostela | Tonino | 31.9 | 0.78 | 0.043 |
| Las Varas uno | 52.59 | 0.152 | 0.088 | |
| Las Varas dos | 45.69 | 0.125 | 0.072 | |
| Chacala uno | 53.45 | 0.125 | 0.091 | |
| Chacala dos | 33.62 | 0.157 | 0.076 | |
| 43.45 ± 4.57 % | 0.127 | 0.074 | ||
| San Blas | El Cocodrilario | 12.93 | 0.05 | 0.031 |
| La Palma | 31.03 | 0.074 | 0.04 | |
| Tecuitata | 18.1 | 0.06 | 0.036 | |
| 20.68 ± 5.38 | 0.061 | 0.036 | ||
| Tepic | Trapichillo | 16.38 | 0.045 | 0.025 |
| V. Carranza | 12.93 | 0.041 | 0.024 | |
| El Limón | 16.38 | 0.043 | 0.024 | |
| 15.23 ± 1.15 % | 0.043 | 0.024 |
P: Porcentaje de loci polimórfico, He: Heterocigocidad esperada, /: índice de Shannon
Se realizó una prueba de exactitud con una significancia de p < 0,05. El valor q de la prueba de exactitud para Nayarit fue de 0,0710 ± 0,0175, valor bajo a nivel zona y poblacional. Un valor análogo a q es el valor estadístico de FST de Wright, el cual fue calculado, obteniendo un valor de 0,00.
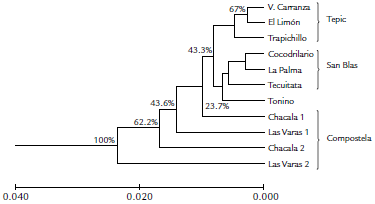
Por otro lado, se realizó un dendograma por el método de agrupación UPGMA (Fig. 2) a partir de la matriz de datos de Identidad/Distancia (Anexo 2) mediante el método de Nei y Feldman (1972). Los resultados mostraron la formación de dos grupos principales, el primero conformado solamente por la población de Las Varas dos y el segundo con las diez poblaciones restantes (Chacal dos, Las Varas uno, Chacala uno, V. Carranza, El Limón, Trapichillo, El Cocodrilario, La Palma y Tecuitata). Sin embargo, la distancia genética entre las poblaciones fue bajo (menores a 0,050), lo que demuestra una escasa diferenciación genética. La distancia genética entre los subgrupos formados (grupo dos) fue de 0,0080.
Además, se realizó un gráfico de PCA basado en los datos obtenidos (Fig. 3a). Se identificaron tres grupos, el primer y segundo grupo (segundo y tercer cuadrante) formados por las poblaciones que pertenecen a la zona de Compostela y el tercer grupo, formado por las zonas de Tepic, San Blas y algunos huertos de las poblaciones de Compostela (cuarto cuadrante). El primer y segundo grupo se encontró más alejado en comparación con el tercer grupo. Esto coincide con lo obtenido por el dendograma de distancias genéticas, donde se observó que la población de Las Varas dos (Compostela) formó un grupo independiente. Estos resultados indican que algunos huertos de la zona de Compostela poseen mínimas diferencias genéticas en comparación con los de Tepic y San Blas.
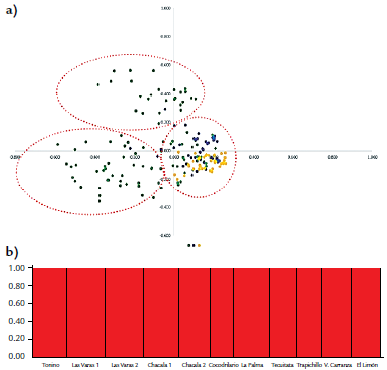
Figura. 3 a). Análisis de PCA de las zonas productoras de guanábana. Amarillo: Tepic; Azul: San Blas; Compostela: Verde. Línea Punteada Roja: Agrupamiento. b) Representación gráfica de la estructura genética de 11 poblaciones de guanábana donde los grupos genéticos (K) están indicados por colores sobre el eje X.
Por otro lado, el tercer grupo formado por PCA coincide con lo observado por el dendograma de distancias genéticas, en el cual se observó que la mayoría de las poblaciones de Tepic, Compostela y San Blas se encuentran agrupadas. Sin embargo, se observó una baja dispersión de todas las poblaciones analizadas en los cuatro cuadrantes. Estos resultados sugieren que las poblaciones de guanábana se agruparon de acuerdo a la zona geográfica analizada.
Por último, se realizó un analisis de estructura genética mediante un análisis bayesiano utilizando el programa STRUCTURE version 2.3.4 (Fig. 3b) con las siguientes condiciones: 250 000 cadenas de Markov Monte Carlo (MCMC), 50 000 réplicas y diez repeticiones para cada K (Evanno et al., 2005). Los resultados del análisis bayesiano mostraron poblaciones puras, lo que significa que todos los individuos presentan las mismas características y pertenecen a una misma población filogenética (Fig. 3b). Esto soporta lo encontrado con los demás análisis.
DISCUSIÓN SSR
Los marcadores microsatélites se caracterizan por ser altamente polimórficos; el alto grado de polimorfismo es debido a una elevada tasa de mutación, que se atribuye a eventos de inserción y deleción durante la replicación del ADN (Carneiro et al., 2016). En anonáceas se han utilizado los marcadores microsatelitales para analizar la diversidad genética, obteniéndose un alto grado de polimorfismo entre las poblaciones analizadas, siendo una herramienta poderosa para la caracterización genética (Pereira, 2007; Gwinner et al., 2016). En una investigación con 23 accesiones de chirimoya, provenientes de diferentes áreas geográficas (Bolivia, Colombia, Ecuador, México, Perú, Portugal y España), se reportaron 52 nuevos marcadores SSR, concluyendo que la mayoría son transferibles para otras especies de anonas (Escribano et al., 2008). Por otro lado, Anuragi et al. (2016), analizaron la diversidad genética mediante el uso de SSR y RAPDs en 20 genotipos de annonas provenientes de cinco especies de annonas (A. cherimola, A. reticulata, A. muricata, A. atemoya, A.squamosa) de distintos lugares de India, encontrando un alto polimorfismo (93 %) entre los genotipos de las especies de annonas analizadas. Sin embargo, como puede observarse, estos resultados difieren con lo obtenido en este estudio debido a que no se observó polimorfismo (Fig. 1), esto posiblemente puede ser atribuido a la temperatura y humedad relativa que predomina en cada región respecto al país de estudio. De acuerdo con lo reportado por Escribano et al. (2007) sus investigaciones en México se llevaron a cabo en regiones de clima templado subhúmedo (Coatepec harinas y Michoacán) con temperaturas promedio de 21° Cy38%de humedad relativa. Azcón-Bieto et al. (2008) reportó que la polinización se ve afectada por las características ambientales, sobre todo por la temperatura y la humedad relativa; el efecto de la temperatura puede ser indirecto, al alterar la actividad de las abejas (principales polinizadoras de los árboles frutales); las cuales permiten la libre polinización mediante el transporte del polen a una temperatura media de 20-22 °C. Para el caso de la región productora de guanábana en Nayarit, la temperatura oscila entre los 28 a 34 °C (en algunos meses la temperatura es hasta 37 °C) y una humedad relativa en la escala de 85 al 92 %; lo que dificulta la libre polinización, que para fines comerciales se realiza manualmente, por lo que se puede dar la autogamia debido a la polinización directa y con ello, la consanguinidad.
SRAP
Los SRAP se utilizaron para detectar variaciones entre poblaciones ya que es un sistema de marcadores basado en PCR, capaz de detectar polimorfismos en secuencias de codificación, a partir de la identificación de las bandas más informativas (Luan et al., 2015).
Salazar et al. (2010), analizaron la diversidad genética en 14 poblaciones de A. squamosa de diferentes zonas socioeconómicas de Yucatán y Quintana Roo, reportando que los marcadores enzimáticos fueron polimórficos con tres y cuatro alelos. Además, encontraron valores de varianza del 85 % entre los genotipos dentro de las poblaciones, 12 % dentro de una misma zona socioeconómica y 2 % entre zonas socioeconómicas, demostrando que la estructura genética no está relacionada con la distancia entre huertos en las entidades geográficas (Salazar et al., 2010). Talamantes et al. (2019) analizaron cinco poblaciones de guanábana del municipio de Compostela, Nayarit, mediante marcadores SRAP, encontrando una variación del 88 % dentro de las poblaciones. Los resultados de las investigaciones mencionadas coinciden con los obtenidos en este estudio, donde se observó un alto porcentaje de varianza dentro de las poblaciones analizadas.
Por otro lado, se encontraron valores mínimos (cero) en la prueba de exactitud para el valor de q y la prueba FST de Wright, lo que indica que las frecuencias alélicas son iguales en todas las poblaciones y posee un tipo de reproducción endogámica. Éstos resultados son significativamente menores a los encontrados en otros huertos de A. muricata, ya que Brown et al. (2003), reportaron valores de 0,5038 en árboles de guanábana de Venezuela utilizando marcadores RAPDs, lo que refleja mayores distancias genéticas entre sus poblaciones.
Salazar et al. (2010) reportaron que la edad de los árboles puede ser un factor que influya en la homogeneidad genética. Asimismo, Parraguirre Lezama et al. (2002) reportan que la diferenciación genética de las poblaciones establecidas en la región puede estar asociada al tamaño de ésta.
Otro factor importante que pudiera estar involucrado en la baja diversidad genética es el polen. En A. squamosa la dispersión de polen es a corta distancia y los principales polinizadores son coleópteros por lo que sugiere que el flujo de polen es limitado (Endres et al., 2007; Salazar et al., 2010). En ese sentido, para poder obtener una gran cantidad de frutos, los agricultores realizan como actividad principal la dispersión del polen entre los huertos, lo que conlleva a una baja diferenciación entre poblaciones. Sin embargo, se han reportado resultados que difieren con los obtenidos en este estudio. Brisibe et al. (2014) analizaron la diversidad genética de 42 accesiones de guanábana de diferentes zonas de Nigeria mediante marcadores RAPD; los resultados de esa investigación mostraron altos niveles de diversidad genética y grados de divergencia, agrupándose las poblaciones en más de tres grupos.
Asimismo, Sánchez et al. (2018) analizaron la diversidad genética con base a la producción de 71 genotipos de guanábana durante los ciclos de producción del 2000 al 2016 en Colombia. En ese estudio encontraron que los genotipos formaron cinco grupos de acuerdo a su rendimiento. Además, demostraron que algunos genotipos tienen el potencial para generar una población con un alto rendimiento y variabilidad genética.
Con base a los resultados obtenidos en esta investigación, la baja diversidad genética de guanábana puede estar asociada a factores bióticos, abióticos y/o epigenéticos. Sin embargo, la falta de datos genómicos en plantas no-modelo ha dificultado conocer la asociación genética entre el genotipo, fenotipo y los factores previamente mencionados (Richards et al., 2017).
Otra posible explicación a los resultados encontrados puede estar asociada a que la mayoría de los árboles provienen de la propagación por semilla, lo que conlleva a un ancestro en común. Guimarães et al. (2013) encontraron baja variabilidad genética entre accesiones provenientes del norte de Minas Gerais, Brasil. Los autores atribuyen este resultado a que los árboles de los huertos pudieron estar establecidos a partir de semillas de frutos del mercado central de la ciudad de Monte Claros, por lo que probablemente los árboles provinieran de un ancestro en común, lo que explicaría la baja diversidad genética entre las accesiones. Por lo tanto, la diversidad genética puede variar como resultado de múltiples factores como la dispersión del polen, condiciones ambientales y tipo de propagación.