Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Revista Salud Uninorte
versión impresa ISSN 0120-5552
Salud, Barranquilla vol.28 no.1 Barranquilla ene/jun. 2012
ARTÍCULO ORIGINAL / ORIGINAL ARTICLE
Detección molecular y tipificación del virus dengue por RT-PCR y PCR anidada usando oligonucleótidos mejorados
Molecular detection and typing of dengue virus by RT-PCR and nested PCR using degenerated oligonucleotides
José A. Usme-Ciro1,2, Alba M. Gómez-Castañeda2, Juan C. Gallego-Gómez1,2
1 Viral Vector Core and Gene Therapy, Área de Neurobiología Celular y Molecular, Grupo de Neurociencias, Sede de Investigación Universitaria – SIU, Facultad de Medicina, Universidad de Antioquia. Medellín (Colombia).
2 Grupo de Medicina Molecular y de Translación, Facultad de Medicina, Universidad de Antioquia. Medellín (Colombia).
Correspondencia: Juan Carlos Gallego Gómez. Departamento de Microbiología y Parasitología, Facultad de Medicina, Universidad de Antioquia. Edificio Histórico Manuel Uribe Ángel. Cra. 51D nº 62 -29, Piso 3. Teléfono: 57 4 + 219 6014 / 02. Fax: 57 4 + 219 6444. Medellín (Colombia). juanc.gallegomez@gmail.com
Fecha de recepción: 25 de agosto de 2011
Fecha de aceptación: 14 de noviembre de 2011
Resumen
Objetivos: Modificar los oligonucleótidos comúnmente utilizados para la detección y tipificación del virus dengue y evaluar su desempeño sobre ARNs provenientes de muestras clínicas.
Materiales y métodos: A partir del alineamiento de secuencias disponibles en el GenBank para la región C-prM/M se determinó la variabilidad genética dentro de cada serotipo del virus dengue, lo cual permitió realizar modificaciones a los oligonucleótidos convencionalmente usados en la tipificación. Tales modificaciones incluyeron la adición y eliminación de nucleótidos en el extremo 3', teniendo en cuenta la posición del codón, así como la inclusión de sitios degenerados en posiciones altamente variables. Los oligonucleótidos se evaluaron a diferentes concentraciones sobre ARNs extraídos a partir de cultivos celulares infectados y posteriormente de sueros de pacientes con diagnóstico probable de dengue.
Resultados: Los oligonucleótidos amplificaron específicamente cada serotipo del virus dengue, tanto desde sobrenadantes de cultivo como desde muestras clínicas en prueba piloto.
Conclusiones: El rediseño de los oligonucleótidos consideró la variabilidad genética acumulada en más de 15 años, mostrándose experimentalmente su valor y utilidad en la detección y tipificación del virus dengue.
Palabras clave: RT-PCR, PCR anidada, oligonucleótidos degenerados, tipificación, virus dengue, detección molecular.
Abstract
Objective: To modify the commonly used primers for detecting and typing of dengue viruses and evaluate their performance on RNAs derived from clinical samples.
Materials and methods: To determine the genetic variability within each dengue virus serotype, sequences of C-prM/M region available on GenBank were aligned allowing modifications to the primers conventionally used for virus typing. Modifications include nucleotide insertion and deletion in the 3' end taking into account the codon position, and also the inclusion of degenerated sites in highly variable positions. Primers were evaluated at different concentrations on extracted RNAs from infected cell cultures and subsequently from sera of probable dengue diagnosed patients.
Results: All primers specifically amplified each dengue virus serotype from cell culture supernatants and clinical samples in a pilot test.
Conclusions: The re-designed primers took into account the accumulated variability during more than 15 years, experimentally showing their value and utility for detecting and typing of dengue viruses.
Keywords: RT-PCR, nested PCR, degenerated primers, typing, dengue virus, molecular detection.
INTRODUCCIÓN
Los países en desarrollo ubicados en el cinturón tropical tenemos una realidad muy paradójica, pues vivimos en sitios privilegiados por la alta biodiversidad y recursos naturales pero al mismo tiempo esta abundancia es también válida para los agentes biológicos que producen y/o están implicados en enfermedades.
Los trópicos podrían ser considerados como los laboratorios evolutivos del mundo, y de forma experimental hemos sido testigos de la rápida diversificación del virus dengue (DENV) a nivel local (1,2).
El dengue es una enfermedad viral transmitida por artrópodos predominante en países tropicales y subtropicales, con un estimado de 50 - 100 millones de casos y alrededor de 25 000 muertes cada año a nivel mundial (3). Esta enfermedad es causada por el DENV, un miembro del género Flavivirus perteneciente a la familia Flaviviridae, conformada por virus envueltos de estructura icosahédrica con genoma ARN de cadena sencilla y polaridad positiva (ssARN+) 4.
Existen 4 serotipos de DENV estrechamente relacionados: DENV-1, -2, -3 y -4, los cuales ocasionan diferentes grados de severidad de la enfermedad (5, 6), haciendo necesaria su identificación no solo para garantizar un tratamiento oportuno previo al inicio de las complicaciones de la enfermedad, sino también útil en los programas de vigilancia epidemiológica. El DENV es el agente causal del dengue clásico (DF), así como de las manifestaciones severas: el dengue hemorrágico (DHF) y el síndrome de choque por dengue (DSS) (6), ahora consideradas dengue grave (7).
Teniendo en cuenta la importancia de la potenciación dependiente de anticuerpos (ADE), como resultado de infecciones secundarias con serotipos heterólogos (8 - 10), se hace necesario mantener y reforzar los programas de vigilancia epidemiológica que permitan conocer la dinámica de circulación de los distintos serotipos y, por ende, la historia de retos inmunológicos de las diferentes poblaciones que las predisponen a efectos adversos ante la entrada de un serotipo diferente; de esta forma es posible anticipar las medidas de control y tratamiento a la población afectada o potencialmente vulnerable ante el ingreso de un nuevo serotipo.
Desafortunadamente, el aislamiento viral no permite realizar un diagnóstico en la fase aguda de la enfermedad, debido a que requiere más de 7 días para la obtención de los resultados (11-12). Por otro lado, aunque las pruebas serológicas permiten obtener datos en menor tiempo, su uso se limita a la fase convaleciente y depende de la capacidad de respuesta del sistema inmune del individuo y el tiempo de infección para poder obtener títulos detectables de anticuerpos (13-14), siendo aún muy difícil serotipificar el DENV debido a la posibilidad de reacción cruzada (15).
Debido principalmente a la infraestructura, equipamiento y personal requerido para la implementación de técnicas moleculares, el diagnóstico de dengue se realiza principalmente a partir de pruebas serológicas y aislamiento viral en cultivos celulares, técnicas que proveen información cuando los pacientes ya han pasado el cuadro clínico, limitándose su relevancia a la vigilancia epidemiológica y estudios virológicos posteriores. Existe, por lo tanto, una necesidad urgente de implementar métodos de diagnóstico molecular para uso temprano en la enfermedad, conducentes a la adopción de medidas adecuadas para el tratamiento y manejo del paciente.
En países como Colombia, donde la investigación y adquisición de nueva tecnología han cobrado importancia, el uso de técnicas moleculares se convierte en una alternativa al alcance no solo para el diagnóstico temprano de la enfermedad sino también para análisis evolutivos y epidemiológicos (1,2, 16-19), derivando en nuevas estrategias de prevención y control. Las técnicas de RT- PCR y PCR convencional, si bien requieren personal capacitado y adquisición de equipos para su implementación en el laboratorio clínico, son una excelente alternativa de detección y tipificación del DENV, debido a que permiten obtener resultados en pocas horas, siendo su sensibilidad mayor al detectar pocas copias del genoma viral en el suero del paciente, inclusive provenientes de partículas virales inactivas o unidas a anticuerpos (20).
La Organización Panamericana de la Salud (PAHO/OPS) recomienda el uso de la técnica de Lanciotti y colaboradores (21), o su variante de Harris y colaboradores (22), para la detección y tipificación del DENV en toda su área de influencia (11). El funcionamiento de estos oligonucleótidos ha sido avalado durante años; sin embargo, debido a que el DENV está constituido por un genoma de tipo ARN, con una ARN polimerasa dependiente de ARN carente de actividad correctora (23), la acumulación de cambios, y por ende, la desactualización de los oligonucleótidos es relativamente rápida, lo cual conlleva un incremento en la probabilidad de generar resultados falsos negativos por ineficiencia en la hibridación de oligonucleótidos con las secuencias de las cepas circulantes. En vista de ello, este estudio pretende hacer uso de las herramientas bioinformáticas actuales, así como de la creciente disponibilidad de secuencias de las cepas de DENV que circulan mundialmente, para el rediseño racional de los oligonucleótidos convencionalmente usados en diagnóstico y tipificación del DENV (21); así como la estandarización y evaluación de los oligonucleótidos sobre muestras de ARN obtenidas a partir de sobrenadantes de cultivos celulares infectados y en prueba piloto sobre sueros de pacientes con diagnóstico clínico de dengue.
MATERIALES Y MÉTODOS
Selección de secuencias y procesamiento bioinformático
Por medio del programa ClustalW (24) integrado al paquete MEGA versión 3.1 (25) se realizó el alineamiento de la región C-PrM/M de las secuencias del DENV correspondientes a los cuatro serotipos, disponibles en la base de datos GenBank (http://www.ncbi.nlm.nih.gov/Genbank/) (Release 155.0), para posteriormente rediseñar los oligonucleótidos y mejorarlos teóricamente por comparación con los obtenidos por el programa HintPCR versión 3.0 (26).
A partir del alineamiento se analizaron las posiciones de los oligonucleótidos previamente reportados (21) en el contexto de toda la variabilidad presente y se verificaron diferentes parámetros: la posición de codón en el nucleótido del extremo 3' de cada oligonucleótido, respecto al marco de lectura de la poliproteína del DENV, la frecuencia de cada polimorfismo en el conjunto total de secuencias analizadas, Tm (temperatura de melting), estructuras secundarias y especificidad de secuencia.
Muestras de virus
Para realizar los ensayos de estandarización de la RT-PCR y PCR anidada se utilizaron cepas correspondientes a los cuatro serotipos del DENV (M1, M2, M3 y M4), previamente crecidas en células de mosquito Aedes albopictus clon C6/36 (27).
Muestras clínicas
Se incluyeron en el análisis un total de 15 muestras clínicas (sueros) de pacientes con diagnóstico presuntivo de dengue, las cuales fueron colectadas en el área metropolitana del Valle de Aburrá entre 2005 y 2007. Por otro lado, para soportar los hallazgos en la tipificación molecular de DENV a partir de ARN obtenido directamente de sueros y ARN extraído de sobrenadantes de cultivos de C6/36 inoculados con dichos sueros se realizó intento de aislamiento viral e inmunofluorescencia a cada una de las 15 muestras en células C6/36 con 500 µL de una dilución 1/10 de cada muestra en D-MEM y un periodo de incubación de 9 días. Finalmente, los fragmentos correspondientes a muestras positivas fueron purificados y secuenciados para corroborar nuevamente la correspondencia de los serotipos.
Extracción de ARN viral
El ARN viral de las cepas de DENV-1 a -4 y de las muestras de suero de pacientes fue extraído usando el estuche comercial QIAamp Viral RNA minikit (Qiagen®), siguiendo las recomendaciones del fabricante. El ARN extraído fue almacenado a -20ºC hasta su posterior utilización.
RT-PCR y PCR anidada
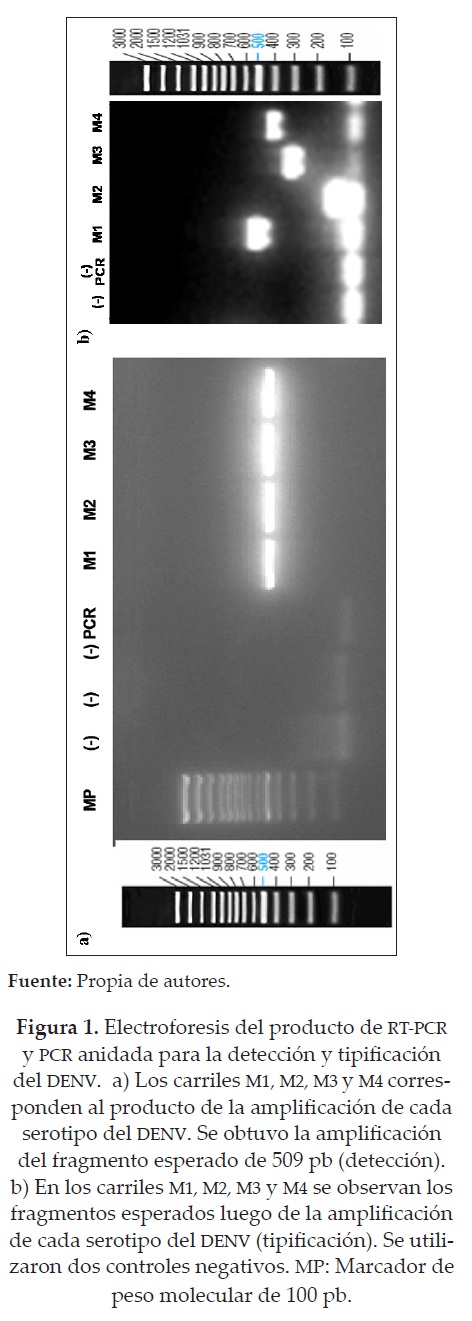
La RT-PCR dirigida a la región C-PrM/M se llevó a cabo utilizando el estuche QIAGEN One-Step RT-PCR kit (Qiagen®) y los oligonucleótidos D1_Modif y D2_Modif, diseñados en este estudio (Tabla 1). El perfil térmico consistió en un paso de transcripción reversa a 41ºC por 45 minutos, un paso de activación de la Hot Start Taq DNA polimerasa a 94ºC por 15 minutos, 40 ciclos (94ºC por 30 segundos para desnaturalización, 58ºC por 1 minuto para la hibridación de oligonucleótidos y 72ºC por 30 segundos para la extensión), seguido por una extensión final a 72ºC por 5 minutos. El volumen final de la reacción fue de 25 µL. El producto amplificado corresponde a un fragmento de 509 pb.
El producto de la RT-PCR fue diluido (1/100) con agua libre de nucleasas, con el fin de disminuir las amplificaciones inespecíficas por exceso de ADN durante la PCR anidada. La PCR anidada fue realizada a partir de 1 µL de la dilución, utilizando los oligonucleótidos D1_Modif, TS1_Modif, TS2_Modif, TS3_Modif y TS4_Modif. El volumen final de la reacción fue 25 µL. Se utilizaron 2.5 µL de tampón Taq DNA polimerasa 10x, 1 mM de MgCl2, 0.4 mM de cada dNTP, 10 pmoles de cada oligonucleótido y 1.25 unidades de la enzima Taq DNA polimerasa (Fermentas®). El perfil térmico fue de 2 minutos a 94ºC, 40 ciclos (30 segundos a 94ºC, 1 minuto a 55ºC y 30 segundos a 72ºC), finalizando con una extensión a 72ºC durante 5 minutos.
Análisis de los productos amplificados
Para la visualización del producto de amplificación del DENV (detección - RT-PCR) y los productos correspondientes a cada uno de sus serotipos (tipificación - PCR anidada) se prepararon geles de agarosa al 1.5% en tampón TAE, utilizando 10 µL del producto obtenido de la RT-PCR y/o PCR anidada y bromuro de etidio para la visualización. Para diferenciar las bandas obtenidas en la RT-PCR (509 pb) y PCR anidada: DENV-1: 487 pb; DENV-2: 122 pb; DENV-3: 299 pb y DENV-4: 391 pb, se utilizó un marcador de peso molecular de 100 pb (Fermentas®).
RESULTADOS
Rediseño de oligonucleótidos
En el diseño de los oligonucleótidos D1_Modif y D2_Modif para la RT-PCR se utilizaron 795 y 910 secuencias obtenidas del GenBank respectivamente. Para el diseño de los oligonucleótidos específicos de serotipo se utilizaron 85, 151, 335 y 201 secuencias para TS1_Modif, TS2_Modif, TS3_Modif y TS4_Modif respectivamente.
El oligonucleótido D2_Modif fue modificado en el extremo 3', de tal forma que coincidiera con la primera posición de codón.
El nucleótido en el extremo 3` de los oligonucleótidos TS1, TS3 y TS4 coincidió con la tercera posición de codón, la cual presenta una alta variabilidad reflejada en los alineamientos (datos no mostrados). Sus versiones aquí modificadas, TS1_Modif, TS3_Modif y TS4_Modif, por su parte, coinciden con la primera o segunda posición de codón, garantizando su hibridación con una mayor probabilidad. El oligonucleótido TS2_Modif fue modificado, de tal modo que su nucleótido en el extremo 3` coincidiera con la segunda posición de codón, la cual presenta significativamente menor variabilidad, debido a que una sustitución a este nivel conllevaría a un cambio de aminoácido con importantes implicaciones en el fenotipo del virus.
Una vez se obtuvo el rediseño preliminar de los oligonucleótidos basado en la variabilidad genética reflejada en los alineamientos, se procedió a la evaluación in silico de los mismos, teniendo en cuenta algunos parámetros termodinámicos importantes (energía libre, formación de homodímeros, heterodímeros, estructuras secundarias y temperatura de melting), y se mejoró teóricamente el diseño por comparación con los candidatos hallados por medio del paquete HintPCR versión 3.0 (26) para la misma región, usando siempre una secuencia (serotipo) como blanco y las secuencias de los otros tres serotipos utilizadas simultáneamente durante la búsqueda para excluir candidatos presentes en al menos una de ellas. En la tabla 1 se muestran en detalle las modificaciones en cada uno de los oligonucleótidos luego de todo el proceso de optimización.
Detección específica de cada serotipo de DENV
Con el propósito de demostrar que la especificad de cada oligonucleótido no ha sido afectada con el rediseño de los mismos, se realizó la RT-PCR (figura 1a) y PCR anidada del DENV (figura 1b) usando ARNs extraídos de cultivos celulares infectados. Cuando se utilizó como molde para la PCR anidada el producto de la RT-PCR sin dilución se obtuvo una amplificación con diferentes bandas inespecíficas, que podrían interferir con la correcta serotipificación del DENV (datos no mostrados). Sin embargo, al hacer dilución 1/100 del producto de la RT-PCR, bajo las condiciones normales de reacción, la técnica mejoró considerablemente y se logró tipificar correctamente cada uno de los serotipos del DENV (figura 1b).
Al excluir el oligonucleótido TS1_Modif de la reacción se obtuvo el resultado esperado, con una ausencia de amplificación correspondiente al DENV-1, lo cual indica que los oligonucleótidos TS2_Modif, TS3_Modif y TS4_Modif presentes en la mezcla de PCR anidada no hibridan inespecíficamente en la secuencia de DENV-1 (figura 2a). Resultados similares fueron obtenidos cuando se eliminaron los oligonucleótidos TS2_Modif, TS3_Modif y TS4_Modif, con ausencia de amplificación del serotipo correspondiente (figura 2b-d). Los controles negativos no mostraron bandas de amplificación.
Optimización de la cantidad de oligonucleótidos
La cantidad final de los oligonucleótidos fue modificada con el propósito de disminuir su exceso al final de la reacción, y así evitar interferencia en la detección de la banda correspondiente al serotipo DENV-2 (122 pb). Para ello se trabajó con tres diferentes cantidades de cada oligonucleótido (18,75 pmoles, 10 pmoles y 5 pmoles (correspondientes a 0,75 µM, 0,4 µM y 0,2 µM respectivamente). En todas las concentraciones se obtuvo amplificación específica de cada serotipo viral (figura 3a-c), sin embargo, el uso de 10 pmoles permitió eliminar el exceso de oligonucleótidos sin sacrificar o disminuir la sensibilidad de la técnica (figura 3b).
Tipificación de DENV en muestras clínicas
De las 15 muestras con diagnóstico presuntivo de dengue incluidas en la prueba piloto, 2 pudieron ser detectadas directamente a partir del producto de RT-PCR (#13 y #27) (figura 4a), 3 permitieron la tipificación de DENV luego de la PCR anidada, correspondiendo a los serotipos DENV-2 (#26) y -3 (#13 y #27) (figura 4b). Adicionalmente, 3 muestras positivas pudieron ser detectadas únicamente al evaluar la técnica con ARNs obtenidos a partir de sobrenadantes de células C6/36 inoculadas con los sueros, correspondiendo a los serotipos DENV-2 (#1291 y #28) y -3 (#25) (datos no mostrados). La correcta tipificación fue confirmada por inmunofluorescencia utilizando anticuerpos específicos para cada serotipo y mediante secuenciación (datos no mostrados).
DISCUSIÓN
En este estudio se muestra el rediseño de los oligonucleótidos para la detección y tipificación del DENV con el propósito de aumentar la cobertura en la detección, de acuerdo con la variabilidad que han acumulado las cepas durante un periodo de más de 15 años. Los oligonucleótidos reportados por Lanciotti y colaboradores (21) actualmente son recomendados por la OPS, y su desempeño en muestras clínicas ha demostrado su efectividad (28, 29).
Aunque se han desarrollado diferentes técnicas con el mismo propósito, estas se basan en el uso de sondas, fluoróforos y equipos de PCR en tiempo real (30-38), los cuales aún no están disponibles en muchos laboratorios de salud pública. Por lo tanto, técnicas simples como la PCR convencional continuarán demostrando su utilidad, sobre todo en países en desarrollo, donde acceder a equipos de última tecnología puede ser difícil.
Estudios anteriores han realizado modificaciones a los oligonucleótidos utilizados convencionalmente (22, 30, 39), logrando incluso la detección y tipificación en el mismo tubo de reacción y disminuyendo, por ende, la probabilidad de contaminación (22); sin embargo, se hicieron necesarias modificaciones adicionales que permitieran adaptarlos a las secuencias actualmente circulantes a nivel mundial.
Los oligonucleótidos D1_Modif y D2_Modif mostraron un buen desempeño luego de realizarles las modificaciones correspondientes. La presencia de una única banda en el producto de la RT-PCR de muestras de los cuatro serotipos es una clara evidencia de que la especificidad se mantiene a pesar de las degeneraciones (figura 1a). El extremo 3' de los oligonucleótidos de D1_Modif y D2_Modif coincide con la segunda y primera posición de codón respectivamente, por lo cual, los frecuentes cambios en la tercera posición de codón en las diferentes cepas no alterarían su hibridación y amplificación.
El extremo 3' de los oligonucleótidos específicos de serotipo TS1, TS3 y TS4 descritos previamente (21) coincide con la tercera posición de codón, mientras que TS2 hibrida en la primera posición de codón. Aproximadamente, el 30% de los cambios en la tercera posición de codón son no sinónimos, cambios en la primera posición son no sinónimos en el 96% de los casos y en la segunda posición de codón los cambios son siempre no sinónimos, llevando, por ende, a un cambio en el aminoácido codificado (40).
Los cambios de aminoácidos pueden tener un efecto en las propiedades de las proteínas (fenotipo) y pueden estar sujetos a presión selectiva, lo que conlleva restricciones al cambio. Aunque la región utilizada (C-PrM/M) se considera conservada, es una región codificante con un comportamiento típico, presentando alta variabilidad en sitios sinónimos como son las terceras posiciones de codón. El gen de la cápside, así como gran parte del genoma de DENV, está sujeto a una fuerte selección negativa (purificadora) (41), reflejada en pocos cambios en la secuencia de aminoácidos, sin que esto impida la ocurrencia de cambios sinónimos en la secuencia de nucleótidos. Los nucleótidos en segundas posiciones de codón (y en menor medida en primeras posiciones) permanecen relativamente constantes a través del tiempo, lo cual puede ser evidenciado en los alineamientos de cualquier región codificante del genoma viral.
Los oligonucleótidos fueron modificados de tal forma que el extremo 3' coincidiera con la segunda o primera posición de codón, con respecto al marco de lectura, logrando, de este modo, disminuir la probabilidad de incompatibilidad con secuencias de cepas que posean alta variabilidad, representada principalmente en la tercera posición de codón; de esta forma, el extremo 3' de los oligonucleótidos TS1_Modif, TS2_Modif y TS4_Modif corresponde a la segunda posición de codón, quedando para TS3_Modif en la primera posición de codón por razones de estabilidad del oligonucleótido.
Como se esperaba, las modificaciones realizadas en estos oligonucleótidos no alteraron su desempeño en la serotipificación del DENV, logrando amplificar específicamente la región de cada serotipo sin presentar reacciones cruzadas entre ellos que conlleven a hibridación de alguno de los oligonucleótidos con dos o más serotipos (figura 2a-d).
Debido a que el tamaño esperado del producto amplificado correspondiente a DENV-2 es de 122 pb, la optimización de la cantidad de oligonucleótidos en la reacción es un factor decisivo, ya que la presencia de una banda a este nivel podría ser confundida con un exceso de los mismos y/o la formación de dímeros. La concentración final de oligonucleótidos de 0,4 µM (10 pmoles) permitió obtener una buena amplificación de la región C-prM/M del DENV, facilitando la identificación de la banda correspondiente a DENV-2. La correcta detección y tipificación del DENV utilizando 5 pmoles de cada oligonucleótido garantiza la utilización de 10 pmoles sin llevar al límite la técnica.
Por otro lado, el desempeño de la técnica en la prueba piloto, al utilizar ARNs provenientes de sueros, es similar al obtenido en el proceso de estandarización, validando, por ende, su utilidad en el diagnóstico de la infección por el DENV. El incremento en la sensibilidad, al realizar la RT-PCR con ARNs obtenidos a partir de sobrenadantes de cultivos inoculados con las muestras clínicas, es acorde con el proceso de amplificación que pueden sufrir los virus presentes en la muestra luego de su inoculación en la línea celular permisiva C6/36. La detección de 2 serotipos (DENV-2 y -3) en muestras clínicas de una misma área geográfica y tiempo es de gran relevancia si se tiene en cuenta que las infecciones secundarias con serotipos heterólogos podrían ser responsables de la mayoría de casos severos (8-10). Este resultado es esperado en países hiperendémicos como Colombia, donde es frecuente la cocirculación de serotipos en gran parte del territorio (1, 2,41 - 43).
Teóricamente, el rediseño tiene en cuenta la variabilidad acumulada en los últimos 15 años y la evaluación experimental demuestra su correcto funcionamiento sobre muestras clínicas. En estudios posteriores, sin embargo, el uso de cepas altamente variables (pertenecientes a los diferentes genotipos dentro de cada serotipo) permitirá determinar su ventaja en la detección y serotipificación respecto a los oligonucleótidos usados convencionalmente. De igual forma, se abre una nueva oportunidad para la implementación de la técnica sobre muestras de mosquitos como complemento a los programas de vigilancia entomológica con miras a determinar las zonas de mayor prioridad para las fumigaciones e instauración de programas de prevención (44).
A pesar de las limitaciones en cuanto al número de muestras clínicas analizadas, la información obtenida con este trabajo deja ver la importancia de la evaluación y seguimiento constante de las técnicas moleculares, más aun cuando se trata de identificación de virus ARN, cuyas rápidas tasas de evolución pueden llevar a la disminución de la eficiencia de dichas técnicas diagnósticas. Adicionalmente, se convierte en un modelo del diseño e implementación de técnicas similares para cualquier otro tipo de virus, principalmente con genomas ARN.
Para 2010, en Colombia el Sistema de Vigilancia Nacional (SIVIGILA) reportó un total de 157 152 casos probables de dengue, de los cuales 9482 casos fueron clasificados como dengue grave (42), de acuerdo con la nueva clasificación de casos avalada por la OMS (7). Esta situación, sumada a la identificación de al menos dos serotipos en casi todo el territorio colombiano, hace necesaria la intensificación de acciones de vigilancia epidemiológica, donde el uso de herramientas moleculares de rápida detección y tipificación sería de rápida y oportuna ayuda para la puesta en marcha de planes de contingencia, dirigidos a organizar los servicios de salud y brindar una atención adecuada al paciente con dengue.
Agradecimientos
A la Dra. Elizabeth Hunsperger (Centers for Disease Control and Prevention, Puerto Rico) por la donación de anticuerpos monoclonales para cada serotipo de DENV.
Conflicto de intereses: Ninguno.
Financiación: Este trabajo fue financiado por el Instituto Colombiano para el Desarrollo de la Ciencia y la Tecnología Francisco José de Caldas –Colciencias– mediante los proyectos 11150416336, 111540820511, 11150418079 y 111549326198.
REFERENCIAS
(1) Méndez JA, Usme-Ciro JA, Domingo C, Rey GJ, Sánchez JA, Tenorio A, Gallego-Gómez JC. Phylogenetic history demonstrates two different lineages of dengue type 1 virus in Colombia. Virology journal 2010; 7: 226. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=20836894 [ Links ]
(2) Usme-Ciro JA, Méndez JA, Tenorio A, Rey GJ, Domingo C, Gallego-Gómez JC. Simultaneous circulation of genotypes I and III of dengue virus 3 in Colombia. Virology journal 2008; 5: 101. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=18764951 [ Links ]
(3) Gubler DJ. Dengue/dengue haemorrhagic fever: history and current status. Novartis Foundation symposium 2006; 277: 3 -16; discussion 16-22, 71-13, 251-253. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=17319151 [ Links ]
(4) Lindenbach BD, Thiel H-J, Rice CM. Flaviviridae: The Viruses and Their Replication. In: Knipe DM, Howley PM, eds. Fields Virology. 5th ed. Philadelphia: Wolkers Kluwer/ Lippincott Williams and Wilkins; 2007. p. 1101-1152. [ Links ]
(5) Balmaseda A, Hammond SN, Pérez L, Téllez Y, Saborio SI, Mercado JC, Cuadra R et al. Serotype-specific differences in clinical manifestations of dengue. The American journal of tropical medicine and hygiene 2006; 74: 449- 456. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=16525106 [ Links ]
(6) Gubler DJ. Dengue and dengue hemorrhagic fever. Clinical microbiology reviews 1998; 11: 480-496. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=9665979 [ Links ]
(7) WHO. Dengue: guidelines for diagnosis, treatment, prevention and control. New edition. Geneva: World Health Organization; 2009. [ Links ]
(8) Goncalvez AP, Engle RE, St Claire M, Purcell RH, Lai CJ. Monoclonal antibody-mediated enhancement of dengue virus infection in vitro and in vivo and strategies for prevention. Proceedings of the National Academy of Sciences of the United States of America 2007; 104: 9422-9427. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=17517625 [ Links ]
(9) Halstead SB. Observations related to pathogenesis of dengue hemorrhagic fever. VI. Hypotheses and discussion. The Yale journal of biology and medicine 1970; 42: 350-362. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=5419208 [ Links ]
(10) Kliks SC, Nimmanitya S, Nisalak A, Burke DS. Evidence that maternal dengue antibodies are important in the development of dengue hemorrhagic fever in infants. The American journal of tropical medicine and hygiene 1988; 38: 411-419. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=3354774 [ Links ]
(11) Balmaseda A, Matute ML, Mira PL. Manual de Procedimientos de Técnicas para el Diagnóstico del Dengue. Nicaragua: Organización Panamericana de la Salud (OPS); 2002. [ Links ]
(12) Teles FR, Prazeres DM, Lima-Filho JL. Trends in dengue diagnosis. Reviews in medical virology 2005; 15: 287-302. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=15672450 [ Links ]
(13) Schwartz E, Mileguir F, Grossman Z, Mendelson E. Evaluation of ELISA-based sero-diagnosis of dengue fever in travelers. J Clin Virol 2000; 19: 169-173. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=11090753 [ Links ]
(14) Takasaki T, Nawa M, Yamada KI, Harada M, Takeda A, Kurane I. Evaluation of dengue IgM detection tests using sera from patients with autoimmune diseases. Journal of virological methods 2002; 102: 61-66. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=11879693 [ Links ]
(15) Soler M. [Laboratory diagnosis to dengue virus infections]. Acta científica venezolana 1998; 49 (Supl 1): 25-32. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=10030051 [ Links ]
(16) Gallego-Gómez JC, Rey GJ, Usme-Ciro JA, Méndez JA, Gómez-Castañeda AM, Domingo C, Tenorio A. Epidemiología Molecular del Virus Dengue en Colombia 1977-2004 y Vigilancia de Genotipos Circulantes 2005- 2006. Acta Biol Col 2006; 11: 158-159. [ Links ]
(17) Méndez JA, Usme-Ciro JA, Rey GJ, Gallego-Gómez JC, Domingo C, Tenorio A. Genotipificación de cepas colombianas del Virus Dengue tipo 3. Acta Biol Col 2006; 11: 161. [ Links ]
(18) Méndez JA, Bernal MP, Calvache D, Boshell J. Genotipificación y análisis filogenético de cepas colombianas del virus dengue tipo 2. NOVA 2003; 1: 37-43. [ Links ]
(19) Ospina MC, Díaz FJ, Osorio JE. Prolonged co-circulation of two distinct Dengue virus Type 3 lineages in the hyperendemic area of Medellin, Colombia. The American journal of tropical medicine and hygiene 2010; 83: 672-678. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=20810837 [ Links ]
(20) Duebel V, Laille M, Hugnot JP, Chungue E, Guesdon JL, Drouet MT, Bassot S et al. Identication of Dengue Sequences by Genomic Amplification: Rapid Diagnosis of Dengue Virus Serotypes in Peripheral Blood. Journal of virological methods 1990; 30: 41-54. [ Links ]
(21) Lanciotti RS, Calisher CH, Gubler DJ, Chang GJ, Vorndam AV. Rapid detection and typing of dengue viruses from clinical samples by using reverse transcriptase-polymerase chain reaction. Journal of clinical microbiology 1992; 30: 545-551. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=1372617 [ Links ]
(22) Harris E, Roberts TG, Smith L, Selle J, Kramer LD, Valle S, Sandoval E et al. Typing of dengue viruses in clinical specimens and mosquitoes by single-tube multiplex reverse transcriptase PCR. Journal of clinical microbiology 1998; 36: 2634-2639. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=9705406 [ Links ]
(23) Steinhauer DA, Domingo E, Holland JJ. Lack of evidence for proofreading mechanisms associated with an RNA virus polymerase. Gene 1992; 122: 281-288. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=1336756 [ Links ]
(24) Thompson JD, Higgins DG, Gibson TJ. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic acids research 1994; 22: 4673-4680. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=7984417 [ Links ]
(25) Kumar S, Tamura K, Nei M. MEGA3: Integrated software for Molecular Evolutionary Genetics Analysis and sequence alignment. Briefings in bioinformatics 2004; 5: 150-163. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=15260895 [ Links ]
(26) Dopazo J, Rodriguez A, Saiz JC, Sobrino F. Design of primers for PCR amplification of highly variable genomes. Comput Appl Biosci 1993; 9: 123-125. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=8386978 [ Links ]
(27) Igarashi A. Isolation of a Singh's Aedes albopictus cell clone sensitive to Dengue and Chikungunya viruses. J Gen Virol 1978; 40: 531-544. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=690610 [ Links ]
(28) Camacho DE, Álvarez M, Rodríguez-Henríquez F, de Quintana M, Soler M, Chiarello A, Sierra G et al. [Laboratory diagnosis of dengue virus infections in Aragua State, Venezuela: October 1997-December 1998]. Investigación clínica 2003; 44: 91-103. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=12822553 [ Links ]
(29) Rosario D, Álvarez M, Díaz J, Contreras R, Rodriguez R, Vázquez S, Guzmán MG. [Polymerase chain reaction for rapid detection and serotyping of dengue virus in clinical samples]. Revista panamericana de salud publica = Pan American journal of public health 1998; 4: 1-5. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=9734221 [ Links ]
(30) Chien LJ, Liao TL, Shu PY, Huang JH, Gubler DJ, Chang GJ. Development of real-time reverse transcriptase PCR assays to detect and serotype dengue viruses. Journal of clinical microbiology 2006; 44: 1295-1304. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=16597854 [ Links ]
(31) Kong YY, Thay CH, Tin TC, Devi S. Rapid detection, serotyping and quantitation of dengue viruses by TaqMan real-time one-step RT-PCR. Journal of virological methods 2006; 138: 123-130. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=17000012 [ Links ]
(32) Lai YL, Chung YK, Tan HC, Yap HF, Yap G, Ooi EE, Ng LC. Cost-effective real-time reverse transcriptase PCR (RT-PCR) to screen for Dengue virus followed by rapid single-tube multiplex RT-PCR for serotyping of the virus. Journal of clinical microbiology 2007; 45: 935-941. http://www.ncbi.nlm.nih.gov/entrez/ query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=17215345 [ Links ]
(33) Munoz-Jordan JL, Collins CS, Vergne E, Santiago GA, Petersen L, Sun W, Linnen JM. Highly sensitive detection of dengue virus nucleic acid in samples from clinically ill patients. Journal of clinical microbiology 2009; 47: 927-931. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=19225099 [ Links ]
(34) Naze F, Le Roux K, Schuffenecker I, Zeller H, Staikowsky F, Grivard P, Michault A et al. Simultaneous detection and quantitation of Chikungunya, dengue and West Nile viruses by multiplex RT-PCR assays and dengue virus typing using high resolution melting. Journal of virological methods 2009; 162: 1-7. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=19773088 [ Links ]
(35) Shu PY, Chang SF, Kuo YC, Yueh YY, Chien LJ, Sue CL, Lin TH et al. Development of group- and serotype-specific one-step SYBR green I-based real-time reverse transcription- PCR assay for dengue virus. Journal of clinical microbiology 2003; 41: 2408-2416. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=12791857 [ Links ]
(36) Leparc-Goffart I, Baragatti M, Temmam S, Tuiskunen A, Moureau G, Charrel R, de Lamballerie X. Development and validation of real-time one-step reverse transcription- PCR for the detection and typing of dengue viruses. J Clin Virol 2009 ; 45: 61-66. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=19345140 [ Links ]
(37) Das S, Pingle MR, Muñoz-Jordan J, Rundell MS, Rondini S, Granger K, Chang GJ et al. Detection and serotyping of dengue virus in serum samples by multiplex reverse transcriptase PCR-ligase detection reaction assay. Journal of clinical microbiology 2008; 46: 3276- 3284. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=18685000 [ Links ]
(38) Lo CL, Yip SP, Cheng PK, To TS, Lim WW, Leung PH. One-step rapid reverse transcription- PCR assay for detecting and typing dengue viruses with GC tail and induced fluorescence resonance energy transfer techniques for melting temperature and color multiplexing. Clinical chemistry 2007; 53: 594-599. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=17317885 [ Links ]
(39) Seah CL, Chow VT, Tan HC, Can YC. Rapid, single-step RT-PCR typing of dengue viruses using five NS3 gene primers. Journal of virological methods 1995; 51: 193-200. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=7738139 [ Links ]
(40) Vandamme AM. Basic Concepts of Molecular Evolution. In: Salemi M, Vandamme AM, eds. The Phylogenetics Handbook: A Practical Approach to DNA and Protein Phylogeny. Cambridge: Cambridge University Press; 2003. p. 1-23. [ Links ]
(41) Usme-Ciro JA. Patrón evolutivo y filogeográfico del virus dengue tipo 1 y 2 en Colombia. [Tesis de Maestría]. Instituto de Biología. Medellín: Universidad de Antioquia; 2008. [ Links ]
(42) INS. Boletín de Vigilancia Epidemia por Dengue en Colombia. Boletín 48. ed. Bogotá: Instituto Nacional de Salud; 2011. [ Links ]
(43) Méndez JA, Usme-Ciro JA, Domingo C, Rey GJ, Sanchez JA, Tenorio A & Gallego-Gómez JC. Phylogenetic reconstruction of dengue virus type 2 in Colombia. Virology Journal 2012; 9:64. http://www.ncbi.nlm.nih.gov/pubmed/22405440/. [ Links ]
(44) Costa CA, Santos IG, Barbosa Mda G. Detection and typing of dengue viruses in Aedes aegypti (Diptera: Culicidae) in the City of Manaus, State of Amazonas. Revista da Sociedade Brasileira de Medicina Tropical 2009; 42: 677-681. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&dopt=Citation&list_uids=20209354 [ Links ]