Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Salud Uninorte
Print version ISSN 0120-5552
Salud, Barranquilla vol.29 no.2 Barranquilla May/Aug. 2013
Artículo de revisión / REVIEW ARTICLE
Aplicaciones e inconvenientes de la técnica Hibridación in situ Fluorescente (FISH) en la identificación de microorganismos
Applications and inconvenient of Fluorescence in situ hybridization technique (FISH) in the identification of microorganism
Raúl Rodríguez Martínez1, Gina Suescún Otero2
Correspondencia: Raúl Rodríguez Martínez. Universidad de Pamplona, Facultad de Salud, Pamplona (Norte de Santander). Ciudadela Universitaria, km 1, vía a Bucaramanga (Colombia). rrodriguez@unipamplona.edu.co
Fecha de recepción: 24 de mayo de 2013
Fecha de aceptación: 11 de julio de 2013
Resumen
Durante el transcurso de los últimos años se ha reportado un gran número de aplicaciones de la técnica FISH, la cual es utilizada en la detección de microorganismos en su propio hábitat sin que requieran de su previo aislamiento y purificación. La importancia de FISH radica en la capacidad que tiene la sonda de ADN de detectar una región específica del ácido nucleico de la célula microbiana y ser visualizada por microscopía de epifluorescencia. En esta revisión se describe los diversos usos que tiene FISH, que van desde la identificación de la microbiota en ambientes acuáticos y su empleo en la biorremediación hasta la detección de patógenos en el diagnóstico clínico. Asimismo, se presentan algunas limitaciones, y los posibles correctivos que se deben tener encuenta cuando se aplica esta técnica.
Palabras clave: Hibridación, fluorescencia, sonda, FISH, fluorocromos, aplicaciones.
Abstract
During these recent years, a large number of FISH technique applications have been reported. These techniques have been used in the detection of microorganisms in their own habitat without requiring their previous isolation and purification. The importance of FISH lies in the ability of the DNA probe to detect a specific region of the nucleic acid of microbial cells and to be visualized by epifluorescence microscopy. This review describes the various FISH uses ranging from the identification of the microbiota in aquatic environments and their use in bioremediation, to the detection of pathogens in clinical diagnosis. It also presents some limitations as well as the potential solutions to be applied when the FISH technique is used.
Key words: Hybridization, Fluorescence, Probe, FISH, Fluorochromes, Application.
Introducción
FISH o Hibridación in situ Fluorescente es una técnica que detecta secuencias de ácidos nucleicos en células o tejidos preservados mediante el empleo de una sonda marcada con un fluorocromo, la cual va dirigida hacia un lugar específico del cromosoma y que emite fluorescencia que puede ser observada por medio de un microscopio. La técnica de hibridación in situ se fundamenta en la capacidad que poseen los ácidos nucleicos para hibridarse entre sí, es decir, la existencia de determinada secuencia de ADN o ARN, que resulta complementaria con otra secuencia a través de puentes de hidrógeno formados entre las bases adenina- timina (DNA) o uracilo (RNA) y citosina-guanina (DNA y RNA).
Los métodos tradicionales de identificación microbiana basados en medios de cultivo en muchos casos requieren de tiempos largos de incubación y en ocasiones se deben emplear medios selectivos complejos, especialmente cuando se pretende aislar bacterias de difícil crecimiento (1); además, estos métodos no reflejan la población exacta o la mezcla de las comunidades bacterianas presentes en el microhábitat (2). Por su parte, la identificación empleando la técnica de FISH combina la precisión de la genética molecular con la información visual de la microscopía, lo cual permite la identificación y visualización de la célula microbiana individual dentro de su microhábitat natural o tejido en el que se encuentre presente (3-4).
Desde sus primeras aplicaciones la ténica de FISH se ha empleado en una gran cantidad de estudios tendientes a identificar microorganismos presentes en muestras tanto ambientales como clínicas. Se estima que únicamente el 0,3 % de bacterias del suelo y < 0,1 % de agua marina son cultivables; por eso, uno de los usos de FISH es el recuento microscópico de células totales, el cual supera al número de bacterias que logran crecer en un medio de cultivo; además permite apreciar la variación filogenética y geográfica de las bacterias presentes en una comunidad microbiana (5).
Debido a la importancia que adquiere día a día la técnica de FISH en el campo de la identificación molecular de los microorganismos, este trabajo hace una revisión de las aplicaciones en áreas del conocimiento como la clínica, medio ambiente, simbiosis entre planta - microorganismo y biorremediación; de igual manera, describe los inconvenientes que se presentan al aplicar esta técnica. Esta información servirá de base para los trabajos que se realicen y que estén orientados a reconocer la microbiota asociada a los diferentes ecosistemas.
Metodología de fish
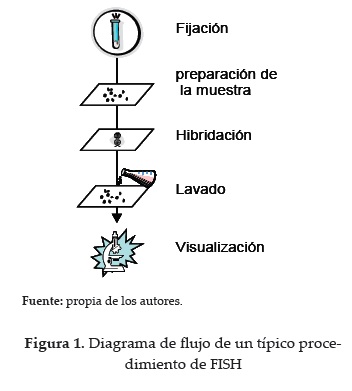
Un protocolo de la técnica de FISH incluye 4 pasos (Figura 1): (i) fijación y permeabilización de la muestra, (ii) hibridación, (iii) lavado y (iv) la detección de las células marcadas a través del microscopio de epifluorescencia o confocal (6). Antes de la hibridación, el cultivo, la muestra o tejido que contiene microorganismos deben ser fijados y permeabilizados para facilitar la penetración de la sonda fluorescente dentro de la célula y para proteger al ARN de la degradación por ribonucleasas endógenas. La hibridación es el proceso en el que a la muestra desnaturalizada se le añade la sonda de interés que se unirá a la secuencia escogida del ARNr. Pasado el tiempo de hibridación, las láminas son lavadas con agua destilada para remover la sonda que no se unió. Finalmente, se realiza la visualización de la muestra, la cual requiere de un microscopio de epifluorescencia equipado con diversos filtros para los diversos espectros de color.
Aplicaciones de FISH
a. Identificación de microorganismos de interés clínico
Para su crecimiento y posterior identificación, muchas bacterias patógenas requieren de medios de cultivos con características nutricionales bastante exigentes, y otras necesitan periodos largos de incubación, lo cual dificulta el suministro de un tratamiento antimicrobiano efectivo en corto tiempo. Afortunadamente, en los últimos años se ha incursionado el empleo de diversas técnicas de diagnóstico molecular, y en conjunto con FISH han sido útiles para identificar o caracterizar rápidamente los microorganismos de importancia médica (7, 8, 9), desde aquellos asociados a enfermedades de la boca hasta los que generan infecciones internas en órganos, y de esta manera dar un manejo adecuado del paciente cuando se trata de establecer la terapia antimicrobiana específica (10).Respecto a la microbiota que ataca los dientes, las fases de la infección del tejido pulpar por microorganismos de la dentina siguen siendo un misterio; por esto diversos estudios se han enfocado a dilucidar estos agentes microbianos mediante la hibridación fluorescente in situ, empleando secciones del tejido, el cual es embebido en resina. De esta manera, se ha logrado identificar los consorcios bacterianos asociados con caries avanzada de la dentina. Los principales grupos microbianos aislados están dominados por las bacterias de las familias Lactobacillaceae, Streptococcaceae, Veillonellaceae, Eubacteriaceae, Lachnospiraceae, Coriobacteriaceae, Bifidobacteriaceae, Propionibacteriaceae y Prevotellaceae, así como fusobacterias. Las sondas de ARNr 16S correspondientes a los taxones principales de las bacterias en la dentina cariosa se han utilizado para proporcionar información sobre las características de la infección de la pulpa dental (11).
La relación entre las bacterias periodontopatogénicas y el desarrollo de la aterosclerosis ha estado bajo investigación durante muchos años, y ha proporcionando evidencia creciente de que la inflamación crónica de la enfermedad periodontal podría actuar como un factor adicional para la aterogénesis. El uso de la técnica de FISH apoya la hipótesis de que patógenos periodontales metabólicamente activos, como Treponema denticola y Porphyromonas gingivalis aislados a partir de biopsias de pacientes periodontopáticos o desdentados que sufren de arteriosclerosis, pueden estar ubicados dentro de la pared de la arteria en la capa íntima a la lesión aterosclerótica (12).
Otra de las enfermedades muy comunes en nuestro medio es la faringitis, la cual es una infección causada por diversos virus o bacterias, siendo el estreptococo betahemolítico del grupo A (EBHGA) el principal agente causal. Sin embargo, el cuadro clínico de la faringoamigdalitis es inespecífico, debido a que los casos de infección estreptocócica moderada son indistinguibles de una infección vírica. Dado que no es posible realizar un estudio microbiológico completo de los potenciales microorganismos responsables, las pruebas deben dirigirse a identificar el EBHGA, por el riesgo de complicaciones supuradas e inmunológicas que implica su presencia. Es aquí cuando las técnicas moleculares, y entre ellas FISH, son indispensables para la identificación rápida de S. pyogenes por su alta sensibilidad y especificidad (13) y para iniciar lo antes posible la terapia con antibióticos que permitan evitar complicaciones y detener la transmisión de la infección a otras personas.
El tracto digestivo puede ser infectado por varios patógenos, y entre estos la bacteria Helicobacter pylori, la cual es conocida por su capacidad de colonizar el estómago humano, y aunque la mayoría de los portadores son asintomáticos, la colonización puede conducir al desarrollo de varias enfermedades gástricas, como la enfermedad de úlcera péptica, la mucosa gástrica asociada a tejido linfoide (MALT) y el carcinoma gástrico. La infección puede ser diagnosticada mediante sondas fluorescentes de oligonucleótidos que van dirigidas a regiones específicas del H. pylori. Además, mediante la hibridación se puede detectar la resistencia de la bacteria al antibiótico claritromicina cuando la sonda se une al gen ribosómico 23S (ARNr 23S), lo cual permite obtener resultados al cabo de tres horas, garantizando de esta manera un tratamiento efectivo (14).
La infección de las vías digestivas también es causada por espiroquetas y se asocia con el crecimiento excesivo de bacterias del género Brachyspira en el intestino grueso. El diagnóstico microbiológico mediante cultivo se ve obstaculizado por el lento crecimiento y, a su vez, por la naturaleza exigente de Brachyspira spp., por lo que en la práctica clínica la infeccón intestinal es diagnosticada histopatológicamente, y aún así, una porción significativa de los casos analizados pueden presentar falsos positivos. Debido a que FISH permite la visualización e identificación de bacterias individuales en secciones de tejido, se han diseñado sondas que permiten identificar todas las especies de Brachyspira con base en la secuencia de datos del gen ARNr 16S actualmente disponibles (15).
La presencia de parásitos también es detectada mediante FISH. Un ejemplo es el parásito del género Cryptosporidium, que puede causar infección gastrointestinal en el hombre, la cual presenta un cuadro de diarrea acuosa y voluminosa con moco, sin sangre ni leucocitos, tras una semana de incubación. De manera característica, la identificación de las especies de este género a partir de la morfología del ooquiste constituye una gran dificultad, ya que las diferencias en algunos casos son indetectables. En la actualidad se recurre a la biología molecular con técnicas de hibridación para identificar especies y genotipos de este y otros parásitos (16).
En infecciones sanguíneas, el hallazgo de cocos Gram positivos dispuestos en racimos (CGPR) en una muestra de hemocul-tivo es sugestivo de bacteriemia, pero la relevancia del diagnóstico depende de la identificación correcta del microorganismo y su evaluación dentro del contexto clínico de cada paciente. En estos casos, el uso de la Hibridación in situ ha mostrado ser un método rápido, fidedigno y practicable para la identificación directa de bacterias que se han desarrollado en hemocultivos, mediante sonda dirigida, por ejemplo, al gen ribo-sómico de Staphylococcus aureus (17).
Se ha reportado el empleo de FISH para detectar biopelículas formadas por bacterias en diferentes procesos infecciosos. De esta manera, ha permitido esclarecer cuál es la distribución espacial de las bacterias formadoras de biofilms que predominan en las heridas crónicas de la piel humana y para obtener la medida de la respuesta inflamatoria celular contra las bacterias en dichas heridas (18). Otros estudios están enfocados a la búsqueda de bacterias causantes de infecciones del tracto urinario. En esta caso, en comparación con los métodos convencionales de identificación, el método de FISH produce resultados positivos para el> 90 % de las muestras analizadas y tiene el potencial de convertirse en una herramienta de diagnóstico muy útil en este tipo de infecciones, por tener una alta precisión y un tiempo de respuesta rápido (3-4 h) (19). De igual manera, la rapidez y sensibilidad se pueden apreciar en otro tipo de muestras, como en la detección de bacterias que no crecen en cultivos a partir de muestras de lesiones de tejido blando (20) y de biopelículas que se forman en fracturas de huesos en proceso de cicatrización (21).
Las infecciones intrahospitalarias (IIH) son un problema de salud pública en nuestro país por su frecuencia, severidad y alto costo, de manera que la identificación fiable y rápida de los microorganismos es de suma importancia para dar el tratamiento adecuado y comprender su papel en la patogénesis de las infecciones. En la práctica clínica, FISH puede ser utilizado en situaciones en las que una identificación rápida es necesaria para un tratamiento óptimo del paciente, y por otra parte, para conocer la abundancia, distribución espacial y la morfología de las células bacterianas que puedan presentarse en las diversas salas, como urgencias, obstetricia, pediatría o cirugía, entre otras, y determinar los tipo de microbios que puedan estar adheridos a material como vendas, ropas, sondas, monitores o instrumentales.
b. Ambientes acuáticos
FISH permite detectar los microorganismos individuales en su microhábitat sin ningún paso de purificación selectiva o amplificación, por lo que puede ayudar a desvelar la función ecológica que cumplen allí. Se han realizado revisiones del empleo de esta técnica en el estudio de la diversidad de las poblaciones ambientales como en hábitats acuáticos (22) y en los grupos microbianos específicos que participan en la eutroficación del agua marina, y más recientemente ha permitido estudiar la comunidad bacteriana presente en el fitoplancton del noroeste del océano Pacífico con el apoyo de la técnica inmunocitoquímica con bromodesoxiuridina (BIC-FISH) (23).
Las arqueobacterias cuyo hábitat son fuentes termales, depósitos profundos de petróleo caliente, fumarolas marinas, lagos salinosos o ambientes polares han sido estudiadas mediante FISH, y en algunos casos con el uso combinado de microsensores, lo cual permite el análisis simultáneo de la comunidad bacteriana y su actividad metabólica (2-24). Se han diseñado sondas para cuantificar miembros específicos de una comunidad microbiana que habita en el hielo, como Octadecabacter, Glaciecola y Polaribacter, y se ha determinado la influencia de los cambios estacionales en la identidad y la actividad in situ de las asociaciones microbianas que habitan en el ártico (25).
c. Plantas de tratamiento
Los primeros trabajos realizados empleando FISH se enfocaron a determinar la cantidad de microorganismos presentes en las aguas residuales de las plantas de tratamiento. Posteriormente se orientó su uso a evaluar la relación entre el volumen de lodos activados y la presencia de bacterias filamentosas; asimismo, ha sido útil en el monitoreo microbiano de procesos de degradación de una mezcla de lodos activados a partir de desechos vegetales (26).
Las recientes investigaciones se han orientado a analizar la distribución in situ y la función de las bacterias sulfato-reductores presentes en tapetes microbianos de aguas termales y su participación en el ciclo del azufre (27). De igual manera, FISH ha sido útil para determinar la asociación sintrófica que se presenta en la comunidad de bacterias que oxidan el amonio (AOB) y el nitrito (28).
d. Biorremediación
Se destacan trabajos con FISH en procesos de tratamiento de aguas de desecho con residuos tóxicos y recalcitrantes como los compuestos fenólicos (29), en el empleo de bacterias Gram positivas, útiles en la degradación anaeróbica del benceno (30), así como en la identificación de microorganismos empleados en la biorremediación de compuestos xenobióticos (31) y de hidrocarburos poliaromáticos (32).
Otras aplicaciones están orientadas a evaluar los procesos metabólicos que realizan ciertas bacterias empleadas en la oxidación de hierro (33) y el uso de bacterias magnetotácticas (34). El empleo de oligos específicos ha sido útil en la detección de cepas que colonizan superficies de las rocas y están involucradas en procesos de biodeterioro de monumentos (35).
e. Simbiosis
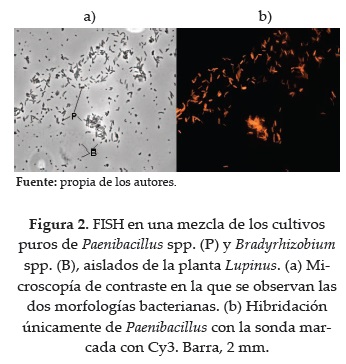
Algunos microorganismos que realizan simbiosis son difíciles de aislar en cultivos puros. en estos casos FISH se convierte en una herramienta que facilita la identificación de microorganismos asociados a plantas, ya que permite la localización del microorganismo dentro del huésped. Esta técnica se ha aplicado a la detección de bacterias intracelulares de yemas y raíces de plantas; de igual manera, para mostrar la presencia de bacterias fijadoras de nitrógeno en la caña de azúcar (36) y en trabajos de identificación de Micromonospora spp., y Paenibacillus spp. en nodulos de la planta Lupinus (Figuras 2) (37) y de bacterias endofíticas del cactus (38).
La interacción de bacterias con otros organismos ha sido tema de muchos trabajos en los que la técnica de FISH se convierte en una enorme herramienta de utilidad. Por citar algunos ejemplos, se ha aplicado para determinar el arreglo espacial de las bacterias en tejidos de esponjas (39), en la identificación de endosimbiontes de amebas de vida libre (40), en hongos micorrizas arbusculares (41), en el estudio de la microbiota intestinal de gusanos marinos (42) y de termitas (43), así como de bacterias quimiosintéticas hospedadas en vertebrados marinos (44).
Inconvenientes al aplicar la técnica fish
Aun cuando es una técnica bastante específica y sus resultados son altamente confiables, es necesario tener en cuenta algunos problemas que se pueden presentar durante el desarrollo de la técnica:
a. Autofluorescencia
Algunos microorganismos producen autofluorescencia, la cual enmascara la señal que emite la propia muestra en estudio. Es el caso de algunas especies bacterianas como Salmonella, arqueobacterias como las metanógenas y de una variedad de mohos y levaduras (45), cianobacterias (46) y algas verdes como las Chlamydomonas (47).
La fluorescencia también se puede encontrar en el material que rodea las células microbianas; por ejemplo, en el tejido de las plantas, lo cual es una fluorescencia biológica natural (37) (Figura 3). Además se presenta en muestras ambientales como lodos activados o plantas de tratamiento de aguas, donde la fluorescencia es causada por los desechos inorgánicos allí presentes (48).
Son escasos los estudios que se centran en el análisis del fenómeno de autofluorescencia y cómo evitar la interferencia con FISH, sin embargo, se ha encontrado que el medio de crecimiento, los métodos de fijación y el medio de montaje influyen en la intensidad de la señal.
Algunas formas de evitar la autofluorescencia son mediante el empleo de FISH con otra técnica de detección (22), manipulando el sistema de filtros durante la visualización y sistemas que permitan amplificar la señal, o mediante el procesamiento y manejo digital de los espectros obtenidos en la imagen (49) .
b. Especificidad de la sonda
La exactitud y confiabilidad de los resultados obtenidos por FISH depende de lo específica que sea la sonda de oligonucleótidos. El diseño, así como la evaluación exhaustiva de nuevas sondas, son pasos críticos, por lo que las sondas deben ser fabricadas en laboratorios que tengan una amplia experiencia en microbiología y en métodos de biología molecular. Cada experimento debe incluir tanto controles positivos como negativos; en el control negativo se debe emplear sondas dirigidas hacia cepas que estén relacionadas filogenéticamente con la cepa en estudio (50) .
A pesar de que la sonda haya sido bien diseñada y probada se puede presentar la unión a microorganismos que no se hayan descrito hasta el momento, especialmente cuando se estudia algún tipo de bacteria específica a partir de una población. Para permitir que el organismo de interés sea detectado es conveniente emplear 2 sondas específicas marcadas con diferentes fluorocromos que vayan dirigidas a distintas posiciones del ARNr 16S, y de este modo, las células que se detecten con ambas sondas y exhiban doble fluorescencia serán consideradas como los organismos que son objeto de estudio (51).
c. Dificultad de acceso de la sonda al sitio diana
La baja intensidad de la señal puede presentarse como consecuencia de la insuficiente penetración de la sonda dentro de la célula bacteriana, lo cual depende de la estructura de su pared celular. Las bacterias Gram negativas generalmente no tienen ningún problema, ya que su pared es permeable a la sonda de oligonucleótidos.
En algunos casos, por ejemplo, al emplear bacterias Gram positivas se debe realizar un tratamiento enzimático con lisozima o proteinasa K que permita abrir la capa de peptidoglicano (37). En bacterias que contengan en su pared ácido micólico, como en los actinomicetos Mycobacterium o Nocardia, se debe realizar la permeabilización empleando una hidrólisis ácida con HCl 1M o tratarlas con mutanolisina o 1,4 ditio-L-treitol (52).
En el estudio realizado por Yilmaz y su equipo de trabajo desarrollaron un modelo termodinámico de la hibridación, el cual provee los mecanismos para calcular la afinidad de la sonda al sitio específico del ARNr, la cual está definida sobre todo por los cambios en la energía libre de Gibbs (53).
d. Estructuras de Orden Superior
Debido a la forma tridimensional del ARNr, la formación de horquillas, así como las interacciones proteína-ARNr, hacen que la secuencia de oligonucleótidos que conforma la sonda en muchas ocasiones tenga dificultad de acceder al sitio específico, impidiendo de esta manera la hibridación. Esto explica el porqué sondas que han presentado buena hibridación empleando ARN o ADN desnaturalizado no dan resultados satisfactorios en FISH (54).
En un estudio sistemático dirigido a evaluar este problema se crearon más de 200 sondas de oligonucleótidos específicas para diferentes posiciones en el ARNr 16S de E. coli y se midió por citometría de flujo la intensidad de la señal emitida por FISH. Se emplearon "helperoligos", los cuales son oligonucleótidos no marcados que se unen a los sitios cercanos de unión de la sonda marcada y abren la estructura secundaria del ARNr, facilitando de esta manera que la sonda marcada se una al sitio específico e incrementando hasta 25 veces más la señal (55).
e. Bajo contenido de ARNr
Normalmente el contenido de ARN ribosomal de las bacterias puede variar considerablemente no solo entre especies, sino dentro de cepas de una misma especie. Lo anterior depende del estado fisiológico que presenten las células y que a su vez se correlaciona con la tasa de crecimiento. La disminución de la actividad celular debido a factores nutricionales conlleva a una baja cantidad de ARNr que genera pocos sitios de unión de la sonda fluorescente, lo que se traduce en una escasa intensidad luminosa y, por ende, en resultados falsos negativos. Esta limitación puede mejorarse mediante el empleo de un modelo termodinámico de competencia de las sondas, el cual permite conocer el número de copias de ARNr 16S presentes en las bacterias y que son necesarias para detectarlas mediante la técnica CARD-FISH (56).
Por otra parte, la sensibilidad también se puede incrementar utilizando sondas de polirribonucleótidos, así como por medio de la incubación de las células en cloranfenicol, el cual inhibe la síntesis de proteínas y degradación del ARNr. De esta manera, la división celular también se inhibe, lo cual lleva a una acumulación e incremento de ARNr dentro de la célula (57).
En el caso de especies de lento crecimiento se recomienda emplear marcadores que produzcan una gran intensidad de luz fluorescente, como el Cy3- Cy5. También se puede usar 2 o más sondas específicas marcadas con el mismo fluorocromo, que permitan aumentar el número de moléculas fluorescentes por célula. Esta técnica está limitada, por supuesto, a la disponibilidad de sitios específicos para la sonda (51).
f. Pérdida de fluorescencia
Muchos fluorocromos se pueden decolorar al ser excitados por el haz de luz y sufrir el proceso de degradación paulatina e irreversible a través del tiempo. Periodos de exposición de la muestra de solo algunos segundos o minutos pueden tener un efecto crítico, particularmente cuando se desea obtener microfotografías. Para superar este inconveniente es aconsejable el uso de una serie de filtros de banda estrecha, así como del empleo de marcadores fotoestables y de reactivos que evitan la pérdida de color como el citifluor AF1 o Gelvatol.
g. Uso de sondas bacterianas
Una alternativa para evaluar si se presentan problemas metodológicos de la técnica, así como de resultados falsos negativos, es mediante el empleo de una sonda bacteriana universal. Si la hibridación con la sonda universal da resultados satisfactorios en FISH, se puede deducir que la fijación, la penetración de la sonda y contenido de ARNr 16S de células bacterianas no son los factores limitantes.
La sonda universal EUB 338 (de eubacterias) es la sonda de uso común para este propósito, sin embargo, en algunos phyla, como por ejemplo Planctomycetes y Verucomicrobia, esta sonda no es útil. Debido a que la sonda EUB 338 se usa como rutina para cuantificar miembros del dominio bacteria, ha sido mejorada por 2 sondas más: la EUB 338 II y EUB 338 III, que van dirigidas hacia bacterias que no son detectadas por la EUB 338. Para tener un control de la unión no específica de la sonda eubacteria al ARNr 16S o a otros componentes celulares como los ácidos nucleicos, se puede usar la sonda complementaria NON 338, la cual no deberá dar ninguna señal con el FISH. Otra sonda empleada es la GAM42, dirigida al ARNr 23S de la mayoría de los miembros de las gammaproteobacterias (51).
La accesibilidad de la sonda al sitio diana puede mejorarse mediante el empleo de sondas coadyudantes no marcadas, el incremento del tiempo de hibridación hasta 96 horas o el uso de sondas de ácidos nucleicos peptídicos (PNAs).
Conclusiones
Uno de los objetivos del empleo de técnicas microbiológicas moleculares es identificar y cuantificar los microorganismos presentes en un determinado ecosistema de una manera rápida y efectiva sin que se requiera el empleo de métodos de cultivo. En este contexto, una de las técnicas más utilizadas en estos últimos años es la Hibridación in situ Fluorescente (FISH). La hibridación in situ se utiliza en los casos en que los microorganismos por estudiar resultan difíciles o imposibles de cultivar, como en el caso de las bacterias que requieren exigencias nutricionales y ambientales que los medios de cultivo no les proporcionan, y además se utiliza cuando en la muestra el material por evaluar es insuficiente.
Por otra parte, en el trabajo diario con FISH se pueden presentar diversos inconvenientes. Pero más allá de estas limitaciones se deben buscar alternativas que hagan cada día más eficiente la labor, y son esas nuevas opciones de mejora a la técnica las que se ha venido presentando con el transcurso de los años y con el avance de otras tecnologías.
En los últimos años, el uso de FISH con microsensores ha facilitado el estudio y monitoreo de la actividad metabólica, cambios en las poblaciones microbianas y el crecimiento de la biopelícula a través del tiempo. La dirección de la técnica FISH apunta hacia el empleo con las tecnologías "omicas" como la metagenómica, transcriptómica y proteómica. De igual manera, la utilidad de FISH con la espectrofotometría de masa iónica (FISH-NanoSIMS) ha abierto las puertas hacia el análisis metabólico de células individuales a partir de grupos filogenéticos microbianos. La principal ventaja que presenta esta técnica es que permite correlacionar la identidad filogenética del gen ARNr 16S con la función metabólica específica que presenta en la célula (58).
Como se puede apreciar, de manera constante aparecen nuevas publicaciones en las que se muestra la diversidad de campos de aplicación de FISH, la cual se ha extendido hacia muy variadas áreas del conocimiento y en los que se combina con otras técnicas que le sirven de apoyo. De esta manera, FISH se ha convertido en una herramienta poderosa para estudios filogenéticos, ecológicos, ambientales y de diagnóstico debido a que provee información acerca de la presencia, número, morfología y distribución espacial de las células microbianas.
Conflicto de intereses: ninguno.
Financiación: propia de los autores.
Notas
1Bacteriólogo y Laboratorista Clínico MSc, Ph.D. Docente de la Facultad de Salud, Universidad de Pamplona (Colombia). rrodriguez@unipamplona.edu.co
2Investigadora Ondas Colciencias. Pontificia Universidad Javeriana, Departamento de Quimica, Bogotá, D.C. (Colombia). ginas200331@hotmail.com
Referencias
(1) Ishii S, Tago K, Senoo K. Single-cell analysis and isolation for microbiology and biotechnology: methods and applications. Appl Microbiol Biotechnol 2010; 86: 1281-1292. [ Links ]
(2) Amann R, Fuchs BM, Behrens S. The identification of microorganisms by fluorescence in situ hybridisation. Curr Opin Biotechnol 2001; 12(3): 231-236. [ Links ]
(3) Amann R, Fuchs BM. Single-cell identification in microbial communities by improved fluorescence in situ hybridization techniques. Nat Rev Microbiol 2008; 6: 339-348. [ Links ]
(4) Cerqueira L, Fernandes RM, Ferreira RM, Carneiro F, Dinis-Ribeiro M, Figueiredo C, Keevil CW, Azevedo NF, Vieira MJ. PNA-FISH as a new diagnostic method for the determination of clarithromycin resistance of Helicobacter pylori. BMC Microbiol 2011; 11:101. [ Links ]
(5) Straza TR, Cottrell MT, Ducklow HW, Kirchman DL. Geographic and phylogenetic variation in bacterial biovolume as revealed by protein and nucleic acid staining. Appl Environ Microbiol 2009; 75(12): 4028-4034. [ Links ]
(6) Rodriguez MR. Empleo de la técnica hibridación in situ fluorescente para visualizar microorganismos. Salud UIS 2011; 43 (3): 307-316. [ Links ]
(7) Bravo LT, Procop GW. Recent advances in diagnostic microbiology. Semin Hematol 2009; 46(3): 248-258. [ Links ]
(8) Cenciarini-Borde C, Courtois S, La Scola B. Nucleic acids as viability markers for bacteria detection using molecular tools. Future Microbiol 2009; 4(1): 45-64. [ Links ]
(9) Venkatesh M, Flores A, Luna RA, Versalovic J. Molecular microbiological methods in the diagnosis of neonatal sepsis. Expert Rev Anti Infect Ther 2010; 8(9): 1037-1048. [ Links ]
(10) Forrest GN. PNA FISH: present and future impact on patient management. Expert Rev Mol Diagn 2007; 7(3): 231-236. [ Links ]
(11) Nadkarni MA, Simonian MR, Harty DW, Zoellner H, Jacques NA, Hunter N. Lactobacilli are prominent in the initial stages of polymicrobial infection of dental pulp. J Clin Microbiol 2010; 48(5): 1732-1740. [ Links ]
(12) Cavrini F, Sambri V, Moter A, Servidio D, Marangoni A, Montebugnoli L et al. Molecular detection of Treponema denticola and Porphyromonas gingivalis in carotid and aortic atheromatous plaques by FISH: report of two cases. J Med Microbiol 2005; 54: 93-96. [ Links ]
(13) Tajbakhsh S, Gharibi S, Zandi K, Yaghobi R, Asayesh G. Rapid detection of Streptococcus pyogenes in throat swab specimens by fluorescent in situ hybridization. Eur Rev Med Pharmacol Sci 2011; 15(3): 313-317. [ Links ]
(14) Cerqueira L, Azevedo NF, Almeida C, Jardim T, Keevil CW, Vieira MJ. DNA mimics for the rapid identification of microorganisms by fluorescence in situ hybridization (FISH). Int J Mol Sci 2008; (10): 1944-1960. [ Links ]
(15) Schmiedel D, Epple HJ, Loddenkemper C, Ignatius R, Wagner J, Hammer B, Petrich A, Stein H, Göbel UB, Schneider T, Moter A. Rapid and accurate diagnosis of human intestinal spirochetosis by fluorescence in situ hybridization. J clin Microbiol 2009; 47(5): 1393-1401. [ Links ]
(16) Jex AR, Smith HV, Monis PT, Campbell BE, Gasser RB. Cryptosporidium, biotechnological advances in the detection, diagnosis and analysis of genetic variation. Biotechnol Adv 2008; 26: 304-317. [ Links ]
(17) Cómito ML, Vilaró M, Cuestas E, Moscone E. Uso de la técnica de hibridación fluorescente in situ para la identificación rápida de staphylococcus aureus en hemocultivos. Revista de la Facultad de Ciencias Médicas 2009; 66(4): 140-145. [ Links ]
(18) Fazli M, Bjarnsholt T, Kirketerp-M0ller K, J0rgensen A, Andersen CB, Givskov M, Tolker-Nielsen T. Quantitative analysis of the cellular inflammatory response against biofilm bacteria in chronic wounds. Wound Repair Regen 2011; (3): 387-391. [ Links ]
(19) Wu Q, Li Y, Wang M, Pan XP, Tang YF. Fluorescence in situ hybridization rapidly detects three different pathogenic bacteria in urinary tract infection samples. J Microbiol Methods 2010; 83(2): 175-178. [ Links ]
(20) Bjarnsholt T, Tolker-Nielsen T, Givskov M, Janssen M, Christensen LH. Detection of bacteria by fluorescence in situ hybridization in culture-negative soft tissue filler lesions. Dermatol Surg 2009; 2: 1620-1624. [ Links ]
(21) Palmer M, Costerton W, Sewecke J, Altman D. Molecular Techniques to Detect Biofilm Bacteria in Long Bone Nonunion: A Case Report. Clin Orthop Relat Res 2011; 11: 3037-3042. [ Links ]
(22) Pernthaler A. Identification of Environmental Microorganisms by Fluorescence in situ Hybridization. En: Timmis, K N, editor. Handbook of Hydrocarbon and Lipid Microbiology (parte 33). Springer Berlin Heidelberg; 2010. p. 4127-4135. [ Links ]
(23) Tada Y, Taniguchi A, Nagao I, Miki T, Uematsu M, Tsuda A, Hamasaki K. Differing growth responses of major phylogenetic groups of marine bacteria to natural phytoplankton blooms in the western North Pacific Ocean. Appl Environ Microbiol 2011; 77(12):4055-4065. [ Links ]
(24) Moissl-Eichinger C. Archaea in artificial environments: their presence in global space-craft clean rooms and impact on planetary protection. ISME J 2011; 5(2): 209-219. [ Links ]
(25) Alonso-Sáez L, Sánchez O, Gasol JM, Balagué V, Pedrós-Alio C. Winter-to-summer changes in the composition and single-cell activity of near-surface Arctic prokaryotes. Environ Microbiol 2008; (9):2444-2454. [ Links ]
(26) Ivanov VN, Wang JY, Stabnikova OV, Tay ST, Tay JH. Microbiological monitoring in the biodegradation of sewage sludge and food waste. J Appl Microbiol 2004; 96(4):641-647. [ Links ]
(27) Kubo K, Knittel K, Amann R, Fukui M, Matsuura K. Sulfur-metabolizing bacterial populations in microbial mats of the Nakabusa hot spring, Japan. Syst Appl Microbiol 2011; 34(4): 293-302. [ Links ]
(28) Yusof N, Hassan MA, Yee PL, Tabatabaei M, Othman MR, Mori M, Wakisaka M, Sakai K, Shirai Y. Nitrification of high-strength ammonium landfill leachate with microbial community analysis using fluorescence in situ hybridization (FISH). Waste Manag Res 2011; 29(6): 602-611. [ Links ]
(29) Di Gioia D, Sciubba L, Bertin L, Barberio C, Salvadori L, Frassinetti S, Fava F. Nonylphenol polyethoxylate degradation in aqueous waste by the use of batch and continuous biofilm bioreactors. Water Res 2009; 43(12): 2977-2988. [ Links ]
(30) Abu Laban N, Selesi D, Jobelius C, Meckenstock RU. Anaerobic benzene degradation by Gram-positive sulfate-reducing bacteria. FEMS Microbiol Ecol 2009; 68(3): 300-311. [ Links ]
(31) Desai C, Pathak H, Madamwar D. Advances in molecular and "-omics" technologies to gauge microbial communities and bioremediation at xenobiotic/anthropogen contaminated sites. Bioresour Technol 2010; 101(6): 1558-1569. [ Links ]
(32) Nielsen JL, Kragelund C, Nielsen PH. Combination of fluorescence in situ hybridization with staining techniques for cell viability and accumulation of PHA and polyP in microorganisms in complex microbial systems. Methods Mol Biol 2010; 599: 103-116. [ Links ]
(33) Fleming EJ, Langdon AE, Martinez-Garcia M, Stepanauskas R, Poulton NJ, Masland ED, Emerson D. What's new is old: resolving the identity of Leptothrix ochracea using single cell genomics, pyrosequencing and FISH. PLoS One 2011; 6(3): e17769. [ Links ]
(34) Lin W, Jogler C, Schüler D, Pan Y. Metagenomic analysis reveals unexpected subgenomic diversity of magnetotactic bacteria within the phylum Nitrospirae. Appl Environ Microbiol 2011; 77(1): 323-326. [ Links ]
(35) Cappitelli F, Principi P, Pedrazzani R, Toniolo L, Solini C. Bacterial and fungal deterioration of the Milan Cathedral marble treated with protective synthetic resins. Sci of the Total Environ 2007; 385: 172-181. [ Links ]
(36) Franke IH, Fegan M, Hayward C, Leonard G, Sly LI. Molecular detection of Gluconacetobacter sacchari associated with the pink sugarcane mealybug Saccharicoccus sacchari (Cockerell) and the sugarcane leaf sheath microenvironment by FISH and PCR. FEMS Microbiol Ecol 2000; 31(1): 61-71. [ Links ]
(37) Rodríguez MR. Análisis de la población bacteriana endófita presente en nodulos de Lupinus: interacción y localización in situ. Tesis Ph.D., Universidad de Salamanca (España); 2008. [ Links ]
(38) Lopez BR, Bashan Y, Bacilio M. Endophytic bacteria of Mammillaria fraileana, an endemic rock-colonizing cactus of the southern Sonoran Desert. Arch Microbiol 2011; 193(7): 527-541 [ Links ]
(39) Manz W, Arp G, Schumann-Kindel G, Szewzyk U, Reitner J. Widefield deconvolution epifluorescence microscopy combined with fluorescence in situ hybridization reveals the spatial arrangement of bacteria in sponge tissue. J Microbiol Methods 2000; 40(2): 125-134. [ Links ]
(40) Horn M, Wagner M. Bacterial endosymbionts of free-living amoebae. J Eukaryot Microbiol 2004; 51(5): 509-514. [ Links ]
(41) Naumann M, Schüssler A, Bonfante P. The obligate endobacteria of arbuscular mycorrhizal fungi are ancient heritable components related to the Mollicutes. ISME J 2010; 4(7): 862-871. [ Links ]
(42) Ruehland C, Dubilier N. Gamma- and epsilonproteobacterial ectosymbionts of a shallow-water marine worm are related to deep-sea hydrothermal vent ectosymbionts. Environ Microbiol 2010; 12(8): 2312-2326. [ Links ]
(43) Strassert JF, Desai MS, Radek R, Brune A. Identification and localization of the multiple bacterial symbionts of the termite gut flagellate Joenia annectens. Microbiology 2010; 156(7): 2068-2079. [ Links ]
(44) Bates AE, Harmer TL, Roeselers G, Cavanaugh CM. Phylogenetic characterization of episymbiotic bacteria hosted by a hydrothermal vent limpet (lepetodrilidae, vetigastropoda). Biol Bull 2011; 220(2): 118-127. [ Links ]
(45) Wang, Pei. Rapid differentiation of Candida albicans from non-C. albicans directly in a variety of clinical specimens using fluorescent in situ hybridisation. Mycoses 2011; 54(4): 331-336. [ Links ]
(46) Schmidt BF. The Optimization of the Catalyzed Reporter Deposition-Fluorescence in situ Hybridization (Card-Fish) Protocol for Future Use in Enumerating Populations of Cyanobacterial Picoplankton. Thesis, College of Bowling Green State University; 2010. [ Links ]
(47) Uniacke J, Colón-Ramos D, Zerges W. FISH and immunofluorescence staining in Chlamydomonas. In: RNA Detection and Visualization Methods and Protocols. Gerst JE, editor. Methods Mol Biol 2011; 714: 15-29. [ Links ]
(48) Vesey G, Deere D, Gauci MR, Griffiths KR, Williams KL, Veal DA. Evaluation of fluorochromes and excitation sources for immunofluorescence in water samples. Cytometry 1997; 29: 147-154. [ Links ]
(49) De Biasio M, Raimund L, Franz GW, Sergey V, Pierre JE. Enhancement of m-FISH Images using Spectral Unmixing. Int J of Biol and Med Sci 2007; 3:4. [ Links ]
(50) Kim DJ, Lee DI, Keller J. Effect of temperature and free ammonia on nitrification and nitrite accumulation in landfill leachate and analysis of its nitrifying bacterial community by FISH. Bioresour Technol 2006; 97(3): 459-468. [ Links ]
(51) Stoecker K, Dorninger C, Daims H, Wagner M. Double labeling of oligonucleotide probes for fluorescence in situ hybridization (DOPE-FISH) improves signal intensity and increases rRNA accessibility. Appl Environ Microbiol 2010; (3): 922-926. [ Links ]
(52) Carr EL, Eales K, Soddell J, Seviour RJ. Improved permeabilization protocols for fluorescence in situ hybridization (FISH) of mycolic-acid-containing bacteria found in foams. J Microbiol Methods 2005; 61(1): 47-54. [ Links ]
(53) Yilmaz LS, Parnerkar S, Noguera DR. Math-FISH, a web tool that uses thermodynamics-based mathematical models for in silico evaluation of oligonucleotide probes for fluorescence in situ hybridization. Appl Environ Microbiol 2011; 77: 1118-1122. [ Links ]
(54) Frischer ME, Floriani PJ, Nierzwicki-Bauer SA. Differential sensitivity of 16S rRNA targeted oligonucleotide probes used for fluorescence in situ hybridization is a result of ribosomal higher order structure. Can J Microbiol 1996; 42: 1061-1071. [ Links ]
(55) Fuchs BM, Glockner FO, Wulf J, Amann R. Unlabeled helper oligonucleotides increase the in situ accessibility to 16S rRNA of fluorescently labeled oligonucleotide probes. Appl Environ Microbiol 2000; 66: 3603-3607. [ Links ]
(56) Hoshino T, Yilmaz LS, Noguera DR, Daims H, Wagner M. Quantification of target molecules needed to detect microorganisms by fluorescence in situ hybridization (FISH) and catalyzed reporter deposition-FISH. Appl Environ Microbiol 2008; 74: 5068-5077. [ Links ]
(57) Zwirglmaier K. Detection of prokaryotic cells with fluorescence in situ hybridization. Methods Mol Biol 2010; 659: 349-362. [ Links ]
(58) Dekas AE, Orphan VJ. Identification of diazotrophic microorganisms in marine sediment via fluorescence in situ hybridization coupled to nanoscale secondary ion mass spectrometry (FISH-NanoSIMS). Methods Enzymol 2011; 486: 281-305. [ Links ]