Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Salud Uninorte
Print version ISSN 0120-5552On-line version ISSN 2011-7531
Salud, Barranquilla vol.32 no.1 Barranquilla Jan./Apr. 2016
https://doi.org/10.14482/sun.32.1.8598
http://dx.doi.org/10.14482/sun.32.1.8598
Bacterias causantes de enfermedades transmitidas por alimentos: una mirada en Colombia
Bacteria causing of foodborne diseases: an overview at Colombia
Zamira Soto Varela1
Liliana Pérez Lavalle2
Dalidier Estrada Alvarado3
1 Docente investigador, Universidad Simón Bolívar. Barranquilla (Colombia). zsoto1@unisimonbolivar. edu.co
2 Docente investigador, Universidad Simón Bolívar. Barranquilla (Colombia). lperez70@unisimonbolivar.edu.co
3 Estudiante Programa de Microbiología, Universidad Simón Bolívar. Barranquilla (Colombia). zsoto1@unisimonbolivar.edu.co
Correspondencia: Zamira Soto Varela. Universidad Simón Bolívar, Programa de Microbiología, carrera 59 n° 59-92. Barranquilla (Atlántico). Teléfono: 344 4333, Fax: 3682892. zsoto1@unisimonbolivar.edu.co
Fecha de recepción: 23 de abril de 2015
Fecha de aceptación: 25 de noviembre de 2015
Resumen
Objetivo: Las enfermedades de transmisión alimentaria constituyen un grave problema de salud pública a nivel mundial; entre sus causas más frecuentes se encuentran los patógenos bacterianos, los cuales generan desde síntomas gastrointestinales hasta complicaciones que pueden conducir a la muerte. En esta revisión se describen estudios sobre detección de patógenos bacterianos en diferentes alimentos en Colombia publicados entre 2010 y 2013, y se presenta información acerca de las características y prevalencia de los microorganismos encontrados, alimentos implicados y caracterización de los aislados. La búsqueda en bases de datos arrojó un total de 16 artículos enfocados directamente a la detección de cinco patógenos: Salmonella spp., Listeria monocytogenes, Escherichia coli, Aeromonas spp. y Vibrio spp. La mayor parte de los estudios correspondió al género Salmonella. No se hallaron investigaciones relacionadas con otras bacterias causantes de enfermedades de transmisión alimentaria. Los productos analizados fueron principalmente de origen animal, desde alimentos crudos, como pescado y carnes, hasta alimentos listos para el consumo. Esta revisión evidencia que a pesar de la importancia a nivel de salud pública de detectar y caracterizar bacterias patógenas trasmitidas por alimentos existen muy pocos estudios publicados relacionados con esta temática en el periodo revisado. Asimismo, los trabajos se encaminaron primordialmente a la búsqueda del microorganismo en el producto final y no a lo largo de la cadena productiva.
Palabras clave: enfermedades transmitidas por alimentos, bacterias, Colombia.
Abstract
Objetive: Food-borne diseases are a serious public health problem worldwide; with pathogenic bacteria as the most common cause, leading to gastrointestinal disorders which can eventually lead to death. In this review are described studies about the detection of food-borne pathogenic bacteria in Colombia publicized between the years 2010-2013, looks at information on the characteristics and prevalence factor of found pathogens, the foods implicated in the studies and the characterization of isolated microorganisms. The database search yielded a total of 16 articles focused directly to the detection of five pathogens: Salmonella spp., Listeria monocytogenes, Escherichia coli, Aeromonas spp. and Vibrio spp.; nevertheless, most of the studies focused on Salmonella. There were no research projects on any other food-borne diseasecausing pathogens. The products tested were mainly raw or ready-to-eat animal-based foods such as sea food and meat. This review reveals that despite the importance of detecting and characterizing pathogens transmitted by contaminated food, there are very few published studies on this topic for the given review period. Likewise, the research work was directed primarily to the search of microorganisms as a final product rather than contamination along the production line.
Keywords: foodborne diseases, bacteria, Colombia.
INTRODUCCIÓN
La enfermedad trasmitida por alimentos (ETA) es el síndrome originado por la ingestión de alimentos y/o agua que contienen agentes etiológicos en cantidades tales que afectan la salud del consumidor (1). Estas enfermedades se caracterizan por una variedad de síntomas gastrointestinales, como náuseas, vómito, diarrea, dolor abdominal y fiebre; en algunos casos se pueden presentar complicaciones severas, como sepsis, meningitis, abortos, síndrome de Reiter, síndrome de Guillan Barré o la muerte (2, 3).
Ciertas complicaciones son producto de toxinas de origen bacteriano, como por ejemplo, la toxina producida por Clostridium botullinum, que puede llegar a generar fallas respiratorias (4), y la toxina shiga, producida por cepas de Escherichia coli, causante del síndrome hemolítico urémico (5).
De acuerdo con la Organización Mundial de la Salud (OMS), las ETA constituyen uno de los problemas sanitarios más comunes que aquejan la salud de las personas en el mundo, y afectan con mayor severidad a niños, mujeres embarazadas, ancianos y personas con otros padecimientos; sin embargo, estas enfermedades no solo afectan la salud, sino que tienen un impacto socioeconómico negativo, debido a que ocasionan una disminución en la productividad y el comercio e imponen una carga sustancial en los sistemas de salud al generar gastos en hospitalizaciones y medicamentos (6, 7).
En Estados Unidos, el Centro para el Control y Prevención de enfermedades (CDC) reportó en 2013 un total de 19 056 infecciones alimentarias, 4200 hospitalizaciones y 80 muertes (8). Por su parte, la Autoridad Europea de Seguridad Alimentaria (EFSA) y el Centro Europeo para el Control y Prevención de Enfermedades (ECDC) reportaron para 2012 un total de 55 453 casos, 5118 hospitalizaciones y 41 muertes (9). En Colombia en el año 2014 se notificaron al Sistema Nacional de Vigilancia en Salud Pública (SIVIGILA) del Instituto Nacional de Salud, un total de 9.730 casos de Enfermedades transmitidas por alimentos o agua (10).
Entre las ETA más frecuentes están aquellas causadas por una contaminación de tipo biológico, evidenciado por los reportes entre 1993-2010 realizados al sistema de información regional de la Organización Panamericana de la Salud (OPS); en los que se indica que de 9180 brotes reportados, el 69 % por bacterias, el 9,7 % por virus y el 1,8 % por parásitos; el porcentaje restante correspondió a otras causas de origen químico (11).
Entre los patógenos bacterianos más frecuentes en Estados Unidos en 2013 se encontraron, en su orden, Salmonella, Campylobacter, Shigella, Escherichia coli, productora de toxina shiga (STEC) O157, STEC O157, Vibrio, Yersinia y histeria (8).
En el caso de Europa, la mayoría de los brotes de 2012 fueron causados por Salmonella, toxinas bacterianas, virus y Campylobacter (9).
En Colombia, los principales agentes etiológicos identificados en brotes de ETA en 2010 fueron Staphylococcus coagulasa positiva, Escherichia coli y Salmonella spp.; bacterias establecidas en la normativa nacional para los diferentes tipos de alimentos; no obstante, se demostró la presencia de otras bacterias, como Listeria monocytogenes y Shiguella spp. (12); de allí la necesidad de investigar otros patógenos en diferentes tipos de alimentos.
En este estudio se realizó una búsqueda de artículos publicados en las bases de datos y bibliotecas virtuales: EBSCO, OVID, PROQUEST, Biblioteca Virtual en Salud Pública de Latinoamérica (BVS), Biblioteca Virtual para la vigilancia en Salud Pública de Colombia (BVS-VSP Col), Medline-Pubmed, Science Direct, Biblioteca Digital de Colombia (BDCOL), Scientific Electronic Library on Line-SciELO y Literatura Latinoamericana y del Caribe en Ciencias de la Salud (LILACS), utilizando como palabras de búsqueda cada uno de los nombres de los patógenos bacterianos: Aeromonas hydrofila, Bacillus cereus, Brucella mellitensis, Brucella abortus, Campylobacter, Clostridium botulinum, Clostridium perfringens, Coxiella burnetti, Enterobacter sakasaki, E. coli, Staphylococcus aureus, Listeria monocytogenes, Plesiomonas shigueloides, Salmonella, Shiguella, Streptococcus pyogenes, Vibrio, Mycobacterium bovis y Yersinia; junto con las palabras alimento y Colombia dentro de los años 2010-2013.
El objetivo de esta revisión fue mostrar las publicaciones sobre la detección de patógenos bacterianos en diferentes alimentos en Colombia; proporcionando información sobre sus características y datos de importancia en salud pública, lo cual es de relevancia para la evaluación de riesgos microbiológicos.
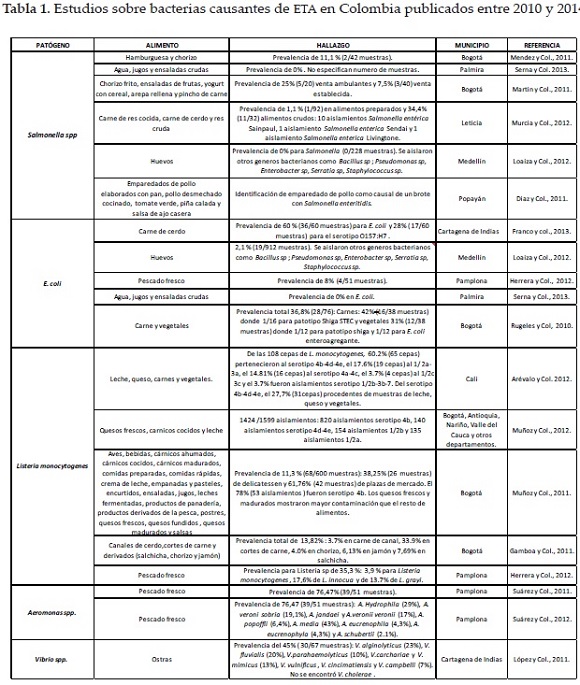
En los resultados obtenidos se encontraron estudios enfocados a los siguientes patógenos ver Tabla 1.
Salmonella spp.
Salmonella spp. es un bacilo Gram negativo, no esporulado y móvil; a excepción de los serotipos S. Gallinarum y S. Pullorum, que no poseen esta última característica, pertenece a la familia Entereobacteriaceae tribu Salmonellae (13). En la actualidad se reconocen dos especies: Salmonella entérica, la cual incluye las subespecie I, II, IIIa, IIIb, IV, y VI, y Salmonella bongori, con la subespecie V (14); dentro de estas existen más de 2500 serotipos, los cuales se clasifican de acuerdo con el antígeno flagelar H y el antígeno somático O (15).
El género Salmonella tiene gran impacto en salud pública; datos epidemiológicos indican que la gastroenteritis y la fiebre tifoidea son de distribución mundial, y ocurren en países desarrollados y subdesarrollados (16, 17).
Las salmonellas no tifoideas (diferentes a Salmonella Typhi y Salmonella Paratyphi), principalmente los serotipos de Salmonella entérica, subespecie entérica, son las que se relacionan con gastroenteritis de origen alimentario (18, 19). Los alimentos en los que se ha detectado principalmente este patógeno son la carne de pollo, carne de cerdo, carne de pavo, productos con carne cruda, huevos y jamón de cerdo (20-22).
Los métodos para la detección de esta bacteria se fundamentan en el cultivo microbiológico, pero su aislamiento se dificulta por el bajo número de células presentes en el alimento y la inhibición por otros microorganismos más competentes que puedan estar presentes; por ello, los métodos moleculares como la Reacción en Cadena de la Polimerasa-PCR se están implementando por su bajo límite de detección, alta sensibilidad y especificidad (23,24).
En la actualidad, una de las principales preocupaciones relacionadas con este patógeno es el aumento de su resistencia a los antibióticos, debido a que cada vez son más frecuentes los aislamientos de origen animal y alimentario resistentes a antibióticos como el ácido nalidíxico, sulfafurazol y la penicilina (25, 26); razón por la cual diversos esfuerzos están enfocándose en la prevención, con el diseño de vacunas para animales (27) y la implementación de tecnologías de control en la industria de alimentos (28).
En Colombia, los estudios realizados entre 2010 y 2013 que buscaron este patógeno en alimentos se centraron en el análisis de muestras provenientes de diferentes establecimientos de ventas ambulantes y restaurantes. Dos de estos fueron realizados en Bogotá. El primero en un sector del norte de la ciudad, el cual incluyó 40 establecimientos de venta establecida y 20 de venta ambulatoria.
Los resultados obtenidos señalan que el 25 % de los alimentos ambulantes y el 7,5 % de los alimentos de venta establecida, como chorizo, fritos, ensaladas de frutas, yogur con cereal, arepa rellena y pincho de carne, fueron positivos para Salmonella spp.
En este estudio también se realizó coprocultivo a los manipuladores de alimentos para la búsqueda de este microorganismo, y se obtuvo una prevalencia del 15 % en ambulantes y 10 % en restaurantes; y se resalta que este personal no presentó manifestaciones gastrointestinales (29).
En el segundo trabajo, realizado en un sector universitario en 2010, se detectaron 2 muestras positivas para Salmonella entérica de 42 muestras analizadas (11.1 %): una muestra fue hamburguesa y otra chorizo. Asimismo, en el proceso se aislaron en 18 muestras otras enterobacterias, como Citrobacter freundii, Edwardsiella tarda, Escherichia coli y Pantotea agglomerans.
En este trabajo también se realizó la prueba de susceptibilidad antimicrobiana, y se encontró que un aislamiento de Salmonella entérica fue sensible a la Ciprofloxacina y Ceftriaxona (30).
En otro sector universitario también se realizó un análisis de muestras de alimentos como ensaladas crudas, jugos, salsas y agua procedentes de 4 expendios, con el objetivo de evaluar el riesgo de adquirir una ETA por la comunidad de este sector; sin embargo, en la búsqueda de Salmonella no se detectó su presencia en ninguna de las muestras analizadas, a diferencia de los coliformes totales y fecales que no estuvieron dentro de los valores permitidos; siendo difícil establecer el impacto de este hallazgo debido a que el estudio no especifica el número de muestras y no incluye análisis microbiológicos a productos de origen animal que también son muy comercializados en el sector universitario y en donde es más probable encontrar este y otros patógenos, como Staphylococcus aureus, el cual tampoco fue detectado (31).
En la ciudad de Leticia (Putumayo, Colombia) en 2011 se analizaron 92 alimentos preparados de origen animal y 32 alimentos crudos provenientes de diferentes establecimientos. En el primer caso solo se aisló Salmonella spp. en una muestra (1,1%) y en el segundo caso en 11 muestras (34,4 %).
En este trabajo se logró la serotipificación de los aislamientos; 10 de estos, procedentes de carne de res cruda, fueron identificados como Salmonella entérica Sainpaul, un aislamiento en carne de cerdo cruda como Salmonella entérica Sendai y otro en carne cocida de res como Salmonella enterica Livingtone (32).
En el mismo año, en Popayán (Cauca, Colombia) un estudio caracterizó aislamientos de Salmonella Enteritidis asociados a un brote; y por análisis fenotípico y genotípico por electroforesis en campo pulsado se confirmó la asociación entre los aislamientos de los pacientes con un emparedado de pollo como la fuente de infección; se observó resistencia de los aislamientos al ácido nalidíxico (33).
Se resalta en estos dos trabajos la caracterización de los aislamientos debido a que muchos estudios solo llegan hasta la identificación de género, lo cual no permite conocer la circulación de serotipos y asociarlos con un brote determinado.
Otro de los productos alimenticios asociados a contaminación con Salmonella spp. es el huevo; sin embargo, en un estudio realizado en Medellín (Colombia) y su área metropolitana no se logró aislar Salmonella en 228 huevos, pero se aislaron otros géneros bacterianos, como Bacillus sp., Pseudomonas sp., Enterobacter sp., Serratia sp., Citrobacter sp., E. coli, Streptococcus viridans, Klebsiella sp., Staphylococcus sp., Aeromonas sp., Sarcinas sp., Acinetobacter sp., E. hermanii, Proteus y Stenotrophomonas maltophilia (34).
Escherichia coli
Escherichia coli es un bacilo Gram negativo, anaerobio facultativo, usualmente móvil por flagelos peritricos, cuyo hábitat es el intestino de animales de sangre caliente (35, 36). Esta bacteria es utilizada como indicador de posible contaminación fecal y presencia de patógenos en agua y alimentos debido a que se encuentra abundantemente en heces de humanos y animales (37).
Aunque Escherichia coli puede ser un residente inocuo del tracto gastrointestinal, varios estudios han documentado que ciertas cepas de E. coli producen diarrea y otras enfermedades extraintestinales en humanos (38-42). Estas cepas de E. coli patogénicas constituyen un grupo heterogéneo de organismos con diferentes propiedades de virulencia, serotipos O:H, epidemiología y enfermedades asociadas. Con base en sus factores de virulencia específicos y rasgos fenotípicos se han subdividido en seis grupos patógenos: E. coli enteropatogénica (EPEC), E. coli ente-roagregante (EAEC), E. coli enterotoxigénica (ETEC), E. coli de adhesión difusa (DAEC), E. coli enteroinvasora (EIEC) y E. coli enterohe-morrágica (EHEC) (43, 44).
Estos grupos han sido detectados en diferentes productos, como cárnicos, lácteos, pescados, mariscos, bebidas, hielo y leguminosas (44-50).
E. coli O157:H7 es reconocido como uno de los serotipos más representativos de E. coli ente-rohemorrágica (EHEC). Esta bacteria es capaz de producir dos tipos de toxina shiga, Stx1 y Stx2, que ocasionan diarrea, colitis hemorrágica y síndrome urémico hemolítico (51-53).
Desde que E. coli O157:H7 fue reportada por primera vez en Estados Unidos en 1982 como agente causal de un brote que afectó al menos a 47 personas en Oregon y Michigan por el consumo de carne de hamburguesa poco cocida, ha sido ampliamente asociada a varios casos y muertes alrededor del mundo (54-57).
Esta bacteria se encuentra regularmente en las heces del ganado sano y es transmitida al hombre principalmente por la ingestión de productos bovinos; aunque se ha relacionado también con la leche no pasteurizada, bebidas contaminadas, verduras frescas y a través del contacto persona a persona (58, 59).
En 2013 se publicó un estudio en el cual se determinó E. coli y se identificó el serotipo O157:H7 en carne de cerdo comercializada en diferentes supermercados de la ciudad de Cartagena de Indias, durante agosto y septiembre de 2008. De 60 muestras analizadas se obtuvo 36 con E. coli en niveles no aceptables según las exigencias del Instituto Nacional de Vigilancia de Medicamentos y Alimentos (INVIMA) para productos cárnicos, y se identificándose el serotipo O157:H7 en 17 muestras; sin embargo, esta caracterización debe considerarse como presuntiva de acuerdo al método utilizado, debido a que en el estudio no se demuestra su confirmación por otros métodos como los moleculares, que además pueden permitir la identificación de genes productores de toxinas (60).
En otro estudio divulgado en 2010 se llevó a cabo una caracterización molecular para la identificación de patotipos de E. coli en 76 muestras de alimentos, conformadas por 38 muestras de carne obtenidas de dos supermercados y 38 muestras de vegetales recolectadas a partir de ocho mercados minoristas de la ciudad de Bogotá.
Se encontró 16 muestras de carne positivas para E. coli, una de estas correspondiente al patotipo productor de toxina shiga (STEC). En el caso de los vegetales 12 muestras presentaron E. coli, una de estas positiva para E. coli productora de toxina shiga (STEC) y otra para E. coli enteroagregante (61).
En relación a estudios limitados solo a la detección de E. coli como indicador de contaminación fecal, sin tener en cuenta el carácter patogénico de las cepas; en el municipio de Pamplona (Norte de Santander, Colombia) se analizaron 51 muestras de pescado fresco, de las cuales el 8 % presentó un nivel inaceptable de E. coli según las recomendaciones establecidas por la Comisión Internacional de Especificaciones Microbiológicas (ICMSF) (62).
En 2011 se detectó la presencia de E. coli en 19 muestras, de un total de 912 procedentes de 228 huevos de graneros de Medellín y el área metropolitana del Valle de Aburrá (34).
En otra investigación no se detectó E. coli en ensaladas y salsas procedentes de cuatro cafeterías de una comunidad universitaria en Colombia; sin embargo, no se relaciona esta ausencia con otros factores intrínsecos como el pH (31).
Histeria monocytogenes
El género Listeria se compone de diecisiete especies, que incluye a Listeria monocytogenes, Listeria ivanovii, Listeria seeligeri, Listeria riparia, histeria grandensis, histeria floridensis, Listeria cornellensis, Listeria aquatica, Listeria innocua, histeria welshimeri, histeria grayi, Listeria marthii, Listeria rocourtiae, Listeria murrayi, Listeria denitrificans, histeria fleischmannii y Listeria weihenstephanensis. Dentro de estas, Listeria monocytogenes se considera patógena tanto para humanos y animales (63).
Listeria monocytogenes es un bacilo Gram positivo, patógeno facultativo intracelular y no formador de esporas. Puede sobrevivir o crecer a valores de pH tan bajos como 4.4 y a concentraciones de sal hasta del 14 %. Esta bacteria es psicrotrófica y crece a temperaturas entre 1 y 45 °C; los alimentos refrigerados son un ambiente ideal para este patógeno (64-66).
Este microorganismo está ampliamente distribuido en el medio ambiente y puede colonizar las plantas de procesamiento de alimentos, persistiendo durante varios meses y años (66-71).
La formación de biopelículas protege a esta bacteria contra la desinfección y favorece su supervivencia en las superficies que están en contacto con los alimentos; lo cual aumenta la contaminación cruzada de estos y los convierte en un vehículo para llegar al hombre y causar la enfermedad (72, 73).
Listeria monocytogenes es el agente causal de la listeriosis, la cual se manifiesta de forma invasiva o no invasiva. La forma invasiva puede provocar mortinatos, abortos, partos prematuros, y en los recién nacidos e inmunodeprimidos puede conducir a septicemia, meningitis y encefalitis. En la forma no invasiva se producen síntomas como fiebre, dolor de cabeza y diarrea.
La mortalidad por listeriosis es del 20 al 30 % en grupos vulnerables (74-76).
La infección en humanos se produce principalmente por el consumo de alimentos contaminados, como leche, quesos, verduras frescas, mariscos, productos cárnicos y alimentos listos para el consumo, los cuales son considerados como importantes fuentes (77-82).
En Colombia, en 2012 se mostraron prevalencias de L. monocytogenes de 3.7 y 33.9 % en carne en canal y cortes de carne, respectivamente, procedentes de 23 plantas de procesamiento de cerdo. Asimismo, se reportó una prevalencia de 4.0 % para chorizo, 6.13 % para jamón y 7.69 % en salchicha (83).
En otro tipo de alimentos, como el pescado fresco, también se ha aislado este microorganismo, así como otras especies de Listeria. Esto se evidenció en un estudio realizado en la plaza de mercado de la ciudad de Pamplona, donde se encontraron prevalencias de L. monocytogenes de 3.9 %, L. innocua de 17.6 % y L. grayi de 13.7 % en este tipo de productos, con una incidencia total del 35.3 % (62).
En estos trabajos solo se llegó hasta la identificación de especie; no realizándose serotipificación. Por otra parte, no se llevó a cabo la búsqueda de esta bacteria en ambientes de procesamiento, donde se recomienda la toma de muestra para determinar una posible fuente de contaminación.
Se han identificado trece serotipos de Listeria monocytogenes; entre ellos, los serotipos 1/2a, 1/2b y 4b son los involucrados en la mayor parte de los casos de listeriosis humana, y el serotipo 4b es el más virulento. Los serotipos 1/2a, 1/2b, 1/2c se aíslan con más frecuencia en alimentos y en ambientes de fábricas (66, 84-89).
En Colombia se determinó la frecuencia de serotipos de L. monocytogenes aislados de alimentos durante 2000-2009. De 1424 aislamientos confirmados como L. monocytogenes, la mayoría pertenecía al serotipo 4b, con 820 aislamientos; seguido por los serotipos 4d- 4e, 1/2b y 1/2a, con 140, 154, y 135 aislamientos, respectivamente; los restantes correspondieron a otros serotipos.
Los aislamientos procedieron principalmente de Bogotá, 1035 (73 %), de Antioquia, 199 (14 %), de Nariño, 109 (8 %), del Valle del Cauca, 50 (3.5 %) y de otros departamentos, 33 (2.3 %).
Los grupos de alimentos con mayor frecuencia de esta bacteria fueron los quesos frescos, los cárnicos cocidos y las leches (90).
En otra investigación realizada entre 2002 y 2008 se identificó la presencia de este patógeno en alimentos listos para el consumo procedentes de plazas de mercado y "delicatessen" de supermercados de cadena en Bogotá. De 600 muestras analizadas, 68 fueron positivas para L. monocytogenes (11.3 %); 26 (38.25 %) correspondían a "delicatesen" y42 (61.76 %) a plazas de mercado. El serotipo aislado con mayor frecuencia fue 4b en 53 (78 %) aislamientos, y entre los alimentos más contaminados estuvieron los quesos frescos y madurados (91).
En 2012 se serotipificaron molecularmente 108 cepas de L. monocytogenes aisladas de diferentes fuentes; de estas, 60.2 % (65 cepas) pertenecían al serotipo 4b-4d-4e, 17.6 % (19 cepas) al 1/ 2a-3a, 14.81 % (16 cepas) al serotipo 4a-4c, 3.7 % (4 cepas) al 1/2c-3c y 1/2b-3b-7. Del serotipo 4b-4d-4e, el 47,7 % (31 cepas) procedieron de muestras de leche, queso, carne y vegetales; el 27,7 % (18 cepas) a ambientes de procesamiento de alimentos y equipos como cortadoras y selladoras; 18,5 % (12 cepas) a muestras clínicas, específicamente líquido cefalorraquídeo y cultivos de sangre, y el 6,2 % restante (4 cepas) correspondió a muestras veterinarias (92).
Vibrio spp.
El género Vibrio comprende a bacterias Gram negativas, incluidas en el grupo gamma de las proteobacterias, con morfología celular de bacilos cortos, que a menudo son curvados o en forma de coma (93, 94).
Las especies de Vibrio están presentes en ambientes acuáticos, principalmente marinos y estuarinos, gracias a su capacidad para adaptarse a los factores físicos y químicos presentes en este tipo de ecosistemas.
En varios estudios se ha encontrado correlación entre la abundancia de varias especies patógenas con diversas condiciones, como salinidad, temperatura, conductividad eléctrica y sólidos disueltos (95, 96).
Otros trabajos evidencian un incremento en la tolerancia de Vibrio cholerae a temperatura, pH y desecación asociado a la colonización de caparazones de camarones; lo cual puede tener implicaciones en la seguridad alimentaria y epidemiología de esta especie (97).
Existen más de 100 especies de Vibrio; solo tres especies: Vibrio cholerae, Vibrio parahaemolyticus y Vibrio vulnificus, se encuentran principalmente asociadas a casos de gastroenteritis; la especie más frecuente es el Vibrio parahaemolyticus (98-103).
Los serogrupos O1 y O139 de Vibrio cholerae causan epidemias de cólera, mientras que los serogrupos restantes pueden causar diarrea esporádica (104).
Vibrio vulnificus puede ocasionar fascitis necrotizante y sepsis por el consumo de mariscos crudos o pocos cocidos, con una tasa de mortalidad mayor del 50 % (105, 106).
Los alimentos en los que se aíslan más frecuentemente estas especies de Vibrio son los productos de origen marino, como pescado, ostras, mejillones, camarones y almejas (107109).
A nivel nacional, en el periodo 2010 - 2013 se encontró un solo estudio en el que se aislaron e identificaron especies de Vibrio en 67 ostras obtenidas en 5 puntos de la Ciénaga de la Virgen en la ciudad de Cartagena de Indias en 2006. Los resultados mostraron positividad para Vibrio spp. en 30 muestras (45 %). Las especies predominantes fueron V. alginolyticus (23 %), V. fluvialis (20 %), V parahaemolyticus (10 %), V. carchariae y V. mimicus (13 %), V. vulnificus, V. cincimatiensis y V. campbelli (7 %).
No se evidenció la presencia de V. cholerae en las muestras analizadas, argumentado por la falta de serotipos halófilos; no obstante, la metodología no describe que fue medido el parámetro de salinidad en los puntos de muestreo (110).
Aeromonas spp.
El género Aeromonas es muy ubicuo; está conformado por bacilos Gram negativos anaerobios facultativos y no esporulados distribuidos en diversos ambientes, sobre todo acuáticos (111). En la actualidad, este género se encuentra clasificado en la familia Aeromonadaceae, a pesar de que por varios años estuvo incluido en la familia Vibrionaceae, pero por análisis de secuencias del gen 16S rRNA lo ubican en una línea diferente dentro de las Gammaproteobacteria; donde fenotípicamente se han propuesto 13 especies pero genotípicamente se consideran 19 (112).
Las bacterias de este género también han sido clasificadas en dos grandes grupos de acuerdo con sus características de crecimiento: el primero de ellos es el grupo mesófilo, que crece entre 35 - 37°C, son móviles y están asociados con enfermedad en seres humanos, como A. hidrófila, A. caviae, A. sobria y A. veronii, (113); el segundo grupo corresponde a los psicrófilos, con crecimiento óptimo entre 22-25 °C, no móviles y asociados a enfermedad en peces, como la especie A. salmonicida (114).
A nivel de salud, estudios indican que Aeromonas es causa potencial de gastroenteritis en seres humanos; aislamientos con genes de virulencia, principalmente relacionados con la producción de enterotoxinas, se han encontrado en pacientes con diarrea (115). Además de la gastroenteristis se pueden presentar otras manifestaciones clínicas, como infección de heridas, septicemia, abcesos en el hígado e infección respiratoria (116).
Las principales fuentes de contaminación con este microorganismo son el agua, los vegetales, comida de mar y comida de origen animal (117).
En Colombia se realizó un estudio en el que se aisló e identificó Aeromonas spp. en diferentes especies de peces comercializados en la ciudad de Pamplona. De un total de 51 muestras de diferentes pescados, como trucha, rampuche, dorada, bagre, bocachico, sierra y mojarra, 39 presentaron crecimiento de Aeromonas spp. (76,47 %). En cuanto a las especies identificadas, se encontró principalmente A. hydrophila (29,7 %) y A. veronii sobria (19,1 %), seguidas de A. jandaei y A. veronii veronii (17 %), A. popoffii (6,4%), A. caviae/A. media (4,3 %), A. eucrenophila (4,3 %) y A. schubertii (2,1 %) (118).
En un estudio posterior, a un total de 47 de estas cepas se le determinó la presencia de factores de virulencia fenotípicos, y se estableció una frecuencia de 87 % en la producción de nucleasas, 83 % con actividad phemolítica sobre eritrocitos humanos, 68 % con producción de lipasas, el 63 % fueron proteolíticas y el 53 % resultaron hemolíticas sobre eritrocitos de cordero. Estos datos indican el posible potencial patógeno de las cepas (119).
Conclusiones
Las enfermedades de transmisión alimentaria constituyen un grave problema de salud pública a nivel mundial; por ello, las autoridades de salud de diferentes países han implementado una serie de acciones con objeto de disminuir la incidencia de estas enfermedades. Adicionalmente, esta problemática ha despertado el interés de varios investigadores, que han llevado a cabo diversos estudios con el fin de buscar patógenos bacterianos causantes de ETA, determinar los alimentos asociados, las prevalencias de estos microorganismos, especies y serotipos predominantes; lo cual es de utilidad para la evaluación de riesgos microbiológicos en alimentos.
En esta revisión se encontraron con los parámetros de búsqueda mencionados 16 artículos entre los año 2010 a 2013 enfocados directamente a la detección de cinco patógenos: Salmonella spp., Listeria monocytogenes, Escherichia coli, Aeromonas spp. y Vibrio spp. cabe resaltar que en algunos estudios se encontraron otros microorganismos indicadores y patógenos como Staphylococcus aureus; sin embargo, no fue el objeto principal del trabajo.
La mayor parte de los estudios correspondieron al género Salmonella; considerada una bacteria de investigación obligatoria para una gran variedad de productos alimenticios según la legislación colombiana. La mitad de los estudios sobre este microorganismo tuvo como factor común el hallazgo de la especie entérica, mientras que los restantes se limitaron a la búsqueda hasta a nivel de género, dejando a un lado la indagación de especies y serotipos circulantes.
En relación con Listeria monocytogenes, a pesar de no ser un patógeno exigido en Colombia, fue objeto de varias investigaciones, en las cuales el queso fue el alimento con mayor frecuencia de este microorganismo entre una variedad de productos. Este dato es relevante para individuos susceptibles, como mujeres embarazadas, las cuales deben tener en cuenta una serie de medidas y precauciones en el consumo de alimentos crudos o poco cocidos de origen animal como los quesos frescos.
Para el caso de E. coli, los estudios se enfocaron principalmente en su aislamiento en productos cárnicos y vegetales, no solo como indicador fecal sino determinándose diferentes patotipos, como E. coli productora de toxina shiga y E. coli enteroagregante; lo cual es importante a nivel epidemiológico para el conocimiento de las cepas circulantes y la definición de la gravedad de los casos.
Otros estudios sobre bacterias de ambientes acuáticos, como Vibrio spp. y Aeromonas spp., en pescados y mariscos, determinaron la frecuencia de algunas especies y factores de virulencia. Para el caso de Vibrio se resalta que no se encontró la especie cholerae, sino otras como V. parahaemolyticus y V. vulnificus, que a nivel mundial se han visto involucrados en casos de gastroenteritis y otras patologías.
Finalmente, en esta revisión la mayoría de los trabajos encontrados se enfocó en determinar el microorganismo en productos elaborados y no a lo largo de la cadena de producción, lo cual es imprescindible en la evaluación de riesgos en alimentos. Adicionalmente, no se hallaron estudios de otros patógenos, como Campylobacter y Shigella, lo cual evidencia un vacío investigativo en esta área.
Conflicto de intereses: ninguno.
Financiación: Universidad Simón Bolívar.
Referencias
1. Instituto Nacional de Salud y Ministerio de la Protección de Colombia. Protocolo de vigilancia en Salud Publica. Enfermedades trasmitidas por alimentos. Bogotá, D. C.: Grupo Enfermedades Transmisibles, Equipo ETA; 2014. Protocolo n° PRO-R02.001. [ Links ]
2. Linscott AJ. Food-Borne illnesses. Clinical microbiology newsletter 2011; 33(6): 41- 45. [ Links ]
3. Steniner T. Treating foodborne illness. Infect Dis Clin North Am 2013; 27(3): 555 -76. [ Links ]
4. Witoonpanich R, Vichayanrat E, Tantisiriwit K, Wongtanate M, Sucharitchan N, Oranrigsupak P et al. Survival analysis for respiratory failure in patients with food-borne botulism. Clin Toxicol 2010; 48(3): 177-183. [ Links ]
5. Mayer C, Leibowitz CL, Kurosawa S, Stearns-Kurosawa D. Shiga Toxins and the Pathophysiology of Hemolytic Uremic Syndrome in Humans and Animals. Toxins (Basel) 2012; 4(11): 1261-1287. [ Links ]
6. Organización Mundial de la Salud (OMS). Estrategia global de la OMS para la inocuidad de los alimentos: alimentos más sanos para una salud mejor. Ginebra: Departamento de Inocuidad de los Alimentos; 2002. [ Links ]
7. Kopper G, Calderón G, Schneider S, Domínguez W, Gutiérrez G. Enfermedades transmitidas por alimentos y su impacto socioeconómico. Estudios de caso en Costa Rica, El Salvador, Guatemala, Honduras y Nicaragua. Informe técnico sobre ingeniería agrícola y veterinaria n° 6. Roma: Organización de las Naciones Unidas para la Agricultura y la Alimentación; 2009. [ Links ]
8. Crim SM, Iwamoto M, Huang JY, Griffin PM, Gilliss D, Cronquist A. Incidence and Trends of Infection with Pathogens Transmitted Commonly Through Food Food-borne Diseases Active Surveillance Network, 10 U.S. Sites, 2006-2013. Morbidity and Mortality Weekly Report (MMWR) [Internet] 2014; 63(15): 328-332. Disponible en:http://www.cdc.gov/mmwr/preview/mmwrhtml/mm6315a3.htm?s_cid=mm6315a3_w [ Links ]
9. European Food Safety Authority y European Centre for Disease Prevention and Control. The European Union Summary Report on Trends and Sources of Zoonoses, Zoonotic Agents and Food-borne Outbreaks in 2012. EFSA Journal 2014; 12(2):3547. Disponible en:http://www.efsa.europa.eu/en/efsajournal/doc/3547.pdf. [ Links ]
10. Instituto Nacional de Salud. Bogotá, D. C.: Sistema de Vigilancia en Salud Pública. Disponible en:http://www.ins.gov.co/lineasdeaccion/SubdireccionVigilancia/sivigila/Paginas/vigilanciarutinaria.aspx. [ Links ]
11. Pires S, Vieira A, Lo Fo Wong D. Attributing human foodborne illness to food sources and water in Latin America and the Caribbean using data from outbreak investigations. Int J Food Microbiol 2012; 152(3): 129-138. [ Links ]
12. Ministerio de Salud y Protección Social, Organización de las Naciones Unidas para la Alimentación y la Agricultura. Documento de Estado Actual de Inocuidad de los Alimentos. Bogotá, D. C.: Departamento de Salud Nutricional, Alimentos y Bebidas; 2013. [ Links ]
13. Hammack T. Salmonella sp. En: Food and Drug Administration, editor. Bad Bug Book, Food borne Pathogenic, Microorganisms and Natural Toxins. 2a ed.; 2012. p. 9-13. [ Links ]
14. Brenner F, Villar R, Angulo F, Tauxe R, Swaminathan. Salmonella Nomenclature. B. J Clin Microbiol 2000; 38 (7): 2465-2467. [ Links ]
15. Grimont PA, Weil FX. Antigenic formulas of the Salmonella serovars, 9th revision, World Health Organization Collaborating Centre for Reference and Research on Salmonella; Pasteur Institute, Paris: 2007. [ Links ]
16. Sánchez FM, Abu-El-Haija MA, Gomez OG, Salmonella infections: An update on epidemiology, management, and prevention. Travel Med Infect Dis 2011; 9: 263-277. [ Links ]
17. Chironna M, Tafuri S, Gallone MS, Sallustio A, Martinelli D, Prato R, Germinario C. Outbreak of Salmonella infantis gastroenteritis among people who had eaten at a hash house in southern Italy. Public health 2014; 128: 438-443. [ Links ]
18. Chambers J, Gong J. The intestinal microbiota and its modulation for Salmonella control in chickens. Food Res Int 2013; 44: 3149-3159. [ Links ]
19. Favier G, Estrada C, Lazarte V, Escudero M. Prevalence, antimicrobial susceptibility, and molecular characterization by PCR and pulsed field gel electrophoresis (PFGE) of Salmonella spp. isolated from foods of animal origin in San Luis, Argentina. Food Control2013; 29: 49-54. [ Links ]
20. Kramarenkoa T, Nurmojaa I, Karssina A, Meremae K, Hórmanc A, Roasto M. The prevalence and serovar diversity of Salmonella in various food products in Estonia. Food Control 2014; 42:43-47. [ Links ]
21. Zhoua Y, Pana Z, Li Y, Kang X, Wang X, Geng S, Liua Z, Jiao X, Liua X. Epidemiological analysis of Salmonella isolates recovered from food animals and humans in eastern China. Food Res Int 2013; 54(1): 223-229. [ Links ]
22. Favier G, Estrada C, Lazarte V, Escudero M. Prevalence, antimicrobial susceptibility, and molecular characterization by PCR and pulsed field gel electrophoresis (PFGE) of Salmonella spp. isolated from foods of animal origin in San Luis, Argentina. Food Control2013; 29: 49-54. [ Links ]
23. González J, Pereira N, Soto Z, Hernández E, Villarreal J. Aislamiento microbiológico de Salmonella spp. y herramientas moleculares para su detección. Salud Uninorte (Barranquilla, Col) 2014; 30(1): 73-94. [ Links ]
24. Villarreal J, Soto Z, Pereira N, Varela L, Jaramillo R, Villanueva, D, Mendoza E. Reacción en cadena de la polimerasa para la detección de Salmonella sp. en leche en polvo: Optimización del método en 12 horas. Salud Uninorte (Barranquilla, Col) 2008; 24 (2): 216225. [ Links ]
25. Clemente L, Correia L, Themudo P, Neto I, Canica M, Bernardo F. Antimicrobial susceptibility of Salmonella enterica isolates from healthy breeder and broiler flocks in Portugal. Vet J 2014; 200 (2) : 276-281 [ Links ]
26. Lai J, Wua C, Wua C, Qi J, Wanga Y, Wang H, Liub Y, Shen J. Serotype distribution and antibiotic resistance of Salmonella in food producing animals in Shandong province of China, 2009 and 2012. Int J Food Microbiol2014; 180: 30-38. [ Links ]
27. Jawale C, Lee J. Characterization of adaptive immune responses induced by anew genetically inactivated Salmonella enteritidis vaccine. Comp Immunol Microbiol Infect Dis2014; 37(3): 159-167. [ Links ]
28. Lerasle M, Guillou S, Simonin H, Anthoine V, Chéret R, Federighi M, Membré JM. Assessment of Salmonella and Listeria monocytogenes level in ready-to-cook poultry meat: Effect of various high pressure treatments and potassium lactate concentrations. Int JFood Microbiol 2014; 186:74-83. [ Links ]
29. Revista Bayona MA. Prevalencia de Salmonella y Enteroparásitos en alimentos y manipuladores de alimentos de ventas ambulantes y restaurantes en un sector del norte de Bogotá, Colombia. U.D.C.A Actualidad & Divulgación Científica 2012; 15(2): 267-274. [ Links ]
30. Méndez I, Badillo C, Ortiz G, Faccini A. Caracterización microbiológica de Salmonella en alimentos de venta callejera en un sector universitario de Bogotá, Colombia. Julio a octubre de 2010. MÉD. UIS 2011; 24(1): 26-33. [ Links ]
31. Serna L, Guarnizo A, Valencia L. Factores de Riesgo de ETAS en una comunidad universitaria en Colombia. Biotecnología en el Sector Agropecuario y Agroindustrial 2012; 10(1):116-26. [ Links ]
32. Murcia L, Sangama V, Algecira K. Determinación de Salmonella spp. en alimentos preparados de origen animal listos para consumo y alimentos crudos del municipio de Leticia, Amazonas, 2011. Inf Quinc Epidemiol Nac 2012;17(1):1-10. [ Links ]
33. Díaz MA, Díaz PL, Rodríguez EC, Montaño LA, Gartner DM, Vernaza ME et al. Brote de Salmonella Enteritidis resistente a ácido nalidíxico en Popayán, Cauca, 2011. Biomédica2013; 33(1): 62-9. [ Links ]
34. Loaiza J, Sánchez M, Henao S, Cardona N. Detección de Bacterias Contaminantes en huevos para consumo en Medellín y su Área Metropolitana. Revista CES Medicina Veterinaria y Zootecnia 2011; 6(2): 21-8. [ Links ]
35. Yang X, Wang H. Escherichia coli /Pathogenic E. coli (Introduction). En: Batt CA., editors. Encyclopedia of Food Microbiology. 2a ed. Oxford: Elsevier; 2014. p. 695-701. [ Links ]
36. Percival SL, Williams DW. Escherichia coli. En: Percival, Yates, Williams, Chalmers, Gray, editors. Microbiology of Waterborne Diseases, Microbiological Aspects and Risks. Oxford: Elsevier; 2014. p. 89-117. [ Links ]
37. Bibek R, Bhunia A. Fundamentos de Microbiología de los alimentos. 4a ed. México: McGrawHill; 2010. p. 202-222. [ Links ]
38. Estrada T, Lopez C, Thompson R, Abonce M, Lopez-Santos J, Rosado J, DuPont H, Long K. Association of diarroheagenic Escherichia coli pathotypes with infection and diarrhea among Mexican children and association of atypical enteropathogenic E. coli with acute diarrhea. J. Clin. Microbiol 2009; 47: 93-98. [ Links ]
39. Buchholz U, Bernard H, Werber D, Bohmer M, Remschmidt C, Wilking H et al. German outbreak of Escherichia coli O104:H4 associated with sprouts N. Engl. J. Med 2011; 365(19): 1763-1770. [ Links ]
40. Portera CK, Riddlea MS, Tribbleb DR, Putnamc SD, Rockabrandd DM, Frenckd R et al. The epidemiology of travelers' diarrhea in Incirlik, Turkey: a region with a predominance of heat-stabile toxin producing ente-rotoxigenic Escherichia coli. Diagn Microbiol Infect Dis 2010; 66(3): 241-247. [ Links ]
41. Chokoshvili O, Lomashvili K, Malakmadze N, Geleishvil M, Brant J, Imnadze P et al. Investigation of an outbreak of bloody diarrhea complicated with hemolytic uremic síndrome. J Epidemiol Glob Health 2014; 4(4): 249-259. [ Links ]
42. Croxen MA, Law RJ, Scholz R, Keeney KM, Wlodarska MM, Finlay B. Recent advances in understanding enteric pathogenic Escherichia coli. Clin Microbiol Rev 2013; 26(4): 822880. [ Links ]
43. Kaper J, Nataro J, Mobley H. Pathogenic Escherichia coli. Nat Rev. Microbiol 2004; 2 (2): 123-140. [ Links ]
44. Canizalez A, Gonzalez E, Vidalc JE, Flores H, León N. Prevalence and antibiotic resistance profiles of diarrheagenic Escherichia coli strains isolated from food items in northwestern Mexico. Int J Food Microbiol 2013; 164 (1): 36-45. [ Links ]
45. Sallam KI, Mohammed MA, Ahdy AM, Tamura T. Prevalence, genetic characterization and virulence genes of sorbitol-fermenting Escherichia coli O157:H-and E. coli O157:H7 isolated from retail beef. Int J Food Microbiol 2013; 165 (3): 295-301. [ Links ]
46. Kagambéga A, Martikainen O, Lienemann T, Siitonen A, Traoré A, Barro N et al. Diarrheagenic Escherichia coli detected by 16-plex PCR in raw meat and beef intestines sold at local markets in Ouagadougou, Burkina Faso. Int J Food Microbiol 2012; 1: 154-158. [ Links ]
47. Charimba G, Hugo C, Hugo A. The incidence of diarrhoeagenic Escherichia coli in minced beef and boerewors. Food Res Int 2012; 47: 353-8. [ Links ]
48. Cerna J, Gómez C, Rangel E, Ramírez E, Castro J. Presence of indicator bacteria, Salmonella and diarrheagenic Escherichia coli pathotypes on mung bean sprouts from public markets in Pachuca, Mexico. Food Control 2013; 31(2): 280-283. [ Links ]
49. Badri S, Filliol I, Carle I, Hassar M, Fassouane A, Cohen N. Prevalence of virulence genes in Escherichia coli isolated from food in Casablanca (Morocco). Food Control 2009; 20 (6): 560-564. [ Links ]
50. Bandyopadhyay S, Lodh C, Rahaman H, Bhattacharya D, Bera AK, Ahmed FA et al. Characterization of shiga toxin producing (STEC) and enteropathogenic Escherichia coli (EPEC) in raw yak (Poephagus grunniens) milk and milk products. Res Vet Sci 2012; 93 (2): 604-610. [ Links ]
51. Cramer JP. Enterohemorrahagic Escherichia coli (EHEC): Hemorrhagic Colitis and Hemolytic Uremic Syndrome (HUS). En: Emerging Infectious Diseases Clinical Case Studies. (Northem Germany) 2014; 213-227. [ Links ]
52. Andrea V. Page W. Conrad L. Enterohemorrhagic Escherichia coli Infections and the Hemolytic-Uremic Syndrome. Med Clin North Am 2013; 97(4): 681-95. [ Links ]
53. Espié E, Mariani-Kurkdjian P, Filliol I, Vaillant V, Valk H. Infections humaines á E. coli producteurs de Shigatoxines en France: aspects cliniques, diagnostiques et épidémiologiques. Revue Francophone des Laboratoires 2008; 2008(400): 59-65. [ Links ]
54. Riley L, Remis R, Helgerson S, McGee H, Wells J, Davis B et al. Hemorrhagic colitis associated with a rare Escherichia coli serotype. N Engl J Med 1983; 308: 681-5. [ Links ]
55. Jones I, Roworth M. An outbreak of Escherichia coli 0157 and campylobacteriosis associated with contamination of a drinking water supply. Public Health 1996; 110(5): 277282. [ Links ]
56. Gelting R, Baloch M, Zarate M, Selman C. Irrigation water issues potentially related to the 2006 multistate E. coli O157:H7 outbreak associated with spinach. Agricultural Water Management 2011; 98 (9):1395-1402. [ Links ]
57. Crump JA, Sulka AC, Langer AJ, Schaben C, Crielly AS, Gage R et al. An Outbreak of Escherichia coli O157:H7 Infections among Visitors to a Dairy Farm. N Engl J Med 2002; 347(8): 555-560. [ Links ]
58. Kuntz T, Kuntz S. Enterohemorrhagic E. Coli infection. Prim Care Update Ob Gyns 1999; 6(6):192-96. [ Links ]
59. Bari ML, Inatsu Y. Escherichia coli 0157 / E. coli O157:H7. Encyclopedia of Food Microbiology. 2a ed. Japan: Elsevier; 2014. p. 735-739. [ Links ]
60. Franco P, Ramírez L, Orozco M, Ugarriza, López L. Determinación de Escherichia coli e identificación del serotipo O157:H7 en carne de cerdo comercializada en los principales supermercados de la ciudad de Cartagena. Rev Lasallista Investig 2013; 10(1): 91-100. [ Links ]
61. Rúgeles L, Bai J, Martínez A, Vanegas M, Gómez-Duarte O. Molecular characterization of diarrheagenic Escherichia coli strains from stools samples and food products in Colombia. Int J Food Microbiol 2010; 138(3): 282-286. [ Links ]
62. Herrera F, Suárez W. Aislamiento e Identificación de histeria spp. a partir de muestras de pescado fresco expendido en Pamplona (Norte de Santander). Rev. U.D.CA Act. & Div. Cient 2012; 15(2): 257-65. [ Links ]
63. Listeria [Internet] [fecha de acceso: septiembre de 2014]. Disponible en:http://www.bacterio.net/listeria.html. [ Links ]
64. Walker S, Archer P, Banks J. Growth of Listeria monocytogenes at refrigeration temperatures. J Appl Bacteriol 1990; 68(2): 157-162. [ Links ]
65. Kramarenko T, Roasto M, Meremae K, Kuningas M, Póltsama P, Elias T. Listeria monocytogenes prevalence and serotype diversity in various foods. Food Control 2013; 30(1): 24-29. [ Links ]
66. Martín M, Perich A, Gómez D, Yangüela J, Rodríguez A, Garriga M et al. Diversity and distribution of Listeria monocytogenes in meat processing plants. Food Microbiol 2014; 44: 119 -127. [ Links ]
67. Nightingale K, Schukken Y, Nightingale C, Fortes E, Ho A, Her Z et al. Ecology and transmission of Listeria monocytogenes infecting ruminants and in the farm environment. Appl Environ Microbiol 2004; 70(8): 4458-4467. [ Links ]
68. Martin B, Garriga M, Aymerich T. Prevalence of Salmonella spp. and Listeria monocytogenes at smallscale Spanish factories producing traditional fermented sausages. J. FoodProt 2011; 74: 812-815. [ Links ]
69. Wulff G, Gram L, Ahrens P, Vogel BF. One group of genetically similar Listeria monocytogenes strains frequently dominates and persists in several fish slaughter and smokehouses. Appl Environ Microbiol 2006; 72 (6): 4313-4422. [ Links ]
70. Carpentier B, Cerf O. Persistence of Listeria monocytogenes in food industry equipment and premises. Int J Food Microbiol 2011; 145(1): 1-8. [ Links ]
71. Ortiz S, López V, Martínez JV. Control of Listeria monocytogenes contamination in an Iberian pork processing plant and selection of benzalkonium chlorideresistant strains.Food Microbiol 2014; 39: 81- 88. [ Links ]
72. Pan Y, Breidt F, Kathariou S. Resistance of Listeria monocytogenes biofilms to sanitizing agents in a simulated food processing environment. Appl Environ Microbiol 2006; 72(12): 7711-7717. [ Links ]
73. Rodriguez A, Autio WR, McLandsborough LA. Effect of biofilm dryness on the transfer of Listeria monocytogenes biofilms grown on stainless steel to bologna and hard salami. J Food Prot 2007; 70: 2480-2484.
74. Swaminathan B, Gerner P. The epidemiology of human listeriosis. Microbes Infect.Epub 2007; 9(10): 1236 -1243. [ Links ]
75. DiMaio H. Listeria infection in women. Primary Care Update for Ob/Gyns 2000; 7(1): 40-45. doi:10.1016/S1068-607X(99)00039-6 [ Links ]
76. Silk B, Date K, Jackson K, Pouillot R, Holt K, Graves L et al. Invasive listeriosis in the foodborne diseases active surveillance network (FoodNet), 2004-2009: further targeted prevention needed for higherrisk groups. Cli Infec Dis 2012; 54 (5): 396-404. [ Links ]
77. Chen M, Wu Q, Zhang J, Yan Z, Wang J. Prevalence and characterization of Listeria monocytogenes isolated from retaillevel ready to eat foods in South China. Food Control 2014; 38:1-7. [ Links ]
78. Beale DJ, Morrison PD, Palombo EA. Detection of Listeria in milk using nontargeted metabolic profiling of Listeria monocytogenes: A proof of concept application. Food Control 2014; 42: 343-346. [ Links ]
79. Schoder D, Stessl B, Szakmary K, Rossmanith P, Wagner M. Population diversity of Listeria monocytogenes in quargel (acid curd cheese) lots recalled during the multinational listeriosis outbreak 2009-2010. Food Microbiol 2014; 39: 68-73. [ Links ]
80. Lundén J, Tolvanen R, Korkeala H. Listeriosis Outbreaks Linked to Dairy Products in Europe. J Dairy Sc 2004, 87: 6-12. [ Links ]
81. Tham W, Ericsson H, Loncarevic S, Unnerstad H, Danielsson ML. Lessons from an outbreak of listeriosis related to vacuumpacked gravad and coldsmoked fish. Inter J Food Microbiol 2000; 62(3): 173 -175. [ Links ]
82. Little CL, Amar CF, Awofisayo A, Grant. Hospital acquired listeriosis associated with sandwiches in the UK: a cause for concern. J Hosp Infec 2012; 82 (1): 13 -18. [ Links ]
83. Gamboa A, Buitrago S, Pérez K, Mercado M, Poutou R, Carrascal A. Prevalencia de Listeria monocytogenes en carnes y derivados de la industria porcícola colombiana. Rev. MVZ Córdoba 2012; 17(1): 2827-2833. [ Links ]
84. Jamali H, Thong K. Genotypic characterization and antimicrobial resistance of Listeria monocytogenes from ready to eat foods. FoodControl 2014; 44:1-6. [ Links ]
85. Yu T, Jiang X. Prevalence and characterization of Listeria monocytogenes isolated from retail food in Henan, China. Food Control 2014; 37: 228-231. [ Links ]
86. Kramarenko T, Roasto M, Meremae K, Kuningas M, Póltsama P, Elias T. Listeria monocytogenes prevalence and serotype diversity in various foods. Food Control 2013; 30(1): 24-29. [ Links ]
87. Makino SI, Kawamoto K, Takeshi K, Okada Y, Yamasaki M, Yamamoto S. An outbreak of food borne listeriosis due to cheese in Japan, during 2001. Int J Food Microbiol 2005; 104(2): 189 -196. [ Links ]
88. Swaminathan B, Gerner-Smidt P. The epidemiology of human listeriosis. Microbes Infect 2007; 9(10): 1236-1243. [ Links ]
89. Gerner-Smidt P, Ethelberg S, Schiellerup P, Christensen JJ, Engberg J, Fussing V et al. Invasive listeriosis in Denmark 1994-2003: a review of 299 cases with special emphasis on risk factors for mortality. Clin MicrobiolInfect 2005; 11(8): 618-624. [ Links ]
90. Muñoz A. Distribución de serotipos de Listeria monocytogenes aislados de alimentos, Colombia, 2000-2009. Biomédica 2012; 32(3): 408-17. [ Links ]
91. Muñoz A, Vargas M, Otero L, Díaz G, Guzmán V. Presencia de Listeria monocytogenes en alimentos listos para el consumo, procedentes de plazas de mercado y delicatessen de supermercados de cadena, Bogotá, D. C., 2002-2008. Biomédica 2011; 31(3): 428-39. [ Links ]
92. Vanegas M, Medrano M, Martínez A, Arévalo S. Molecular serotyping and identification of the 85M gragment in different Colombian isolates of Listeria monocytogenes strains: A descriptive study. Colombia Med 2012; 43(1): 38-45. [ Links ]
93. Percival S, Williams D. Vibrio. En: Percival, Yates, Williams, Chalmers, Gray, editors. Microbiology of Waterborne Diseases, Microbiological Aspects and Risks. Oxford: Elsevier; 2014. p. 237-248. [ Links ]
94. Mandal S, Mandal M. VIBRIO Vibrio cholerae En: Batt CA, editor. Encyclopedia of Food Microbiology. 2a ed. Oxford: Elsevier; 2014. p. 708-716. [ Links ]
95. León A, Acedo FE, Gomez B, Quiñones EI, Nevárez M, Noriega L. Relationship of aquatic environmental factors with the abundance of Vibrio cholerae, Vibrio parahaemolyticus, Vibrio mimicus and Vibrio vulnificus in the coastal area of Guaymas, Sonora, Mexico. J Water Health 2013; 11 (4): 700-712. [ Links ]
96. Thongchankaew U, Mittraparparthorn P, Sukhumungoon P, Tansila N, Nuidate T, Nishibuchi M, and Vuddhaku V. Occurrence of potentially pathogenic vibrios and related environmental factors in Songkhla Lake, Thailand. Can. J. Microbiol 2011; 57: 867- 873. [ Links ]
97. Castro J, Escartín E. Increased tolerance of Vibrio cholerae O1 to temperature, pH or drying associated with colonization of shrimp carapaces. Int J Food Microbiol 2005; 102(2): 195-201. [ Links ]
98. Vibrio [Internet] [fecha de acceso: septiembre 2014]. Disponible en:http://www.bacterio.net/vibrio.html [ Links ]
99. Fuenzalida L, Armijo L, Zabala B, Hernández C, Rioseco ML, Riquelme C, Espejo RT. Vibrio parahaemolyticus strains isolated during investigation of the summer 2006 seafood related diarrhea outbreaks in two regions of Chile. Int J Food Microbiol 2007; 117(3): 270-275. [ Links ]
100. Ottaviania D, Leonia F, Rocchegiania E, Santarellia S, Masinia L, Trania. Prevalence and virulence properties of non-O1 non-O139 Vibrio cholerae strains from seafood and clinical samples collected in Italy. Int J Food Microbiol 2009; 132(1): 47-53 [ Links ]
101. Wu A, Wen B, Ma Y, Ma Y, Chen Y. Epidemiology of foodborne disease outbreaks caused by Vibrio parahaemolyticus, China, 2003-2008. Food Control 2014; 46: 197-202. [ Links ]
102. Johnston JM, Becker SF, McFarland LM. Gastroenteritis in patients with stool isolates of Vibrio vulnificus. The Amer J Med 1986; 80(2): 336-338.
103. Robert A, Copin S, Himber C, Gay M, Quilici ML. Occurrence of the three major Vibrio species pathogenic for human in seafood products consumed in France using real time PCR. Int J Food Microbiol 2014; 189: 75-81. [ Links ]
104. Food, Drug Administration & World Health Organization (FAO & WHO). Risk assessment of choleragenic Vibrio cholerae O1 and O139 in warm water shrimp in international trade Food and Agriculture Organization. Rome, 2005. [ Links ]
105. Matsumoto K, Ohshige K, Fujita N, Tomita Y, Mitsumizo S, Nakashima M et al, Clinical features of Vibrio vulnificus infections in the coastal areas of the Ariake Sea, Japan. 2010. J Inf Chem 2010; 16(4):272-279. doi: 10.1007/s10156-010-0050-z. [ Links ]
106. Cazorla C, Guigon A, Noel M, Quilici M, Lacassin F. Fatal Vibrio vulnificus infection associated with eating raw oysters, New Caledonia Emerg. Infect. Dis 2011; 17(1): 136137. [ Links ]
107. Canigrala I, Morenoa Y, Alonsob JL, Gonzalez A, Ferrusa MA, Detection of Vibrio vulniíicus in seafood, seawater and wastewater samples from a Mediterranean coastal area. Microbiol Res 2010; 165: 657-64. [ Links ]
108. Baffone W, Pianetti A, Bruscolini F, Barbieri E, Citterio B. Occurrence and expression of virulence related properties of Vibrio species isolated from widely consumed seafood products. Int J Food Microbiol 2000; 54 (1):9-18. [ Links ]
109. Yano Y, Hamano K, Satomi M, Tsutsui I, Ban M, Aueumneoy D. Prevalence and antimi crobial susceptibility of Vibrio species related to food safety isolated from shrimp cultured at inland ponds in Thailand. Food Control 2014; 38: 30-36. [ Links ]
110. López AL, Manjarrez G, Herrera L, Montes A, Olascuaga Y, Ortega R. Aislamiento de Vibrio spp. en ostras (Crassostrea rhizophorea) capturadas en la Ciénaga de la Virgen. Ciencia actual 2011; 1(1): 2248-22468. [ Links ]
111. Lampel KA. Aeromonas species. En: Food and Drug Administration, editor. Bad Bug Book, Food borne Pathogenic, Microorganisms and Natural Toxins. 2a ed, 2012: 54-56. [ Links ]
112. Michael AJ, Abbott SL. The Genus Aeromonas: Taxonomy, Pathogenicity, and Infection. Cli Microbiol Rev 2010; 23 (1): 35-73. [ Links ]
113. Vila J, Ruiz J, Gallardo F, Vargas M, Soler L, Figueras M et al. Aeromonas spp. and Traveler's Diarrhea: Clinical Features and Antimicrobial Resistance. Emerg Infect Dis 2003; 9(5): 552-555. [ Links ]
114. Coscelli A, Bermúdez R, Losada A, Faílde L, Santos I, Quiroga M. Acute Aeromonas salmonicida infection in turbot (Scophthalmus maximus L.). Histopathological and immunohistochemical studies. Aquaculture 2014; 430:79-85. [ Links ]
115. Ottaviani D, Parlani C, Citterio B, Masini L, Leoni F, Canonico C et al. Putative virulence properties of Aeromonas strains isolated from food, environmental and clinical sources in Italy: A comparative study. Int J Food Microbiol 2011 5; 144(3): 538-545. [ Links ]
116. Parker J, Shaw J. Aeromonas spp. clinical microbiology and disease. J Infect 2011; 62: 109118. [ Links ]
117. Pablos M, Huys G, Cnockaert M, Rodríguez-Calleja J, Otero A, Santos J et al. Identification and epidemiological relationships of Aeromonas isolates from patientswith diarrhea, drinking water and foods. Int J Food Microbiol 2011 Jun 30; 147(3): 203-210. [ Links ]
118. Suárez W, Herrera F. Aislamiento de Aeromonas spp. en muestras de pescado fresco comercializado en Pamplona (Norte de Santander). Rev. U.D.CA Act. & Div. Cient 2011;14(2): 7-13. [ Links ]
119. William A, Herrera F. Determinación de factores de virulencia en cepas de Aeromonas spp., aisladas a partir de pescado. Rev MVZ Córdoba 2012; 17(1): 2846-2851. [ Links ]