Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Salud Uninorte
Print version ISSN 0120-5552On-line version ISSN 2011-7531
Salud, Barranquilla vol.32 no.2 Barranquilla May/Aug. 2016
Determinación del ensamblaje genético de aislados axénicos colombianos de Giardia intestinalis
Genetic assemblages of axenic Colombian cultures of Giardia intestinalis
Paula C. Hernández Atehortúa1, Jacqueline Chaparro Olaya1, Liliana Morales de La Pava1
1 Docentes investigadoras. Laboratorio de Parasitología Molecular, Universidad El Bosque, Bogotá, D.C. (Colombia).
Correspondencia: Paula C. Hernández A. Av. Cra. 9 n° 131 A-02. Bogotá, D. C. Teléfono: 6489000, ext. 1291. hernandezpaula@unbosque.edu.co
Fecha de recepción: 30 de mayo de 2015
Fecha de aceptación: 10 de abril de 2016
Resumen
Objetivo: Genotipificar muestras de Giardia aisladas de pacientes de tres regiones geográficas de Colombia.
Materiales y métodos: Se examinaron 22 aislados de G. intestinalis, los primeros descritos cultivados axénicamente de origen colombiano por medio del análisis del polimorfismo de longitud de fragmentos de restricción (RFLP) del gen glutamato deshidrogenasa (gdh).
Resultados y conclusión: El patrón del RFLP mostró que todos los aislados pertenecían al ensamblaje A, específicamente al AI; resultado confirmado por secuenciación. Con este estudio se pretende contribuir al conocimiento de los genotipos circulantes de Giardia intestinalis en Colombia.
Palabras clave: Giardia intestinalis, ensamblaje, genotipificación, gdh, RFLP.
Abstract
Objective: To genotype samples of G. intestinalis isolated from patients of three Colombian regions.
Materials and methods: 22 isolates of Giardia (all of them established as axenic cultures) were examined by analyzing restriction fragment length polymorphism (RFLP) of the glutamate dehydrogenase gene (gdh).
Results and conclusion: The RFLP patterns showed that all of the Giardia isolates belonged to the assembly A, specifically to AI. This result was confirmed by sequencing. This study aims to contribute to the knowledge of circulating genotypes of Giardia intestinalis in Colombia.
Keywords: Giardia intestinalis, assemblage, genotyping, gdh, RFLP.
Introducción
Giardia intestinalis es un parásito protozoario flagelado que habita el intestino delgado de humanos y de otros mamíferos y causa una enfermedad conocida como giardiasis.
La población infantil que vive en condiciones sanitarias precarias es la más susceptible al contagio con Giardia.
Según el Instituto Nacional de Salud, en la Investigación Nacional de Morbilidad realizada en 1980 se estableció que en Colombia Giardia tenía una prevalencia del 12 % en la población general y del 28 % en niños entre 1 y 4 años (1).
Los parásitos de G. intestinalis muestran muy pocas diferencias morfológicas entre sí, pero constituyen una especie genéticamente compleja que agrupa ocho distintos ensamblajes o genotipos (A-H) (2, 3). Entre ellos, los parásitos con ensamblaje A, subgrupos AI y AII, y los parásitos con ensamblaje B, subgrupos BIII y BIV, son los únicos que infectan humanos (4). En 2001 apareció el primer reporte en el que se relacionaban estos tipos genéticos con las manifestaciones clínicas de la giardiasis (5); en ese estudio se observó una correlación del 100 % entre la severidad de la enfermedad y el genotipo de Giardia, ya que el ensamblaje A se encontró solo en pacientes con quejas intermitentes de diarrea, mientras que el ensamblaje B fue aislado de pacientes con diarrea persistente. Dos estudios posteriores, en2002y 2005, encontraron también una fuerte relación entre el ensamblaje A e infecciones sintomáticas y entre el ensamblaje B e infecciones asintomáticas (6, 7). Sin embargo, actualmente hay controversia sobre la relación entre el genotipo de Giardia y la sintomatología, ya que otros autores han demostrado que factores intrínsecos al paciente, como por ejemplo la edad, podrían afectar las manifestaciones clínicas de la giardiasis, más que el tipo de ensamblaje per se (8, 9).
En Latinoamérica, principalmente en México, hay reportes de genotipificación de cepas de Giardia donde se encontró una predominancia del ensamblaje A en aislados de humanos (10-12). También hay un estudio de entero-parásitos en niños de Perú, donde todos los aislados de Giardia pertenecían al ensamblaje A, subgrupos AI y AII, y al ensamblaje B. En esta población hubo predominio del ensamblaje AI, y todos los niños infectados con este genotipo presentaron diarrea (13). Igualmente, en Argentina hay un reporte de caracterización genética de aislados de Giardia en niños y adultos, donde se encontró una alta prevalencia del ensamblaje B en individuos polisintomáticos, la mayoría con diarrea, mientras que todos los pacientes sin diarrea tenían parásitos del ensamblaje AII (14). En Colombia, los reportes epidemiológicos que existen de las parasitosis intestinales se circunscriben a colegios, veredas o pequeñas comunidades (15-18). En algunos de estos estudios se muestra una dominancia de parásitos del ensamblaje B (17, 19), mientras que en otros los ensamblajes A y B se encontraron en la misma proporción (20). En este estudio se analizó el ADN de aislados axénicos de G. intestinalis de pacientes de tres diferentes regiones del país, recolectadas entre 1997 y 2001, que presentaron diversos cuadros clínicos de malestar gastrointestinal.
Materiales y métodos
Origen del ADN de Giardia intestinalis
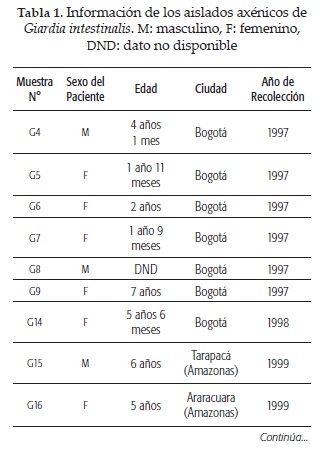
El ADN analizado provenía de parásitos de Giardia obtenidos de 22 aislados de hombres y mujeres de distintas edades, procedentes de tres regiones colombianas: Amazonas, Boyacá y Bogotá (Distrito capital) (tabla 1 y figura 1). Estas muestras fueron recolectadas en un período de 5 años y los aislados fueron establecidos por Ravid y colaboradores en un trabajo anterior (21). En ese estudio, las muestras infectadas con quistes de Giardia fueron usadas para inocular gerbils (Meriones unguiculatus) y los trofozoitos recuperados del intestino delgado de los animales se axenizaron y fueron usados para extraer el ADN (21).
PCR-RFLP
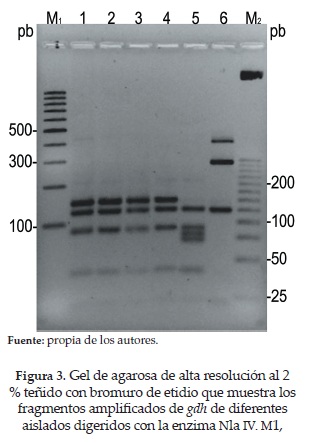
El ADN extraído se usó como plantilla en un PCR en el que se utilizaron los oligonucleótidos GDHiF y GDHiR (22) para amplificar un fragmento de 432 pares de bases (pb) del gen glutamato deshidrogenasa (gdh). Las mezclas de reacción de PCR se ajustaron a un volumen de 30 ul y contenían 0.05 uM de cada oligonucleótido, 1.5 mM de MgCl2, 200 uM de dNTPs, 0.1 U de GoTaq Flexi ADN polimerasa (Promega), buffer de PCR y 50 ng de ADN genómico. El PCR se llevó a cabo en un termociclador GeneAmp 2400 (Perkin Elmer) con las siguientes condiciones de amplificación: un paso inicial de denaturación a 94°C (10 min), 35 ciclos de 94°C (35 s), 61°C (35 s) y 72°C (50 s), con un paso de elongación final de 72°C (7 min). Como control positivo se usó ADN de los siguientes aislamientos de Giardia: de la cepa WBC6 para el ensamblaje A, subgrupo AI; de la cepa Bris-136 para el ensamblaje A, subgrupo AII, y de la cepa Ad-28 para el ensamblaje B, subgrupo BIV. A continuación, 10 ul de cada reacción de PCR fueron sometidos a digestión con NlaIV (NewEngland Biolabs), por 3 horas a 37°C (22). Los productos de PCR y los productos de digestión se sometieron a electroforesis en geles de agarosa de amplio rango (Sigma), se tiñeron con bromuro de etidio y fueron visualizados en un analizador de imágenes UV Gel Doc 1000 (BioRad).
Secuenciación y análisis filogenético
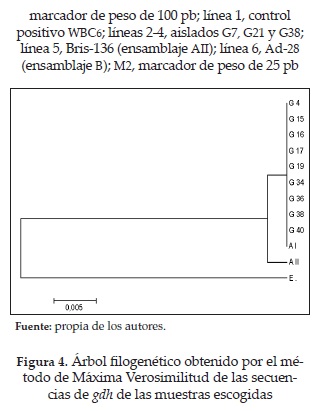
Los productos de PCR de 9 muestras (G4, G15, G16, G17, G19, G34, G36, G38 y G40), escogidas con base en el origen geográfico y el año de recolección (tabla 1), fueron secuenciados en ambas direcciones por MACROGEN (Seoul, Korea). Los cromatogramas y las secuencias se editaron usando BioeditTM. El alineamiento múltiple se realizó con el programa ClustalW 1.6 y como referencia se usó una secuencia de gdh perteneciente al ensamblaje AI (número de acceso en el GenBank: L40509), una secuencia de gdh perteneciente al ensamblaje AII (L40510.1) y una secuencia de gdh perteneciente al ensamblaje E, no infectivo de humanos (U47632.1). El alineamiento fue evaluado para encontrar el modelo evolutivo más adecuado por medio del programa MEGA 6 TM. Se usó un análisis de Máxima Verosimilitud usando el modelo Tamura-3 parameter para generar un árbol filogenético con el más alto valor de logaritmo de verosimilitud. La robustez de este árbol fue evaluada por medio de un boostrap de 1000 réplicas. Todo el análisis evolutivo fue realizado en MEGA 6TM (23).
Resultados
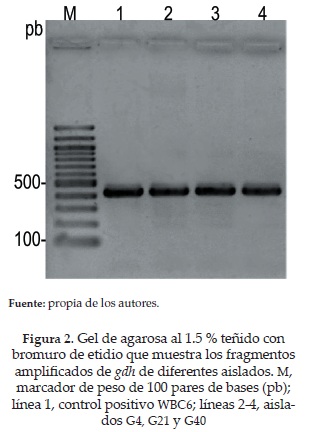
En los ensayos de PCR para amplificar gdh se obtuvo el fragmento esperado de 432 pb para las 22 muestras analizadas. La figura 2 muestra el producto obtenido para tres aislados representativos. En los ensayos de RFLP, los perfiles de restricción generados por la enzima Nla IV permiten discriminar entre los ensamblajes AI (perfil diagnóstico de fragmentos: 40,90, 120 y 150 pb), AII (40, 70, 80, 90 y 120 pb), C (70, 120 y 190 pb), D (120 y 250 pb), E (80, 100 y 220 pb) y BIII/BIV (120 y 290 pb) (22). En la figura 3 se ve que la cepa referencia WBC6 produjo el perfil de restricción diagnóstico para ensamblaje subgrupo AI (línea 1), la cepa referencia Bris-136 produjo el perfil de restricción diagnóstico para ensamblaje A, subgrupo AII (línea 5) y la cepa referenciaAd-28 produjo el perfil de restricción diagnóstico para el ensamblaje B (línea 6). Del mismo modo, la figura muestra tres aislamientos (líneas 2 a 4) cuyo patrón de restricción fue igual al obtenido para la cepa WBC6. Se obtuvo este mismo patrón con las 22 muestras, lo cual indicó que todos los aislamientos axénicos de Giardia que fueron analizados pertenecían al ensamblaje AI. Para confirmar estos resultados se secuenciaron los productos de PCR (del gen gdh) de 9 aislamientos (G4, G15, G16, G17, G19, G34, G36, G38 y G40) y las secuencias resultantes fueron alineadas con secuencias de referencia para los aislamientos AI, AII y E, obtenidas del Genbank. El árbol filogenético de la figura 4 muestra que todos los aislados pertenecen al genotipo AI.
Discusión y conclusiones
La caracterización molecular de aislados de Giardia es importante no solo porque se conoce muy poco acerca de la estructura poblacional de las cepas circulantes en Colombia, sino también porque se ha asociado la variabilidad genética de los parásitos con la virulencia, y por tanto, con los diversos cuadros clínicos que pueden presentarse durante el transcurso de la giardiasis.
En Colombia, tres reportes han mostrado la prevalencia de los ensamblajes de G. intestinalis en tres áreas del país. En un primer estudio en niños de Ibagué se reportó una distribución similar de los ensamblajes A (subensamblaje AII) y B, con un 48 y 52 %, respectivamente (20). En un segundo estudio, en muestras de niños y perros de la costa atlántica, se encontró gran predominancia del genotipo B (92.3 %) sobre el ensamblaje A (5.1 %) (19). Finalmente, en un tercer estudio en una población de Cundinamarca, el 90 % de las infecciones fueron causadas por el genotipo B, mientras que el ensamblaje A, o las infecciones mezcladas A+B, solo se detectaron en el 3 y 7 % de las muestras (17).
Este trabajo analizó muestras de ADN de Giardia con el fin de determinar los ensamblajes genéticos a los que pertenecían. Las muestras habían sido analizadas anteriormente por medio de la técnica de RAPD (21), y según los resultados de ese estudio, todas las muestras pertenecían al ensamblaje A. Debido a esa homogeneidad y teniendo en cuenta los estudios previos en los que se encontró que en muestras colombianas el ensamblaje B podía estar en la misma o incluso en mayor proporción que el ensamblaje A (19-21), nos preguntamos si al utilizar una técnica con una mejor discriminación podríamos encontrar resultados diferentes.
La genotipificación de aislados de Giardia por medio de la técnica RFLP usando el gen gdh ha demostrado ser sensible, confiable y reproducible para distinguir entre aislados de Giardia de origen animal o humano. Sin embargo, también se ha visto que para los mismos aislamientos no siempre hay concordancia entre el ensamblaje asignado al genotipificar con gdh y al genotipificar con otros marcadores moleculares (22).
En nuestro estudio, los tipos genéticos fueron determinados por PCR-RFLP basada en el análisis del gen gdh, y se hizo confirmación por secuenciación. En ningún caso se obtuvieron patrones de restricción mezclados, y tampoco se evidenciaron picos dobles en los cromatogramas que delataran la potencial presencia de infecciones mixtas.
La concordancia de resultados con el estudio de 2008 demuestra que no hubo ensamblajes no detectados en ese trabajo y que al menos para este grupo de muestras en particular, el ensayo de RFLP (usando el gen gdh) estableció correctamente el tipo de ensamblaje en todos los casos. Basados en estos resultados, la conclusión es que todos estos parásitos, adaptados a cultivo, corresponden específicamente al ensamblaje A, subensamblaje AI.
Los resultados que obtuvimos son similares a los de un estudio en el que se usaron 26 aislados mexicanos, también axenizados (11), en el que igualmente se encontró que todos ellos pertenecían al ensamblaje AI.
A pesar de la homogeneidad genética de los aislados examinados en este estudio, ellos se derivan de un período de 5 años de recolección y de un grupo heterogéneo de individuos, con diferentes sintomatologías. En efecto, las muestras provenían de niños y adultos de regiones separadas por una gran distancia geográfica, y aunque no se tenía registro de la sintomatología de todos los pacientes en el momento de la toma de la muestra, para aquellos que sí existía esa información, las manifestaciones clínicas fueron desde diferentes grados de gastroenteritis hasta giardiasis crónica. Con base en esto, es evidente que no se puede hacer ninguna correlación entre el tipo de ensamblaje, que fue el mismo para todos, y la sintomatología de los pacientes, que fue muy diversa. Por supuesto, es necesario suponer que debe haber otros factores determinantes del resultado de la infección, y que esos factores, asociados al huésped o al parásito, no son detectados por la genotipificación del gen gdh.
En este punto es necesario anotar que se han encontrado diferencias entre los perfiles genéticos de quistes aislados de muestras clínicas, y de los parásitos que se establecen en cultivos axénicos a partir de esos quistes. De hecho, se ha visto que la axenización puede favorecer el crecimiento de parásitos del ensamblaje A, y esta evidencia ha permitido plantear que los diferentes genotipos en una muestra pueden ser seleccionados durante la infección en el animal o en el cultivo "in vitro" (24). En la misma vía están los resultados del trabajo de Bénéré y colaboradores, quienes rastrearon por PCR el tipo de ensamblaje de los quistes purificados de una muestra clínica, de los trofozoitos que obtuvieron al infectar gerbils con esos quistes y de los cultivos axénicos establecidos "in vitro" a partir de esos trofozoitos. Para los quistes de la muestra original, en el PCR únicamente se obtuvieron bandas para el ensamblaje B, mientras que una vez establecido el cultivo axénico, el PCR solo identificó parásitos con el ensamblaje A (25).
Los autores explican este resultado proponiendo que la muestra original contenía una mezcla de ambos genotipos, con una mínima proporción de parásitos con ensamblaje A, pero que fueron precisamente estos últimos los que crecieron más eficientemente "in vitro".
Del mismo modo, un estudio reciente analizó por PCR dos poblaciones de parásitos obtenidas de dos pacientes, y encontró que uno de los aislados tenía pequeñas trazas de ensamblaje B, mientras que el otro contenía exclusivamente el ensamblaje A. Sin embargo, la misma prueba después de 10 pasajes de cultivo "in vitro" detectó únicamente el genotipo A para ambos aislamientos (26).
Estos trabajos refuerzan resultados previos que mostraron que el ensamblaje B tenía más dificultades para adaptarse al cultivo "in vitro" y que eso favorecía el crecimiento del ensamblaje A (27).
Por todo lo anterior, no puede descartarse que en las muestras originales, de donde provienen los aislamientos que se usaron en este estudio, hubiera habido parásitos con ensamblaje B, que luego desaparecieron durante la adaptación al cultivo "in vitro". Sin embargo, también es cierto que hay disponibles aislados de origen humano que son referencia para el ensamblaje B (BAH12, BRIS/694, Ad-19, Ad-22, Ad-28, Ad-45, Ad-52), los cuales fueron exitosamente establecidos como cultivo axénico (BAH12, BRIS/694), o propagados infectando ratones (Ad-19, Ad-22, Ad-28, Ad-45, Ad-52) (2). Del mismo modo, hay algunos reportes que van en contravía de esta desventaja adaptativa adjudicada a los parásitos del ensamblaje B. Por ejemplo, en una trabajo en el que se establecieron cultivos axénicos de siete aislados de Giardia (tres humanos y cuatro de animales), la genotipificación por PCR mostró que todos pertenecían al ensamblaje B y que mantuvieron ese genotipo incluso después de años de cultivo (28).
Asimismo, en un trabajo reciente se establecieron cultivos "in vitro" de dos aislamientos de G. intestinalis: uno de ciervo (Cervus elaphus) y el otro de gacela (Gazela thomsonii) (29). En este caso, el aislamiento del ciervo fue clasificado como ensamblaje A, y solo pudo ser mantenido en cultivo por 30 días, debido a que el número de parásitos decreció constantemente. Por su parte, el aislamiento de la gacela fue clasificado como ensamblaje B, y ya había permanecido 3 años en cultivo al momento de la publicación. La evidencia sugiere entonces que aunque la axenización de G. intestinalis puede llevar a la selección de algunos genotipos, no forzosamente son los parásitos con ensamblaje A los más estables.
En resumen, aunque hay que tener siempre presente que trabajar con cultivos establecidos "in vitro" puede enmascarar la diversidad genética de la población de parásitos existente en la muestra original; es evidente que tampoco podemos asumir como cierto que la no detección del ensamblaje B en este trabajo se haya debido a que se analizaron aislados axenizados. Indudablemente, todo esto nos lleva a plantear la gran importancia de hacer los rastreos genéticos también sobre los aislamientos originales.
Debido a la homogeneidad genética encontrada entre los aislamientos analizados, no es posible hacer una asociación entre la frecuencia de los ensamblajes y el origen geográfico, los factores socioeconómicos de la población o la sintomatología de los pacientes. Para eso son indispensables trabajos de diagnóstico y tipificación molecular de mayor envergadura y con un abordaje diferente para poder establecer la dinámica real de la transmisión de G. intestinalis en Colombia.
Actualmente el laboratorio desarrolla estudios en aislados colombianos en los que la genotipificación se hace directamente sobre el ADN extraído de las muestras fecales y tenemos a nuestra disposición la historia clínica del paciente. Con estos dos elementos esperamos llegar a conclusiones contundentes que no sean sesgadas por falta de información o por las condiciones de las muestras que se analicen.
Agradecimientos
Al doctor Moisés Wasserman del Laboratorio de Investigaciones Básicas en Bioquímica (LIBBIQ) de la Universidad Nacional de Colombia, quien gentilmente nos facilitó las muestras de ADN. A la doctora María do C. Sousa, de la Universidad de Coimbra (Portugal), quien nos donó la cepas WBC6, Bris-136 y Ad-28 de Giardia.
Conflicto de intereses: ninguno.
Financiación: Universidad El Bosque.
Referencias
1. Giraldo-Gómez JM, Lora F, Henao LH, Mejía S, Gómez-Marín JE. Prevalence of giardiasis and intestinal parasites in pre-school children from homes being attended as part of a state programme in Armenia, Colombia. Rev Salud Pública 2005; 7(3):327-38. [ Links ]
2. Monis PT, Andrews RH, Mayrhofer G, Ey PL. Genetic diversity within the morphological species Giardia intestinalis and its relationship to host origin. Infect Genet Evol 2003; 3: 29-38. doi:10.1016/S1567-1348(02)00149-1. [ Links ]
3. Lasek-Nesselquist E, Welch DM, Sogin ML. The identification of a new Giardia duodenalis assemblage in marine vertebrates and a preliminary analysis of G. duodenalis population biology in marine systems. Int J Parasitol 2010; 40(9): 1063-74. doi: 10.1016/j. ijpara.2010.02.015. [ Links ]
4. Monis PT, Mayrhofer G, Andrews RH, Homan WL, Limper L, Ey PL. Molecular genetic analysis of Giardia intestinalis isolates at the glutamate dehydrogenase locus. Parasitology 1996; 112 (Pt 1): 1-12. [ Links ]
5. Homan WL, Mank TG. Human giardiasis: genotype linked differences in clinical symptomatology. Int J Parasitol 2001; 31: 822-826. doi:10.1016/S0020-7519(01)00183-7. [ Links ]
6. Read C, Walters J, Robertson ID, Thompson RC. Correlation between genotype of Giardia duodenalis and diarrhoea. Int J Parasitol 2002; 32(2): 229-31. doi:10.1016/S0020-7519(01)00340-X. [ Links ]
7. Haque R, Roy S, Kabir M, Stroup SE, Mondal D, Houpt ER. Giardia assemblage A infection and diarrhea in Bangladesh. J Infect Dis 2005; 192(12): 2171-3. doi: 10.1086/498169. [ Links ]
8. Yason JADL, Rivera WL. Genotyping of Giardia duodenalis isolates among residents of slum area in Manila, Philippines. Parasitol Res 2007; 101: 681-87. doi: 10.1007/ s00436-007-0533-8 [ Links ]
9. Sahagún J, Clavel A, Goñi P, Seral C, Llorente MT, Castillo FJ, Capilla S, Arias A, Gómez-Lus R Correlation between the presence of symptoms and the Giardia duodenalis genotype. Eur J Clin Microbiol Infect Dis 2008; 27(1): 81-3.doi: 10.1007/s10096-007-0404-3 [ Links ]
10. Ponce-Macotela M, Martínez-Gordillo MN, Bermúdez-Cruz RM, Salazar-Schettino PM, Ortega-Pierres G, Ey PL. Unusual prevalence of the Giardia intestinalis A-II subtype amongst isolates from humans and domestic animals in Mexico. Int J Parasitol 2002; 32(9): 1201-2. doi:10.1016/S0020-7519(02)00086-3. [ Links ]
11. Cedillo-Rivera R, Darby JM, Enciso-Moreno JA, Ortega-Pierres G, Ey PL. Genetic homogeneity of axenic isolates of Giardia intestinalis derived from acute and chronically infected individuals in Mexico. Parasitol Res 2003; 90(2): 119-23. doi: 10.1007/s00436-002-0807-0. [ Links ]
12. Lalle M, Jimenez-Cardosa E, Cacció SM, Pozio E. Genotyping of Giardia duodenalis from humans and dogs from Mexico using a betagiardin nested polymerase chain reaction assay. J Parasitol 2005 Feb; 91(1): 203-5. http://dx.doi.org/10.1645/GE-293R. [ Links ]
13. Peréz Cordón G, Cordova Paz Soldan O, Vargas Vásquez F, Velasco Soto JR, Sempere Bordes L, Sánchez Moreno M, Rosales MJ. Prevalence of enteroparasites and genotyping of Giardia lamblia in Peruvian children. Parasitol Res 2008; 103(2): 459-65. doi: 10.1007/s00436-008-1007-3. [ Links ]
14. Minvielle MC, Molina NB, Polverino D, Basualdo JA. First genotyping of Giardia lamblia from human and animal feces in Argentina, South America. Mem Inst Oswaldo Cruz 2008; 103(1): 98-103. http://dx.doi.org/10.1590/S0074-02762008000100015. [ Links ]
15. Carmona-Fonseca J, Uscátegui R, Correa A. Parasitosis intestinal en niños de zonas palúdicas de Antioquia (Colombia). Iatreia 2009; 22: 27-46. Disponible en: www.scielo.org.co/scielo.php?script=sci_arttext&pid=S0121-07932009000100004&ln g=en&tlng=es. [ Links ]
16. Suescún S. Prevalencia de parásitos intestinales y factores de riesgo en escolares del colegio Chicamocha Kennedy I del municipio de Tuta, Boyacá - Colombia. Rev Univ salud 2013; 15(2): 218 -224. Disponible en: http://www.scielo.org.co/scielo.php?script=sci_arttext&pid=S0124-71072013000200012&lng=en. [ Links ]
17. Ramírez JD, Heredia RD, Hernández C, León CM, Moncada LI, Reyes P, Pinilla AE, López MC. Molecular diagnosis and genotype analysis of Giardia duodenalis in asymptomatic children from a rural area in central Colombia. Infec Genet Evol 2015; 32:208-13. doi: 10.1016/j.meegid.2015.03.015 [ Links ]
18. Arias JA, Guzmán GE, Lora-Suárez FM, Torres E, Gómez JE. Prevalencia de protozoos intestinales en 79 niños de 2 a 5 años de edad de un hogar infantil estatal en Circasia, Quindío. Infectio 2010; 14(1): 31-38. Disponible en: http://www.scielo.org.co/scielo.php?script=sci_arttext&pid=S0123-93922010000100004&lng=es. [ Links ]
19. Arroyo-Salgado B, Buelvas-Montes Y, Villalba-Vizcaíno V, Salomón-Arzuza O. Genetic profiling of Giardia intestinalis by polimerase chain in human and dogs samples of Colombian Caribean Coast. Enferm Infecc Microbiol Clin 2014; 32(7): 424-7. doi: 10.1016/j.eimc.2013.07.016. [ Links ]
20. Rodríguez V, Espinosa O, Carranza J, Duque S, Arévalo A, Clavijo J, Urrea D, Vallejo G. Genotipos de Giardia duodenalis en muestras de niños de las guarderías del Instituto Colombiano de Bienestar Familiar y de perros en Ibagué, Colombia. Biomédica 2014; 34: 271-81. doi: 10.1590/S0120-41572014000200013. [ Links ]
21. Ravid Z, Duque S, Arévalo A, Nicholls RS, Wasserman M. Genetic diversity of Giardia intestinalis populations in Colombia. Biomedica 2007; 27(1): 34-41. Disponible en: http://www.scielo.org.co/scielo.php?script=sci_arttext&pid=S0120-41572007000100004&ln g=en&tlng=en. [ Links ]
22. Read CM, Monis PT, Thompson RC. Discrimination of all genotypes of Giardia duodenalis at the glutamate dehydrogenase locus using PCR-RFLP. Infect Genet Evol 2004; Jun;4(2): 125-30. doi:10.1016/j.mee-gid.2004.02.001 [ Links ]
23. Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6: Molecular Evolutionary Genetics Analysis version 6.0. Mol Biol Evol 2013; 30: 2725-2729. doi: 10.1093/molbev/ mst197. [ Links ]
24. Andrews RH, Chilton NB, Mayrhofer G. Selection of specific Giardia intestinalis by different growth conditions. Parasitology 1992; 105: 375-386. [ Links ]
25. Bénéré E, Geurden T, Robertson L, Van Assche T, Cos P, Maes L. Infectivity of Giardia duodenalis Assemblages A and E for the gerbil and axenisation of duodenal trophozoites. Parasitol Int 2010; 59(4): 634-637. doi: 10.1016/j.parint.2010.08.001. [ Links ]
26. Ankarklev J, Franzén O, Peirasmaki D, Jerlstróm-Hultqvist J, Lebbad M, Andersson J, et al. Comparative genomic analyses of freshly isolated Giardia intestinalis assemblage A isolates. BMC genomics 2015; 16(1): 697. doi: 10.1186/s12864-015-1893-6. [ Links ]
27. Binz N, Thompson RCA, Lymbery AJ, Hobbs RP. Comparative studies on the growth dynamics of two genetically distinct isolates of Giardia duodenalis in vitro. Int J. Parasitol 1992; 19: 183-90. doi:10.1016/0020-7519(92)90101-P [ Links ]
28. Solarczyk P. Occurrence of Giardia species and genotypes in humans and animals in Wielkopolska region, Poland. Wiadomosci parazytologiczne 2008; 55(4): 459-462. [ Links ]
29. Solarczyk P, Majewska A, Slodkowicz-Kowalska A. Axenic in vitro culture and molecular characterization of Giardia duodenalis from red deer (Cervus elaphus) and Thomson's gazelle (Gazella thomsonii). Acta Parasitologica 2014; 59(4): 763-766. doi: 10.2478/s11686-014-0279-2. [ Links ]