Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Salud Uninorte
Print version ISSN 0120-5552On-line version ISSN 2011-7531
Salud, Barranquilla vol.32 no.3 Barranquilla Sept./Dec. 2016
Estudio y análisis comparativo de interacciones entre la proteina integrina con fragmentos de la proteína fibrilina-1 y fragmentos mutados de esta utilizando la metodología de docking molecular
Study and comparative analysis of interactions protein between the integrina with fragments fibrillin-1 and mutated fragments of this by using molecular docking
Ricardo Vivas-Reyes1, Yamiris Tirado2, Verónica Valdiris3
1 Grupo de Química Cuántica y Teórica, Universidad de Cartagena, Facultad de Ciencias Naturales y Exactas, Programa de Química. Campus de San Pablo. Cartagena (Colombia). Grupo de investigación CIPTEC, Facultad de Ingeniería, Programa de Ingeniería de Procesos, Fundación Universitaria Tecnológico Comfenalco. Cartagena (Colombia).Profesor titular Universidad de Cartagena. Químico, máster en Química, doctor en Química.
2 Grupo de Química Cuántica y Teórica, Universidad de Cartagena, Facultad de Ciencias Naturales y Exactas, Programa de Química. Campus de San Pablo. Cartagena (Colombia). Química de la Universidad de Cartagena. Máster en Bioquímica de la misma universidad.
3 Grupo GISIBEC. Corporación Universitaria Rafael Núñez, Facultad de Salud, Programa de Enfermería. Cartagena (Colombia). Enfermera Universidad de Cartagena, estudiante de Maestría en Enfermería de la misma universidad.
Correspondencia: Ricardo Vivas-Reyes. Universidad de Cartagena (Cartagena, Colombia). rvivasr@unicartagena.edu.co.
Fecha de recepción: 22 de junio de 2016
Fecha de aceptación: 12 de julio de 2016
Resumen
Objetivo: Evaluar las interacciones proteína-proteína que pueden generarse entre fragmentos de la proteína fibrilina-1, cuyas mutaciones causan el síndrome de Marfan (SM).
Materiales y métodos: Se realizó una serie de cálculos docking proteína-proteína entre las macromoléculas de interés; se empleó el programa Molsoft ICM; se utilizaron las estructuras cristalinas de la proteína integrina αVβ3 y los fragmentos de la proteína fibrilina-1; además se generó una sucesión de mutaciones en la fibrilina-1, las cuales son características de pacientes con síndrome de Marfan, y posteriormente se realizó el acoplamiento molecular. Adicionalmente se determinó los aminoácidos que con mayor frecuencia estaban presentes en el sitio de interacción y su hidrofobicidad.
Resultados: Se cuantificó la cantidad de aminoácidos hidrófobos presentes en las zonas de interacción producidas por los acoplamientos, teniendo en cuenta la energía del sistema; esta ponderación estuvo entre el 40 y 50 % de los aminoácidos de la zona de interacción, con un porcentaje mayor con respecto a aminoácidos neutros o cargados. En los resultados obtenidos utilizando las mutaciones realizadas sobre los fragmentos cbEGF22-TB4-cbEGF23 y cbEGF9-hyb2-cbEGF10 de la fibrilina-1 se encontró que no se ubicaron en zonas cercanas al sitio de interacción en la mayoría de los casos.
Conclusiones: Las interacciones entre los fragmentos de fibrilina-1, y estos con respecto a la integrina, mostraron en sus zonas de interacción la presencia mayoritariamente de aminoácidos hidrofóbicos, que es lo esperado normalmente.
Palabras clave: fibrilina-1, docking proteína-proteína, síndrome de Marfan, dominios cbEGF, integrina.
Abstract
Objective: To assess protein-protein interactions between fragments of fibrillin-1 protein, whose mutations cause Marfan syndrome (MS).
Materials and Methods: We performed a series of protein-protein docking calculations between the macromolecules of interest for this purpose was used Molsoft ICM program. We used the crystal structures of αVβ3 Integrin protein and fragments of fibrillin-1 protein also were generated mutations in the fibrillin-1, which are characteristic in patients with Marfan syndrome and subsequently to the molecular docking. We determined the amino acids most often present at the site of interaction and its hydrophobicity.
Results: The amount of hydrophobic amino acids present in the areas of interaction given by the couplings was quantified. Given the energy of the system, was between 40 and 50% of the amino acids of the interaction zone, with a higher proportion relative to charged or neutral amino acids. In the results obtained using the mutations performed on fragments cbEGF23 cbEGF22-TB4-and-cbEGF10 cbEGF9-HYB2 of fibrillin-1, was found they were not placed in areas near the site of interaction in most cases.
Conclusion: The interaction between fragments of fibrillin-1, and those with respect to their integrin showed the presence interaction zones mostly hydrophobic amino acids, which are normally expected.
Keywords: fibrillin-1 protein-protein docking, Marfan syndrome, cbEGF domains, integrin.
INTRODUCCIÓN
El síndrome de Marfan (SM) es considerado una enfermedad rara o huérfana, catalogada como un desorden sistémico del tejido conectivo de los humanos con manifestaciones clínicas en el sistema esquelético, ocular y cardiovascular, el cual fue descrito por primera vez por Antoine-Bernard Marfan en 1896, quien reportó la asociación de extremidades delgadas y otras anormalidades esqueléticas en una niña de 5 años (1-3).
Existe una serie de factores y síntomas que están ligados al síndrome de Marfan; entre estas manifestaciones se observa que los pacientes con SM presentan con frecuencia un aumento desproporcionado en su sistema óseo que les causa malformaciones en las extremidades y la pared torácica anterior. También estos pacientes presentan anormalidades cráneofaciales, escoliosis e hípermovilidad articular(4-6) .En la mayoría de los casos se produce miopía temprana y severa, así como dislocación de uno o ambos lentes del ojo; las manifestaciones cardiovasculares incluyen dilatación progresiva de la raíz aortica y anormalidades en las válvulas del corazón, principalmente la válvula mitral y la aórtica. Aneurismas de la aorta ascendente pueden generar complicaciones tales como insuficiencia aortica, disección o ruptura (7-11).
En el tratamiento vascular de los pacientes se busca principalmente controlar la progresión de aneurismas mediante beta bloqueadores y la cirugía profiláctica para prevenir complicaciones aorticas (7, 12, 13).
El gen FBN1 es el causante de que las personas presenten el síndrome de Marfan, y sus mutaciones son las causantes de las principales características anatómicas, fisiológicas y del mal funcionamiento del organismo, como se describió arriba; contiene 65 exones que abarcan 235 kb de DNA genómico. El FBN1 codifica a una glicoproteína de 350 kDa, llamada fibrilina-1, la cual está altamente conservada entre las diferentes especies.
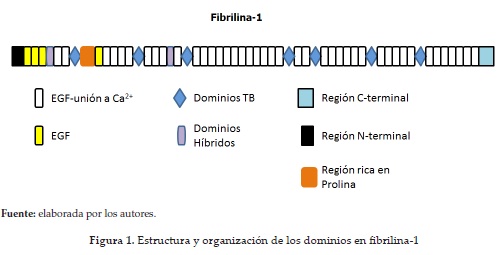
Las fibrilinas se caracterizan por la presencia de dominios de factores de crecimiento epidermal enlazados a calcio (cbEGF), los cuales son intercalados por dominios de factor de crecimiento β transformante que contiene en su estructura 8 cisteínas (TB) a través del polipéptido. También se encuentran dominios relacionados, llamados dominios híbridos (hyb), que tienen secuencias similares a dominios TB y cbEGF, y son encontrados cerca al N-terminal de estas moléculas; en la estructura de la fibrilina-1 se encuentran además EGF como dominios y una zona rica en prolina (4, 14, 15). La figura 1 muestra la estructura de la fibrilina; en la misma se puede ver claramente lo antes expuesto, es decir, las diferentes regiones que se encuentran en ella.
Los dominios TB se diferencian del resto porque en su estructura hay motivos de ocho cisteínas que forman cuatro enlaces disulfuros con un arreglo así: 1-3, 2-6, 4-7, 5-8, lo cual estabiliza el plegamiento junto con un pequeño centro hidrofóbico. Además se ha demostrado que estos dominios actúan en interacciones específicas proteína-proteína, importantes para las funciones de regulación asociadas a la familia de proteínas fibrilinas/LTBP (proteínas latentes de unión a factor de crecimiento transformante β). Entre las interacciones en las que participan podemos mencionar aquellas con la superficie celular mediante integrinas, lo cual se logra por la secuencia RGD (arg-gly-asp), presente en el dominio TB4 de la fibrilina (16).
En la estructura de la proteína fibrilina-1 están presentes 47 módulos de EGF, 43 de ellos tienen una secuencia de unión a calcio y son llamados módulos de unión a calcio EGF (cbEGF).
El calcio unido es importante desde el punto estructural, debido a que estabiliza la interacción entre los dominios de la molécula, lo cual puede facilitar las interacciones proteína-proteína (17, 18). Como se mencionó antes, en la estructura de las fibrilinas encontramos otros cuatro dominios no enlazantes a calcio (dominios EGF) y dos dominios híbridos; estos últimos tienen secuencias similares a los dominios TB y cbEGF, y son encontrados cerca del N-terminal de la proteína. Se considera también que los dominios híbridos participan en el mantenimiento de la integridad estructural de las microfibrillas (14, 19).
La mayoría de las mutaciones en la fibrilina-1 que generan las características físicas en los pacientes con el síndrome de Marfan pueden ser ubicadas a lo largo de los 47 dominios EGF que están en tándem en dicha proteína; además muchas interrupciones de los seis residuos de cisteína que interactúan a través de enlaces disulfuros para determinar el plegamiento del dominio o residuos afectan la unión de la fibrilina-1 al calcio. Dichas perturbaciones conducen a una mayor segmentación y degradación proteolítica (1, 20-22). Las mutaciones que se presentan en estos dominios por lo general consisten en la sustitución de aminoácidos que afecta a los residuos de cisteína en los cbEGF, TB y dominios híbridos (23).
La familia de proteínas fibrilinas/LTBP (proteínas latentes de unión a factor de crecimiento transformante β) de la matriz extracelular (ECM) realizan diferentes funciones en el tejido conectivo, y son importantes en el desarrollo y la homeostasis tisular.
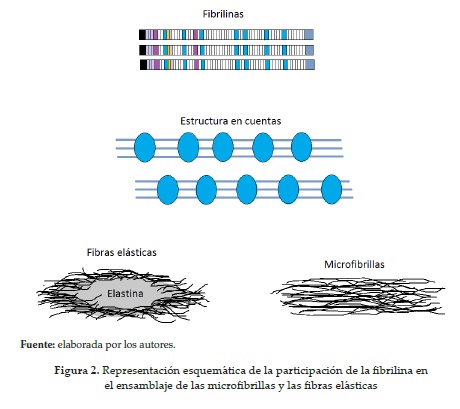
Las fibrilinas se ensamblan y generan las estructuras de orden superior, conocidas como microfibrillas de 10-12 nm. Antes de la formación de las microfibrillas, la fibrilina se somete a una serie de modificaciones posttraduccionales, incluyendo la glicosilación; una vez ensambladas, se las encuentra involucradas en una variedad de interacciones célula-matriz y procesos de desarrollo. Estas pueden localizarse tanto en asociación con la elastina en las fibras elásticas (por ejemplo, en las paredes de las arterias elásticas) y también sin elastina (por ejemplo, en las zónulas ciliares), que conectan la lente del ojo a los músculos ciliares, como lo vemos de manera esquemática en la figura 2 (16, 24).
Hoy en día, y debido al gran desarrollo que en los últimos años ha tenido la informática se cuenta con computadores con más poder de cálculo y softwares con mayor capacidad para hacer estos cálculos con aplicación a moléculas de importancia biológica, tales como proteínas y otro tipo de macromoléculas de interés biológico. De esta forma, la informática se encuentra muy ligada a la biología, principalmente por su gran apoyo en distintas ramas de esta.
La técnica del docking molecular se empleó en este estudio para simular las interacciones entre fragmentos de fibrilina-1 y las posibles interacciones de estos con la integrina αVβ3. La interacción entre fibrilina, especialmente el dominio TB4 con integrinas, ha sido estudiado con las integrinas αVβ3, αVβ6 y α5β1, las cuales son importantes para la adhesión celular. Para que se produzcan estas interacciones debe estar presente el motivo RGD en la proteína fibrilina, ubicándose en el dominio TB4 (15, 16); esta es la razón de nuestro interés del docking entre la integrina αVβ3 y fragmentos de la fibrilina-1 en esta propuesta.
La meta principal de esta contribución es determinar y analizar las interacciones que pueden generarse entre los fragmentos de la proteína fibrilina-1, además de aquellas interacciones de estos con la integrina αVβ3, la cual se encuentra en la superficie celular e interviene en el anclaje de proteínas de adhesión extracelular a componentes del citoesqueleto. Se simularon también algunas de las mutaciones que pueden presentarse en los fragmentos de fibrilina-1, los cuales tienen como consecuencia las manifestaciones del síndrome de Marfan. Las mutaciones que se realizaron a las estructuras fueron las obtenidas de la Base de datos de Mutación Universal (Universal Mutations database: UMD) para síndrome de Marfan (4, 25).
MATERIALES Y MÉTODOS
Mediante cálculos tipo docking utilizando el programa Molsoft ICM basado en el método ODA (Optimal Docking Areas) (26) se logró simular y evaluar las interacciones entre proteína-proteína. Esto con el fin de profundizar en los fenómenos involucrados en dichas interacciones (27, 28).
La asociación proteína-proteína representa un hecho significativo en muchos procesos biológicos, de los cuales podemos mencionar la transducción de señal, regulación del crecimiento celular, metabolismo y adhesión, respuesta inmune, entre otros.
Entender como se producela unión de estos complejos macromoleculares y las condiciones que determinan su especificidad no solo es fundamental para establecer y explicar la forma cómo se generan los procesos biológicos, sino también útil en el desarrollo de nuevas estrategias terapéuticas (29).
Datos moleculares
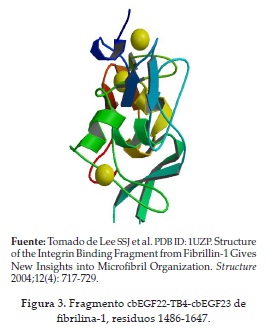
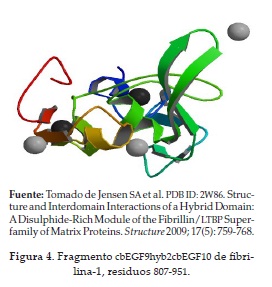
Para el desarrollo de este trabajo se emplearon las estructuras cristalinas de la proteína integrina αVβ3 (30) y de los fragmentos de la proteína fibrilina-1 (fragmentos cbEGF22-TB4-cbEGF23 y cbEGF9-hyb2-cbEGF10). En las figuras 3 y 4 se muestran las estructuras secundarias de estos dos fragmentos (31, 32). Estas estructuras fueron tomadas de la base de datos Protein Data Bank (PDB).
Los fragmentos de fibrilina-1 asimismo fueron mutados y se llevaron a cabo procedimientos de docking; las mutaciones se tomaron de la base de datos UMD (25).
Acoplamiento molecular
Para la realización del docking proteína-proteína entre las macromoléculas de interés se empleó el programa Molsoft ICM basado en el método ODA (26), el cual predice sitios de interacción proteína-proteína sobre superficies proteicas, como el mostrado en la figura 5, donde interaccionan dos fragmentos de la proteína fibrilina-1. Adicionalmente se identificaron zonas superficiales óptimas con los más bajos valores de energía de acoplamiento de desolvatación, dando a estas zonas las características mínimas y esenciales, así como una mayor probabilidad para participar en procesos de enlaces o interacciones inter-proteínas.
Estos datos son calculados por parámetros de solvatación atómica (ASP) derivadas de los experimentos de transferencia de octanol/agua y se ajustan para el acoplamiento proteína-proteína.
El programa ODA tiene como objetivo principal predecir zonas de interacción (26) como punto de referencia en 66 estructuras consolidadas no homólogas, con el fin de validar el método. Se encontró que los puntos de interacciones identificados están correctamente situados en el 80 % de los casos (26).
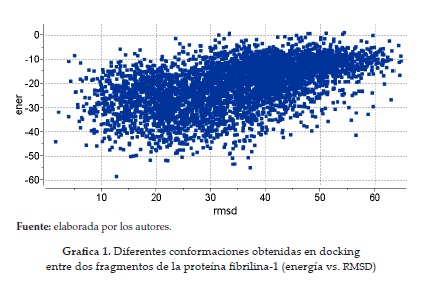
Al finalizar el docking, el programa arrojó una serie de datos de energía y valores de RMSD (grafica 1), los cuales ayudan a escoger las conformaciones más factibles de todas aquellas arrojadas durante el procedimiento. (33).
Como se muestra en la gráfica 1, se encontró que aproximadamente 5 puntos con una RMSD menor que 10 y valores de energía menores que -40 kcal/mol:
Teniendo en cuenta las mejores conformaciones en cada uno de los acoplamientos realizados, se analizaron dichas interacciones para determinar los aminoácidos que con mayor frecuencia estaban presentes en el sitio de interacción y su hidrofobicidad.
RESULTADOS Y DISCUSIÓN
Debido a que las proteínas están formadas por aminoácidos, las características de aquellos que estén en la superficie, así como su posición relativa, determinan la capacidad de interacción con otras proteínas o moléculas y les dan su funcionalidad y características particulares dentro de la proteína.
Para encontrar las características que determinen de mejor manera una interacción es necesario evaluar los criterios fisicoquímicos presentes en las proteínas. Esto quiere decir que se deben evaluar aquellas que participan en la interacción como tal. Alguna de estas características que han sido utilizadas para la determinación de las zonas de interacción incluyen:
- Porcentaje de área hidrofóbica: corresponde al porcentaje del área total aportada por los residuos hidrofóbicos.
- Número de aminoácidos polares: es el número de residuos polares que forman parte del área de interacción.
- Número aminoácidos apolares: es el número de residuos apolares que forman parte del área de interacción.
En este caso se tuvo en cuenta el porcentaje de aminoácidos hidrofóbicos y polares neutros que encontramos en la superficie de interacción de los acoplamientos entre las proteínas de interés. Entre los aminoácidos considerados hidrofóbicos están la Valina, Leucina, Isoleucina, Alanina, Metionina, Fenilalanina, Tirosina y Triptófano.
El índice de hidropatía indica qué tan hidrofóbico es un aminoácido; es utilizado para clasificarlos en las zonas de interacción entre las proteínas.
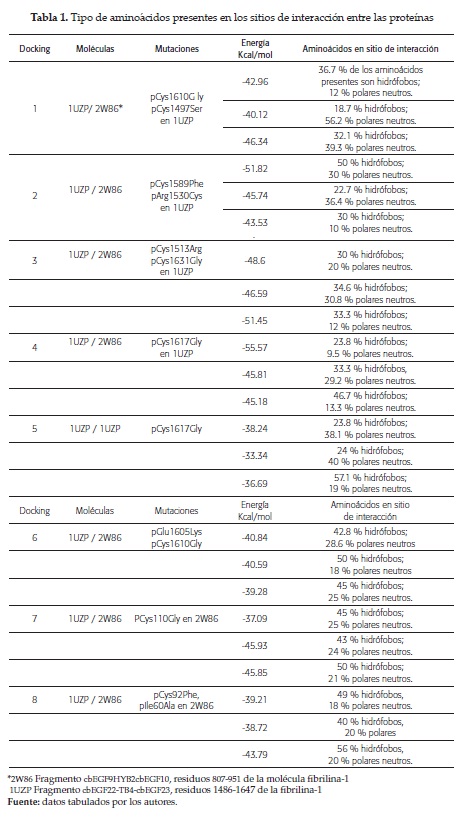
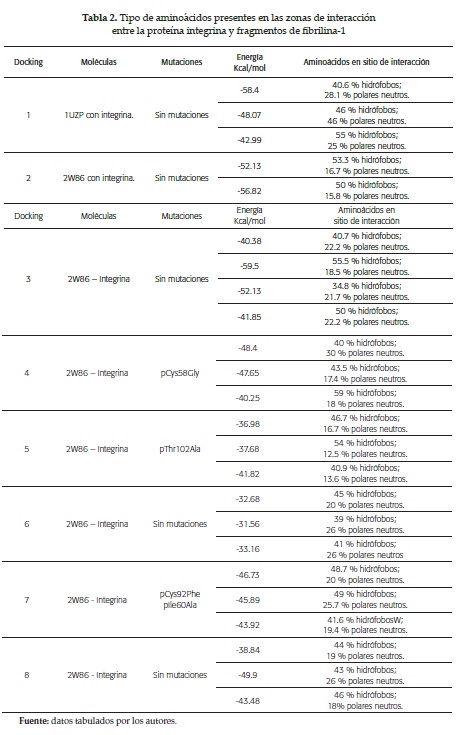
Las tablas 1 y 2 muestran los resultados expuestos anteriormente, de donde se tomaron las tres mejores energías de acoplamiento para cada par de moléculas interaccionando.
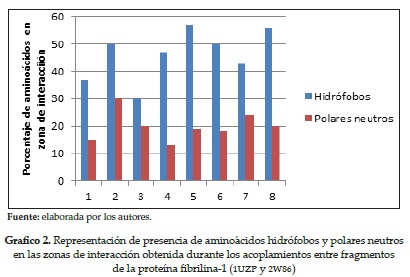
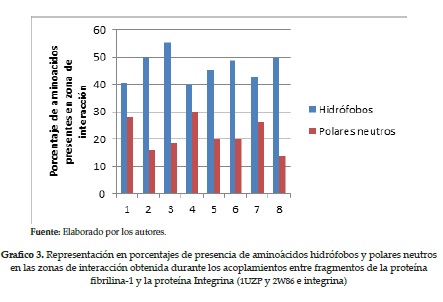
En tanto en los gráficos 2 y 3 se presentan de una manera más clara el porcentaje de aminoácidos hidrófobos y polares neutros en las zonas de interacción para ocho de los acoplamientos realizados durante el estudio, con el fin de mostrar la marcada presencia de moléculas hidrófobas en dichos sitios.
Es aceptado comúnmente que los sitios de interacción proteína-proteína se componen principalmente de un núcleo hidrófobo; en este caso, esto se ve reflejado en la cantidad de aminoácidos hidrófobos presentes en las zonas de interacción dadas por los acoplamiento; teniendo en cuenta la energía del sistema, vemos que entre el 40 y 50 % de los aminoácidos de la zona de interacción son hidrófobos. En una menor proporción se encontraron aminoácidos polares neutros y aminoácidos cargados, cuya interacción puede generarse a través de enlaces de hidrógeno o puentes salinos. Estos residuos suelen actuar como fuertes puntos de anclaje, y de esta manera mantener la integridad estructural del complejo.
Los resultados obtenidos utilizando las mutaciones realizadas sobre los fragmentos cbEGF22-TB4-cbEGF23 y cbEGF9-hyb2-cbEGF10 de la fibrilina-1 muestran que estos no se ubicaron en zonas cercanas al sitio de interacción en la mayoría de los casos. Debe tenerse en cuenta que contamos solo con fragmentos de la fibrilina-1 y no con la molécula completa, lo cual puede minimizar la posibilidad de encontrar las mutaciones en los sitios de interacción proteína-proteína.
También se observó que las mutaciones no indujeron grandes cambios estructurales en los fragmentos; sin embargo, a nivel global, es decir, viendo los cambios teniendo en cuenta la proteína completa, no solo los fragmentos, y la importancia de esta estructura en su estado normal en las interacciones con otras proteínas, puede ser la respuesta a las manifestaciones clínicas de la patología presentada por el síndrome de Marfan.
Los resultados obtenidos en esta propuesta resultan ser útiles en la predicción de propiedades moleculares utilizando métodos semiempíricos tales el como PM6 para los complejos proteína-proteína derivados del docking; además se puede obtener más información a partir de simulación de dinámica molecular en escalas espaciales que son difíciles de acceder experimentalmente, y de esta manera profundizar de manera más profunda y amplia en la explicación a los posibles efectos que tienen las mutaciones en la estructura de la proteína fibrilina-1, las cuales son la principal causa de los signos y síntomas presentes en los pacientes con Marfan.
CONCLUSIONES
La proteína fibrilina-1, cuyas mutaciones causan el síndrome de Marfan, interactúa con moléculas de la matriz extracelular, tales como integrinas, glicoproteínas asociadas a microfibrillas (MAGP), y proteínas LTBPs.
En las simulaciones de acoplamientos moleculares entre los fragmentos de fibrilina-1 con la integrina αVβ3 se evidencia de manera significativa la presencia de aminoácidos hidrófobos, y de aminoácidos neutros polares en una menor proporción, los cuales de forma conjunta contribuyen en el sitio de interacción con la formación de puentes de hidrogeno, así como con las interacciones hidrófobas esperadas.
Las interacciones entre los fragmentos de fibrilina-1 siguieron el mismo comportamiento, así como aquellos en los que se realizaron mutaciones que generalmente están presentes en el síndrome de Marfan.
Con base en los resultados obtenidos en esta propuesta investigativa se encontraron las conformaciones más probables y se predijeron las propiedades moleculares mediante el método semiempírico PM6 para los complejos proteína-proteína derivados del docking. Así mismo, se realizó la simulación de dinámica molecular, y se obtuvo información del sistema biomolecular en escalas espaciales que son difíciles de acceder de forma experimental.
Agradecimientos
Los autores agradecen a la Universidad de Cartagena y a Colciencias por el soporte económico prestado para el desarrollo de este trabajo, en especial al Programa de Jóvenes Investigadores, financiado por las dos instituciones.
Conflicto de intereses: ninguno.
Financiación: Universidad de Cartagena.
REFERENCIAS
1. Judge DP, Dietz HC. Marfan's syndrome. The Lancet 2005;366(9501):1965-76. [ Links ]
2. Battle RW. Edgar Allan Poe: A Case Description of the Marfan Syndrome in an Obscure Short Story. The American Journal of Cardiology 2011;108(1):148-9. [ Links ]
3. Pyeritz RE. The Marfan syndrome. Annual Review Of Medicine 2000;51:481-510. [ Links ]
4. Ling-Gen G, Fang L, Ru-Tai H, Xian-Liang Z. Recent molecular biological progress in Marfan syndrome and Marfan-associated disorders. Ageing Research Reviews 2010;9:363-8. [ Links ]
5. Strider D, Keeling AW, Tullmann DF, Reigle J, Cherry KJ. Marfan Syndrome teaching algorithm: Does it make a difference? Journal of Vascular Nursing 2013;31(1):21-31. [ Links ]
6. Roll K. The influence of regional health care structures on delay in diagnosis of rare diseases: The case of Marfan Syndrome. Health Policy 2012;105(2-3):119-27. [ Links ]
7. Franken R, den Hartog AW, Singh M, Pals G, Zwinderman AH, Groenink M et al. Marfan syndrome: Progress report. Progress in Pediatric Cardiology 2012;34(1):9-14. [ Links ]
8. Herrera RN, Miotti JA, Pereyra AS, Lobo MV, Ibarra MT, Tomé Guzmán AF. Síndrome de Marfan con disección aórtica asociada tromboembolismo venoso e hiperhomocisteinemia. Medicina 2012;72(6):478-80. [ Links ]
9. Villaseñor CP, Amezcua Guerra LM. Síndrome de Marfan. Archivos de Cardiología de México 2004;74:S482-S4. [ Links ]
10. Nemet AY, Assia EI, Apple DJ, Barequet IS. Current Concepts of Ocular Manifestations in Marfan Syndrome. Survey of Ophthalmology 2006;51(6):561-75. [ Links ]
11. Nollen GJ, Mulder BJM. What is new in the Marfan syndrome? International Journal of Cardiology 2004;97, Supplement 1(0):103-8. [ Links ]
12. Ramirez F, Dietz HC. Marfan syndrome: from molecular pathogenesis to clinical treatment. Current Opinion in Genetics & Development 2007;17(3):252-8. [ Links ]
13. Detaint D, Aegerter P, Tubach F, Hoffman I, Plauchu H, Dulac Y et al. Rationale and design of a randomized clinical trial (Marfan Sartan) of angiotensin II receptor blocker therapy versus placebo in individuals with Marfan syndrome. Archives of Cardiovascular Diseases 2010;103(5):317-25. [ Links ]
14. Handford PA, Downing AK, Reinhardt DP, Sakai LY. Fibrillin: from domain structure to supramolecular assembly. Matrix Biology 2000;19(6):457-70. [ Links ]
15. Jensen Sacha A, Robertson Ian B, Handford Penny A. Dissecting the Fibrillin Microfibril: Structural Insights into Organization and Function. Structure 2012;20(2):215-25. [ Links ]
16. Robertson I, Jensen S, Handford P. TB domain proteins: evolutionary insights into the multifaceted roles of fibrillins and LTBPs. Biochem J 2011;433:263-76. [ Links ]
17. Werner JM, Knott V, Handford PA, Campbell ID, Downing AK. Backbone dynamics of a cbEGF domain pair in the presence of calcium. Journal of Molecular Biology 2000;296(4):1065-78. [ Links ]
18. Handford PA. Fibrillin-1, a calcium binding protein of extracellular matrix. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research 2000;1498(2-3):84-90. [ Links ]
19. Jensen SA, Iqbal S, Lowe ED, Redfield C, Handford PA. Structure and Interdomain Interactions of a Hybrid Domain: A Disulphide-Rich Module of the Fibrillin/LTBP Superfamily of Matrix Proteins. Structure 2009;17(5):759-68. [ Links ]
20. Dietz HC. Marfan Syndrome: From Molecules to Medicines. The American Journal of Human Genetics 2007;81(4):662-7. [ Links ]
21. Segade F. Functional evolution of the microfibril-associated glycoproteins. Gene 2009;439(1-2):43-54. [ Links ]
22. Lu Y, Jeremy R, Kekic M, Yin J, Hambly BD. Marfan Syndrome Mutations Predominantly Alter Fibrillin Domain Folding. Biophysical Journal 2012;102(3, Supplement 1):251a. [ Links ]
23. Mellody KT, Freeman LJ, Baldock C, Jowitt TA, Siegler V, Raynal BDE et al. Marfan Syndrome-causing Mutations in Fibrillin-1 Result in Gross Morphological Alterations and Highlight the Structural Importance of the Second Hybrid Domain. Journal of Biological Chemistry 2006;281(42):31854-62. [ Links ]
24. Sherratt MJ, Wess TJ, Baldock C, Ashworth J, Purslow PP, Shuttleworth CA et al. Fibrillin-rich microfibrils of the extracellular matrix: ultrastructure and assembly. Micron 2001;32(2):185-200. [ Links ]
25. Stheneur C, Collod-Béroud G, Faivre L, Buyck JF, Gouya L, Le Parc J-M et al. Identification of the minimal combination of clinical features in probands for efficient mutation detection in the FBN1 gene. European Journal Of Human Genetics: EJHG 2009;17(9):1121-8. [ Links ]
26. Fernandez-Recio J, Totrov M, Skorodumov C, Abagyan R. Optimal docking area: A new method for predicting protein-protein interaction sites. Proteins: Structure, Function, and Bioinformatics 2005;58(1):134-43. [ Links ]
27. Gray JJ, Moughon S, Wang C, Schueler-Furman O, Kuhlman B, Rohl CA et al. Protein-Protein Docking with Simultaneous Optimization of Rigid-body Displacement and Side-chain Conformations. Journal of Molecular Biology 2003;331(1):281-99. [ Links ]
28. Fernández-Recio J, Totrov M, Abagyan R. Identification of Protein-Protein Interaction Sites from Docking Energy Landscapes. Journal of Molecular Biology 2004;335(3):843-65. [ Links ]
29. Fiorucci S, Antonczak S, Golebiowski J. Prediction and Calculation of Protein-Protein Binding Affinities and Mutation Effects. In Zacharias M, editor. PROTEIN-PROTEIN COMPLEXES. Analysis, Modeling and Drug Design. Imperial College Press; 2010. [ Links ]
30. Xiong J-P, Mahalingham B, Alonso JL, Borrelli LA, Rui X, Anand S et al. PDB ID: 3IJE. Crystal structure of the complete integrin alphaVbeta3 ectodomain plus an alpha/beta transmembrane fragment. The Journal Of Cell Biology 2009;186(4):589-600. [ Links ]
31. Lee SSJ, Knott V, Jovanović J, Harlos K, Grimes JM, Choulier L et al. PDB ID: 1UZP. Structure of the Integrin Binding Fragment from Fibrillin-1 Gives New Insights into Microfibril Organization. Structure 2004;12(4):717-29. [ Links ]
32. Jensen SA, Iqbal S, Lowe ED, Redfield C, Handford PA. PDB ID: 2W86. Structure and Interdomain Interactions of a Hybrid Domain: A Disulphide-Rich Module of the Fibrillin/LTBP Superfamily of Matrix Proteins. Structure 2009;17(5):759-68. [ Links ]
33. Kozakov D, Brenke R, Comeau SR, Vajda S. PIPER: An FFT-based protein docking program with pairwise potentials. Proteins: Structure, Function, and Bioinformatics 2006;65(2):392-406. [ Links ]