Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Revista Salud Uninorte
versão impressa ISSN 0120-5552versão On-line ISSN 2011-7531
Salud, Barranquilla vol.32 no.3 Barranquilla set./dez. 2016
Validación técnica de una PCR: Reacción en cadena de la polimerasa para la detección de Chlamydia trachomatis
Technical validation of a polymerase chain reaction for Chlamydia trachomatis detection
Diana Jurado-Orejuela1, Claudia Paredes-Amaya2, Gerardo Libreros-Zúñiga3
1 Profesora Escuela de Bacteriología y Laboratorio Clínico, Universidad del Valle, Cali (Colombia). dijurado@yahoo.com
2 Bacterióloga Departamento de Microbiología, Universidad del Valle, Cali (Colombia). ccparedes09@gmail.com
3 Profesor Departamento de Microbiología, Universidad del Valle, Cali (Colombia). gerardo.Kbreros@correounivalle.edu.co
Correspondencia: Gerardo Andrés Libreros Zúñiga. Av. 1N n° 3N-35, oficina 2301, Centenario, Cali (Valle del Cauca, Colombia). Teléfono 57-2-6670326, Fax 57-2-6670329. gerardo.Kbreros@correourtívalle.edu.co
Resumen
Objetivo: Estandarizar una técnica de PCR para la detección de Chlamydia trachomatis.
Materiales y método: Estudio experimental en el que se optimizaron las condiciones de PCR para la detección in vitro de C. trachomatis. Se utilizó ADN de C. trachomatis VR885D y los cebadores CtP15'-TAGTAACTGCCACTTCATCA-3'y CtP2 5'- TTCCCCTTGTAATTCGTTGC-3, que amplificaron un segmento de 201 pb del plásmido clamidial. Se realizaron diluciones logarítmicas de ADN clamidial y fueron empleados para determinar la sensibilidad analítica expresada como copias de plásmidos y/o de cuerpos elementales. La especificidad analítica de la prueba se evaluó usando los cebadores CtP1 y CtP2 y ADN de microorganismos colonizadores y/o patógenos urogenitales. La variabilidad intraensayo fue evaluada sobre muestras por triplicado, mientras que la variabilidad interensayo se determinó mediante comparación de los resultados obtenidos por tres técnicos en diferentes días.
Resultados: Las condiciones de PCR para la amplificación del gen de interés fueron establecidas (94°C/4 min; 40 ciclos de 94°C/1 min, 56°C/1 min. y 72°C/1.5 min; 72°C/4 min); 1.5 mM MgCl2 y 1 U/pL Taq polimerasa. La sensibilidad analítica de la PCR fue de 10-17 g de ADN, equivalentes a una copia del plásmido o menos de un cuerpo elemental de C. trachomatis. Los cebadores CtP1 y CtP2 amplificaron específicamente el ADN de C. trachomatis bajo las condiciones experimentales evaluadas. La repetitibilidad y reproducibilidad de la PCR se determinó con experimentos de variabilidad intra e interensayo respectivamente.
Conclusiones: La estandarización de esta PCR es el primer paso para su utilización en el diagnóstico de infecciones por C. trachomatis. Se requieren estudios adicionales de validación clínica de ésta prueba.
Palabras clave: Chlamydia trachomatis, diagnóstico molecular, PCR.
Abstract
Objective: To standardize a PCR for Chlamydia trachomatis detection.
Materials and methods: An experimental study was designed to standardize a C. trachomatis PCR test. Genomic DNA from C. trachomatis serovar D ATCC VR885D was used for the PCR standardization. An amplicon of 201 bp from clamidial plasmid was obtained using primers CtP15'-TAGTAACTGCCACTTCATCA-3' and CtP2 5'- TTCCCCTTGTAATTCGTT-GC-3'. Serial dilutions of clamidial DNA were used to determine the analytical sensitivity. Analytical specificity was tested using DNA from several urogenital microorganisms. Intra assay variability was assessed on triplicate DNA samples, while inter assay variability was assessed comparing the results by three technicians on different days.
Results: Established (94°C/4 min; 40 cycles at 94 °C/1 min, 56°C/1 min and 72°C/1.5 min; 72°C/4 min; 1.5 mM of MgCl2 and 1U/pL of Taq polymerase. The analytical sensitivity was 10-17 g of DNA equivalent to one plasmid or less than one elementary body from C. trachomatis. Primers CtP1 and CtP2 amplified specifically C. trachomatis in the experimental conditions evaluated. Intra and inter assay variability demonstrated the repeatability and reproducibility of the PCR respectively.
Conclusions: We standardized the experimental conditions for a C. trachomatis PCR that can be used for diagnostic purposes. Other studies are required for further clinical evaluation of this test.
Keywords: Chlamydia trachomatis, molecular diagnosis, PCR.
INTRODUCCIÓN
Chlamydia trachomatis es la bacteria que causa el mayor número de infecciones de transmisión sexual en el mundo, con un estimado de 100 millones de casos por año, constituyéndose en un problema de salud para la población sexualmente activa (1, 2). C. trachomatis infecta principalmente células del epitelio mucoso y, simultáneamente, se asocian con un estado de inflamación local que favorece la co-infección de otros microorganismos, como por ejemplo, el Virus de laInmunodeficiencia Humana (VIH) (3).
En Colombia, la transmisión de C. trachomatis se favorece por la carencia de un programa de vigilancia de infecciones de transmisión sexual específicamente enfocado hacia el control de este patógeno y a las dificultades para su diagnóstico de laboratorio. Adicio-nalmente, la naturaleza asintomática y crónica de la infección por Chlamydia facilita su diseminación entre las poblaciones de mayor riesgo. Por lo anterior, la implementación de nuevas técnicas diagnósticas tendría gran impacto en el tratamiento oportuno de los pacientes y en el control de la infección antes del desarrollo de una vacuna. (4).
El cultivo en células MacCoy, a pesar de ser considerado como el método de referencia para el diagnóstico de laboratorio de C. trachomatis, presenta baja sensibilidad y es de difícil implementación en laboratorios clínicos. (5).
Por otro lado, como alternativa al cultivo, se encuentran las pruebas de detección de antígenos bacterianos (LPS o MOMP), que a pesar de su bajo costo y fácil implementación presentan baja especificidad comparadas con el cultivo (6).
Actualmente, las recomendaciones mundiales para el diagnóstico de laboratorio de las infecciones por C. trachomatis se inclinan por el uso de las pruebas de amplificación de ácidos nucleicos como la Reacción en Cadena de Polimerasa (PCR), debido a su elevado desempeño diagnóstico (sensibilidad y especificidad superiores a 90 % comparadas con el cultivo o la detección de antígenos) (7); adicionalmente la PCR también puede ser estandarizada para el análisis de diferentes tipos de muestras clínicas, facilitando así el diagnóstico de infecciones genitales y extragenitales (8, 9). Por lo anterior, este estudio pretende validar una PCR para la detección de C. trachomatis que pueda ser usada con fines diagnósticos.
MATERIALES Y MÉTODOS
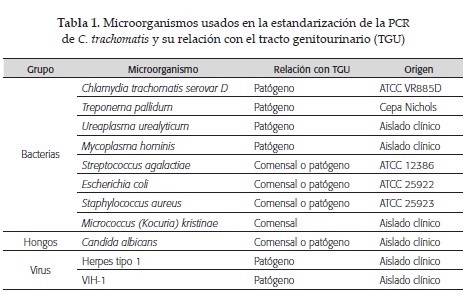
Microorganismos y material genético: Todos los microrganismos o ADN usados en este trabajo se presentan en la tabla 1.
Extracción y cuantificación de ADN: A partir de un cultivo puro en agar trispticasa soya se suspendieron 2 a 5 colonias en 1.5 ml de agua Milli-Q. Se centrifugó a 12.000 g y el precipitado se utilizó para la extracción del ADN genómico empleando el estuche Wizard Genomic ADN Purification (Promega). Todas las muestras de ADN fueron cuantificadas en el Qubit 2.0 (Invitrogen).
PCR para ARN ribosomal 16S: Como un control interno de las PCR bacterianas se amplificó un segmento de 602 pb del gen que codifica para el ARN ribosomal 16S usando los cebadores Ub1 5'-GATTAGATACCCTGGTAGTCCAC-3' y Ub2 5'-CCCGGGAACGTATTCACCG-3'. La PCR se realizó de acuerdo con Ashimoto et al. (10).
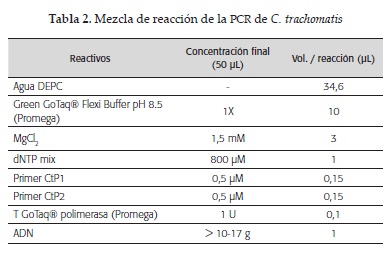
PCR para Chlamydia trachomatis: Se realizaron experimentos de optimización de la PCR variando las concentraciones de MgCl2 (1.0 a 3.0 mM con variaciones de 0.5 mM), Taq polimerasa (0.5 a 2 U/uL con variaciones de 0.5 U/uL) y temperaturas de alineamiento de cebadores (52 a 57°C con variaciones de 1°C). Se empelaron los cebadores CtP1 5'-TAGTAACTGCCACTTCATCA-3' y CtP2 5'-TTCCCCTTGTAATTCGTTGC-3'(11). La composición y concentración final de los reactivos en la mezcla reacción se muestra en la tabla 2. Una vez estandarizada la prueba, todos los experimentos se realizaron en termoci-clador Applied Biosystems 2720 empleando el programa: 95°C/4 min, 40 ciclos (95°C/1 min; 56°C/1 min; 72°C/1.5 min) y 72°C/4 min. Todos los experimentos incluyeron un control positivo y blanco de reactivo.
Sensibilidad analítica: Se realizaron diluciones logarítmicas del ADN de C. trachomatis y se procesaron por PCR. Se estimó el número de plásmidos y de CE considerando un plásmido de 7500 pb y 10 copias por cada CE. La sensibilidad analítica de la prueba se definió como la mínima concentración de ADN clamidial o menor número de plásmidos o CE detectados por la PCR.
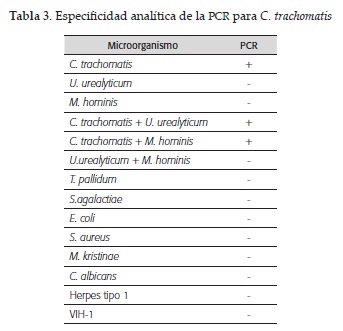
Especificidad analítica: Se realizaron PCR por triplicado con los cebadores CtP1 y CtP2 y ADN de cada uno de los microorganismos o mezclas de organismos mostrados en la tabla 1. La especificidad analítica se definió como la capacidad de los cebadores CtP1 CtP2 de amplificar exclusivamente el gen de interés, evidenciado como una banda de 201 pb en el gel de agarosa.
Análisis de los productos de PCR: Se adicionaron 10 uL de cada producto de PCR en los pozos del gel de agarosa 2 % con SYBR-Safe (Invitrogen). La electroforesis se realizó en tampón TBE 1X, pH 8.3 a 7 V/cm por 60 minutos. Los geles se documentaron con el transiluminador Safe-Imager 2.0 (Invitrogen). Se usó el marcador de peso molecular 100 pb DNA ladder (Invitrogen).
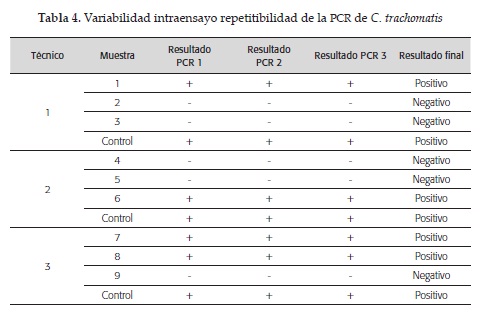
Variabilidad intraensayo: Diferentes técnicos realizaron la PCR por triplicado usando diferentes muestras de origen urogenital que previamente habían sido procesadas y diagnosticadas como positivas o negativas para C. trachomatis con el estuche comercial de PCR en tiempo real (qPCR) múltiplex C. trachomatis, Ureaplasma y Mycoplasma (Sacace Biotechnologies). La variabilidad intraensayo se definió como la repetitibilidad de los triplicados realizados por cada operador bajo las mismas condiciones experimentales (tabla 4).
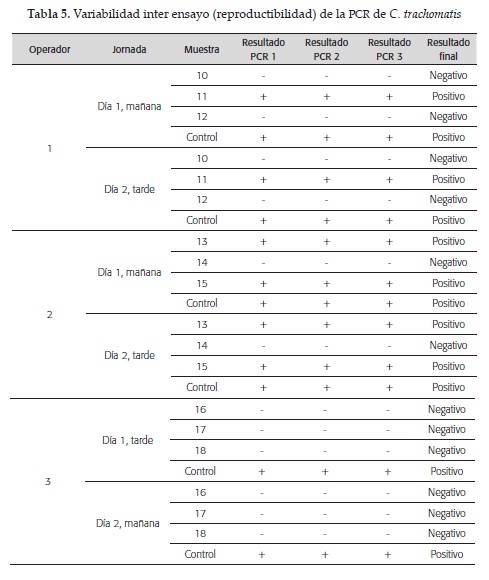
Variabilidad interensayo: Se procesaron por PCR muestras de origen urogenital variando las condiciones experimentales (por ejemplo, diferentes técnicos y días de ensayo). La variabilidad interensayo se definió como la reproducibilidad de los resultados obtenidos bajo diferentes condiciones experimentales (tabla 5).
Consideraciones éticas: Este estudio fue aprobado por el Comité Institucional de Revisión Ética Humana de la Universidad del Valle. Acta 045-012.
RESULTADOS
Los cebadores CtPl y CtP2 flanquean una secuencia de 201 pb comprendida entre la timina 3584 (T3584) y la Adenina3784 (A3784) del plásmido críptico de C. trachomatis. Esta región génica no está incluida dentro de las ocho secuencias codificantes (CDS) conocidas para el plásmido de C. trachomatis, por lo que, el fragmento génico amplificado en esta PCR correspondería a una posible región intergénica con función desconocida hasta el momento (GenBank: AAB02587.1) (figura l).
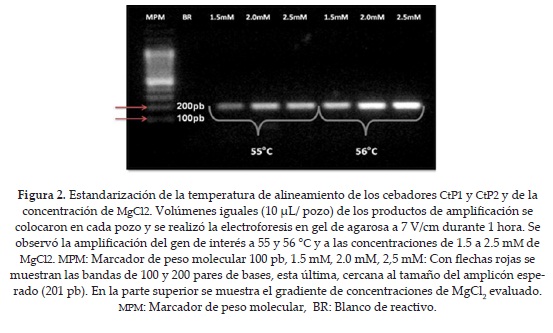
Para estandarizar las condiciones experimentales de la PCR se realizaron variaciones en la temperatura de alineamiento de los cebadores CtP1 y CtP2 (entre 52 a 57°C) y en las concentraciones de MgCl2 (1 a 3 mM). Se observó amplificación de una banda de 201 pb entre 55 y 56 °C y entre 1.5 a 2.5 mM de MgCl2 (figura 2). A temperaturas de alineamiento inferiores a 55°C o superiores a 57°C no se observó la banda esperada (datos no mostrados). Adicionalmente se realizaron experimentos evaluando diferentes concentraciones de Taq polimerasa (0.5 a 2 U/ uL). Considerando que la concentración de enzima recomendada por el fabricante era de 1 U/uL, esta PCR consiguió amplificar el fragmento génico de interés en todas las concentraciones de Taq polimerasa evaluadas, inclusive a 0.5 U/uL (datos no mostrados).
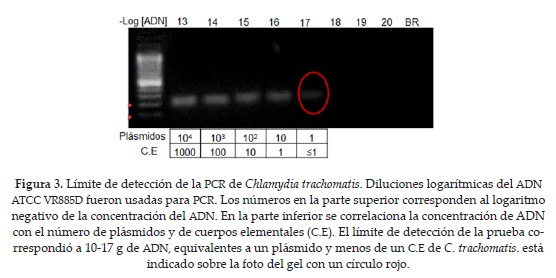
Con el fin de establecer el límite de detección de la prueba se realizaron PCR de 40 ciclos a 56 °C, usando diluciones logarítmicas del ADN de C. trachomatis, 1 U de Taq polimerasa, 1.5 mM de MgCl2 y los cebadores CtP1 y CtP2.
Esta PCR detectó hasta 10-17 g de ADN, lo que equivale a un plásmido de 7500 pb o menos de un cuerpo elemental de C. trachomatis (figura 3), o menos de un cuerpo elemental de C. trachomatis (figura 3).
Con el propósito de examinar la especificidad de los cebadores CtP1 y CtP2 se realizaron PCR de 40 ciclos a 56° C, usando aproximadamente 1 ng de ADN proveniente de microorganismos patógenos o comensales del tracto genitourinario. Solo se observó amplificación del gen de interés en las muestras que contenían ADN de C. trachomatis, o en las mezclas (C. trachomatis + U. urealyticum) y (C. trachomatis + M. hominis).
Para determinar la variabilidad intra e inte-rensayo se compararon los resultados de los triplicados de las PCR realizadas por triplicado y variando las condiciones experimentales (operadores y días de ensayo) respectivamente. La PCR para C. trachomatis evaluada en este estudio demostró la repetitibilidad y reproducibilidad esperada (tablas 4 y 5).
DISCUSIÓN
Chlamydia trachomatis es el patógeno que causa el mayor número de infecciones urogenitales en el mundo cada año, produciendo enfermedades de tipo agudo como uretritis y cervicitis e infecciones persistentes que frecuentemente son asintomáticas y pueden llegar a producir complicaciones crónicas. La infección natural con C. trachomatis no protege contra la reinfección y actualmente no existe una vacuna contra esta bacteria, por lo tanto, el diagnóstico y tratamiento oportuno de los individuos infectados es la principal herramienta para el control de la transmisión.
Este trabajo realizó la validación técnica de una PCR para la detección de Chlamydia trachomatis que podría servir como punto de partida para la implementación clínica de esta prueba en nuestro medio. Debido a las dificultades para el diagnóstico de C. trachomatis en Colombia, se espera que la aplicación de esta PCR pueda beneficiar a la población de mayor riesgo (individuos jóvenes sexualmen-te activos) y simultáneamente, impactar en el control de esta bacteria de transmisión sexual.
A pesar de que el cultivo celular es considerado como el método de referencia para el diagnóstico de laboratorio de C. trachomatis, este presenta grandes inconvenientes para su implementación en los laboratorios clínicos, dentro de los que se destacan el elevado costo y la complejidad operacional (5); así como las pruebas inmunoenzimáticas, que se basan en la detección de antígenos bacteriano y tienen la ventaja de ser económicas y de fácil ejecución, pero presentan una baja especificidad comparadas con el cultivo (12). Por lo anterior, los métodos basados en amplificación de ácidos nucleicos, como la PCR, son la alternativa de elección para el diagnóstico de C. trachomatis, debido a su alta sensibilidad, especificidad y fácil estandarización dentro del laboratorio (7).
La gran versatilidad de las pruebas de amplificación de ácidos nucleicos ha permitido su aplicación en el diagnóstico de C. trachomatis por medio de diferentes estrategias, como LCR (Ligase Chain Reaction), PCR (Polymerase Chain Reaction) y PCR en tiempo real o PCR cuantitativo (qPCR), entre otros. Actualmente, existen en el mercado estuches de qPCR fácilmente automatizables y con excelente desempeño diagnóstico (9); sin embargo, los altos costos de los equipos y estuches comerciales de qPCR pueden ser el principal inconveniente para su implementación en países en desarrollo. Por este motivo, este trabajo se estandarizó una PCR convencional In house, con una capacidad diagnóstica similar a una qPCR pero que podría resultar más económica.
La enzima Taq polimerasa cataliza la adición de mononucleótidos, derivados de desoxinu-cleósidos 5' trifosfatos, al grupo OH 3' de la cadena creciente de ADN. Esta reacción solo es posible bajo la presencia de iones Mg2+ que inducen ajustes conformacionales en la enzima necesarios para su actividad (13).
La estandarización de la concentración de Mg2+es clave durante la PCR porque así como estos iones son esenciales para garantizar la actividad enzimática, el exceso de Mg2+ disminuye la especificidad y fidelidad de la enzima, además de afectar la Tm de los híbridos cebador-ADN, ADN-ADN y cebador-cebador formados durante la PCR (14).
Nuestros datos mostraron amplificación del gen de interés a partir de 1.5 mM y hasta 3 mM de MgCl2; sin embargo, para minimizar el efecto del exceso de Mg+2 Taq polimerasa y la Tm de los híbridos, todas las PCR se realizaron con la menor concentración de MgCl2 que permitió amplificar el gen de interés (1.5 mM).
C. trachomatis es un patógeno intracelular con un cromosoma de 7 Mb y 10 copias de un plásmido de 7.5 kb (15). Este plásmido no conjugativo contiene cinco secuencias codificantes (CDS) que comparten identidad con genes de mantenimiento episomal y tres CDS que codifican por proteínas, posiblemente relacionadas con virulencia (16).
El plásmido es un blanco común de las pruebas de PCR usadas en el diagnóstico de infecciones por Chlamydia (incluyendo la PCR de este estudio), que han sido fundamentales para el seguimiento y control de las infecciones causadas por esta bacteria en Europa y Estados Unidos.
En 2006 se describió por primera vez en Suecia una nueva variante de C. trachomatis que no era detectada por algunas pruebas de PCR disponibles en el mercado (17). Posteriormente se determinó que estas variantes tenían una deleción de 377 pb, justo en la región usada para la amplificación génica en estas pruebas (18).
La secuencia del plásmido amplificada por nuestra PCR no está incluida en la deleción reportada en la variante sueca, lo cual significa que la PCR estandarizada en este estudio podría potencialmente detectar tanto los aislados clásicos como la variante sueca de C. trachomatis.
La emergencia de la variante sueca de C. trachomatis representa un ejemplo de la gran versatilidad del genoma bacteriano y muestra, simultáneamente, la importancia de la selección de regiones génicas conservadas como blancos para la detección de patógenos en las pruebas de amplificación de ácidos nucleicos.
Un estudio que comparó las ocho CDS del plásmido de once aislados de C. trachomatis, incluyendo la nueva variante sueca, determinó las regiones CDS2, CDS6, CDS7 y CDS8 como las más conservadas dentro de los plásmidos secuenciados (19). Por lo tanto, la utilización de cebadores que flanqueen simultáneamente estas secuencias conservadas del plásmido junto con genes cromosómicos, como ompA, pueden disminuir la probabilidad de falsos negativos en las pruebas de detección de C. trachomatis.
La sensibilidad analítica de la PCR estandarizada en este estudio, usando 40 ciclos, fue de 10-17 g de ADN; este valor corresponde a una copia del plásmido o menos de un cuerpo elemental de C. trachomatis (figura 3). Estos resultados fueron similares a los reportados por otros (20) y demuestran el potencial diagnóstico de esta PCR.
Un estudio que mostró la carga de C.E de C. trachomatis excretados por individuos infectados en diferentes muestras clínicas (21) determinó en promedio, 1200 C.E/100 uL y 162 C.E/100 uL en orina de hombres y mujeres respectivamente. De la misma manera, determinaron cargas de 2231 y 773 C.E/100uL en muestras endocervicales y vaginales, respectivamente. Basados en el número de CE excretados por individuos sintomáticos y asumiendo que los individuos con infecciones asintomáticas tienen una menor carga de CE que los sintomáticos, se puede sugerir que esta PCR tendría el potencial de detectar ambos grupos considerando su sensibilidad analítica (1 plásmido « < 1 CE) (Figura 4). La identificación de una bajo número de CE, e inclusive de ADN plasmidial (liberado después de la muerte bacteriana), es fundamental para el diagnóstico y tratamiento de individuos asintomáticos y por consiguiente para el control de la transmisión.
Para la estandarización de esta prueba se usó ADN de Chlamydia trachomatis serovar D (asociada con infecciones genitales no invasivas), para amplificar una región del plásmido críptico de gran estabilidad genética y amplia distribución dentro del género Chlamydia (16, 19, 22); por lo tanto, esta PCR podría emplearse para detectar otras serovares genitales no invasivas como E-K, así como los biovares tracoma y linfogranuloma venéreo.
Aunque este trabajo utilizó unas pocas muestras de exudados vaginales, para los ensayos de especificidad y reproducibilidad se requiere de un estudio adicional que incluya individuos sintomáticos y asintomáticos para realizar validación clínica de esta prueba.
CONCLUSIONES
A pesar de que la terapia con antibióticos es efectiva para el tratamiento de las infecciones por Chlamydia trachomatis, la prevención de las secuelas post inflamatorias, las reinfecciones y co-infecciones por otros patógenos, constituyen en la actualidad los principales desafíos que debemos afrontar antes de la implementación de una vacuna contra este patógeno.
La naturaleza asintomática de las infecciones por C. trachomatis interfiere con el diagnóstico y tratamiento de los individuos infectados y a la vez facilita la inflamación crónica y el daño tisular irreversible (23, 24).
En Colombia, el acceso limitado a las pruebas de diagnóstico de C. trachomatis, sumado a la ausencia de un programa de vigilancia de infecciones por este microorganismo, ofrecen el escenario ideal para la transmisión de esta bacteria, así como de otros microorganismos de transmisión sexual, como VIH, que puede aprovechar el estado de inflamación crónica inducido por C. trachomatis para infectar células blanco reclutadas en las mucosas urogenitales (24, 25).
La implementación de pruebas diagnósticas, tal como la PCR estandarizada en este estudio, podría contribuir con el diagnóstico y tratamiento temprano de las infecciones por C. trachomatis de individuos sintomáticos y asintomáticos (26).
Agradecimientos
Los autores agradecen la donación de material genético de Mycoplasmataceae, Dra. Natalia Basto (Lab. Celagem); Treponema pallidum, Dra. Lady Ramírez (CIDEIM); PBMC-VIH-1 y Herpes simples-1, Dra. Beatriz Parra (Universidad del Valle). Y a los profesionales Leonor Oviedo y Javier Bustamante, del Departamento de Microbiología de la Universidad del Valle, por su apoyo técnico.
Financiación: Universidad del Valle. Convocatoria interna 2011. CI 1671.
Conflicto de intereses: ninguno.
REFERENCIAS
1. ECDC. ECfDPaC. Sexually transmitted infections in Europe 2013 Stockholm2015. Available from: http://ecdc.europa.eu/en/publications/Publications/sexual-transmitted-infections-europe-surveillance-report-2013.pd. [ Links ]
2. CDC CfDCaP. Sexually Transmitted Disease Surveillance 2012 Atlanta 2013. [Available from: http://www.cdc.gov/sTD/stats12/Surv2012.pdf. [ Links ]
3. Senior K. Chlamydia: a much underestimated STI. Lancet Infect Dis 2012;12(7):517-8. [ Links ]
4. Gottlieb SL, Deal CD, Giersing B, Rees H, Bolan G, Johnston C et al. The global roadmap for advancing development of vaccines against sexually transmitted infections: Update and next steps. Vaccine 2016;34(26):2939-47. [ Links ]
5. Gordon FB, Woolridge RL, Quan AL, Gill-more JD, Arm HG, Yang YF et al. Field studies on Mccoy cell cultures for detection of Chlamydia trachomatis. Southeast Asian J Trop Med Public Health 1972;3(1):69-78. [ Links ]
6. Lefebvre J, Laperrière H, Rousseau H, Massé R. Comparison of three techniques for detection of Chlamydia trachomatis in en-docervical specimens from asymptomatic women. J Clin Microbiol 1988;26(4):726-31. [ Links ]
7. Boyadzhyan B, Yashina T, Yatabe JH, Pat-naik M, Hill CS. Comparison of the AP-TIMA CT and GC assays with the APTIMA combo 2 assay, the Abbott LCx assay, and direct fluorescent-antibody and culture assays for detection of Chlamydia trachomatis and Neisseria gonorrhoeae. J Clin Microbiol 2004;42(7):3089-93. [ Links ]
8. Loeffelholz MJ, Lewinski CA, Silver SR, Pu-rohit AP, Herman SA, Buonagurio DA et al. Detection of Chlamydia trachomatis in en-docervical specimens by polymerase chain reaction. J Clin Microbiol 1992;30(11):2847-51. [ Links ]
9. Peuchant O, de Diego S, Le Roy C, Frantz-Blancpain S, Hocké C, Bébéar C et al. Comparison of three real-time PCR assays for the detection of Chlamydia trachomatis and Neisseria gonorrhoeae in young pregnant women. Diagn Microbiol Infect Dis 2015;83(4):335-7. [ Links ]
10. Ashimoto A, Chen C, Bakker I, Slots J. Po-lymerase chain reaction detection of 8 putative periodontal pathogens in subgingival plaque of gingivitis and advanced periodontitis lesions. Oral Microbiol Immunol 1996;ll(4):266-73. [ Links ]
11. Lan J, Walboomers JM, Roosendaal R, van Doornum GJ, MacLaren DM, Meijer CJ et al. Direct detection and genotyping of Chlamydia trachomatis in cervical scrapes by using polymerase chain reaction and restriction fragment length polymorphism analysis. J Clin Microbiol 1993;31(5):1060-5. [ Links ]
12. Watson EJ, Templeton A, Russell I, Paavo-nen J, Mardh PA, Stary A et al. The accuracy and efficacy of screening tests for Chlamydia trachomatis: a systematic review. J Med Microbiol 2002;51(12):1021-31. [ Links ]
13. Vinod K, Beard W, Shock D, Krahn J, Pendersen L, Wilson S. Magnesium induced assembly of a complete DNA polymerase catalytic complex. Structure 2006;14(4):757-66. [ Links ]
14. Eckert K, Kunkel T. High fidelity DNA synthesis by the Thermus aquaticus DNA polymerase. Nucleic Acids Res 1990(13):3739-44. [ Links ]
15. Stephens RS, Kalman S, Lammel C, Fan J, Marathe R, Aravind L et al. Genome sequence of an obligate intracellular pathogen of humans: Chlamydia trachomatis. Science 1998;282(5389):754-9. [ Links ]
16. Rockey DD. Unraveling the basic biology and clinical significance of the chlamydial plasmid. J Exp Med 2011;208(11):2159-62. [ Links ]
17. Unemo M, Clarke IN. The Swedish new variant of Chlamydia trachomatis. Curr Opin Infect Dis 2011;24(1):62-9. [ Links ]
18. Ripa T, Nilsson PA. A Chlamydia trachomatis strain with a 377-bp deletion in the cryptic plasmid causing false-negative nucleic acid amplification tests. Sex Transm Dis 2007;34(5):255-6. [ Links ]
19. Seth-Smith HM, Harris SR, Persson K, Marsh P, Barron A, Bignell A et al. Co-evolution of genomes and plasmids within Chla-mydia trachomatis and the emergence in Sweden of a new variant strain. BMC Genomics 2009;10:239. [ Links ]
20. Ostergaard L, Birkelund S, Christiansen G. Use of polymerase chain reaction for detection of Chlamydia trachomatis. / Clin Microbiol 1993;31(11):3081. [ Links ]
21. Michel CE, Sonnex C, Carne CA, White JA, Magbanua JP, Nadala EC et al. Chlamydia trachomatis load at matched anatomic sites: implications for screening strategies. / Clin Microbiol 2007;45(5):1395-402. [ Links ]
22. Thomas NS, Lusher M, Storey CC, Clarke IN. Plasmid diversity in Chlamydia. Microbiology 1997;143 ( Pt 6):1847-54. [ Links ]
23. Mascellino MT, Boccia P, Oliva A. Immu-nopathogenesis in Chlamydia trachomatis Infected Women. ISRN Obstet Gynecol.2011;2011:436-936. [ Links ]
24. Molano M, Meijer CJ, Weiderpass E, Arslan A, Posso H, Franceschi S et al. The natural course of Chlamydia trachomatis infection in asymptomatic Colombian women: a 5-year follow-up study. J Infect Dism 2005;191(6):907-16. [ Links ]
25. Paredes MC, Gómez YM, Torres AM, Fernández M, Tovar MB. Prevalence of infections by Chlamydia trachomatis and Neisseria gonorrhoeae among high school students in the Sabana Central area of Cundinamarca, Colombia. Biomedica 2015;35(3):314-24. [ Links ]
26. Fredlund H, Falk L, Jurstrand M, Unemo M. Molecular genetic methods for diagnosis and characterisation of Chlamydia trachomatis and Neisseria gonorrhoeae: impact on epidemiological surveillance and interventions. APMIS 2004;112(11-12):771-84. [ Links ]