INTRODUCCIÓN
Las enfermedades infecciosas fueron la principal causa de muerte del ser humano antes del descubrimiento de la penicilina en los años 40; pese a que la medicina y otras ciencias presentan avances, las infecciones bacterianas siguen siendo un grave problema en muchos países del mundo 1. Aunque la resistencia bacteriana es un fenómeno natural, el uso irracional de antimicrobianos tanto en el ser humano, animales y agricultura están acelerando este proceso, dejándonos sin alternativas para el tratamiento de las infecciones 2.
Las infecciones por bacterias resistentes pueden afectar a cualquier persona, sin importar edad, religión o región donde se encuentre. A nivel hospitalario, las infecciones asociadas a la atención de salud (IAAS) cada vez se vuelven más difíciles de tratar, por lo cual se han convertido en una de las mayores amenazas para la salud pública 3. En hospitales de alta complejidad, el problema de la resistencia presenta un mayor impacto por las pocas medidas de prevención y control, higiene de manos, protocolos de limpieza y desinfección, elevado número de pacientes críticamente enfermos, presencia de múltiples enfermedades concomitantes y uso frecuente de dispositivos invasivos, el uso inapropiado de antibióticos, mecanismos de resistencia intrínsecos o adquiridos por la bacteria que conllevan a la aparición de multirresistencia, capacidad que tienen las bacterias de eludir la acción antibacteriana como fenómeno inagotable, así como su alta tasa de mutaciones y su amplia gama de transferencia de material genético intra- o interespecífico 1,4.
En estos ambientes hospitalarios, uno de los factores que más altas tasas de mortalidad y morbilidad producen son las infecciones por enterobacterias productoras de β-lactamasas de espectro extendido (BLEE), debido a la capacidad de hidrolizar los antibióticos β-lactámicos empleados; dichos antibióticos son los más empleados a nivel mundial 5.
El panorama de la resistencia de las bacterias en Colombia es complejo, y gracias a los múltiples estudios de caracterización microbiológica y molecular llevados a cabo por diferentes grupos de vigilancia e investigación del país, se ha logrado conocer la prevalencia, las variantes, la distribución y las implicaciones clínicas de las betalactamasas en los hospitales de diferentes ciudades del país 6.
El departamento de Boyacá tiene un perfil de resistencia muy diverso a nivel hospitalario, lo que hace necesario ampliar los estudios de estos microorganismos aislados, buscando factores relacionados con la diseminación de bacterias resistentes, sumando al ya referido uso inapropiado de antibióticos y la aplicación deficiente en las medidas de prevención y control 7. Por lo tanto, el objetivo de este estudio fue determinar la presencia de los genes de resistencia en cepas bacterianas asociados a infecciones en una IPS de segundo nivel del municipio de Duitama (Boyacá), de tal manera que se identifiquen y confirmen estos fenotipos de resistencia y se aporte datos relevantes a la vigilancia de este evento de interés en dicho departamento.
MATERIALES Y MÉTODOS
Se llevó a cabo un estudio observacional, descriptivo, de corte transversal. Para ello se caracterizaron cepas provistas por el laboratorio de la IPS de segundo nivel del municipio de Duitama que presentaron algún fenotipo de resistencia. El muestreo se realizó durante febrero a octubre de 2017; y para esto no se tuvo acceso a las historias clínicas ni a la información de los pacientes. La identificación bacteriana inicial se realizó mediante el sistema PHOENIX 100 BD®, al igual que los perfiles de susceptibilidad.
Para la confirmación fenotípica de bacterias Gram negativas y Gram positivas se emplearon métodos convencionales (BBL Crystal GP / GN ID (BD)) en el Laboratorio de Microbiología de la Universidad de Boyacá 8. Para las cepas Gram negativas se realizó prueba de tamizaje a las entero-bacterias aisladas, empleando sensidiscos de cefotaxime (CTX30), ceftazidime (CAZ30), cefepime (FEP30), ceftriaxona y aztreonam (ATM30). Una vez obtenidos los resultados de los antibiogramas, se confirmó la presencia de BLEE con el test de sinergia de doble disco, utilizando la cepa Klebsiella pneumoniae ATCC 700603 como control positivo y la cepa Escherichia coli ATCC 25922 como control negativo 9. El resultado positivo se interpreta por un halo de inhibición mayor o igual a 5 mm de la cefalosporina en combinación con el ácido clavulánico respecto de la cefalosporina; en el total de las cepas se confirmó en el 100 % (n=36) el fenotipo de resistencia.
Para las cepas Gram positivas, los sensidiscos de antibióticos utilizados fueron: Oxacilina (OX, 1μg), Eritromicina (ERI, 15 μg), Clindamicina (CLI, 2 μg), Tobramicina (TOB, 10 μg), Tetraciclina (TCY, 30 μg), Ciprofloxacina (CIP, 5 μg) y Vancomicina (VAN, 30 μg). A todas las cepas identificadas como S. aureus que presentaron resistencia a oxacilina (OX, 1μg) se les realizó el método de difusión del disco con cefoxitina (FOX, 30μg), acorde con los criterios del CLSI/NCCLS 2017 10. Toda cepa de S. aureus oxacilina-resistente y cefoxitina-resistente, fue considerada como S. aureus resistente a meticilina (cepa SARM). Todos estos ensayos fueron realizados por duplicado. Como control de calidad se utilizaron dos cepas: Staphylococcus aureus ATCC 43300 como control positivo y S. aureus ATCC 29213 como control negativo.
Pruebas moleculares
Para la identificación de los genes de resistencia, se realizó una extracción de ADN empleando el kit de extracción de Promega "Wizard" Genomic DNA Purification, siguiendo las instrucciones del fabricante 11. El ADN obtenido fue cuantificado empleando un protocolo basado en un marcador fluorescente intercalante 12; para esto se empleó el equipo de QuantusTM Fluorometer (Promega) y el kit QuantiFluor® dsDNA System (Promega), siguiendo las recomendaciones del fabricante.
Todas las amplificaciones se realizaron mediante PCR convencional en un termociclador labconco de doble bloque; para estas se empleó el reactivo premezclado 2X PCR Taq MasterMix with dye de Applied Biological Materials (ABM), que contiene una concentración balanceada de ADN Taq polimerasa, dNTPs, MgCl2 y demás componentes necesarios para la PCR, según se muestra en la tabla 1. Las amplificaciones fueron realizadas en un volumen final de 25 uL (master master mix 1X; 0.2 uM de cada primer y 2 ng/uL de ADN) en el termociclador Labconco, según los protocolos especificados para cada gen. Todos Los iniciadores fueron sintetizados por Macrogen Korea.
Tabla 1 Componentes de 2X PCR Taq MasterMix with dye de ABM
| Componente | |
|---|---|
| Tris-HCl | 20 mM |
| Tween® 20 | 0.1% |
| MgCl2 | 3 mM |
| KCl | 100 mM |
| dNTPs | 0.4 mM |
| AND Taq Polimerasa | 50 unidades/ml |
Caracterización de genes AmpC, blaTEM, blaCTXM1, blaSHV en cepas Gram negativas:
Para la detección molecular de los genes de resistencia en Escherichia coli y Klebsiella pneumoniae se utilizó el protocolo 005 del Laboratorio de Epidemiología Molecular de la Universidad de Boyacá, empleando iniciadores y condiciones establecidas por Paterson et al. 13. Para los genes tipo AmpC (550pb) se emplearon los iniciadores F5'-ATCAAAACTGGCAGCCG-3' y R5'-GAGCCCGTTTTAT-GCACCCA-3', amplificados bajo las siguientes condiciones: desnaturalización inicial de 94 °C 30s, seguido por 35 ciclos de 94 °C 30s, alineamiento a 56.9 °C 1 min y extensión 72 °C 1 min, con una extensión final de 72 °C 10 min. Para la detección de los genes blaTEM (900pb) se emplearon los iniciadores F5'-AAACGCTGGTGAAAGTA-3' y R5'-AGCGATCTGTCTAT-3' y las siguientes condiciones: desnaturalización inicial de 94 °C 30s seguido por 35 ciclos de 94°C por 30 s, alineamiento a 49 °C por 1 min y extensión 72 °C por 1 min, con una extensión final de 72 °C por 10 min. Para blaCTXM1 (500pb) se utilizaron los inciadores F5'-GACGATGTCACTGGCTGAGC-3' y R5'-AGCCGCC-GACGCTAATACA-3', amplificados bajo las condiciones: desnaturalización inicial de 94 °C por 30 s, seguido por 35 ciclos de 94 °C por 30 s, alineamiento a 58 °C por 1 min, y extensión 72 °C por 1 min, con una extensión final de 72 °C por 10 min. Para blaSHV (700pb) se emplearon los iniciadores F5'ATGCGTTATATTCGCCTGTG-3' y R5'-TGCTTTGTTATTCGGGCCAA-3', amplificados bajo las siguientes condiciones: desnaturalización inicial de 94 °C por 30 s, seguido por 35 ciclos de 94 °C por 30 s, alineamiento a 56 °C por 1 min, y extensión 72 °C por 1 min, con una extensión final de 72 °C por 10 min.
Para el control de amplificación se utilizó la cepa Klebsiella pneumoniae ATCC 700603 (control positivo) y la cepa Escherichia coli ATCC 25922 (control negativo).
Caracterización de genes blaZ y mecA en cepas Gram positivas:
Para la caracterización molecular de cepas Gram positivas se utilizó la presencia o ausencia de los genes de resistencia blaZ y mecA. La extracción del ADN se realizó empleando el protocolo 003 para extracción de ADN en bacterias Gram Positivas del Laboratorio de Epidemiología Molecular de la Universidad de Boyacá 14. Como control se emplearon cepas de Staphylococcus aureus ATCC43300.
El gen blaZ (861pb) se evaluó mediante reacción en cadena de la polimerasa con los iniciadores F5' TAC AAC TGT AATATC GGA GGG y R5' CAT TAC ACT CTT GGC GGT TTC. En el esquema de amplificación se tuvieron en cuenta las siguientes condiciones: desnaturalización inicial de 94 °C 5 min, seguido por 35 ciclos de 94 °C por 30 s, alineamiento a 55 °C por 30 s, y extensión 72 °C por 30 s, con una extensión final de 72 °C por 5 min. Como control se emplearon cepas de Staphylococcus aureus ATCC43300. En cuanto al gen mecA (533 pb); las cepas meticilino resistentes fueron detectadas utilizando los iniciadores F5' AAA ATC GAT GGT AAA GGT TGG y R5' AGT TCT GCA GTA CCG GAT TTG. La amplificación se realizóde la siguiente mmanera : desnaturalización 94 °C por 30 s, 30 ciclos de alineamiento a 55 °C por 30 s y extensión a 72 °C por 1 min, y una etapa de extensión final de 72 °C por 5 min.
Los productos de amplificación se evidenciaron mediante electroforesis en gel de agarosa al 1 % en TAE 1 %, empleando safeview classic (ABM) como agente intercalante, y opti DNA Marker 1Kb (ABM) como marcador de peso molecular. Las bandas se evidenciaron en un transiluminador Ul-traSlim Led Illuminator Maestrogen.
RESULTADOS
Se evaluaron un total de 76 cepas, las cuales cumplieron con el criterio de inclusión de presentar algún fenotipo de resistencia. Dentro de los aislamientos de los microorganismos Gram negativos, de las 36 cepas, el 55.6 % (n= 20) correspondió a Escherichia coli (E. coli) y el 41.7 % (n=15) a Klebsiella pneumoniae (K. pneumoniae). El 2.8 % restante está representado por una cepa de Klebsiella oxytoca (n=1). Se evidenció que el servicio con mayores aislamientos bacterianos fue Urgencias, con predominio de la cepa de E.coli, seguido de Hospitalización y Unidad de Cuidado Intensivo (UCI), con predominio de aislamientos de K. pneumoniae.
Se incluyeron un total de 40 cepas Gram positivas identificadas como S. aureus en el 100 % de los aislados. Con respecto a la procedencia de la infección según el servicio del hospital, se determinó que 52.5 % (n=21) correspondía a Consulta por Urgencias; 20 % (n=8) a Hospitalización; 10 % (n=4) a Quirófanos; 7.5 % (n=3) a Consulta de Observación; 7.5 % (n=3) Unidad de Cuidado Intensivo - UCI Adultos, y por último, 2.5 % (n=1) a Consulta Externa.
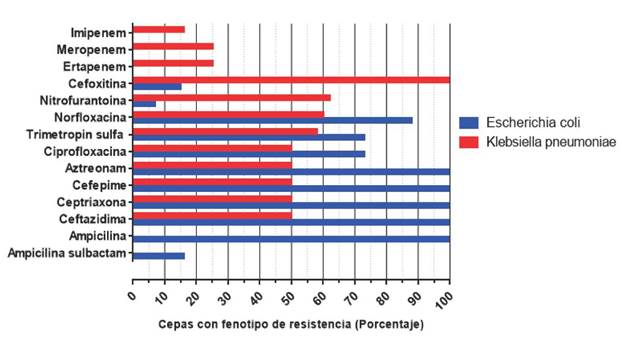
Los resultados de la evaluación de los fenotipos de resistencia para los aislados de E.coli (n= 20) y K. pneumoniae (n=16) se presentan en la figura 1.
El patrón de resistencia para las 40 cepas de S. aureus mostró que el 100 % de las cepas presentó resistencia frente a Penicilina G; el 47.5 % a la oxacilina. En cuanto a esta última, la literatura refiere que se debe realizar la confirmación empleando un sensidisco de cefoxitin que determinará la presencia presuntiva de la resistencia a oxacilina mediada por el gen mecA 15,16. El 15 % de las cepas presentó resistencia a la eritromicina; 5 % resistencia frente a clindamicina; 2.5% son resistentes al trimetropin sulfa, y ningún aislamiento presentó resistencia a Vancomicina.
Caracterización molecular de genes de resistencia en bacterias Gram negativas y Gram positivas. El estudio mostró una prevalencia de genes de resistencia en el 91,7 % de las muestras evaluadas (33/36), siendo blaTEM el más frecuente, presente en 33 cepas (91,7 %), seguido por blaCTXM1, presente en el 36,1 % y blaSHV, presente en el 27,8 % de las cepas. Al evaluar el gen Ampc en las cepas Gram negativas, se determinó una frecuencia de 5,6 %. Un análisis más detallado revela que el 26,8 % de las cepas presentaba tanto el gen blaTEM como el gen blaSHV; adicionalmente, las dos cepas que presentaron el gen Ampc también presentaron el gen blaTEM, y solo una de las cepas analizadas, que corresponde a una cepa aislada de Klebsiella pneumoniae, presentó tres genes: Ampc, blaSHV y blaTEM (tabla 2).
Tabla 2 Caracterización molecular de los genes en cepas Gram negativas
| Ampc | blaSHV | blaCTXM1 | blaTEM | ||||||
| Cepas evaluadas | pos | % | pos | pos | pos | % | |||
| Escherichia coli | 20 | 0 | 0 | 2 | 10 | 11 | 55 | 18 | 90 |
| Klebsiella pneumoniae | 16 | 2 | 13 | 8 | 50 | 2 | 13 | 15 | 94 |
| Total Cepas | 36 | 2 | 10 | 13 | 33 | ||||
Pos.: cepas positivas para el gen.
Fuente: Elaboración propia.

A. Carril 1: marcador de peso molecular 1kb. Carril 2: control negativo E. coli. Carril 3: control positivo K. pneumoniae. Carril 4 - 17 muestras (positivas: muestras 3, 10, 12 y 14 Banda 500pb) B. Carril 1: Marcador de peso molecular 1kb. Carril 2: control negativo E. coli. Carril 3 - 10 muestras (positivas muestra: 7, banda 550pb). C. Carril 1: marcador de peso molecular 1kb. Carril 2: control negativo E. coli. Carril 3 - 13 Mmestras (positivas: muestras 3, 5, 6, 7 y 9, banda 700pb). D. Carril 1: marcador de peso molecular 1kb. Carril 2: control positivo K. pneumoniae. Carril 3 - 17 muestras (positivas: muestras 1 - 15, banda 900pb muestras 10 y 12, banda adicional 1500 kb).
Fuente: Laboratorio de Epidemiología Molecular de la Universidad de Boyacá.
Figura 2 Gel de Electroforesis genes de resistencia en bacterias Gram negativas
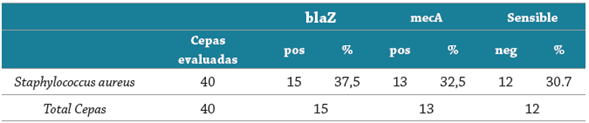
Caracterización de genes blaZ y mecA en cepas Gram positivas. Los resultados de los estudios de presencia de genes de resistencia en S. aureus se muestran en la figura 3.
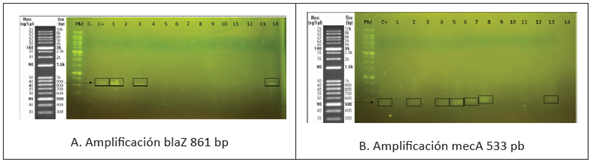
A. Carril 1: marcador de peso molecular 1kb. Carril 2: control negativo S. aureus. Carril 3: control positivo S. aureus. Carril 4 - 17 muestras (positivas: muestras 1, 3 y 14, banda 861pb) B. Carril 1: Mmarcador de peso molecular 1kb. Carril 2: control positivo S. aureus. Carril 3 - 16 muestras (positivas: muestras 2, 4, 5 y 6, banda 533pb, muestra 8 y 13, banda 550pb)
Fuente: Laboratorio de Epidemiología Molecular de la Universidad de Boyacá.
Figuras 3 Gel de Electroforesis genes de resistencia en bacterias Gram positivas
DISCUSIÓN
Los resultados de este estudio se asemejan a los reportados en otras investigaciones en las que E. coli es el uropatógeno con mayor número de aislamientos Gram negativos, seguidos por K. pneumoniae17-19. Así mismo, se evidenció que el servicio con mayor número de aislamientos fue urgencias, con predominio de la cepa de E.coli, seguido de Hospitalización y Unidad de Cuidado Intensivo (UCI), con predominio de aislamientos de K. pneumoniae, lo cual sugiere una grave problemática, debido a que el mayor número de muestras aisladas en pacientes son ambulatorios. Esto demuestra la existencia de este patógeno a nivel de comunidad, y se pudo establecer un uso indiscriminado de antibióticos, que motiva a realizar más trabajos de este tipo para vigilar el uso correcto de los mismos 20,21. Entre agosto y diciembre de 2011 se realizó un estudio de casos y controles en 3 instituciones de salud de tercer nivel en Colombia. En este se tomaron todos los pacientes admitidos por urgencias con diagnóstico probable de infección de vías urinarias. De los 2124 pacientes seleccionados, 629 tuvieron un urocultivo positivo; en 431 de estos se aisló E. coli; 54 fueron positivos para BLEE y 29 corresponden a CTXM. Dicho estudio mostró resultados compatibles con los de este estudio en cuanto a cepa y mecanismo de resistencia predominante 22.
Una de las cepas identificada como K. pneumoniae presenta en su resultado del antibiograma resistencia a la mayoría de los antibióticos, incluso a la penicilina; este patrón de resistencia es compatible fenotípicamente con el mecanismo de resistencia KPC (Klebsiella pneumoniae Carbapenemasa) 23, que no pudo ser confirmado molecularmente debido a que aún no se cuenta con su estandarización, dejando la confirmación para próximas investigaciones.
La cepa catalogada como posible portadora de este mecanismo de resistencia proviene de muestra de orina aislada del servicio de urgencia, lo cual puede sugerir que no se asocia a la estancia prolongada sino, por el contrario, a un mal manejo de la terapia antibiótica por parte del personal de salud, o la falta de apego al tratamiento por parte del paciente, lo cual pudo generar la falla terapéutica, y así favorecer la expresión del mecanismo de resistencia 24.
K. pneumoniae ha venido en aumento considerable; en un estudio a nivel nacional se tomaron 23 hospitales entre 2009 y 2012 y se analizaron 38 048 aislamientos durante el periodo descrito. Se describieron perfiles de resistencia para Escherichia coli, Klebsiella pneumoniae, Pseudomonas aeruginosa y Acinetobacter baumannii. E. coli fue el microorganismo más frecuente (promedio=14,8 %); seguido de K. pneumoniae, con un 15 %. De los aislamientos de K. pneumoniae evaluados, 68,4 % fue positivo para KPC. Porcentaje elevado que requiere de vigilancia y monitoreo por las entidades encargadas. Así mismo, se sugiere la realización de investigaciones enfocadas a este mecanismo de resistencia principalmente 9.
En cuanto a los Gram positivos de las cepas evaluadas, el 47.5 % presenta resistencia a la meticilina (oxacilina), que presuntivamente podrían catalogarse como Staphylococcus aureus resistente a la meticilina (SARM), patógeno de gran importancia en salud pública, debido a la rápida diseminación y diversificación de linajes pandémicos que presentan perfiles variables de virulencia y sensibilidad antimicrobiana 25-28, comparado con los resultados reportados por Nancy Amaya entre enero y diciembre de 2008, en los que se obtuvo una resistencia meticilino resistente de 64 %, pero al igual que la presente investigación, no se detectó resistencia a la Vancomicina 29.
En cuanto al análisis molecular, se muestra una prevalencia de genes de resistencia en el 91,7 % de las muestras evaluadas (33/36), siendo blaTEM el gen más frecuente identificado en 33 cepas (91,7 %), lo cual coincide con el rango de frecuencia reportada para Klebsiella pneumoniae30; blaCTXM1 fue el segundo más prevalente, con 36,1 %, seguido por blaSHV, con 27,8 %. Al comparar estos resultados con el trabajo de Ting y colaboradores en 2013, la frecuencia de estos genes es mayor en sus casos de estudios, lo cual indica que considerando las condiciones ambientales, las cepas bacterianas pueden presentar diferentes perfiles y mecanismos de resistencia. En el caso particular de E. coli, solo se encontraron 2 cepas que expresaban blaSHV, mientras que Ting no encontró cepas que presentaran este mecanismo de resistencia 31. Al evaluar el gen Ampc en las cepas Gram negativas, se encontró una frecuencia de 5,6 %. Un análisis más detallado revela que el 26,8 % de las cepas presentaban tanto el gen blaTEM como blaSHV, las dos cepas que presentaron el gen Ampc también presentaron el gen blaTEM; solo una cepa, que corresponde a una K. pneumoniae presentó tres genes: Ampc, blaSHV y blaTEM.
Al comparar los resultados obtenidos con los resultados de caracterización fenotípica se pudo apreciar que el 100 % de las cepas presentaron fenotipo BLEE, mientras que molecularmente se identificó el gen blaZ en un 40 % de ellas. En el caso de fenotipo meticilino resistente, se observó en el 50 %, mientras que solo un 30 % presentó el gen mecA.
En Sudamérica, una de las primeras publicaciones de infecciones por SARM en pacientes ambulatorios adultos con infección de piel fue en Brasil, en 2002 32; posteriormente, fueron apareciendo otros reportes, entre ellos un estudio multicéntrico en Argentina, realizado entre noviembre de 2006 y noviembre de 2007, en el que se diagnosticaron 840 infecciones por S. aureus, de las cuales 447 fueron adquiridas en la comunidad, representando el 62 % del total de las infecciones adquiridas en la comunidad (CA-SARM) 33. Con el aumento en la prevalencia de SARM en pacientes ambulatorios con infecciones de piel y partes blandas (IP y PB) hubo un cambio epidemiológico en los últimos años respecto a las infecciones producidas por S. aureus, lo cual lo convierte en un hecho de importancia clínica y terapéutica, de gran trascendencia en la salud pública, por lo que se presenta más en comunidad que no ha sido hospitalizada ni recibido tratamiento antibiótico 25,34.
CONCLUSIONES
La genotipificación bacteriana permitió confirmar la presencia de clones con resistencia tipo BLEE en el 92 % de las cepas Gram negativas (E. coli y K. pneumoneae) y en el 37 % de las cepas Gram positivas (S. aureus), por lo cual es necesario mantener la vigilancia de estas cepas a fin de evitar posibles brotes causados por estos microorganismos resistentes.
Las consecuencias de ignorar la presencia de las bacterias productoras de BLEE en nuestra realidad puede condicionar al fracaso del tratamiento, lo que puede conllevar a aumentar la resistencia y diseminación de este tipo de microorganismos desde el espacio intrahospitalario a la comunidad.
Ante el incremento de microorganismos Gram negativos multirresistentes y la amplia distribución de las carbapenemasas en esta Institución prestadora de servicios de salud, es de gran importancia la articulación de la biología molecular a los sistemas de vigilancia epidemiológica tradicionales que permita el análisis integral de la resistencia; ya que estos hallazgos podrían servir para el desarrollo de programas de uso adecuado de antimicrobianos y la elaboración de guías para la prevención y contención de bacterias multirresistentes no solo en esta institución sino en Colombia.